基于DNA条形码技术的海参物种鉴定
2019-07-10胡冉冉邢冉冉葛毅强
胡冉冉,邢冉冉,王 楠,葛毅强,陈 颖,*
(1.中国检验检疫科学研究院,北京 100123; 2.中国农业大学食品科学与营养工程学院,北京 100083; 3.中国农村技术开发中心,北京 100045)
海参是一种名贵海产品,富含多种营养物质,在世界各地特别是亚洲国家深受消费者的喜爱,尤其在中国的消费量最大[1-2]。目前全球可食用海参大概有60种[3],不同种的海参价格相差可高达数十倍。由于海参的自溶特性,鲜活海参的运输较为困难,因此海参多以干制品的形式在贸易中流通。但一般加工后的海参产品形态学特征会全部或部分消失,使得通过形态学鉴定海参种类变得困难。在经济利益的驱动下,不法商人通过贴错标签等形式以次充好,扰乱海参市场秩序。市场上最常见的是以刺参属海参冒充高值仿刺参。此外,部分市售海参产品根据产地或颜色贴标,如美国海参、黑海参等,混淆了海参的物种来源。
针对海参物种的鉴别,目前主要有形态学鉴定[4]、理化鉴定[5]和分子生物学鉴定[6]方法。形态学鉴定对于形态学特征消失的海参加工制品的物种鉴定具有局限性,理化鉴定结果容易受加工过程中酸、碱的影响。以DNA为基础的分子生物学检测方法,如DNA条形码技术(DNA barcoding)[7]、多重PCR[8]、实时荧光PCR技术[9]、聚合酶链反应-限制性片段长度多态性(polymerase chain reaction-restriction fragment length polymorphism,PCR-RFLP)[10]等不受季节、加工方法、产品形态的影响,鉴别结果准确度高。多重PCR和实时荧光PCR技术局限于对已知样品的检测。PCR-RFLP虽然在物种鉴定中已成为一种重要的方法,但种内变异可能使目标序列某一特定的酶切位点消失或产生新的酶切位点,PCR产物酶切片段长度差异不大,可能造成结果假阴性。DNA条形码技术是近几年发展迅速的一项分子生物学鉴定技术,其利用通用引物扩增一段标准的、较短的DNA序列,将测序得到的序列在生命条形码数据系统(The Barcode of Life Data System,BOLD)和美国国家生物技术信息中心网站(National Center for Biotechnology Information,NCBI)数据库中进行比对,进而实现物种鉴定[11]。与其他分子生物学技术不同,DNA条形码技术是一种通用的检测方法,具有操作简单、快速可靠等优势,可广泛应用于未知样品的物种鉴定[12-15]。高通量测序技术的出现为混合样品的物种鉴定提供了新的工具,将DNA条形码和高通量测序技术结合可检出混合样品中的未知成分,使得DNA条形码技术在多物种混合样品的物种鉴定中具有更广阔的应用前景[16]。
在动物物种鉴定中,一般将线粒体细胞色素氧化酶亚基I(cytochrome coxidase subunit I,COI)基因作为DNA条形码研究的靶基因[17-19]。律迎春等[7]计算7种海参基于COI基因的遗传距离并建立系统发育树,发现海参COI种间差异较大而种内差异较小,不同海参在系统发育树中分别形成各自独立的分支,说明COI用于海参物种鉴定的可行性。除COI外,16S核糖体RNA(16S ribosomal RNA,16S rRNA)基因亦可作为DNA条形码使用[20-21]。Wen等[22]以16S rRNA基因为分子标记进行海参物种鉴定时发现,5种市售刺参科(Stichopodidae)海参中有1个仿刺参(Apostichopusjaponicus)被暗刺参属(Isostichopus)海参所替代。
本研究选取线粒体COI基因和16S rRNA基因片段作为海参物种鉴定的通用DNA条形码,比较两个靶基因的物种鉴别能力,旨在建立基于DNA条形码技术的海参物种鉴定方法,并应用于市售海参样品的标签符合性判别,为市售海参的品种鉴别提供有效的技术方法。
1 材料和方法
1.1 材料与仪器
16种食用干海参:玉足海参(Holothurialeucospilota)、糙海参(Holothuriascabra)、墨西哥海参(Holothuriamexicana)、短刺乌爪参(Holothuriapoli)、长刺乌爪参(Holothuriatubulosa)、黑海参(Holothuriaatra)、黄乳海参(Holothuriafuscogilva)、图纹白尼参(Bohadschiamarmorata)、蛇目白尼参(Bohadschiaargus)、仿刺参(Apostichopusjaponicus)、绿刺参(Stichopuschloronotus)、糙刺参(Stichopushorrens)、加州拟刺参(Parastichopuscalifornicus)、梅花参(Thelenotaananas)、大西洋瓜参(Cucumariafrondosa)、智利瓜参(Athyonidiumchilensis),分属3科8属。其中,玉足海参、糙海参、仿刺参由渤海大学提供,其余海参由青岛海康水产发展有限公司提供。以上实验样品均经过形态学鉴定,具体信息见表1。2017年3月到4月从水产市场(9份)、海参专卖店(3份)、超市(2份)和电子商务平台(10份)共计购买24份市售海参样品;海洋动物组织基因组DNA提取试剂盒 天根生化科技北京有限公司;PCR Mix预混液 北京百泰克生物技术有限公司;6×上样缓冲液、DL2000 DNA分子标记 购自宝生物工程(大连)有限公司;琼脂糖 购自北京擎科新业生物技术有限公司;PCR产物回收试剂盒 购自美国Sigma公司;PCR产物纯化试剂盒 购自美国Affymetrix公司;5×测序缓冲液、DNA测序试剂盒BigDye 3.1、测序产物纯化试剂盒 购自美国Applied Biosystems公司;PCR扩增引物 均由英潍捷基(上海)贸易有限公司合成。
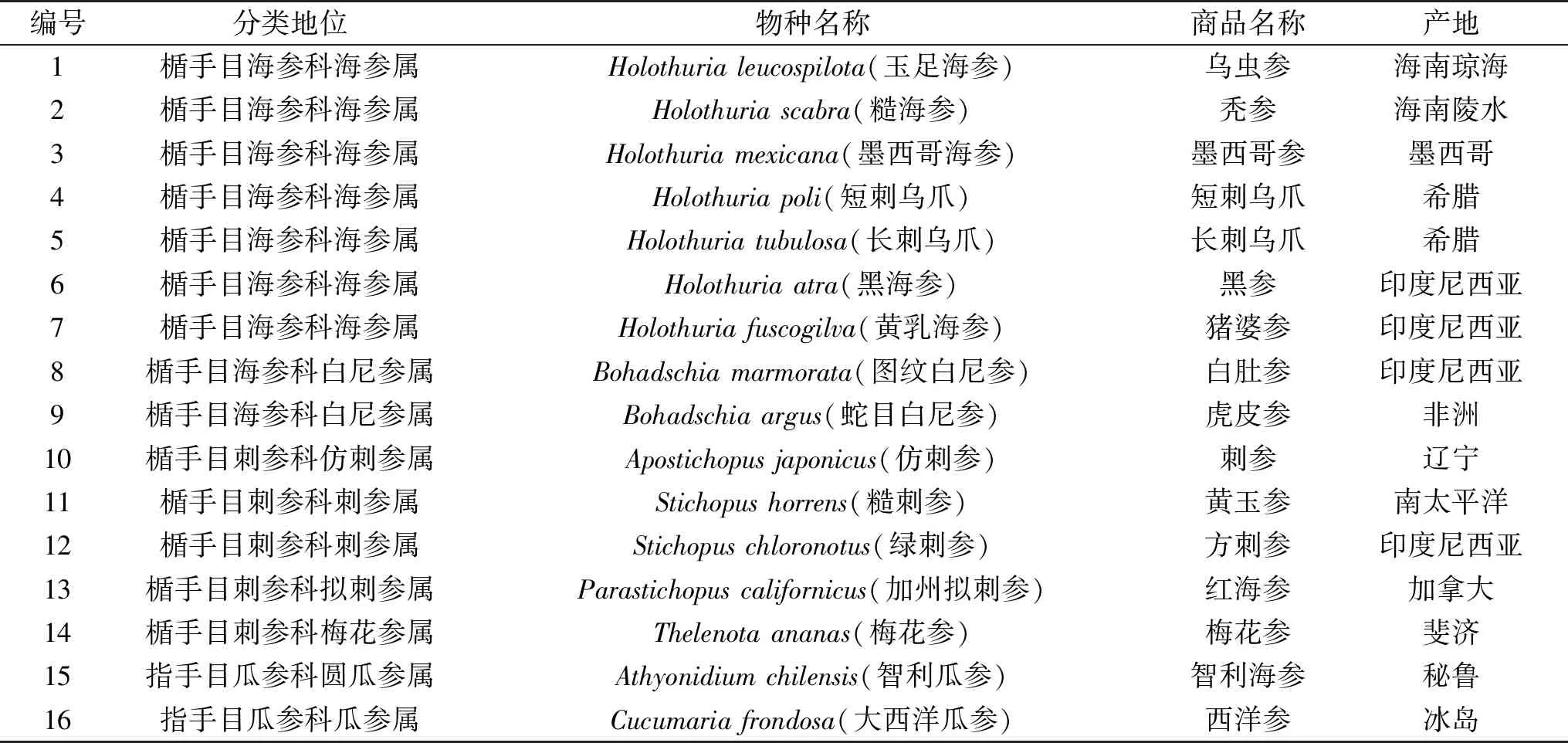
表1 收集的海参样品信息Table 1 Information of collected sea cucumber samples
BT323S电子天平 赛多利斯科学仪器有限公司;Venticell烘箱 德国MMM公司;PICO17台式离心机 美国Thermo Fisher Scientific公司;Research可调量程移液器、BioPhotometer plus紫外分光光度计 德国Eppendorf公司;200/2.0 Power Supply电泳仪、Versa Doc 4000MP凝胶成像系统 美国BIO-RAD公司;Veriti 96孔热循环梯度PCR仪、ABI3500基因分析仪 美国Applied Biosystems公司。
1.2 实验方法
1.2.1 DNA提取 收集的干海参和市售海参分别取2~3个整块样品在液氮中研磨成细小微粒,收集到干净离心管中。分别称取30 mg的海参组织样品,用海洋动物组织基因组DNA提取试剂盒提取DNA,按照试剂盒说明书操作,对个别不易裂解的样品适当延长裂解时间,最后用去离子水代替洗脱缓冲液TE溶解DNA。取DNA原液5 μL溶于45 μL去离子水中,以去离子水作为空白对照,使用紫外分光光度计在波长260 nm紫外线下检测DNA的浓度,并以OD260/OD280比值判断DNA纯度。一般DNA纯品的OD260/OD280为1.7~2.0,若OD260/OD280>2.0,表明有RNA的污染,OD260/OD280<1.7则表明DNA中有蛋白质、酚等污染。
1.2.2 PCR扩增COI通用引物[23]为COIe-F(5′-ATAATGATAGGAGGRTTTGG-3′)和COIe-R(5′-GCTCGTGTRTCTACRTCCAT-3′)。16S rRNA基因通用引物[24]为16Sar(5′-CGCCTGTTTATCAAAAACAT-3′)和16Sbr(5′-CTCCGGTTTGAACTCAGATCA-3′)。
PCR反应体系总体积为25 μL:上、下游引物各1 μL(引物浓度10 μmol/L),2×PCR Mix预混液12.5 μL,DNA模板10 ng,用灭菌双蒸水补足体积至25 μL。扩增程序:94 ℃ 5 min;94 ℃ 50 s,引物COIe-F/R退火温度46 ℃ 1 min,16Sar/Sbr退火温度52 ℃ 1 min,72 ℃ 60 s,35个循环;最后72 ℃延伸10 min;4 ℃保存。PCR产物均用2%的琼脂糖凝胶进行电泳,电压110 V 30 min,将目的片段电泳条带单一且明亮的PCR产物纯化回收。
1.2.3 测序 将纯化后的PCR产物进行双向测序,测序反应体系总体积10 μL:PCR纯化产物1 μL,5×测序缓冲液1 μL,BigDye 3.1 1 μL,引物1.6 μL(1 μmol/L),无菌水5.4 μL。PCR扩增反应条件为:96 ℃ 2 min;96 ℃ 30 s,50 ℃ 40 s,60 ℃ 4 min,30个循环;4 ℃保存。采用测序产物纯化试剂盒进行纯化,每管吸取10 μL上清液于96孔板中,于ABI 3500基因分析仪进行测序。
1.3 数据处理
1.3.1 序列比对及分析 使用DNAStar7.0[25]软件中的SeqMan程序对测序成功的序列进行拼接,将拼接好的序列经过MEGA6.0软件进行多重序列比对,删除两端引物序列。将处理过的COI序列分别在BOLD中的公共DNA条形码数据库(Public Record Barcode Database)和NCBI的GenBank数据库进行比对鉴定物种来源;16S rRNA基因序列在GenBank数据库进行比对得到物种信息。
1.3.2 系统发育树分析 根据序列比对结果,从NCBI网站下载部分相关海参物种的COI参考序列与16S rRNA基因参考序列。将样本序列与下载的基因序列采用MEGA6.0软件进行多重序列比对后,基于K-2-P(Kimura-2-parameter)[26]模型构建邻接法(N-J法)系统发育树,Bootstrap自展检测1000,验证DNA条形码鉴定结果的准确性。
2 结果与分析
2.1 海参COI、16S rRNA基因片段扩增
用紫外分光光度计测得海参样品DNA的A260 nm/A280 nm比值在1.6~1.8之间,说明所提取的海参基因组DNA纯度好,可用于常规PCR扩增。扩增结果显示被测的16种海参样品均扩增出长度为690 bp的COI目的片段和长度为560 bp的16S rRNA基因目的片段(图1),PCR产物可用于下一步的测序。
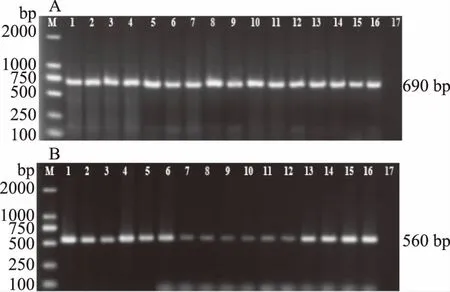
图1 海参样品COI和16SrRNA基因的扩增结果Fig.1 PCR amplification of COI and 16S rRNA gene of sea cucumbers samples注:A:海参样品COI目的片段的扩增B:海参样品16S rRNA基因目的片段的扩增;M:DL 2000 分子量标准;1:H. leucospilota(玉足海参);2:H. scabra(糙海参);3:H. mexicana(墨西哥海参);4:H. poli(短刺乌爪);5:H. tubulosa(长刺乌爪);6:H. atra(黑海参);7:H. fuscogilva(黄乳海参);8:B. marmorata(图纹白尼参);9:B. argus(蛇目白尼参);10:A. japonicus(仿刺参);11:S. horrens(糙刺参);12:S. chloronotus(绿刺参);13:P. californicus(加州拟刺参);14:T. ananas(梅花参);15:A. chilensis(智利瓜参);16:C. frondosa(大西洋瓜参);17:空白对照。
2.2 COI和16S rRNA基因对海参的物种鉴别能力分析
本研究对收集的16种海参先进行形态学鉴定,然后基于DNA条形码技术,以线粒体COI和16S rRNA基因分别作为靶基因,通过序列比对得到海参的物种信息,评价COI与16S rRNA基因对食用海参的物种鉴别能力。
目前用于物种鉴定的16S rRNA基因数据库只有GenBank数据库,而COI基因数据库有GenBank数据库[27]和BOLD数据库[28],考虑到许多科学家并不同时向BOLD和GenBank数据库提交COI序列信息[29],综合两个数据库的比对结果更能保证COI基因鉴定结果的准确性。研究表明基于COI和16S rRNA基因的海参种内遗传距离均低于2%[26,30],因此在进行序列比对时相似度≥98%的可判定为同一物种,低于95%的认定为不同种,而相似度介于95%~98%的需结合其他方法进一步验证。根据COI序列相似度结果,16种海参中除糙刺参(Stichopushorrens)外,其余15种的鉴定结果与形态学鉴定结果一致,物种序列相似度均≥99%,其中6种海参的结果结合了BOLD数据和GenBank数据库的鉴定结果。基于16S rRNA基因14种海参能够得到准确鉴定,标注为黄乳海参(Holothuriafuscogilva)和糙刺参(Stichopushorrens)目前已将本研究获得的BOLD数据库缺少的海参COI序列信息上传至BOLD数据库。的海参无法鉴定出明确物种(表2)。
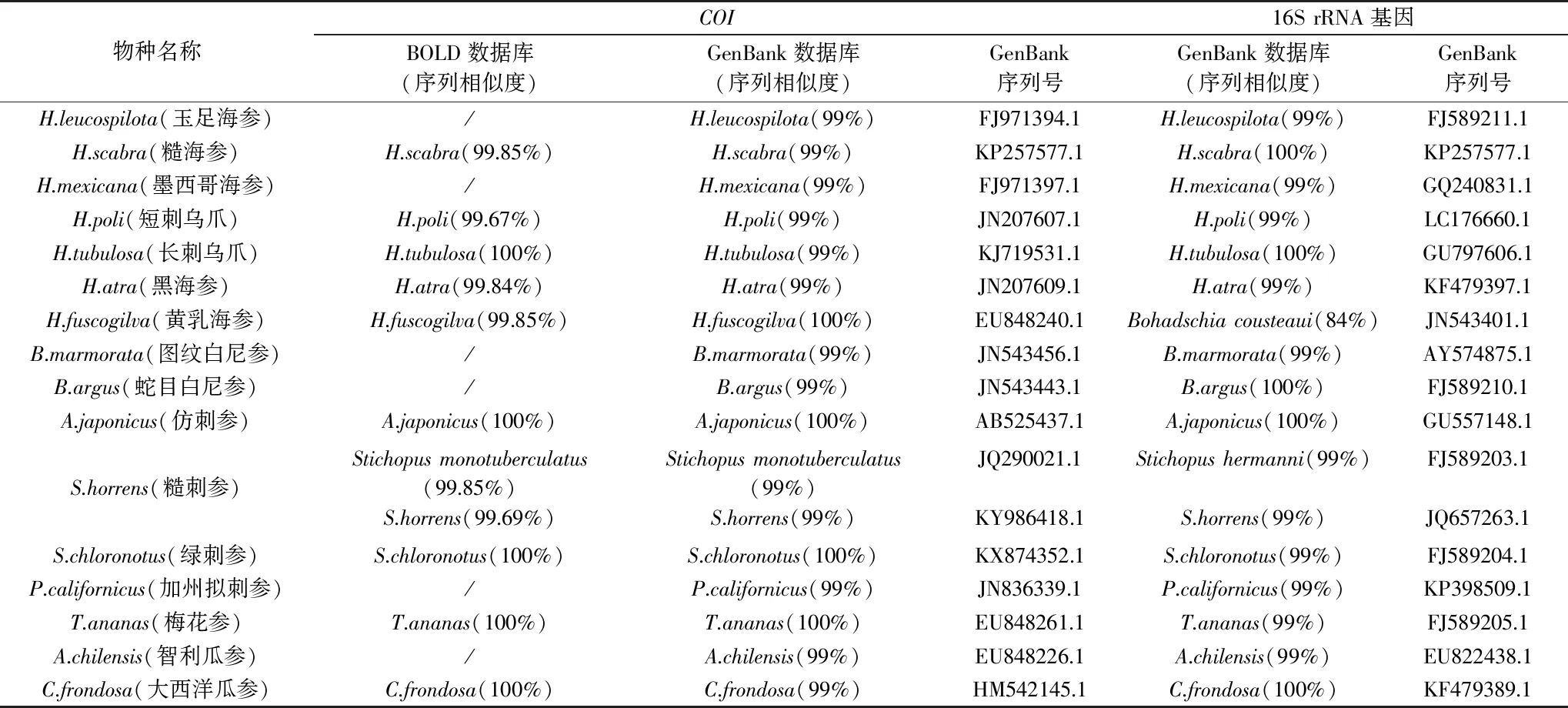
表2 海参样品基于COI和16S rRNA基因的物种鉴定结果Table 2 Identification results of sea cucumber samples based on COI and 16S rRNA gene
COI与16S rRNA基因都是目前用于海参系统进化和遗传多样性研究的重要目的基因。在进化速率上,16S rRNA基因相对COI来说在线粒体基因中更为保守[31]。COI序列没有插入或缺失位点,16S rRNA基因序列有部分插入或缺失位点,而且COI的变异位点大于16S rRNA基因[32]。比较COI和16S rRNA基因的鉴定结果发现,两种靶基因均能较好的鉴定出海参物种,14种海参的鉴定结果一致,两个例外是糙刺参(Stichopushorrens)和黄乳海参(Holothuriafuscogilva)。糙刺参(Stichopushorrens)的COI序列和16S rRNA基因序列分别与单疣刺参(Stichopusmonotuberculatus)、花刺参(Stichopushermanni)的物种序列相似度达99%,因此无法将糙刺参、单疣刺参以及花刺参区分开。黄乳海参(Holothuriafuscogilva)则是基于COI序列可准确鉴别但其16S rRNA基因序列在GenBank数据库的物种序列相似度低于90%,仅鉴定到科,无法有效鉴定到种名,可能与GenBank数据库中缺乏黄乳海参的16S rRNA基因参考序列有关。DNA条形码技术用于物种准确鉴定需要完整和准确的DNA序列数据库作为支撑。目前海参DNA条形码数据库并不完整,部分海参品种的基因序列未上传数据库,在一定程度上限制了DNA条形码技术的应用。因此,必须进一步完善DNA条形码数据库,提高数据库中DNA序列的数量和质量。
鉴于本研究中扩增得到的部分海参COI序列在BOLD数据库无匹配(表2),为了丰富海参品种的线粒体DNA序列信息,为今后对海参品种进行更精确的分析、鉴定奠定基础。根据BOLD数据库要求,已将本研究获得的BOLD数据库缺少的海参COI序列信息上传至BOLD数据库。
2.3 系统发育树分析
基于K-2-P模型分别得到海参COI序列的N-J系统发育树(图2)和16S rRNA基因序列的N-J系统发育树(图3),在进化树中样品以序号-物种种名表示。两个靶基因的系统发育树均显示海参科白尼参属的图纹白尼参和蛇目白尼参亲缘关系最近;刺参科的加州拟刺参和仿刺参的亲缘关系较近;糙刺参与花刺参、单疣刺参的亲缘关系较近聚为单支(100%的置信度),然后与其他刺参属海参再聚在一起;其他海参物种都分别聚为独立的一支。基于种内和种间遗传差异的系统发育树验证了DNA条形码鉴定结果的准确性。
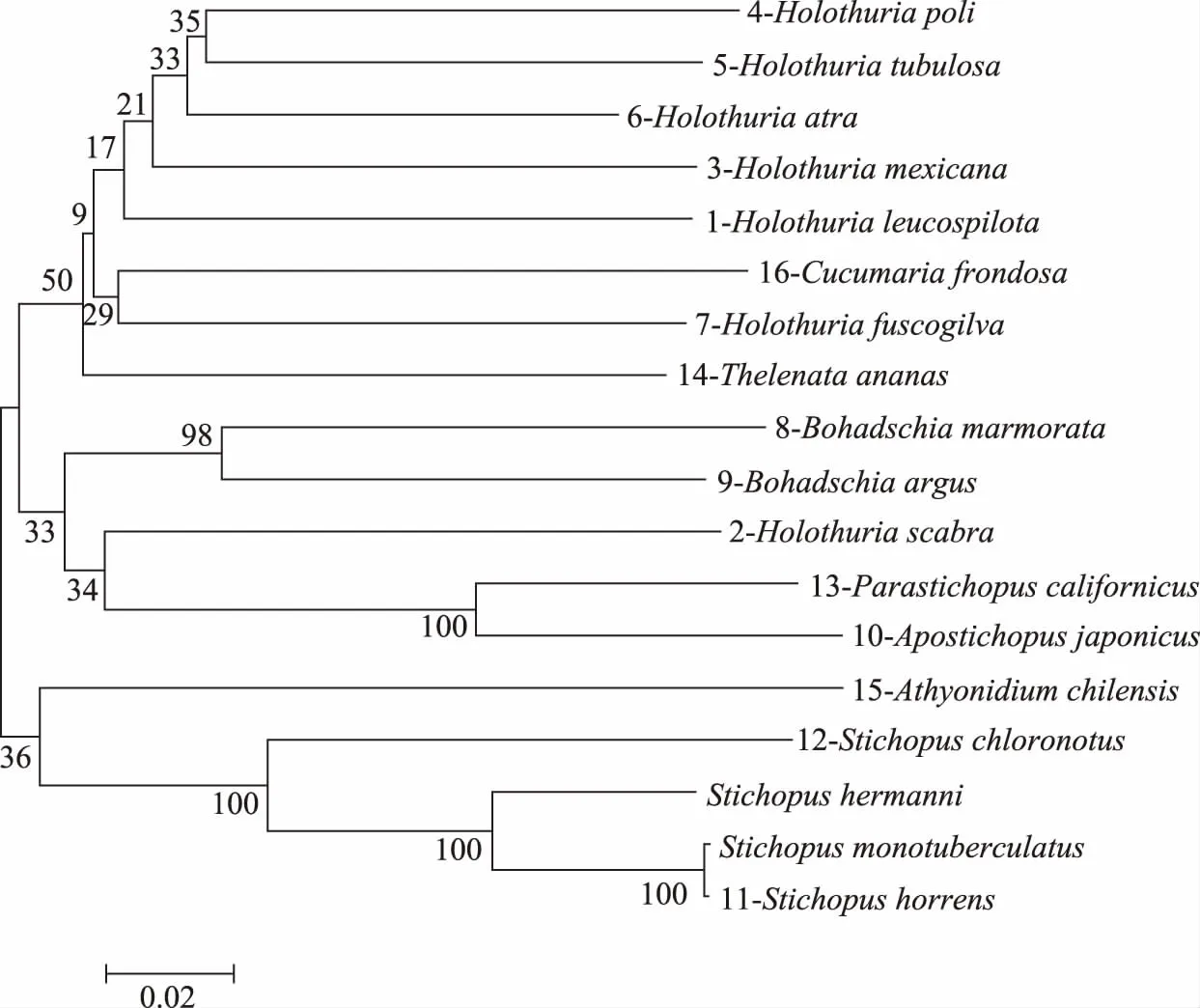
图2 基于COI构建的海参物种N-J系统发育树Fig.2 Neighbor-joining phylogenetic tree of sea cucumbers based on COI
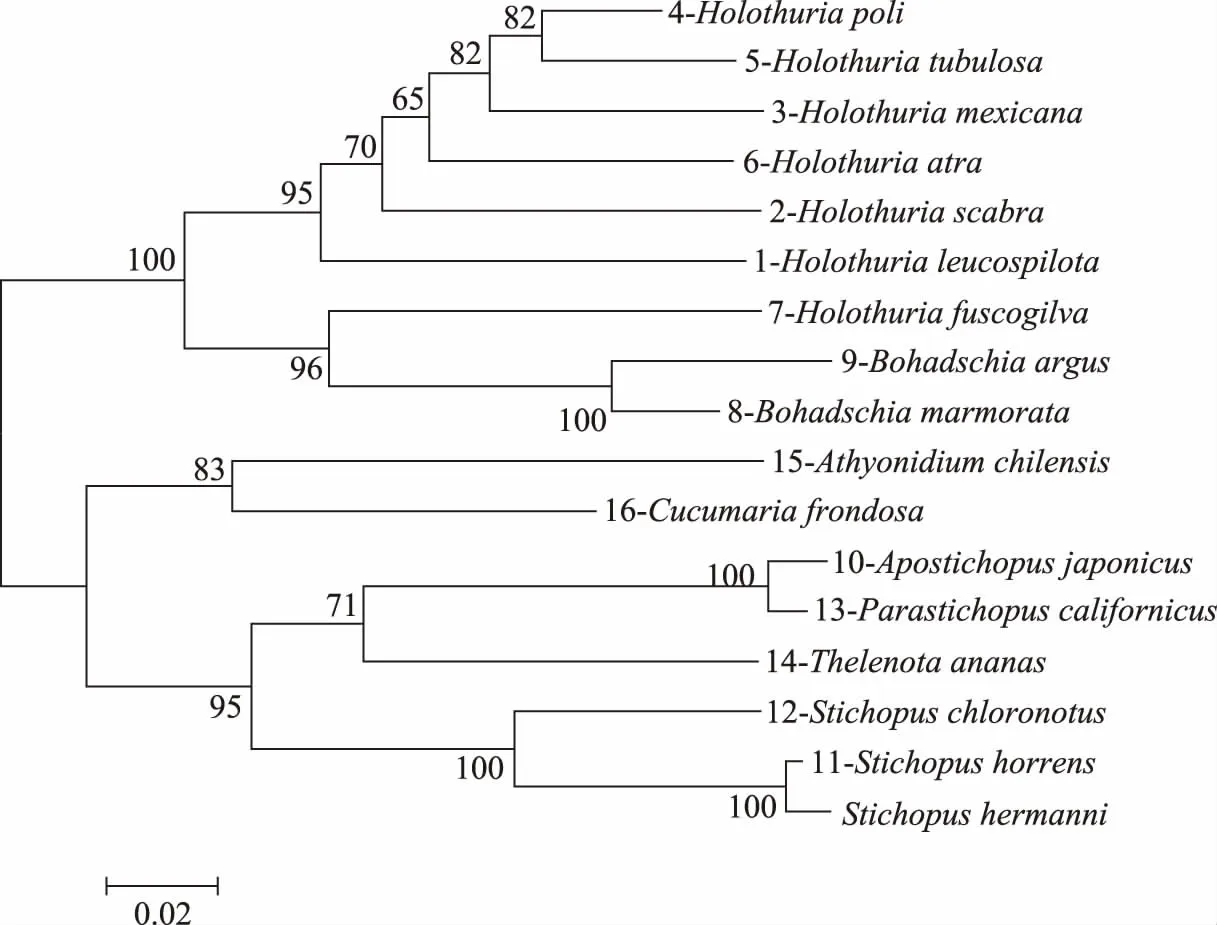
图3 基于16S rRNA基因构建的海参物种N-J系统发育树Fig.3 Neighbor-joining phylogenetic tree of sea cucumbers based on 16S rRNA gene
本研究基于COI和16S rRNA基因的系统发育树显示糙刺参、单疣刺参和花刺参的亲缘关系较近,与Uthicke等[26]和文菁等[33]的研究结果一致。Uthicke等[26]研究了47种海参的COI序列和系统进化关系,丰富了海参COI序列数据,基于遗传距离的系统发育树也表明COI可用于大部分海参的物种鉴定,系统发育树中糙刺参和单疣刺参聚为单支。文菁等[33]基于16S rRNA基因序列对我国15种重要经济海参物种的亲缘关系进行了研究,分子系统发育的结果符合传统的形态学分类结果,在系统发育树中花刺参与糙刺参两者聚为一簇。因此COI或16S rRNA基因对于亲缘关系较近的部分海参物种,即种间序列差异较小的海参物种无法实现准确鉴定,说明单一靶标在DNA条形码技术应用于海参物种鉴定方面存在一些不足。未来DNA条形码技术的发展趋势之一是多个基因联合标记的DNA条形码物种鉴定方法[34],以鉴定进化关系近、序列差异小的近源种。本研究建议在海参物种鉴定中,综合COI和16S rRNA两种靶基因,然后在GenBank和BOLD两个数据库进行比对,选择一致的物种作为鉴定结果。同时,本研究首次基于16S rRNA基因序列分析海参属的黄乳海参与其他海参物种的亲缘关系,与COI序列的系统发育树不同,在16S rRNA基因序列的系统发育树中黄乳海参与白尼参属先聚为一簇,再与海参属聚为一簇,其原因还有待进一步研究。
2.4 市售海参产品标签名称符合性分析
采用建立的DNA条形码方法对24份市售海参进行鉴定,结果表明基于COI序列24份海参产品中21份可鉴别出明确物种(3号墨西哥小有刺参、9号猪婆参、18号刺参除外);而基于16S rRNA基因序列可鉴别出22份海参(9号猪婆参和10号黑香参样品除外)(表3)。将两个靶基因的鉴定结果结合起来,除了9号猪婆参样品无法得到鉴定,其余23份海参均可得到海参种名。9号猪婆参COI和16S rRNA基因的鉴定结果均显示其物种序列相似度小于90%,可能与两个数据库中COI序列和16S rRNA基因序列缺乏有关。

表3 市售海参样品的鉴定结果Table 3 Identification results of commercial sea cucumber samples
在被测的24份市售海参产品中,大部分海参标注的为俗名。将DNA条形码鉴定结果与标签名称比对,10份样品标签名称与DNA条形码鉴定结果一致,6份样品与DNA条形码鉴定结果不一致(8号黑海参、9号猪婆参、11号靴参、18号刺参、20号花刺参和21号白刺参),如11号样品将阿氏辐肛参(Actinopygaagassizi)标为价格更高的白底辐肛参(Actinopygamauritiana),18号样品将经济价值较低的、形态类似的糙刺参(Stichopushorrens)标记为经济价值较高的仿刺参(Apostichopusjaponicus),21号样品将挪威拟刺参(Parastichopustremulus)标为花刺参(Stichopusherrmanni),存在虚假标识,以次充好,标签混乱的情况。24份样品中,有8份只标注了某类海参的总称,既非俗名,也非分类学上的物种名称,如1号秃参、2号和3号小有刺参、4号小黑刺参、7号米刺参、10号黑香参、12号海参丝和23号梦贝海参,利用DNA条形码技术对其鉴定可得到海参物种的准确信息(表3)。
3 结论
以COI和16S rRNA基因作为靶基因建立的DNA条形码方法可有效适用于海参物种鉴定。对于某些亲缘关系较近的海参物种,应结合COI和16S rRNA的鉴定结果,加强多基因条形码技术的研究,弥补单一靶标的局限性,使鉴定结果更加准确。此外,DNA条形码数据库的完整性直接关系DNA条码方法的适用性,应进一步完善DNA数据库,以充分发挥其在海参物种鉴定中的作用。基于建立的DNA条形码方法对市售海参样品鉴定,结果大部分市售海参的标签符合性较差,存在标识混乱、标签错误的现象。由于消费者对海参品种的认识较少,加之海参市场监管的不完善,经销商以贴错标签的形式用价格相对便宜的低值物种替代高值物种欺骗消费者或者滥用海参的方言名称迷惑消费者,通过不同种海参间的价格差获取巨大的经济利益。鉴于上述原因,海产品正确的标签对海鲜贸易的经销商、消费者和食品监管部门来说都是必不可少的,因此必须要加强海参的标签符合性监管。本研究中的DNA条形码方法可对海参品种进行鉴定,为海参进出口检测和市场监管提供科学依据。
