定远猪CATSPER1基因多态性及与产仔数的相关分析
2019-06-19李庆岗田广友吴义景苏世广
李庆岗,田广友,吴义景,苏世广,许 娟
(1.安徽省农业科学院畜牧兽医研究所,安徽 合肥 230031;2.定远县安康农牧有限公司,滁州 233200)
CATSPER离子通道蛋白是由Ren等(2001)首次发现的[1],后来发现这个离子通道是一种复杂的蛋白质复合物,由至少6个亚单位,其中4个α亚单位(CATSPER 1-4)形成钙选择孔[2],2个附加辅助子单元CATSPERB和CatSperG是跨膜蛋白,具有较大的细胞外结构域[3]。人和小鼠的研究表明,CATSPER家族主要表达于睾丸组织[4],而猪CATSPER1基因的表达不仅仅限于睾丸组织,在猪附睾、下丘脑、子宫颈和子宫角都有表达[5-6],因此CATSPER1除了对雄性个体精子运动和活力有关外[7],可能对雌性的繁殖功能及生长性能有关。猪的CATSPER1基因位于2号染色体上,包含12个外显子,共编码722个氨基酸,本课题组在猪的重测序过程中发现该CATSPER1基因第一外显子存在1个错义突变,c.A779G(NM_001244257),该位点在 SNP 数据库中的编号为rs324850126,可导致第227位氨基酸H突变为R。因此,本研究以CATSPER1基因作为母猪繁殖性能的候选基因,研究定远猪母猪c.A779G位点多态性,并分析该位点多态性与初产母猪的产仔数相关性,以期寻找影响产仔数的主效位点。
1 材料与方法
1.1 试验材料
定远猪猪群饲养在定远县安康农牧有限公司,统计2018年5—12月期间264头初产母猪的产仔数,并采集基础群264头母猪耳组织样品,迅速放入装有75%酒精的离心管中,送回实验室,并冷冻保存,用于提取基因组DNA。基因型分析于2019年2—3月完成。
1.2 试验方法
1.2.1 DNA提取 采用苯酚-氯仿抽提法,TE溶解,-20℃冷冻保存。
1.2.2 引物设计合成 根据CATSPER1基因DNA序列,应用Primer premier 5.0软件在多态位点(rs324850126)两侧,设计一对引物,突变位点可用限制性内切酶HhaI识别,由于该突变位点下游15 bp处仍有2个酶切位点,因此将下游引物进行了两处编辑(引物CATSPER1-R第15碱基由G改为T,第30位G碱基改为A),使HhaI只有突变位点一个识别位点,并在5'端增加了7个A,引物序列见表1。引物由上海生工生物工程技术服务有限公司合成。
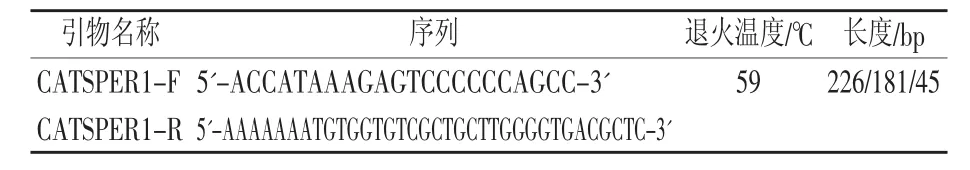
表1 CATSPER1基因引物序列
1.2.3 PCR扩增 PCR反应体系均为(20 μL):2×PCR Mix 10 μL,上游引物和下游引物各 0.5 μL,DNA模板 1 μL,加dd H2O至20 μL。PCR反应条件为:变性和延伸温度分别为95℃和72℃;退火温度为59℃,变性、退火、延伸时间均为30 s,35个循环;最后72℃延伸5 min。
1.2.4 限制性内切酶酶切(RFLP) CATSPER1基因可用限制性内切酶HhaI进行酶切。酶切反应体系均为 10 μL,其中内切酶 0.3 μL(3 U),10×Buffer 1 μL,PCR产物5 μL,加dd H2O至10 μL。反应温度均为37℃,酶切过夜。
1.2.5 统计分析
(1)基因效应估计如下[8]。
显性效应:d=AG-(AA+GG)/2;加性效应:a=(AA-GG)/2;显性度:D=d/a。
A 等位基因的平均效应:α1=q[a+d(q-p)];G 等位基因的平均效应:α2=-p[a+d(q-p)]。
A等位基因替代G等位基因的平均效应值α=α1-α2=a+d(q-p)。
以上各式中,p为A等位基因频率,q为G等位基因频率,AA、AG、GG为各基因型对应性状的最小二乘均值。
(2)模型构建。
用于基因型效应分析的母猪初产繁殖性状包括总产仔数、产活仔数,采用PASW Statistics 18软件中的一般线性模型(GLM)对CATSPER1基因多态性与部分繁殖性状的相关性进行最小二乘分析,所用模型如下:
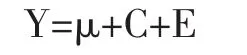
其中Y为性状观察值(总产仔数或产活仔数);μ为群体均值;C为CATSPER1基因型效应;E为随机残差效应。
2 结果与分析
2.1 CATSPER1基因PCR扩增
CATSPER1基因PCR扩增见图1,PCR扩增后为226 bp单一条带,无杂带,可用于RFLP酶切。
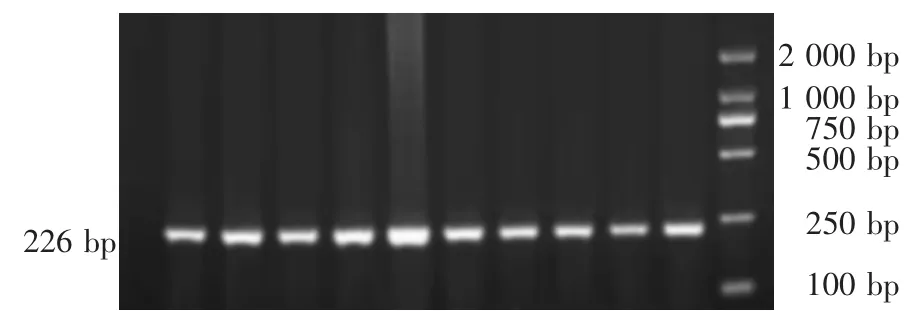
图1 CATSPER1基因PCR扩增电泳
2.2 CATSPER1基因型判断
CATSPER1基因PCR扩增产物经HhaI限制性内切酶酶切后1.5%琼脂糖凝胶电泳,见图2,第1道为DL 2 000 Marker,酶切后会出现3条带,分别为226 bp、181 bp和45 bp,由于45 bp条带太短,无法通过琼脂糖凝胶进行检测,因此利用226 bp和181 bp两条带即可判断基因型。含有226 bp条带的个体为AA型,如图2中第13泳道;含有181 bp和45 bp(图中未显示)的个体为 GG型,如图2中2、3、5、6、10、11、12 泳道;同时出现 226 bp、181 bp 和 45 bp条带的个体为AG型,如图2中第4、7、8、9泳道。
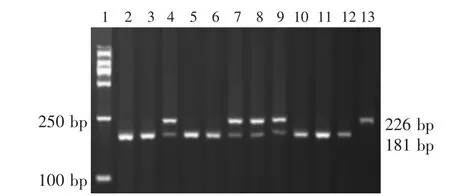
图2 CATSPER1基因HhaI酶切电泳
2.3 CATSPER1在定远猪中的基因型和等位基因频率
CATSPER1基因在定远猪中的等位基因和基因型频率分布情况见表2。由表2可知,CATSPER1基因A 和 G 的等位基因频率分别为 0.166 7、0.833 3,3 种基因型AA、AG和GG的检测频率分别为0.030 3、0.272 7 和 0.697 0,而它们的期望频率分别为 0.027 8、0.277 8和0.694 4,基因型频率检测值和期望值经χ2适合性检验,差异不显著(P>0.05),表明 CATSPER1基因在该群体中分布处于哈德-温伯格平衡状态。

表2 定远猪CATSPER1基因频率及基因型频率分布
2.4 CATSPER1对定远猪初产繁殖性状的效应分析
CATSPER1基因的AA型比AG、GG型个体的初产总产仔数分别高1.74、2.08头,均差异极显著(P<0.01),AG 型个体总产仔数高于 GG 型个体,但差异不显著(P>0.05);AA 型个体初产活仔数比 AG、GG型个体分别高 1.61、1.66头,均达到显著水平(P<0.05),而 AG 和 GG 型个体间差异不显著(P>0.05),详见表3。CATSPER1基因对总产仔数、产活仔数表现出负的显性效应和正的加性效应;等位基因A对总产仔数、产活仔数具有正效应,分别为0.478和0.258;等位基因G对总产仔数和产活仔数具有负效应,分别为-0.096 和-0.052;该基因座上基因型对总产仔数和产活仔数的加性效应值分别为0.573和0.310,因此选择AA基因型个体的留种可提高群体的总产仔数和产活仔数,等位基因A为该群体的优良等位基因。

表3 CATSPER1基因多态性与产仔数相关及基因效应分析
3 讨论
定远猪是中国江淮流域优良的猪种之一,由于其生长速度慢、瘦肉率低、饲料报酬低,导致社会饲养量锐减。近几年,定远猪不断进行产业创新模式的探索,形成了规模化养殖和产业开发相结合,形成了从原种、祖代和父母代的繁育体系。繁殖性能好一直以来都是我国地方品种的优良特性,定远猪初产总产仔数 10.9~11.3 头,初产活仔数为 10.4~10.7头[8-9],而本研究中统计分析了264窝未经选育的初产定远猪产仔情况,发现初产总产仔数为10.45头,产活仔数仅为9.65头。产仔数性状是微效多基因控制的复杂数量性状,遗传力低,通过表型选育,进展缓慢,因此开发更多的分子标记,根据标记进行选择显得尤为重要。CATSPER1基因是一种钙离子通道蛋白,不仅表达于公猪睾丸组织中,在下丘脑和母猪子宫颈、子宫角中也有表达,因此推广与母猪的繁殖性能可能存在一定的关系。本研究发现c.A779G位点在定远猪群体中存在多态性,并处于哈德-温伯格平衡状态,说明该群体对该位点未进行选择。多态性与产仔数间的相关分析表示,AA型个体总产仔数和产活仔数最高,AG型次之,GG型个体最少。AA型总产仔数均极显著高于AG和GG型(P<0.01),AG和GG型个体间差异不显著(P>0.05);AA型产活仔数均显著高于 AG 和 GG 型(P<0.05),AG 和GG型间差异不显著(P>0.05)。等位基因A对定远猪总产仔数、产活仔数具有正效应,等位基因A替代G的效应值分别为0.573和0.310,因此等位基因A是定远猪的优良等位基因,AA型是定远猪的优良基因型。选择AA型个体留种可提高群体的产仔数,在今后选育中可适当提高等位基因A的频率。
