5种楠木木材DNA的提取与条形码鉴定
2019-06-05乔梦吉陈柏旭符韵林
乔梦吉 陈柏旭 符韵林
( 1. 广西大学林学院,广西 南宁 530004;2. 广西高校林业科学与工程重点实验室, 广西 南宁 530004;3. 广西壮族自治区木材产品质量监督检验站, 广西 南宁 530022)
长期以来,木材识别技术对于木材的合理加工和使用,保护珍稀濒危树种,规范市场秩序和维护消费者利益等方面一直具有重要意义。传统的木材解剖识别方法,通过观察木材的宏观及微观构造进行识别,要求木材鉴定者具有深厚的木材专业知识和丰富的经验。近年来,随着分子生物学的发展,一些学者开始尝试运用以DNA条形码为代表的分子生物学手段进行木材的树种识别[1-5]。由于木材本身DNA含量低且降解严重,高质量木材DNA的提取一直是一个很难突破的技术难题,阻碍了分子生物学在木材识别中的应用。1998年,De Filippis等[6]利用改良的CTAB法成功提取出刺槐(Robinia pseudoacacia)的木材DNA。之后,其他学者陆续采用改良的CTAB法或商业试剂盒从不同木材中提取出可满足PCR扩增的DNA,利用分子标记技术实现了一些木材材种的鉴定[7-13]。近年来,我国也有一些研究人员把DNA条形码技术应用到了木材的鉴定工作中,并取得一定的成果[14-19]。以上研究结果均证明了分子生物学技术在材种识别领域应用的可行性。
楠木作为我国非常重要且珍贵的木材,普遍流通于木材市场中,是名贵的家具和建筑用材[20]。目前,人们对“楠木”的概念主要持有2种观点:第1种观点认为楠木包括樟科(Lauraceae)中楠属(Phoebe)的所有树种和润楠属(Machilus)的部分树种[21-22]。第2种观点则认为楠木是指楠属的所有树种[23]。楠属的树种约有94种,在亚洲和非洲均有分布,我国约有34种,主要分布于长江以南地区。润楠属的树种多达100多种,主要在亚洲的热带及亚热带地区分布,我国约有84种。我国楠属和润楠属的树种种类虽然很多,但数量其实很少,尤其是楠属中的一些树种已经成为濒危树种,被列为国家II级保护植物[22,24]。这些木材的材性和解剖结构特性非常相似,准确识别它们一直以来都具有一定的难度。因此,本研究通过对比不同DNA提取方法的提取效果,探索适合楠木木材DNA提取的方法,同时尝试用DNA条形码的手段识别几种木材,为楠木木材的分子鉴定提供参考。
1 材料与方法
1.1 实验材料
本研究的实验材料选取闽楠(Phoebe bournei)、桢 楠 (Phoebe zhennan)、 紫 楠 (Phoebe sheareri)、建润楠(Machilus oreophila)和刨花润楠(Machilus pauhoi)5个树种。闽楠、桢楠、紫楠属于樟科(Lauraceae)楠属(Phoebe),建润楠和刨花润楠属于樟科润楠属(Machilus),材料来源见表1。每个树种选择4~5株树,砍下侧枝并锯成段,装袋保存好带回实验室。
1.2 实验方法
1.2.1 木材样品的处理及DNA提取
新鲜木材:取适量的木材样品,剥除树皮,用70%乙醇进行表面消毒,用消毒过的工具刀片将样品削成木屑,在研钵中加入液氮后快速反复研磨,使木屑充分粉碎至粉末状。随后采用以下3种方法分别进行木材DNA提取,每种方法3个重复。

表 1 实验样品来源Table 1 The list of experimental materials and its collection sites
干燥木材:为了比较不同干燥温度对木材DNA提取效果的影响,根据实际生产和实验中常用的木材干燥温度,设置了30、70、103 ℃不同的干燥温度。选择闽楠作为实验材料,首先将试材切割为规格3 cm×3 cm×3 cm的小方块,然后分别采用3个不同的温度对木材进行干燥处理,每个温度干燥9个木块。待木材达到绝干状态后,分别采用以下3种方法提取DNA,每种方法3个木块作为重复,提取前的处理方法同前。
1)DNeasy Plant Mini Kit(QIAGEN)提取法。采用Qiagen公司的DNeasy Plant Mini Kit试剂盒进行DNA提取。快速称取约100 mg刚研磨好的木材粉末至1.5 mL的离心管中,加入400 μL Buffer AP1和4 μL RNase A,涡旋混匀,65 ℃水浴2 h,水浴期间颠倒混匀4~5次;加入130 μL Buffer P3,混合均匀,用冰孵育5 min;将混合物以14 000 r/min的速度离心5 min;吸取上清液至QIAshredder spin column管中,将管置于2 mL收集管上,以14 000 r/min的速度离心2 min;将收集管中的液体转移至新管,加入1.5倍的Buffer AW1,吸打混匀;吸取650 μL混合液至放置于2 mL收集管中的DNeasy Mini Spin Column管中,以10 000 r/min的速度离心1 min,弃收集液。将剩余的混合液重复此步骤。将DNeasy Mini Spin Column放置于 2 mL的收集管中,加入500 μL Buffer AW2,以10 000 r/min的速度离心1 min,弃收集液;加入 500 μL Buffer AW2,以 14 000 r/min 的速度离心2 min;将DNeasy Mini Spin Column放入一个新的1.5 mL的离心管中;加入60 μL Buffer AE,室温放置5 min,以10 000 r/min的速度离心1 min,收集溶液,重新加到DNeasy Mini Spin Column中,重复此步骤1次,收集溶液,于-20 ℃保存。
2)CTAB法。本研究采用的CTAB法参照刘金良[25]的方法并略有改动。65 ℃预热2% CTAB提取缓冲液。快速称取约150 mg刚研磨好的木材粉末至2 mL的离心管中,加入800 μL 2% CTAB缓冲液和0.02 g PVP,混匀;65 ℃水浴2 h,水浴期间混匀4~5次;冷却至室温,加入等体积氯仿/异戊醇(24∶1)混合溶液,颠倒混匀;12 000 r/min离心5 min;吸取上清液,重复前2步;吸取上清液至新的离心管中,加入2/3体积冷冻异丙醇,轻缓颠倒混匀,冰箱冷冻4 h以上;10 000 r/min离心10 min,弃上清;用2 mL冷冻70%乙醇溶液洗涤沉淀2次;在超净工作台上晾干至无乙醇残留,加入90 μL TE溶液溶解DNA,-20 ℃保存。
3)SDS-CTAB法。本研究采用的CTAB法参照刘金良等[16]的方法并略有改动。65 ℃预热2%CTAB提取缓冲液。快速称取约150 mg刚研磨好的木材粉末至2 mL的离心管中,加入900 μL 2% CTAB 溶液、0.02 g PVP、20 μL β-巯基乙醇,2 μL 蛋 白 酶 K(20 mg/ mL), 混 匀 ; 加 入 35 μL 20% SDS,再次混匀,65 ℃水浴2 h,水浴期间混匀4~5次;冷却至室温,加入900 μLV(酚)∶V(氯)∶V(异戊醇)为25∶24∶1的混合溶液,颠倒混匀5 min;13 000 r/min离心20 min,吸取上清液至新的离心管中;加入等体积V(氯仿)∶V(异戊醇)为24∶1的混合溶液,颠倒混匀,13 000 r/min离心20 min,吸取上清液至新的离心管中;加入140 μL 10% CTAB溶液和280 μL 5 mol/L NaCl溶液,颠倒混匀,13 000 r/min离心20 min,吸取上清液至新的离心管中;重复步骤5。加入等体积冷冻的异丙醇,轻轻晃动,于-20 ℃冷冻4 h以上;13 000 r/min离心20 min,弃上清液;用2 mL冷冻70%乙醇溶液洗涤沉淀2次;在超净工作台上晾干至无乙醇残留,加入90 μLTE溶液溶解DNA,-20 ℃保存。
1.2.2 PCR扩增、产物纯化及测序
选取4段DNA条形码作为候选条形码,分别是叶绿体基因组上的matK基因片段、rpoB基因片段、trnL-trnF片段(trnL基因与trnF基因之间的非编码区序列)和trnL-intro(trnL基因内含子区)。以各样品的木材总DNA为模板,分别以4段DNA条形码的对应引物进行PCR扩增,引物序列见表2。反应体系(25 μL)为:10×PfuBuffer with MgSO42.5 μL,PfuDNA 聚合酶 (2.5 U/μL)0.5 μL,正反向引物 (10 μmol/L)各 1 μL,模板DNA 1 μL,无核酸酶水 19 μL。反应程序如下:94 ℃预变性5 min,接着每个循环94 ℃变性30 s,54 ℃退火30 s,72 ℃延伸1 min,共35个循环,最后72 ℃延伸10 min。PCR产物首先采用1%琼脂糖凝胶电泳进行检测,然后采用Biospin PCR产物纯化试剂盒对PCR产物进行纯化,纯化后的PCR产物送至北京六合华大基因科技股份有限公司进行测序。
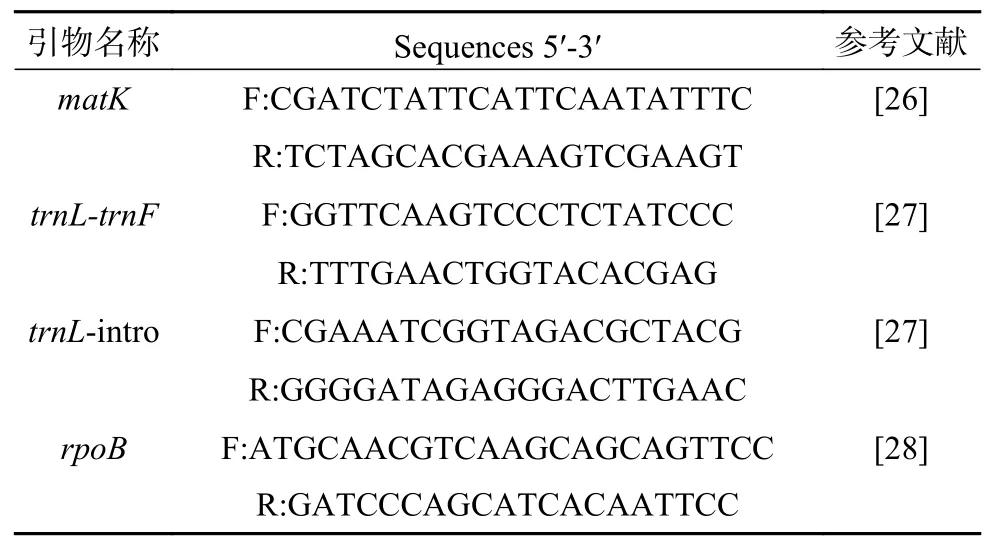
表 2 所用引物序列Table 2 The list of primers
1.2.3 序列分析及系统发育树的构建
测序得到的序列采用Vector NTI 10软件进行DNA序列分析及多重比对。将所得序列中碱基差异较大的DNA条形码序列用MEGA 5.2进行系统发育分析和进化树的构建,采用邻接(NJ)法构建系统发育树,各分支的置信度采用1 000次抽样自举检验分析(boot-strap test)。
2 结果与分析
2.1 不同提取方法的DNA提取结果
由图1和表3可以看出,对于新鲜木材而言,3种方法都可以提取出质量良好的DNA。DNeasy Plant Mini Kit试剂盒提取的DNA完整性好,电泳条带清晰,没有降解。CTAB法和SDSCTAB法提取的DNA整体上比试剂盒提取的量多,电泳条带明亮,略有降解。从表2中测得的OD260/280数据(3个重复的平均值)可以看出,DNeasy Plant Mini Kit试剂盒提取的DNA,OD260/280值多小于1.6,说明有蛋白残留;而CTAB法和SDS-CTAB法提取的DNA多介于1.8~2.0之间,说明有RNA存在。
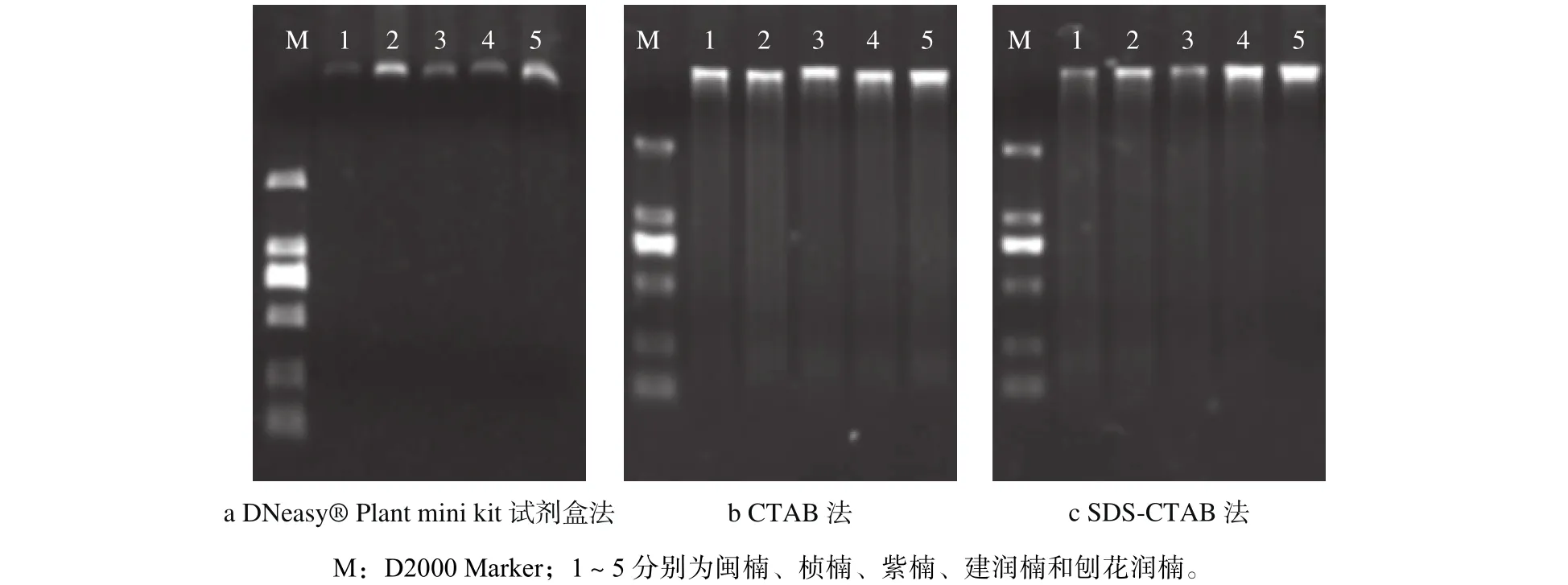
图 1 不同方法提取的楠木木材DNA电泳Fig. 1 The DNA from timbers extracted by different methods
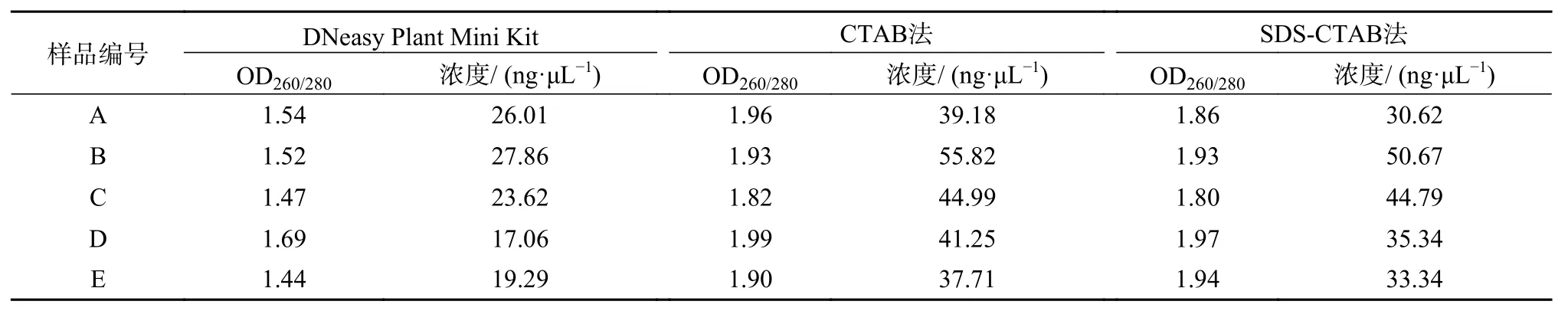
表 3 不同方法提取的楠木木材DNA浓度及纯度Table 3 Concentration and purity of DNA from timber extracted by different methods
而对于干燥木材,由图2和表4可以看出,30 ℃和70 ℃干燥处理后,3种方法都可以提取出质量较好的木材DNA。相对于新鲜木材而言,干燥木材DNA的浓度明显下降,DNA略有降解,但整体质量较好。CTAB法和SDS-CTAB法提取的DNA浓度比试剂盒高,这与新鲜木材提取的DNA情况相同。从测得的OD260/280数据也可以看出,在30 ℃和70 ℃干燥处理后,3种方法提取的DNA质量与新鲜木材提取的DNA质量接近。而经过103 ℃干燥的木材则较难提取到质量良好的DNA。从电泳图(图2)可以看出,3种方法提取的DNA浓度都非常低,在电泳图上几乎看不到。为了检测DNA是否能够满足PCR的要求,对103 ℃干燥后提取的DNA进行了matK片段的PCR扩增。PCR结果显示(图3),电泳条带明亮,特异性好,证明DNA的完整性尚好,虽然模板DNA浓度很低,但仍可以满足分子鉴定的需求。

图 2 干燥处理后采用不同方法提取的木材DNA电泳图Fig. 2 The DNA extracted by different methods after drying at different temperature

表 4 干燥处理后木材DNA的纯度和浓度Table 4 The purity and concentration of DNA from timbers after drying
3种方法对比而言,DNeasy Plant Mini Kit试剂盒提取更为方便快捷且效果较好,但DNA浓度相对较低,且有蛋白残留。CTAB法和SDS-CTAB法提取的DNA量较多,总体质量较好,但步骤较为繁琐。从测定结果和电泳结果来看,CTAB法和SDS-CTAB法提取的DNA差异不明显。鉴于实际鉴定工作的需求,综合考虑提取效果、操作步骤和成本因素,CTAB法是最理想的提取方法。

图 3 以103 ℃干燥木材DNA为模板的matK片段PCR扩增Fig. 3 PCR-amplified product of matK sequence using the DNA from 103 ℃ drying timbers as templates
2.2 PCR扩增结果
PCR扩增结果(图4)显示,所有木材DNA样品均能扩增出清晰且单一的条带。matK序列的PCR产物长度为750~1 000 bp,trnL-trnF序列的PCR产物长度为250~500 bp,trnL-intro和rpoB序列的PCR产物长度为500~750 bp。
2.3 PCR产物测序与序列分析
将PCR产物测序结果进行整理分析,在NCBI上进行BLAST相似性检索,每段序列都有很高的匹配程度,表明测序结果准确可靠,所测序列为目标序列。将每个树种的几个样品之间的测序结果比对,发现每个样品的序列都完全一致,即种内没有发现变异。将5个树种的各段DNA条形码PCR产物测序结果进行比对分析,得出以下结果,见表5。
从matK序列的测序及比对结果中可得,楠属的闽楠、桢楠、紫楠的matK序列长度为882 bp,润楠属的建润楠和刨花润楠的matK序列长度分别为883 bp和884 bp,共有3个特异性位点。在第168位点和849位点处可以将楠属和润楠属区分开来,在第14个位点处可以将刨花润楠单独区分出来。但是其他树种不能区分。
从trnL-trnF序列的测序及比对结果中可得,5个树种的trnL-trnF序列长度均为375 bp,有2个特异性位点。在第97位点处和第267位点处,可以将两个属区分开,种间无法区分。

图 4 4段DNA条形码的PCR结果电泳Fig. 4 Agarose gel electrophoresis of DNA extracts and PCR products
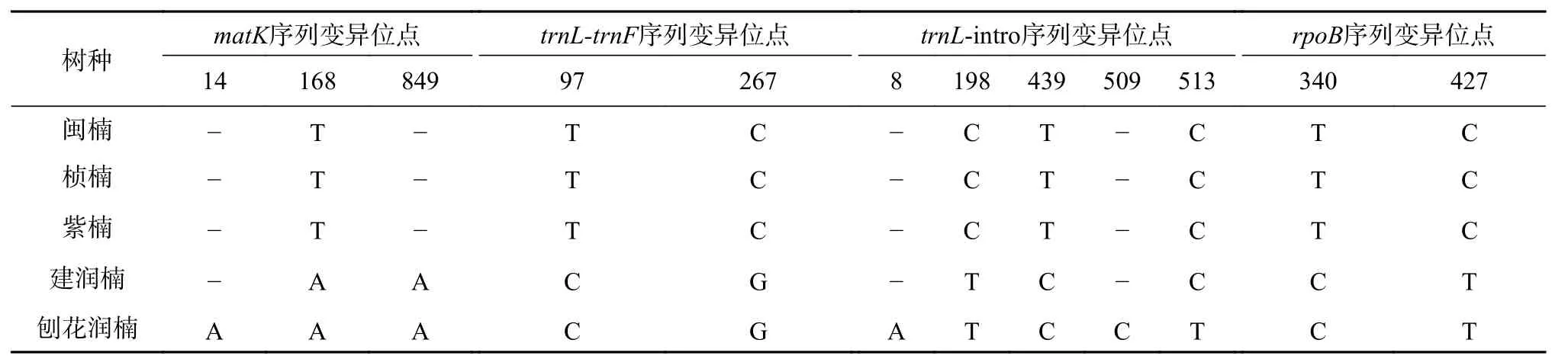
表 5 各序列变异位点Table 5 Variation sites of sequences
从trnL-intro序列的测序及比对结果中可得,闽楠、桢楠、紫楠、建润楠在trnL-intro扩增的DNA序列长度为均为514 bp,刨花润楠在trnL-intro扩增的DNA序列长度为516 bp,共有5个特异性位点。在第198位点和第439位点处,可以将楠属与润楠属之间区分开来。在第8位点、第509位点和第513位点处,可以将刨花润楠单独区分出来。但其他几个树种不能单独区分。
rpoB序列的测序及比对结果得出,5个树种的DNA序列长度均为498 bp,有2个特异性位点。在第340和第427这两个特异性位点处,能对5个树种进行属间的区分,不能进行种间的区分。
整体来看,本研究所选取的几段DNA条形码都不能单独区分开5种楠木。即使将几段DNA条形码结合起来分析,也只能区分开两个属,其中润楠属的2个树种能够区分开,但楠属的3个树种之间仍旧难以区分。
2.4 系统发育树的构建
将樟科樟属植物三桠乌药(Lindera obtusiloba)作为外类群,将5个树种测序所得的4段序列及三桠乌药的4段序列(GenBank:KU645668.1;AB259067.1;AB237276.1;MH220737.1)结合起来用MEGA5.2进行多重比对后采用邻接法(neighbor joining,NJ)构建系统发育树(图5)。结果表明,5种楠木总体聚为3个分支,润楠属聚为1个分支,楠属的桢楠和闽楠聚为一个分支,而紫楠单独为一支。外类群植物三桠乌药单独聚为1枝,这与传统分类结果相一致。说明将4段序列结合起来,能够对这几个树种正确聚类,作为区分几个树种的参考。

图 5 基于trnL-intro、trnL-trnF、matK和rpoB序列构建的系统发育树Fig. 5 The phylogenetic tree obtained by the NJ method based on the trnL-intro, trnL-trnF, matK and rpoB sequence
3 结论与讨论
质量良好的木材DNA是实现分子手段鉴定木材材种的关键。本实验采用了楠木的新鲜木材和干燥后的木材进行DNA提取,结果发现使用3种方法均可从楠木木材中提取出满足PCR扩增需求的楠木木材DNA,以CTAB法和SDS-CTAB法提取效果更理想,去除蛋白质和其他杂质的效果较好。这与刘金良等[16]对针叶木木材的提取结果和伏建国等[17]对黄檀属木材的提取结果一致。余敏等[19]研究发现PTB(N-phenacylthiazolium bromide)试剂对于提取降香黄檀干燥木材的效果良好,在今后的研究中可以借鉴。鉴于实际鉴定工作的需求,综合考虑提取效果、操作步骤和成本因素,CTAB法是目前最理想的提取方法。
樟科植物的起源比较古老,虽然经过漫长的进化,但其遗传变异速率相对较慢。本研究选取了4段DNA条形码序列对5种楠木木材进行分子鉴定,每段条形码在2个属之间都显示出明显的差异,可区分开2个属;但在树种之间差异很小,单个条形码只能区分部分树种,不足以完全区分这5种树种。陈云霞等[29]对30种楠木及其近缘属植物的叶绿体matK基因序列进行了扩增和序列比对,发现matK基因在樟科植物中反映出较低的变化速率,其差异基本可以用于对属间进行鉴定,而种间的鉴定则不可行[25],这与本研究的结果基本一致。除了这些树种之间本身较小的遗传差异之外,扩增的DNA条形码片段并不是完整的基因可能也是一个原因,这也有可能造成信息不够完整,错失了一些可能的变异位点。此外,植物的DNA条形码还有很多,除了本研究选取的几段条形码之外,叶绿体基因组上的ycf3、rpoC1、psbC-trnS等基因片段和核糖体DNA转录间隔区ITS序列也是经常被选用的DNA条形码。鉴于本研究选择的树种有限,对于实际的鉴定工作而言还远不够,因此对于更多的楠属和润楠属的其他树种,想要快速准确的识别出来,则还需要对更多的DNA条形码进行扩增和测序,以积累更丰富的数据用于这些树种的鉴定。
