海南东寨港海水和沉积物中抗生素抗性基因污染特征研究
2019-02-26姜春霞李森楠刁晓平王道儒叶翠杏
姜春霞 ,黎 平 ,李森楠 ,刁晓平, *,黄 炜 ,王道儒,叶翠杏
1. 海南大学热带农林学院,海南 海口 570228;2. 海南大学南海海洋资源利用国家重点实验室,海南 海口 570228;3. 热带岛屿生态学教育部重点实验室,海南 海口 571158;4. 海南省海洋与渔业科学院,海南 海口 570125
抗生素作为养殖业防治细菌性疾病和刺激生长的重要助力,常作为防病药物及饲料添加剂而被广泛应用于水产养殖中。有研究报道,抗生素被摄入动物体内后,有近85%以上的原药会随着动物粪便或尿液排除体外,进入自然环境(Hartmann et al.,1998)。残留在环境中的抗生素会对环境中微生物产生选择压力,从而诱导抗生素抗性基因(Antibiotic Resistance Genes,ARGs)产生。ARGs可以通过转座子、质粒等可移动遗传元件(Mobile Genetic Elements,MGEs)在不同菌株之间进行水平转移(Gogarten et al.,2005;Schlüter et al.,2007),从而使得更多微生物获得抗生素抗性。相关研究表明,在使用过抗生素的养殖环境中,携带ARGs的耐药性菌株的种类和数量日益增加,ARGs将通过食物链最终进入人体,这将给人类健康带来风险隐患。Pruden et al.(2006)认为ARGs是一种新型污染物,世界卫生组织(WHO)也已将 ARGs列为21世纪威胁人类健康的最重大挑战之一(王丽梅等,2010),其污染问题也越来越受到人们的关注。
针对磺胺类、四环素类、氯霉素类、喹诺酮类抗生素的耐药菌通常携带有一种或多种耐药机制的ARGs。包括介导药物靶位点改变机制的基因 sul1、sul1、dfrA1,介导抗生素外排机制基因cmle1、cmle3、tetA、tetC、tetG,介导药物靶位点保护机制基因tetM、qnrS,介导药物活性位点失活的耐药基因cata1、cata2。这些基因在人体、畜禽及多重环境介质分离菌中均被定性检出(李壹等,2016)。研究表明,水产养殖是ARGs的重要来源,许多ARGs在中国乃至世界各地的水产养殖环境或饲养对象中都有检出(Muziasari et al.,2016;Stalin et al.,2016)。然而,目前的研究大多针对畜禽及淡水水产养殖环境中抗生素及ARGs的检测(Wang et al.,2016;Qian et al.,2017;Czekalski et al.,2014;Harnisz et al.,2015;Marti et al.,2018),而对海水水产养殖环境关注较少。
海南省抗生素排放在中国处于中等偏高水平(Zhang et al.,2015),抗生素的高水平排放将会给当地生态环境带来挑战。东寨港位于海南省东北部,是国家级红树林湿地自然保护区,已列入世界重要湿地保护名录,具有重要的生态及经济价值。同时,东寨港海水养殖区是海南重要的滩涂养殖基地,其周边陆地区域也存在大量的养殖塘,该区域抗生素及其引起的ARGs污染不容小觑。在以往的研究中,有关该区域抗生素及ARGs的研究未见报道。基于此,本研究将对该区域抗生素及ARGs进行调查,具体内容包括:(1)分析沉积物和海水中12种 ARGs的丰度、分布特征及;(2)16种抗生素的残留状况;(3)比较东寨港海水和沉积物中ARGs的分布差异;(4)初步分析ARGs丰度与抗生素残留的关系。本研究旨在为规范当地水产养殖管理和评估生态环境现状提供数据基础。
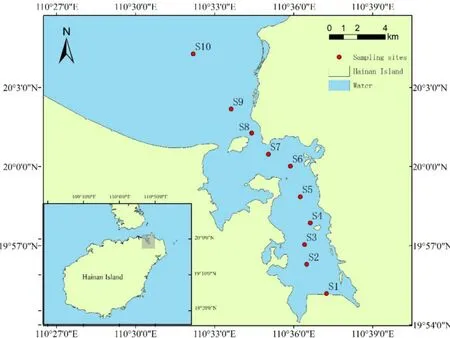
图1 采样点分布图Fig.1 Location of sampling sites
1 材料与方法
1.1 样品采集
东寨港形似漏斗,属溺谷型港湾,海岸线总长84 km,滩涂水域面积5400 hm2(李仕平等,2017)。本研究于2016年6月进行样品采集,采样点如图1所示,共设置 10个采样点(S1-S10),采集海水和沉积物样品。其中,S1靠近内陆;S2-S5浅海区域均有滩涂养殖区,且周围海岸均存在养殖塘;S6、S7靠近养殖塘;S8、S9靠近铺前码头;S10靠近外海,是铺前出海要塞。采样区域由内港向入海口方向延伸,可完整呈现该区域内港至外港抗生素及ARGs的污染趋势及水平。使用采水器采集表层0-0.5 m海水并装入灭菌采样瓶中;用采泥器采集表层沉积物并装入铝盒(121 ℃湿热灭菌)中;所采集样品用于ARGs及抗生素检测。样品采集完毕后立即运回实验室,水样于4 ℃冷藏,沉积物于-20 ℃保存,尽快进行样品处理。
1.2 抗生素的检测
本研究对东寨港沉积物及海水中4类15种抗生素(磺胺二甲基嘧啶、磺胺二甲氧嘧啶、磺胺甲恶唑、磺胺甲基嘧啶、磺胺吡啶、磺胺异恶唑、四环素、金霉素、土霉素、氯霉素、甲砜霉素、氟苯尼考、恩诺沙星、环丙沙星、诺氟沙星)进行检测,检测方法及分析条件如下:
1.2.1 沉积物抗生素检测
将沉积物样品冷冻干燥48 h,过0.30 mm孔径筛,准确称取2 g沉积物至锥形瓶中,向锥形瓶中加入 V(甲醇):V(EDTA-Mcllvaine缓冲液)=1∶1 混合液(10 mL),振荡30 min,超声提取10min,静置,收集提取液。残渣用上述方法反复提取两次,合并提取液。将提取液进行旋转蒸发(水浴40 ℃)、浓缩至约10 mL。将LC-SAX(500 mg/3 mL)与LC-18(500 mg/3 mL)进行串联,固定在萃取仪上,萃取富集。用3 mL甲醇(色谱纯)洗脱LC-18小柱,收集洗脱液。氮吹浓缩洗脱液至近干。用流动相95%A[0.1%甲酸(1∶1,V/V)-1 g·L-1甲酸铵水溶液]和5%B[甲醇-乙腈(1∶1,V/V)]定容至 1 mL,过 0.22 µm尼龙滤膜,待测。
1.2.2 海水抗生素检测
取1 L已过滤水样,用盐酸-水溶液(1∶1,V/V)调节pH至2.5。依次用20 mL甲醇、6 mL超纯水、6 mL盐酸对SPE柱进行活化。用已活化的SPE柱对水样进行富集,流速控制在 10 mL·min-1左右。富集完成后用氮气轻柔吹干富集小柱,将氮吹干燥后的小柱与填有无水硫酸钠的小柱串联,用 6 mL甲醇进行洗脱,收集洗脱液。洗脱液氮吹(水浴40 ℃)浓缩至近干。用流动相95%A和5%B定容至1 mL,过0.22 µm尼龙滤膜,待测。
1.2.3 抗生素分析条件
色谱柱为:ACQUITY UPLC® BEH C18(2.1×100 mm,1.7 µm,美国waters公司);进样量:10 mL;柱温:40 ℃;流速为0.30 mL·min-1。流动相A为含0.2%甲酸铵的超纯水;流动相B为含有甲醇和乙腈的混合物(1∶1,V/V)。梯度洗脱程序为:起始5% B 3 min,然后在18 min内从5% B线性变化至88% B。质谱离子源为:ESI+/ESI-切换,MRM数据采集模式。
本研究采用外标法对抗生素进行定量,用流动相配制一系列不同浓度的混合标准液进行测定。标准曲线以抗生素浓度为横坐标,峰面积为纵坐标,所得标准曲线判定系数均在0.998之上。水样中抗生素的回收率为71.6%-112%,检出限范围为0.10-4.20 ng·L-1。沉积物中抗生素的回收率为 62.1%-95%,检出限范围为0.12-6.3 ng·g-1。
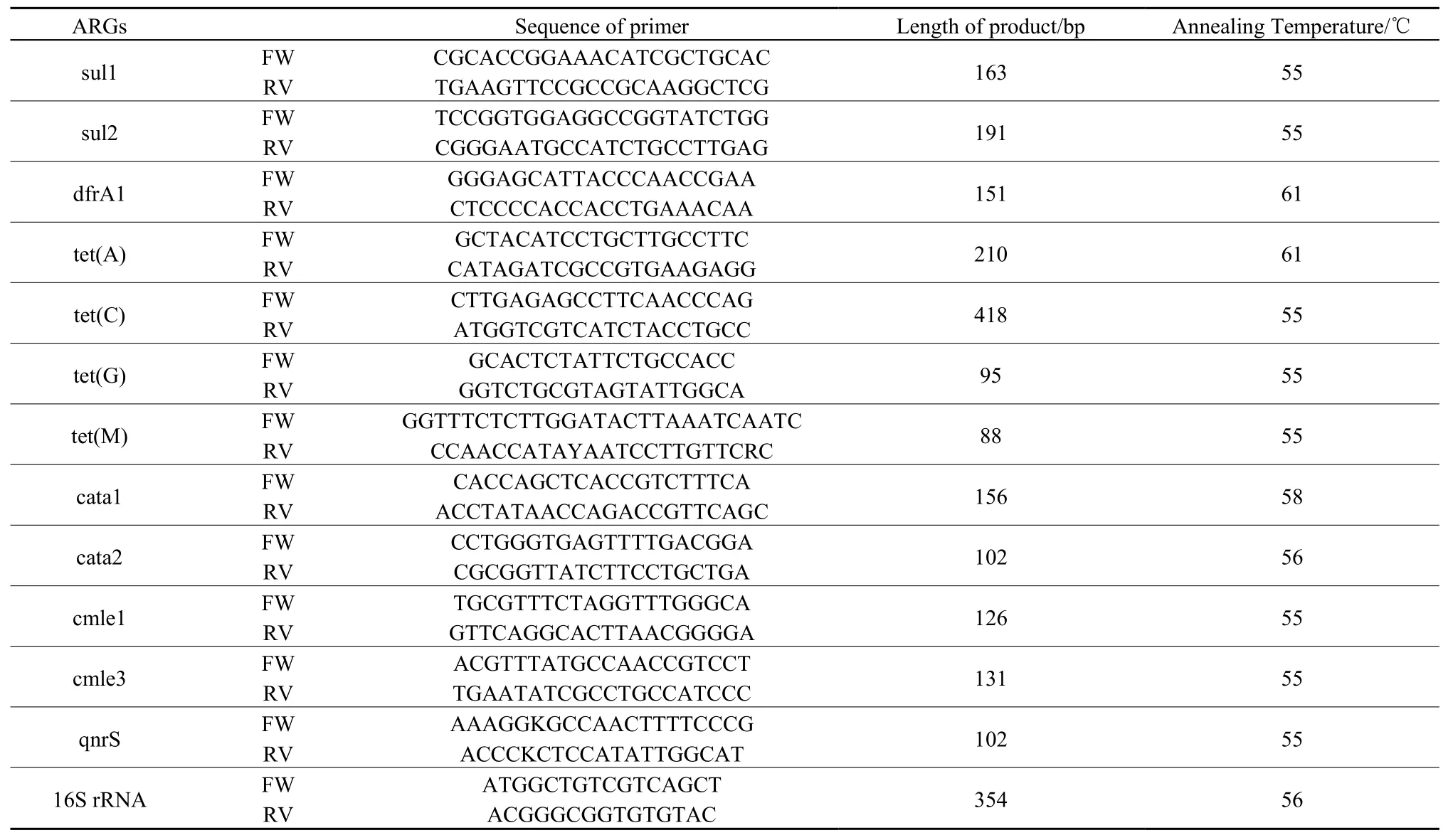
表1 实验所用序列及PCR反应条件Table1 Sequence of primer and condition of PCR reaction in this study
1.3 样品总DNA的提取
1.3.1 沉积物
每个样点设置3个生物学重复,每个重复称取0.25-0.35 g新鲜样品,采用 PowerSoilTMDNA Isolation Kit试剂盒(MoBio,美国)进行沉积物总DNA提取,所得DNA用NanoDrop 2000微量分光光度计(Thermo Fisher Scientific,美国)测其含量及纯度。DNA样品于-80 ℃下保存备用。
1.3.2 海水
于24 h内将获得的水样依次过8 μm、5 μm无菌滤膜以除去杂质,之后用0.22 μm滤膜过滤并收集滤膜,用无菌铝箔包裹,-20 ℃下保存。用液氮将滤膜研磨成粉末状,用PowerSoilTMDNA Isolation Kit试剂盒(MoBio,美国)提取总DNA,并使用NanoDrop 2000微量分光光度计(Thermo Fisher Scientific,美国)测定DNA含量及纯度。DNA样品于-80 ℃下保存备用。
1.4 质粒制备
选取 ARGs(sul1、sul2、dfrA1、tetA、tetC、tetG、tetM、cata1、cata2、cmle1、cmle3、qnrS)和16s rRNA作为目的基因,引物设计详见表1。对样品中12种ARGs目的片段进行PCR扩增,同时进行空白试验。扩增体系为:14.5 μL Green Master Mix,上下游引物各1.5 μL,2 μL DNA模板(质量浓度范围:10-50 mg·L-1),5.5 μL 无酶水。扩增条件为:94 ℃预变性4 min,94 ℃变性30 s,退火温度(见表1)30 s,72 ℃延伸1 min,共35个循环。PCR产物用琼脂糖凝胶电泳进行检测,用SanPrep柱式DNA胶回收试剂盒(生工,上海)对正确的片段进行胶回收。将目的基因连接到PGEM-Teasy载体后导入商品化感受态Trans5α(全氏金,北京)进行克隆。每个平板挑选 40个克隆子进行培养。裂解细菌,使用SanPrep柱式质粒提取试剂盒(生工,上海)进行质粒抽提。抽提得到的质粒使用微量分光光度计测其浓度,保存于-80 ℃下备用。
1.5 RT-PCR
利用一系列稀释后的质粒作为 RT-PCR模板DNA测定其每个反应管内的荧光信号达到设定的阈值时所经历的循环数(Cycle threshold,tC),以拷贝数对数作为横坐标,tC值作为纵坐标构建标准曲线。RT-PCR 反应体系为:5 μL SYBR Green Master Mix,上下游引物(表1)各0.5 μL,2 μL DNA模板(质量浓度范围:10-50 mg·L-1),2 μL 无酶水。反应条件为:95 ℃预孵育10 min,95 ℃变性30 s,退火温度(表1)30 s,共45个循环。溶解条件为:95 ℃ 10 s,65 ℃ 60 s,72 ℃ 30 s。拷贝数计算公式如下:
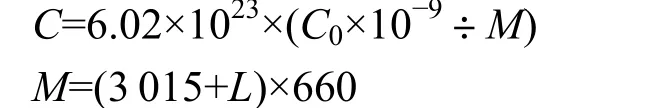
式中,C0为质粒初始浓度;C为质粒拷贝数(copies·g-1或 copies·μL-1);3015 为 PGEM-Teasy载体长度;L代表目的基因长度。
标准曲线结果显示判定系数R2均大于0.99,线性关系良好,可用于各 ARGs拷贝数计算。使用RT-PCR对样品中12种抗性基因和16S rRNA进行绝对定量。将样品得到的tC值带入标准曲线,最终计算出基因绝对丰度。
基因丰度表达有两种表达方式:绝对丰度、相对丰度。绝对丰度表示目的基因在某一环境介质中的含量;而相对丰度表示目的基因的绝对丰度相对于内参基因16S rRNA的变化,可定量表示抗生素对ARGs的诱导率(冀秀玲等,2011),同时ARGs的相对丰度更能反映ARGs在样本中的分布状况。相对丰度计算如下:
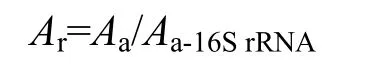
式中,Ar为抗性基因相对丰度;Aa为抗性基因绝对丰度;Aa-16SrRNA为16S rRNA基因绝对丰度。
1.6 数据统计
所有数据运用SPSS 17.0进行统计分析,采用t检验及One-way ANOVA进行差异显著性分析,采用Pearson相关性分析法分析ARGs间或ARGs与抗生素间的相关性。
2 结果
2.1 抗生素残留情况
2.1.1 海水中抗生素的残留及分布
本研究 10个采样点总抗生素质量浓度范围为0.168-12.963 ng·L-1。不同点位抗生素质量浓度高低顺序为:S4 (12.963 ng·L-1)>S5 (7.545 ng·L-1)>S3(6.619 ng·L-1)>S2 (6.274 ng·L-1)>S7 (5.368 ng·L-1)>S9 (5.300 ng·L-1)>S1 (4.910 ng·L-1)>S6 (4.460 ng·L-1)> S8 (3.828 ng·L-1)>S10 (0.168 ng·L-1)。15 种抗生素中有 5种抗生素(诺氟沙星、磺胺二甲基嘧啶、磺胺甲恶唑、磺胺吡啶、磺胺异恶唑)被检出,检出率分别为 70%、80%、100%、60%、20%。其中诺氟沙星质量浓度范围为 ND(未检出)-6.170 ng·L-1;磺胺二甲基嘧啶质量浓度为 ND-0.925 ng·L-1;磺胺甲恶唑质量浓度范围为 0.168-5.735 ng·L-1;磺胺吡啶质量浓度范围为 ND-0.375 ng·L-1;磺胺异恶唑质量浓度范围为ND-0.133 ng·L-1。
2.1.2 沉积物中抗生素的残留及分布
沉积物抗生素检测中,各样点总抗生素质量浓度范围为 ND-1.240 ng·L-1,其中 S5(1.240 ng·L-1)质量浓度最高,S1、S7、S8(低于检出限)最低。15种目标抗生素中,仅有磺胺吡啶、磺胺异恶唑两种抗生素被检出,检出率分别为50%、60%,其浓度范围分别为 ND-0.6、ND-0.64 ng·g-1。
2.2 ARGs丰度及分布特征
2.2.1 海水中ARGs丰度及分布特征
海水中ARGs及16S rRNA绝对丰度及分布特征见图2a。由图可以看出,海水∑ARGs、16S rRNA绝对丰度分别为 8.68×103-1.37×109copies·L-1、1.01×1010-1.37×1011copies·L-1;∑ARGs相对丰度为 8.57×10-7-3.45×10-2,最高∑ARGs 相对丰度(3.45×10-2)出现在S4号采样点,S3次之(2.68×10-2),S10 最小(8.57×10-7)。在 12 个 ARGs中,sul1、sul2、dfrA1、tetA、tetC、tetG、tetM、cata2、cmle1、cmle3检出率为90%,cata1检出率为80%,qnrS检出率为100%,在样点S10处仅有qnrS被检出。在海水样品中,sul2绝对丰度最高(5.13×108copies·L-1),sul1 次之(1.21×108copies·L-1),tetM最低(1.16×105copies·L-1)。
2.2.2 沉积物中ARGs丰度及分布特征
沉积物ARGs绝对丰度以样品干重进行计量,详见图2c。由图可知,沉积物中∑ARGs和16S rRNA绝对丰度分别为 1.13×108-5.34×108copies·g-1、1.33×109-1.24×1010copies·g-1;ARGs 相对丰度为1.57×10-2-1.08×10-1。由图 2c、图 2d 可知,各 ARG在沉积物样品中均有检出,且检出率为 100%,说明检测的 ARGs在沉积物样品中广泛存在。ARGs含量水平表现为:磺胺类抗性基因>氯霉素类抗性基因>四环素类抗性基因>喹诺酮类抗性基因,其中sul2丰度在所有采样点中均为最高,绝对丰度高达5.05×107-2.17×108copies·g-1,cmle3 次之,绝对丰度为 2.73×107-1.40×108copies·g-1。
3 讨论
3.1 东寨港抗生素含量水平及分布
各检出抗生素质量浓度差异不明显(P>0.05)。与高桥红树林区(Li et al.,2016)、渤海湾(Zou et al.,2011)及珠江(He et al.,2012)、汉江(Hu et al.,2018)相比,东寨港抗生素的质量浓度及检出率都处于较低水平。这可能与东寨港区域抗生素间断性输入有关,同时,抗生素自身性质也可能是重要原因之一,比如磺胺类及喹诺酮类抗生素具有光敏感性(Thiele-Bruhn,2003;Park et al.,2002)而四环素类抗生素存在吸附-解析行为(Tenenbaum et al.,2014)。
抗生素在海水和沉积物中的残留量均表现为内港区域高于临海区域,这可能与内湾区域海水养殖和养殖塘排污有关;对各点抗生素质量浓度进行差异分析发现,各采样点抗生素的污染差异不明显(P>0.05)。此外,海水中抗生素残留量高于沉积物,可能与抗生素的水溶性有关。
3.2 东寨港ARGs污染分析
3.2.1 海水中ARGs丰度及分布特征分析
内港区域∑ARGs绝对丰度高于入海口。利用One-way ANOVA对海水中不同点ARGs进行比较分析,发现各采样点之间并无显著性差异(P>0.05),这可能是当地海水频繁交替而加速各样点微生物扩散导致的。采用相对丰度进行比较分析,发现东寨港的sul1、sul2检出丰度(图2b)比山东地区海水养殖区高(李壹等,2016),与海河河水检出丰度相近(Luo et al.,2010),说明该地受到了磺胺类抗生素抗性基因的污染。高拷贝数的sul1和sul2可能是微生物受到磺胺类药物诱导产生的,同时也与 sul基因在环境中代谢速度慢有关(Mckinney et al.,2010)。此外,sul1经常与一类整合子结合在一起(Sköld,2000),两者之间的紧密关系也可能是sul1基因表现出高丰度的原因之一。
3.2.2 沉积物中ARGs丰度及分布特征分析
就绝对丰度而言,内港区域∑ARGs丰度高于入海口区域;而相对丰度呈现相反的趋势(图2c、图2d)。内港区域高水平的ARGs绝对丰度可能与该区域大面积的海水养殖有关;而其较入海口区域低的相对丰度说明内港ARGs在微生物群落中的表达低于入海口区域。对沉积物中不同采样点 ARGs进行差异分析,发现各采样点间的ARGs丰度差异并不明显(P>0.05),这可能与该区域抗生素的污染源分布及当地频繁的水流交换有关。与长江口(Guo et al.,2018)和北江流域(Ling et al.,2013)的研究结果相比,东寨港沉积物中ARGs丰度较高,与灞河相当(Jia et al.,2018)。在研究区域,磺胺类抗性基因(sul1、sul2)含量较其他基因处于较高水平,这与珠江水产养殖区调查结果一致(梁惜梅等,2013)。sul1和 sul2的高水平表达,说明这两种基因的宿主菌对各种环境的耐受能力强,从而促进了这两种基因在环境中的扩散与传播。
本研究表明,东寨港海水和沉积物均受到12种ARGs的影响,与前人研究相比,其丰度处于较高水平。氯霉素是一类广谱性药物,曾被广泛用于畜牧和水产养殖行业,而由于其具有诱发再生障碍性贫血症和致畸等强毒副作用(Turton et al.,2000),中国已于1999年禁止此类抗生素药物在水产养殖行业的使用。但近年来氯霉素乙酰转移酶类耐药基因(cata1、cata2)及编码氯霉素外排泵蛋白类基因(cmle1、cmle3)在海水养殖区仍屡屡被检出(Hu et al.,2008;李壹等,2016),说明历史背景诱导产生的 ARGs仍广泛存在于养殖环境中。在东寨港海水和沉积物中氯霉素类抗性基因(cata1、cata2、cmle1、cmle3)分布极为广泛,与山东地区海水养殖区相比(李壹等,2016),该区域氯霉素类抗性基因相对丰度较高。四环素外排基因(tetA、tetC、tetG)及核糖体保护外排基因(tetM)常常在海水养殖环境及自然环境水体中被检出(Ling et al.,2013;Luo et al.,2010;Mackie et al.,2006),同时也广泛存在于东寨港区域。在东寨港海水和沉积物中,tetC基因含量均比其他四环素类抗性基因丰度高,这一结果与对洪泽湖的研究结果相似(罗方园等,2017)。此外,喹诺酮类抗生素是近年来使用的一类抗生素类药物,但喹诺酮类耐药菌已在多种环境中被检测到。喹诺酮类抗性基因(qnrS)东寨港海水和沉积物中检出率均为100%,其带来的污染不容小觑。
3.2.3 海水和沉积物中ARGs差异分析
采用t检验对两种介质中ARGs进行比较分析,发现两者之间存在明显的差异(P<0.05),其中沉积物的ARGs丰度显著高于海水。这说明抗性基因在沉积物样品中的污染比海水严重,即沉积物中ARGs在微生物群落中的表达高于海水。这一现象可能与沉积物对污染物的强贮存能力和海水的强扩散能力有关,同时也可能与沉积物中较为活跃的微生物基因交流有关。东寨港生态环境复杂,为全面了解当地污染情况,还需进一步研究验证。
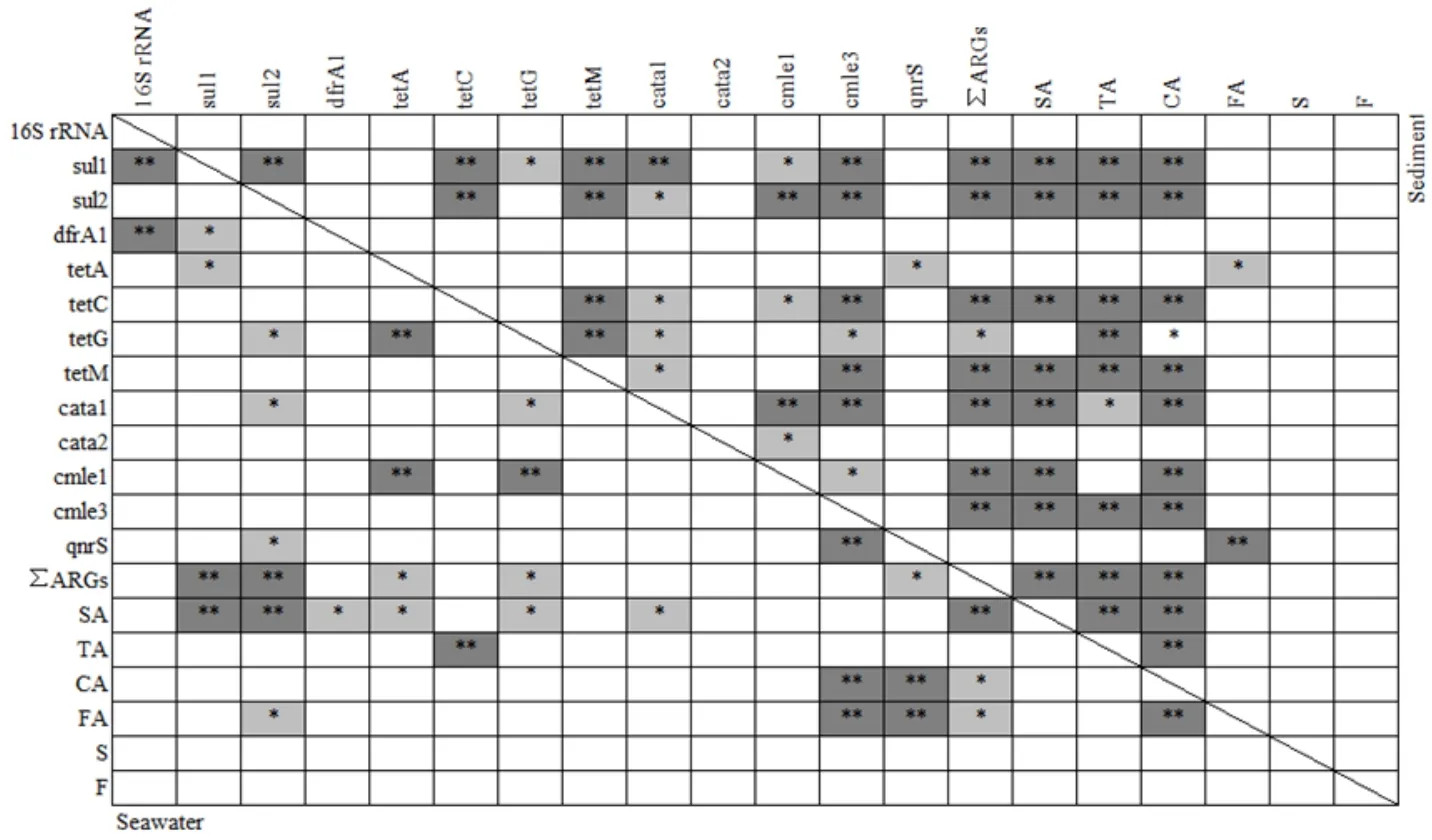
图3 海水及沉积物中ARGs绝对丰度及抗生素含量的相关分析Fig.3 Correlation analysis of ARGs in seawater and sediment.
3.3 海水/沉积物中ARGs、抗生素相关性分析
对东寨港海水/沉积物中ARGs进行相关性分析,结果如图3。海水中SA与∑ARGs之间存在显著的相关性(r=0.990,P<0.01);海水中16S rRNA与sul1和 drfA1均存在显著相关性(r=0.882和 0.799,P<0.01),而与其他基因相关性较弱。在沉积物中,介导抗生素外排机制基因tetC、cmle1、cmle3两两之间存在显著相关性(r=0.731-0.958,P<0.01或P<0.05)。忽略ARGs的作用机制不谈,在海水样品中,sul1、sul2、tetA、tetG与∑ARGs、SA之间显著相关(r=0.657-0.976,P<0.1或P<0.05)。沉积物中sul1及sul2分别与另外5种ARGs(tetC、tetM、cata1、cmle1、cmle3)存在显著相关性(r=0.674-0.964,P<0.01或P<0.05)。此外,sul1、sul2、tetC、tetM、cata1、cmle3与∑ARGs、SA、TA、CA 均存在显著或极显著相关性(r=0.756-0.997,P<0.01或P<0.05),∑ARGs、SA、TA、CA两两之间呈极显著相关(r=0.838-0.986,P<0.01)。根据以上分析结果可知,东寨港海水/沉积物中不同或相同类别的ARGs之间都可能存在相关性,这与灞河的结果类似(Jia et al.,2018),而抗生素产生的选择压力和 ARGs在环境中的转移机制可能是导致这一现象的重要原因。为更全面了解各ARG之间的关系,进一步研究环境中ARGs的转移机制十分有必要。
对已检出的不同类型的抗生素分别进行加和统计,并与ARGs进行相关性分析,结果如图3所示。抗生素与ARGs之间相关性较弱(P>0.05),这可能与抗生素的物理化学性质及ARGs在环境中的转移机制有关(Jia et al.,2018)。此外,天气状况和水温等环境因素也对抗生素及 ARGs造成影响(Cesare et al.,2017;Peak et al.,2007)。同时,抗生素的低水平及ARGs的高水平检出说明:由历史背景诱导产生的ARGs可进行自我扩增而持续存在于环境中(Ling et al.,2013)。
4 结论
(1)东寨港海水和沉积物的ARGs丰度处于较高水平,这将给当地微生物群落稳定性带来考验。
(2)沉积物中抗性基因污染比海水严重,说明沉积物是东寨港区域重要的ARGs储存介质。
(3)东寨港抗生素检出率较低,残留较少,与ARGs相关性较弱。
