2018诺贝尔化学奖:酶定向进化与噬菌体展示技术
2019-02-15蒋迎迎孙周通
曲 戈,张 锟,蒋迎迎,孙周通
(中国科学院 天津工业生物技术研究所,天津 300308)
2018年10月3日,诺贝尔化学奖揭晓。奖金一半授予Frances H.Arnold教授,表彰她在“酶定向进化”(“the directed evolution of enzymes”)领域的卓越贡献;另一半颁给了George P.Smith和Gregory P.Winter教授,奖励他们在“多肽及抗体的噬菌体展示”(“the phage display of peptides and antibodies”)技术方面的开创性工作与应用(图 1)[1]。地球生命的多样性彰显了自然进化的力量,而2018年诺贝尔化学奖的3位得主以“掌控了进化的方式,并用它谋求为人类带来最大福祉”,将自然进化发展为:可控制、可加速、可应用的人工进化[2]——通过定向进化的非天然酶广泛应用于生物燃料、医药中间体等领域;而采用噬菌体展示技术筛选出的抗体可以对抗自身免疫疾病,甚至治愈癌症。本文主要从生命化学的角度出发,综述定向进化以及噬菌体展示技术发展过程中的关键技术及应用,并对未来发展方向进行展望。

图1 2018年诺贝尔化学奖3位得主:Frances H.Arnold(左,现任美国加州理工学院Linus Pauling讲席教授),George P.Smith(中,美国密苏里大学荣誉退休教授)以及Gregory P.Winter(右,现任MRC分子生物学研究所所长)[2]Figure 1 The Nobel Prize in Chemistry 2018 has been awarded to Frances H.Arnold (California Institute of Technology),and George P.Smith (University of Missouri) and Gregory P.Winter (Medical Research Council (MRC) Laboratory of Molecular Biology (LMB))[2]
1 酶的定向进化
1.1 物种进化与酶定向进化
地球生命普遍存在着进化现象。以我们人类为例,就经历了一条“早期猿人→能人→直立人→智人→现代人”的进化路线(图2)。进化需要两个基本要素——突变与选择。基因突变往往是随机发生的,但自然选择却是有方向性的。达尔文在《物种起源》中详细阐述了自然选择是进化的内驱动力,而进化的方向则是更好地适应生存环境。与之类似,酶定向进化也包括两个基本要素——突变与筛选。但突变可以是随机发生的,也可以是定点发生的;而筛选则会施加一个人为偏好的方向,因此称之为“定向”。酶定向进化主要通过蛋白质工程等手段,对编码基因进行反复的突变、表达和筛选,诱使蛋白质等生物分子的结构及功能发生定向改变,从而在短时间内在实验室里完成自然界需要成千上万年的进化,而定向进化的方向则是最终获得性能改进或具有新功能的蛋白质[3]。

图2 人类进化过程示意图[1]Figure 2 The evolution of human being[1]
这种人为模拟自然进化过程既可在活细胞体内(invivo)进行,或是在体外(invitro)进行。通常情况下分为4步:1)突变:通过分子生物学技术,在编码目的蛋白的基因上引入随机突变位点;2)表达:将突变基因转化至宿主菌,并借助宿主体内蛋白表达系统合成突变体蛋白;3)筛选:使用特异的化学反应等手段筛选目标突变体,舍弃不符合要求的突变体;4)循环:基于筛选得到的最优突变体,引入新的突变位点,进入下一轮“突变-筛选”的循环,直至获得预期性能的蛋白质(图 3)。
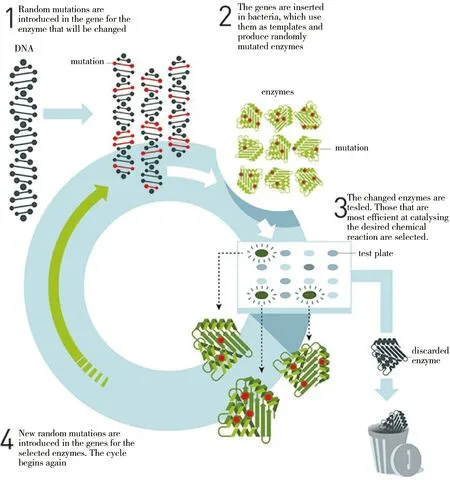
图3 酶定向进化流程示意图[1]Figure 3 The workflow of the directed evolution of enzyme[1]
1.2 酶定向进化技术的发展历程
前文概述了酶的定向进化,然而这项技术本身,又是如何“进化”的呢?时间追溯到1953年,James D.Watson和Francis H.Crick发现了DNA双螺旋结构(两人因此获得1962年诺贝尔生理学或医学奖),开启了分子生物学时代。随后诞生于20世纪70年代的DNA重组技术为利用生物工程手段的研究和应用奠定了重要基石[4]。在此基础上,定向进化领域的先驱们开发了一系列经典的进化策略与技术(图 4):1978年,Michael Smith首次提出定点突变技术(Site-Specific Mutagenesis,SSM,凭此贡献获得1993年诺贝尔化学奖),揭开了蛋白质改造与设计的新篇章[5]。1985年James A.Wells团队开发了寡核苷酸饱和突变(Oligonucleotide-based Saturation Mutagenesis,OSM)技术[6],实现基因序列的单点饱和突变;1989年David W.Leung团队最先提出易错PCR(error-prone Polymerase Chain Reaction,epPCR)概念[7],随后应用于体外抗体改造筛选[8]。1993年,本届诺贝尔化学奖获得者之一的Frances H.Arnold教授提出酶分子定向进化的概念,通过使用多轮epPCR(Sequential epPCR,SepPCR)技术,连续反复地对枯草杆菌蛋白酶E(Subtilisin E)编码基因进行随机突变,并经逐步积累正向突变,最终获得可在高浓度有机溶剂二甲基甲酰胺(DMF)中稳定存在且表现出活性的突变体[9]。定向进化领域的另外一位先驱Willem P.C.Stemmer教授,于1994年开创了用于单基因或多基因间重组的DNA混组(DNA shuffling)技术,可加速有益突变的积累以及组合两个或多个已优化的参数,并成功提高了β-内酰胺酶的活性[10],为定向进化领域做出了开拓性贡献。定向进化领域的第三位开拓者Manfred T.Reetz教授,于1997年首次将定向进化技术应用在酶的不对称催化改造领域[11],将铜绿假单胞菌来源的脂肪酶对映体选择性由2%提升至81%;随后又发现对手性选择有益的突变位点主要集中在底物结合口袋区域,由此提出了组合活性中心饱和突变策略(Combinatorial Active-site Saturation Test,CAST)[12]及迭代饱和突变技术(Iterative Saturation Mutagenesis,ISM)[13],极大精简了突变体库的构建规模,从而提高了筛选效率。Romas J.Kazlauskas团队统计分析并比较了位于活性中心和远离活性中心的氨基酸位点突变对酶的活性、底物选择性、对映体选择性及稳定性的影响,指出前者更有利于改造酶的底物选择性及对映体选择性,从统计学的角度进一步支持了CAST方法的可靠性[14]。因此CAST方法广泛应用于酶的立体/区域选择性、底物谱、催化效率等参数的改造[15-16]。

图4 定向进化技术发展历程Figure 4 Pioneering technologies in the directed evolution
除此之外,还有一些衍生技术也被广泛应用于定向进化领域。比如,Ulrich Schwaneberg教授在单点饱和突变的基础上,开发了序列饱和突变技术(Sequence saturation Mutagenesis,SeSaM),通过引入通用碱基避免了突变偏差,提高了突变效率[17];从进化的角度出发,基于序列保守性分析,Steven A.Benner课题组设计了REAP分析方法(Reconstructed Evolutionary Adaptive Path,REAP),使聚合酶可接受3′端氨基化(dNTP-ONH2)的三磷酸底物[18];Peter J.Schaap教授利用基于结构的多重序列比对技术,开发了3DM策略(3D-Multiple sequence alignment,3DM),应用于酶催化活性和对映体选择性等参数改造[19];Gjalt W.Huisman团队将统计学方法与定向进化技术相结合,提出PROSAR策略(Protein Sequence Activity Relationships,PROSAR),可加速突变体设计优化速度,成功提升了脱卤酶的工作效率[20];为提高随机突变效率,Miguel Alcalde开发了MORPHING技术(Mutagenic Organized Recombination Process by HomologousinvivoGrouping,MORPHING),成功改造了过氧化酶和环化酶等酶的催化性能[21];孙周通教授在CAST方法的基础上,通过理性选择3种氨基酸密码子作为饱和突变的建构单元,开发了三密码子饱和突变策略(Triple Code Saturation Mutagenesis,TCSM)[22-23],通过构建“小而精”的突变体库,可大大降低筛选工作量,成功应用于酶的立体选择性、区域选择性、底物谱拓展及热稳定性等酶参数的定向改造[24-25]。
1.3 酶定向进化技术的具体应用
酶定向进化技术经历了近50年的发展变革,期间Michael Smith、Frances H.Arnold、Willem P.C.Stemmer、Manfred T.Reetz等先驱做了一系列开创性工作,极大丰富了该技术的研究及应用范畴[26-28]。近年来定向进化技术广泛应用于生物催化剂改造。通过定向进化这一人工过程,可有效驯服天然酶在环境(包括酸碱性、温度、有机溶剂等)耐受性、立体/区域选择性、底物特异性、催化效率及产物抑制性等方面存在的缺点,使得自然界成千上万年的进化历程可在实验室短时间内完成。从而拓展了生物催化剂的应用范畴,使其广泛应用于食品工业、医药卫生、精细化工及生物能源等诸多领域[27-28]。
通过定向进化技术,极大拓展了蛋白质的应用领域。以Frances H.Arnold团队近年来的工作成果为例,通过使用定向进化技术改造的细胞色素C氧化酶,可提高碳-硅键形成的催化效率[29],以及实现了传统化学上难以合成的高能高张力小分子碳环的生物合成[30],可应用于制备医药中间体等工业领域。除此之外,表1列举了定向进化技术的其他关键应用,比如赋予30亿年来逆转录酶原本不存在的校对功能[31];实现P450单加氧酶区域选择性、立体选择性和底物适配性等多参数改造[25];增强CRISPR-Cas9核酸酶可识别的序列范围及精准度[32]等。

表1 酶定向进化技术的应用节选Table 1 Applications of the directed evolution of enzymes
2 噬菌体展示技术
2.1 噬菌体展示技术与抗体进化流程
与Frances H.Arnold分享今年诺贝尔化学奖的George P.Smith开发的噬菌体展示技术是一种基于定向进化的高通量筛选技术,大大拓展了定向进化技术在抗体多肽改造筛选方面的应用。该技术通常分为三步:1)基因融合:利用基因工程技术将外源基因片段插入噬菌体特定蛋白基因;2)外源蛋白扩增与展示:带有外源蛋白的噬菌体侵染宿主菌进行扩增,并将外源蛋白展示在噬菌体表面;3)生物淘洗:筛选与靶分子特异性结合的噬菌体(图5)。
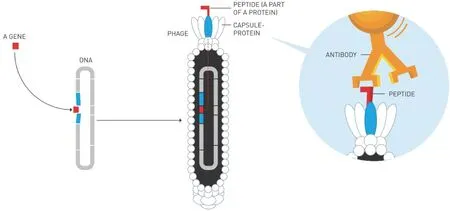
图5 噬菌体展示技术示意图[1]Figure 5 The workflow of phage display[1]
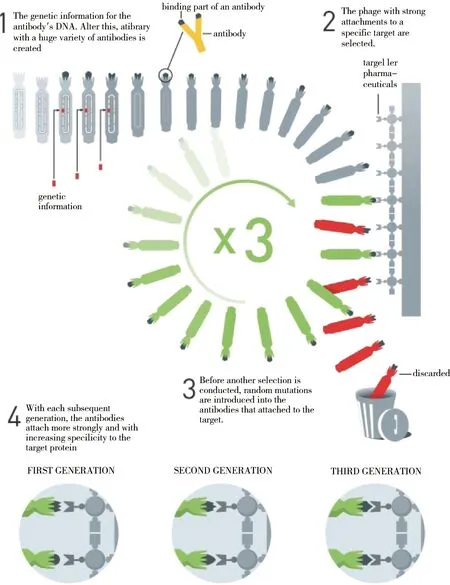
图6 使用噬菌体展示定向进化抗体的原理[1]Figure 6 The principle for the directed evolution of antibodies using phage display[1]
Gregory P.Winter和George P.Smith共享了一半的诺贝尔化学奖。他在George P.Smith教授开发的噬菌体展示技术的基础上,开创了噬菌体展示定向进化抗体技术。该技术通常分为四步:1)将抗体抗原结合位点的遗传信息插入到噬菌体DNA中,创建具有多样性的抗体库;2)通过靶抗原筛选出对特定抗原具有强附着性的噬菌体;3)在进行下一轮筛选之前,将随机突变引入到附着目标抗体的噬菌体上;4)随着新一代抗体突变体的出现,抗体会更特异并更加强力地附着在目标蛋白质上(图 6)。
2.2 噬菌体展示及抗体进化技术发展历程
将噬菌体开发为简单、灵敏的实验载体的构想具有悠久历史。1966年,Joseph Haimovich、Michael Sela和O.Mäkelä提出将小分子半抗原通过化学修饰方法连接到活体噬菌体表面,修饰后的噬菌体能够被相应的抗体灭活,从而简化特异性抗体的检测[49-50]。1982年,Renato Dulbecco表明免疫原性多肽可与噬菌体或其他病毒的外壳蛋白融合并展示在噬菌体表面,得到具有新的抗原性和免疫原性的病毒颗粒,并提出展示(Display)的概念[51]。1985年,George P.Smith将限制性内切酶EcoR I上的一段基因片段(57个氨基酸)插入到丝状噬菌体M13的衣壳蛋白基因III中,该片段成功展示在噬菌体表面,且能够被特异性抗体所识别,George P.Smith将该技术命名为“噬菌体展示”(“Phage display”)[52]。1989年,Richard A.Lerner和同事把噬菌体展示技术和抗体工程结合起来,把鼠源抗体识别抗原的可变区(Fragment of antigen binding,Fab)的DNA片段克隆到噬菌体的衣壳蛋白基因中,从而在噬菌体表面展示了抗体的结构[53]。1990年,Gregory P.Winter等通过在噬菌体表面展示免疫球蛋白轻链和重链可变区基因的单链抗体(Single-chain variable fragment,scFV),筛选了一种溶菌酶的抗体[54]。在前人基础上,Richard A.Lerner和Gregory P.Winter分别于1991年和1992年成功利用噬菌体展示了人源抗体片段(Human antibody libraries,图7)[55-56]。
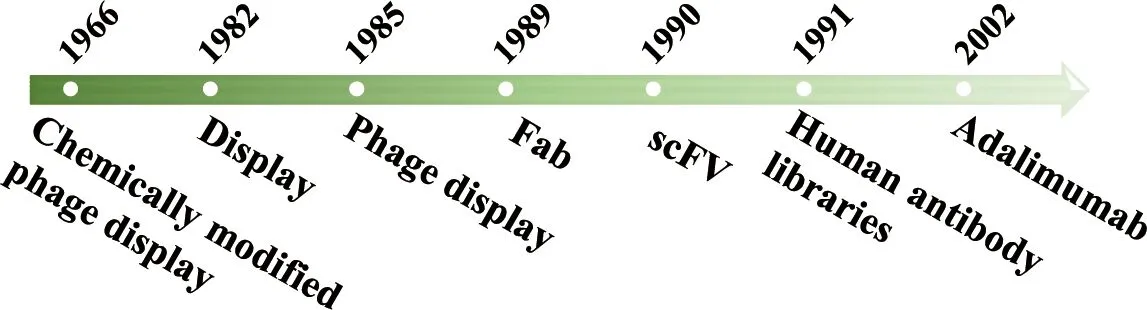
图7 噬菌体展示技术发展历程Figure 7 The development of phage display technology
2.3 噬菌体展示技术的具体应用
噬菌体展示技术可以应用于大小不同、性质不同的结构和序列未知蛋白的表面展示和筛选,从而拓展了它的应用范围,尤其是在疾病的诊断和治疗领域。以噬菌体展示技术在治疗性抗体研发领域的应用为例,在George P.Smith和Gregory P.Winter教授开发噬菌体展示抗体库筛选技术前,抗体研发的方向是获得人鼠嵌合抗体以降低免疫原性[57- 58]。而使用噬菌体展示抗体库筛选技术可以得到全人源抗体,从而彻底解决异源抗体的免疫原性问题,且该技术操作简单,可应用于多种类型抗体的筛选。“阿达木(Adalimumab)”是第一个基于该技术研发的单抗,并于2002年被FDA批准用于治疗类风湿性关节炎(图7)。以阿达木单抗为活性成分的药物修美乐(Humira)是2013年世界最畅销的药物之一,其全球销售额达到96亿美元,表2展示了部分已商业化的抗体[59- 60]。
除此之外,表3节选了噬菌体展示技术的具体应用实例。比如定向进化提高DNA聚合酶的反转录活力[61];研发于2009年被FDA批准用于治疗遗传性血管性水肿的艾卡拉肽(Ecallantide)[62]等。
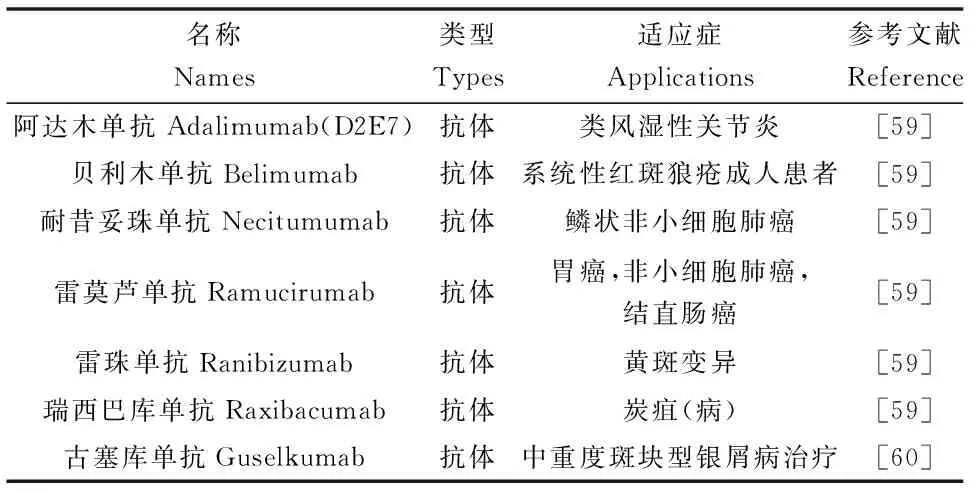
表2 应用噬菌体展示技术研发的部分上市抗体药物Table 2 Phage display-derived drugs approved by FDA or EMA
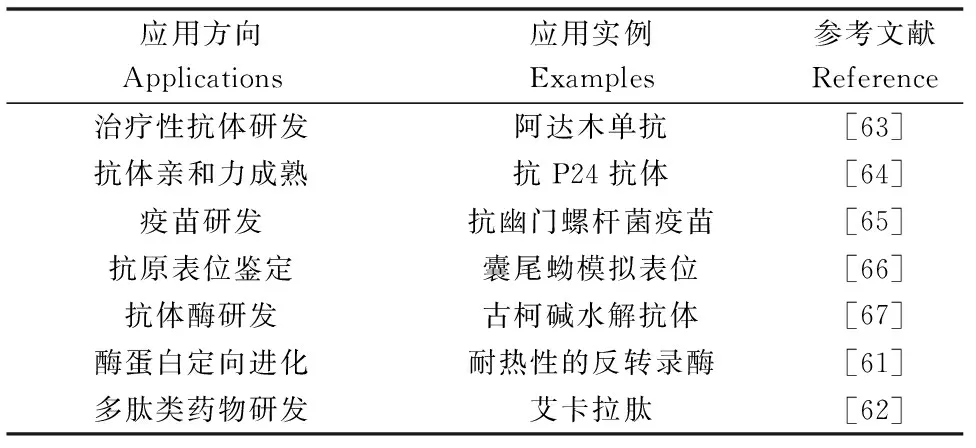
表3 噬菌体展示技术应用实例节选Table 3 Applications of the phage display
3 展示技术在定向进化领域的拓展
前文综述了定向进化技术在酶的改造和多肽及抗体进化领域中的应用,但其潜力还远没有被挖掘,其主要瓶颈在于如何建立高效、快速的筛选方法[16,26,68-70]。许多定向进化策略试图通过建立针对特定改造靶点的高质量突变体文库来解决筛选问题[71],虽然获得了极大成功,然而随着拟突变位点数目的增加,筛选规模呈指数级增长。以NNK(N:A,T,C和G;K:T和G)简并密码子为例,设定95%库覆盖度,如果同时突变10个位点时,需筛选3.4×1015个转化子[16],假如一个蛋白质全长为350个氨基酸,随机突变为20种天然氨基酸,那么突变体库的多样性为20350。筛选是重要瓶颈,因此需要设计开发更加高效的建库策略或者高通量筛选方法来解决酶定向进化的筛选问题。目前常见的筛选方法包括:平板筛选法、微孔板筛选法、荧光活化细胞分选法及液滴微流控等[72]。这几种方法的筛选通量各不相同,平板筛选最高为104,微孔板筛选最高为105,细胞分选法通过流式细胞仪可以超过108[72],而液滴微流控可达1.7×109以上[73]。噬菌体展示技术是一项特异性多肽或蛋白的高通量筛选技术,不仅可以用于抗体或多肽的筛选,也可以用于酶突变库及疫苗等高通量筛选,如1991年,John McCafferty等人在噬菌体表面成功展示了碱性磷酸酶,证明了可以在噬菌体表面展示有活性的酶[74]。除了噬菌体表面展示技术,还包括细菌表面展示技术以及酵母表面展示技术等,基本原理都是利用DNA重组技术,将目标蛋白质富集在宿主(如噬菌体、细菌、酵母)表面进行筛选[75- 76],筛选规模达109~1010 [77]。
4 结语及展望
以Frances H.Arnold、George P.Smith和Gregory P.Winter教授为代表的科学家们通过驯服进化的力量,促进了“绿色化学和医药产业”的快速发展,更好地为人类社会谋求福祉,因此他们获得2018年诺贝尔化学奖实至名归!除了上述3位获奖人外,还有很多科学家也在各自领域取得了开拓性成果,为酶定向进化和噬菌体展示技术做出了不可磨灭的贡献,我们同样也应当向他们致敬!
当今人工智能技术的快速发展,对酶定向进化领域带来了深刻变革,从头设计蛋白质的时代已然来临[78]:过去在实验室进行的湿实验工作流程(图 3),如今可以通过计算生物学开发的算法和程序模拟自然界蛋白质的进化轨迹,预测酶活性位点并考察特定位点突变对其稳定性、蛋白折叠及与底物结合等方面的影响,从而对酶进行精准设计与改造,并将繁重的筛选工作转移至计算机上进行[79]。依赖CPU/GPU高性能计算处理能力,借助机器学习等智能算法,实现计算机虚拟定向进化(insilicodirected evolution),快速准确地预测目标突变体。这种理性设计的策略已经在环氧水解酶[80- 81]及天冬氨酸裂解酶[82]等酶的设计改造上取得了成功。依赖计算机技术,通过人工智能驱动酶定向进化将成为未来发展的重要方向。
