TOMM40基因rs2075650位点多态性与脑梗死的关系
2019-01-24焦雪倩余丹黄伏生
焦雪倩,余丹,黄伏生
(武汉大学中南医院 检验科,湖北 武汉 430071)
脑卒中是遗传和环境共同作用的疾病,具有高发病率和高病死率,其中梗死性脑卒中是最常见的形式,约占全部脑卒中的80%[1]。据报道,高血压、糖尿病及高血脂等是脑梗死的危险因素[2]。此外,一些未知的基因也与脑梗死的发生有关。线粒体外膜转移酶 40(translocase of outer mitochondrial membrane 40,TOMM40)定位于19q13.2,在某些蛋白质如DNA聚合酶、RNA聚合酶及氧自由基清除因子等进入线粒体的过程中起作用[3]。本研究采用高分辨率熔解曲线(high resolution melting, HRM)技术,探索TOMM40基因多态性与武汉地区中老年人群脑梗死发病的相关性。
1 资料与方法
1.1 一般资料
选取2016年9月—2017年7月在武汉大学中南医院就诊的507例汉族脑梗死患者作为脑梗死组。其中,男性305例,女性202例;年龄40~84岁,平均(66.89±11.68)岁;所有患者经过CT或MRI证实。纳入标准:无脑梗死病史,经CT或MRI检查无脑血管病变。排除标准:脑出血、颅内占位、自身免疫性疾病、严重的肝肾疾病、感染性疾病及恶性肿瘤。收集同期在武汉大学中南医院体检中心就诊的510例汉族健康体检者作为对照组。其中,男性313例,女性197例;年龄42~87岁,平均(65.62±11.77)岁;收集受试者的性别、年龄、收缩压(systolic blood pressure, SBP)、舒张压(diastolic blood pressure, DBP)、血糖(blood glucose, GLU)、总胆固醇(total cholesterol,TC)、三酰甘油(Triglyceride, TG)、高密度脂蛋白胆固醇(high density lipoprotein cholesterol, HDL-C)及低密度脂蛋白胆固醇(low density lipoprotein cholesterol, LDL-C)。本研究通过本院伦理委员会批准。
1.2 方法
1.2.1 外周血DNA的提取 收集受试者的静脉血2 ml,用苯酚-氯仿法提取基因组DNA,纯度和浓度采用Nanodrop 2000C分光光度计(美国Thermo Fisher公司)检测。
1.2.2 目的片段的扩增及基因型的检测TOMM40基因rs2075650位点正向引物:5’-AGGAAGAGAT GAGAGTTGGTGTG-3’,反向引物:5’-CTGGAGAA GAGAAACGCTGTC-3’。PCR反应体系为10μl。PCR反应结束后使用LightScanner 96高分辨率分析系统(美国Idaho Technology公司)结合熔解曲线进行基因分型。根据熔解曲线图挑选出不同基因型的样本进行PCR扩增,扩增产物送到武汉天一辉远生物公司进行测序验证。
1.2.3 实时荧光定量聚合酶链反应(quantitative real-time polymerase chain reaction, qRT-PCR) 选取脑梗死组和对照组患者各80例,收集静脉血2 ml,取200μl血用DNA提取试剂盒(北京天根生化科技有限公司)提取DNA,剩余血液用Trizol(美国Invitrogen公司)试剂提取RNA,纯度和浓度用Nanodrop 2000C分光光度计(美国Thermo Fisher公司)检测,将提取的RNA置于-80℃冰箱保存。利用逆转录试剂盒(日本TaKaRa公司)将RNA逆转录为cDNA。TOMM40基因正向引物:5’-CATCCAGACCCAGCAGTCG-3’,反向引物:5’-GAGGTAGTGGGCTACGAGGA-3’,选 取 3- 磷酸甘油醛脱氢酶为内参基因。内参基因正向引物:5’-GGTCTCCTCTGACTTCAACA-3’,反向引物:5’-GTGAGGGTCTCTCTCTTCCT-3’。总反应体系为20μl。qRT-PCR反应条件:95℃预变性10 min,95℃变性30 s,60℃退火30 s,72℃延伸30 s,共40个循环。每个样本重复检测2次,取2次检测的平均值。用2-ΔCt法计算相对表达量。
1.3 统计学方法
数据分析采用SPSS 17.0统计学软件,计量资料以均数±标准差(±s)或中位数和四分位数间距M(P25,P75)表示,比较用t检验或秩和检验,计数资料以率(%)或构成比表示,比较用χ2检验,群体基因型用Hardy-Weinberg遗传平衡检验,P<0.05为差异有统计学意义。
2 结果
2.1 两组一般资料比较
两组年龄、性别及LDL-C比较,差异无统计学 意 义(P>0.05);两 组 SBP、DBP、GLU、TC及TG、HDL-C比较,差异有统计学意义(P<0.05)。见表1。
2.2 两组基因型和等位基因的频率比较
两组TOMM40基因rs2075650位点AA、AG及GG基因型频率比较,差异有统计学意义(P<0.05),两组A、G等位基因频率比较,差异有统计学意义(P<0.05)。脑梗死组显性模型AA与AG+GG基因型频率分别占83.63%和16.37%,对照组分别为77.84%和22.16%,两组AA与AG+GG基因型频率比较,差异有统计学意义0.942)P=0.019],脑梗死组隐性模型GG与AG+AA基因型频率分别占1.18%和98.82%,对照组分别为2.55%和97.45%,两组GG与AG+AA基因型频率比较,差异无统计学意义。见表2和附图。

表1 两组一般资料比较

表2 两组患者基因型和等位基因频率比较 例(%)
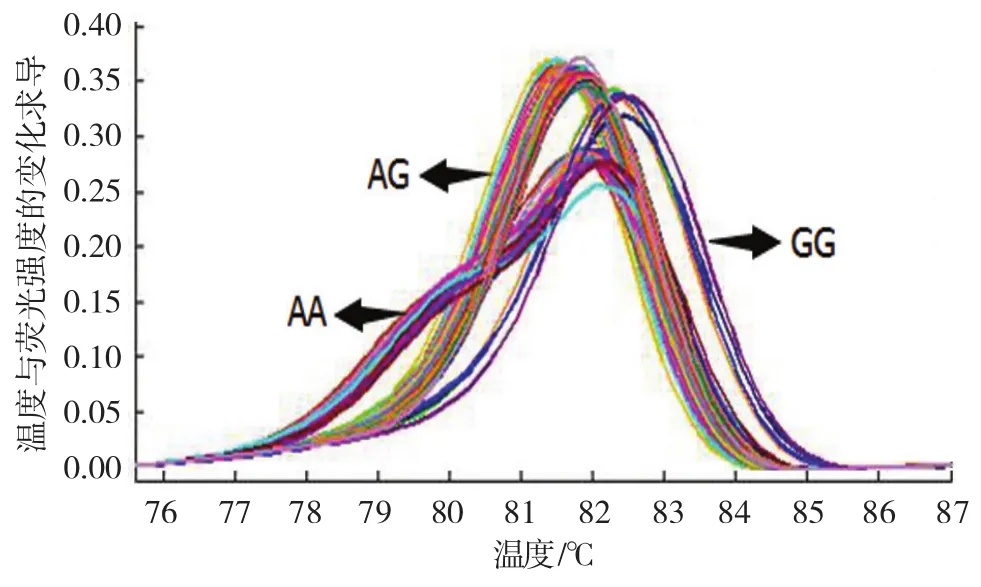
附图 HRM对TOMM40基因rs2075650位点的分型图
2.3 两组TOMM40 mRNA表达水平比较
脑梗死组TOMM40mRNA表达量为3.91(1.71,6.49),对照组为7.84(6.24,10.18),经秩和检验,差异有统计学意义(Z=-6.834,P=0.000),脑梗死组低于对照组。
3 讨论
TOMM40基因位于19q13.2,是外膜转位酶形成的复合体,介导核基因编码的蛋白质从细胞质进入线粒体的传递。TOMM40位于载脂蛋白E(apolipoprotein E,APOE)上游,其中的几个单核苷酸多态性与APOE具有连锁不平衡[4-5]。TOMM40与阿尔茨海默病、冠状动脉粥样硬化性心脏病、长寿及认知障碍等有关[6-8]。APOE可能与TOMM40相互作用而影响线粒体功能[9]。线粒体被认为是所有细胞的动力,在中枢神经系统中发挥重要作用,为神经元的存活提供能量[10]。线粒体中99%蛋白质都是由核基因编码合成再转运到线粒体中,而TOMM40几乎为所有核编码的线粒体蛋白质入口的重要组成部分,TOMM40的减少或功能障碍导致一些重要的蛋白质不能进入线粒体发挥作用,影响线粒体的功能和神经元的存活[11]。HAINES等[12]发现敲除小鼠的解偶联蛋白2(uncoupling protein 2,UCP2)基因能使缺血再灌注小鼠脑组织的梗死面积增加,敲除UCP2后能抑制缺血再灌注后的抗氧化基因。UCP2能够保护血管内皮细胞,改善脑血管功能障碍[13-14]。UCP2的表达下调还与动脉粥样硬化、血管损伤有关,影响神经传递,对神经系统疾病的发生有重要影响[15]。解偶联蛋白属于线粒体载体蛋白超家族,TOMM40的缺乏可能影响解偶联蛋白转运到线粒体发挥作用,从而引起线粒体功能障碍,影响脑组织的功能。
本研究中脑梗死组与对照组TOMM40基因的rs2075650位点之间基因型和等位基因的频率分布比较有差异,对照组中突变等位基因G的频率高于脑梗死组,脑梗死组和对照组的显性模型(AA vs AG+GG)基因型频率也有差异,说明该基因位点可能与脑梗死的发病相关。YAMASE等[16]发现TOMM40的rs2075650位点与脑梗死有相关性,且次要等位基因G是脑梗死的保护性基因,与本研究结果一致。对相关指标的分析表明,血压、GLU及TG是脑梗死发病的危险因素,HDL-C可能是脑梗死的保护性指标,这符合传统的研究结果。然而本研究中两组LDL-C比较无差异,可能是脑梗死患者所用药物及人群选择差异导致。qRT-PCR结果显示,对照组的TOMM40 mRNA相对表达量高于脑梗死组。机制可能是脑梗死患者TOMM40 mRNA表达量下调影响核基因编码的蛋白质进入线粒体,导致一些关键性蛋白质不能发挥作用,线粒体的功能障碍、脑组织能量不足等可能导致脑梗死的发生。LEE等[17]发现TOMM40 mRNA在阿尔茨海默病患者中表达下调,该疾病的发生可能也与TOMM40相关蛋白质无法正常发挥作用有关。
综上所述,本研究用HRM技术对脑梗死组和对照组TOMM40的rs2075650位点进行基因分型,并用qRT-PCR检测基因的相对表达量,发现武汉地区中老年汉族人群中该基因位点与脑梗死的发病相关。
