DNA条形码在西藏水系裂腹鱼亚科鱼类鉴定中的研究
2019-01-22周建设刘海平王万良曾本和牟振波
周建设,张 驰,刘海平,马 波,王万良,曾本和,牟振波
(1.西藏自治区农牧科学院水产科学研究所,拉萨 850032;2.中国水产科学研究院黑龙江水产研究所,哈尔滨 150000;3.河南农业大学牧医工程学院,郑州 450002)
裂腹鱼亚科(Schizothoracinae)隶属于鲤形目(Cypriniformes)、鲤科(Cyprinidae),全球约有100多种[1],中国共计76个种和亚种,占全球裂腹鱼亚科鱼类的80%以上[2],广泛分布于西藏水系,是西藏鱼类区系的优势群体,具有明显的区域特性[3],曹文宣[4]首次对中国裂腹鱼亚科鱼类进行了较系统的整理,也为西藏水系裂腹鱼的分类奠定了基础。
DNA条形码技术自用于物种鉴别体系以来,在鱼类的鉴定应用方面越来越多,尤其是在鱼卵、仔稚鱼等[5]通过外部形态难以辨识的物种上。Ariagna等[6]用DNA条形码技术分析了古巴126尾淡水鱼,认为COI基因序列能够区分本地的淡水鱼,Alba等[7]对亚马逊河大范围销售的“Acará”进行了分析,发现是由7个物种组成,Brito等[8]用DNA条形码对137个石首鱼科进行了鉴定,鉴定率达到90%。在西藏部分及其毗邻地区以裂腹鱼亚科属间系统发育学、生物地理学和进化中的隔离事件为主线的研究多以线粒体细胞色素b(Cytb)基因为基础[9],比较系统、全面、客观地探讨了裂腹鱼亚科鱼类的起源、演化、扩散等与青藏高原的不断隆升事件的关系,而涉及分类学和物种鉴定的讨论并不多,尤其是限定在西藏水系裂腹鱼亚科属间鱼种的鉴定鲜有报道,产久林等[10]对采自拉萨市羊卓雍措的60尾5种高原裂腹鱼进行了基于COI和16 S rRNA基因部分序列的物种鉴定,表明COI基因能有效鉴定高原裂腹鱼鱼种。
西藏水系格局复杂,河流纵横交错,高原湖泊星罗棋布,裂腹鱼亚科鱼类作为西藏水系优势种群,鱼类可能存在不同水域迁移和同一水域种间杂交,出现形态相似不同种和同种鱼不同形态的现象。近年来,雅鲁藏布江鱼类资源受到严重威胁[11],对其鱼类资源的养护迫在眉睫,摸清西藏水系鱼类资源现状,确定优势物种、普通物种和濒危物种,制定合理的养护措施,对西藏水系所有鱼类进行准确的物种鉴定显得尤为重要,而传统的鱼类鉴定方法具有一定的局限性[12],历史上在裂腹鱼亚科鱼类各属的界定中也不尽相同[2,13],事实也说明西藏水系现存裂腹鱼种类繁多,一部分种间形态学差异不明显,尤其在幼鱼期和裸鲤属内种间表型极其相似。因此,从分子角度对西藏水系裂腹鱼亚科鱼类的鉴定显得尤为重要,应用DNA条形码技术对西藏水系的裂腹鱼亚科鱼类进行种类鉴定鲜有报道。本研究是基于线粒体细胞色素C氧化酶1(COI)基因序列的DNA条形码技术,以目前能捕获的西藏水系裂腹亚科鱼类为研究对象,展开DNA条形码研究,验证COI基因序列对采自中国西藏水系的裂腹鱼亚科鱼类样本进行物种鉴定的适用性,对于西藏水系裂腹鱼亚科鱼类种类鉴定,摸清西藏地区鱼类资源现状,制定合理的鱼类养护进程具有一定的指导意义。
1 材料与方法
1.1 样品采集
本研究中,所采集到的裂腹鱼亚科鱼类形态鉴定主要依据乐佩琦等[13]的分类体系及其对各物种的形态描述。在西藏水域共采集到裂腹鱼亚科包括5属20种的260个样本,其中包括裂腹鱼属(Schizothorax)的107个样本,尖裸鲤属(Oxygymnocypris)30个样本,裸鲤属(Gymnocypris)32个样本,叶须鱼属(Ptychobarbus)22个样本,裸裂尻属(Schizopygopsis)69个样本,代表了西藏水域现能采集到的大部分裂腹鱼亚科的鱼种。本研究中所测物种均采集于2014—2017年间,野外采样时,取鳍条组织保存在70%的酒精中,带回实验室后保存于-70 ℃备用。
1.2 样品DNA提取
采用传统氯仿法从鳍条中提取DNA。
1.3 线粒体基因组COI基因片段扩增和测序
COI基因片段PCR扩增的引物为F(5′-TCAACCAACCACAAAGACATTGGCAC-3′),L(5′-TAGACTTCTGGGTGGCCAAAGAATCA-3′)[10]。引物由美因基因公司合成。PCR反应体系为5×FastPfu Buffer 4 μL,2.5 mmol/L dNTPs 2 μL,Forward Primer(5 μmol/L)0.8 μL,Reverse Primer(5 μmol/L)0.8 μL,FastPfu Polymerase 0.4 μL,Template DNA 10 ng,ddH2O补至20 μL。PCR扩增程序为95 ℃,5 min,95 ℃,30 s,55 ℃,30 s,72 ℃,45 s,27个循环,最后延伸72 ℃,10 min。PCR扩增仪为ABI GeneAmpR9700型。PCR产物通过琼脂糖凝胶(2%)电泳5 V/cm,20 min,凝胶图片经凝胶成像系统检测阳性后送美因基因公司进行测序。
1.4 数据处理
分段进行正反链双向测序的数据经CHROMAS软件比对后,适当进行人工校对,然后用DNAMAN软件进行分段数据的拼接,所有COI序列利用CLUSTAL X2.1软件的默认参数设置为基础进行同源序列的多重比对,并实施人工校对。比对后的序列在NCBI数据库中进行Blast比对分析,检索相似性,确定物种分类地位。依据物种COI序列相似度达到99%以上认定为同一种,相似度92%~99%为同一属,83%~92%为同一科[14]。通过Mega5.0软件分析序列的碱基组成及其变异位点等统计数据,计算序列Kimura双参数种内及种间个体间遗传距离,在物种鉴定中,遗传距离以0.02作为种内及种间的鉴定标准[15,16],采用邻接法(Neighbor-Joining method,NJ)构建分析系统发育树,并通过Bootstrap检验聚类树各分支的置信度,系统发育树中的断线脂鲤(Phenacogrammusinterruptus)和墨西哥丽脂鱼(Astyanaxmexicanus)为外群[17]。
2 结果
2.1 传统形态学鉴定
对所采集的西藏水系裂腹鱼亚科260个样本经形态学鉴定(表1),包括裂腹鱼属10个种的拉萨裂腹鱼(S.racomawaltoni)31尾,异齿裂腹鱼(S.schizothoraxo'connori)38尾,巨须裂腹鱼(S.macropogon)31尾,长丝裂腹鱼(S.schizothoraxdolichonema)、澜沧江裂腹鱼(S.racomalantsangensis)、怒江裂腹鱼(S.schizothoraxnukiangensis)、西藏裂腹鱼(S.labiatus)、墨脱裂腹鱼(S.molesworthi)、横口裂腹鱼(S.plagiostomus)、弧唇裂腹鱼(S.curvilabiatus)各1尾;尖裸鲤属1个种的尖裸鲤(O.stewaritii)30尾;裸鲤属4个种的兰格胡裸鲤(G.chui)29尾,纳木错裸鲤(G.namensis)、硬刺裸鲤(G.firmispinatus)、色林错裸鲤(G.selincuoensis)各1尾;叶须鱼属3个种的双须叶须鱼(P.dipogon)20尾,锥吻叶须鱼(P.conirostris)、裸腹叶须鱼(P.kaznakovi)各1尾;裸裂尻鱼属2个种的拉萨裸裂尻(S.younghusbandi)34尾,高原裸裂尻(S.stoliczkai)35尾。
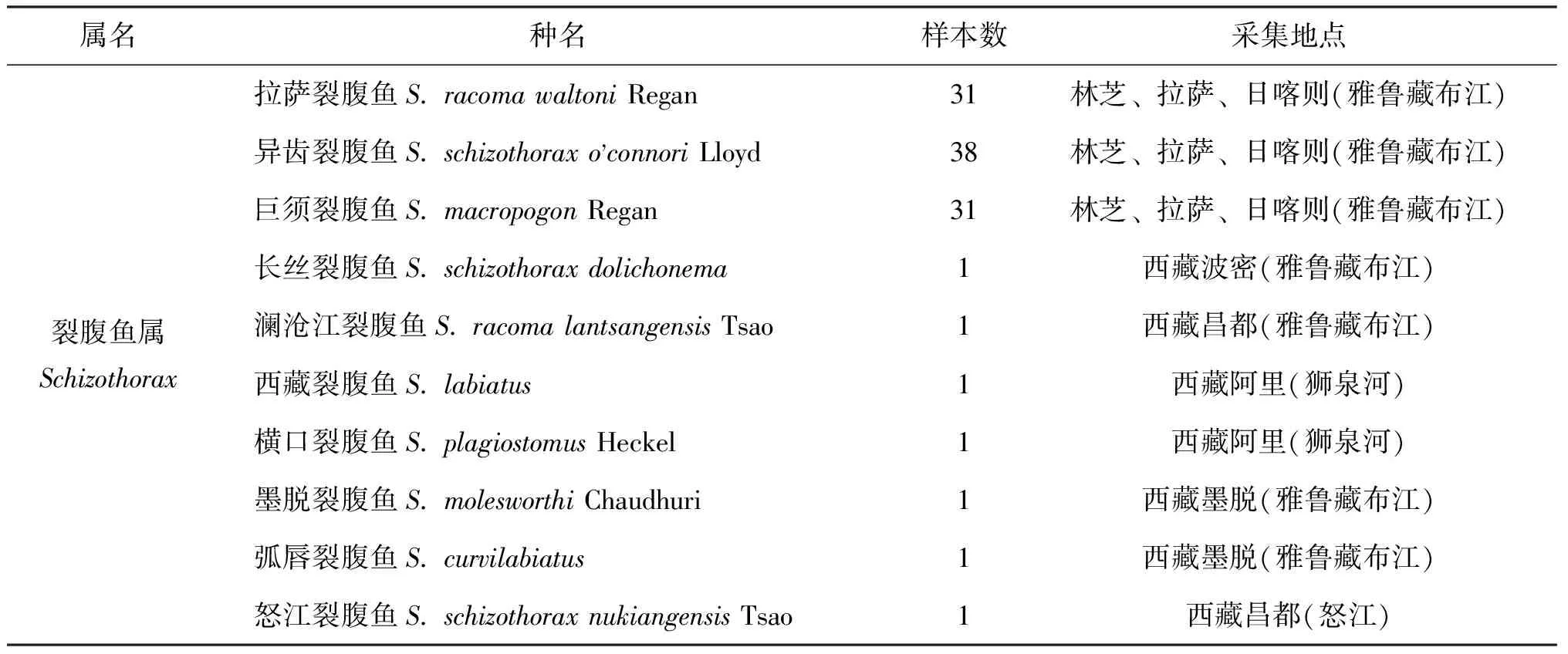
表1 样本采集信息Tab.1 The collection information of samples
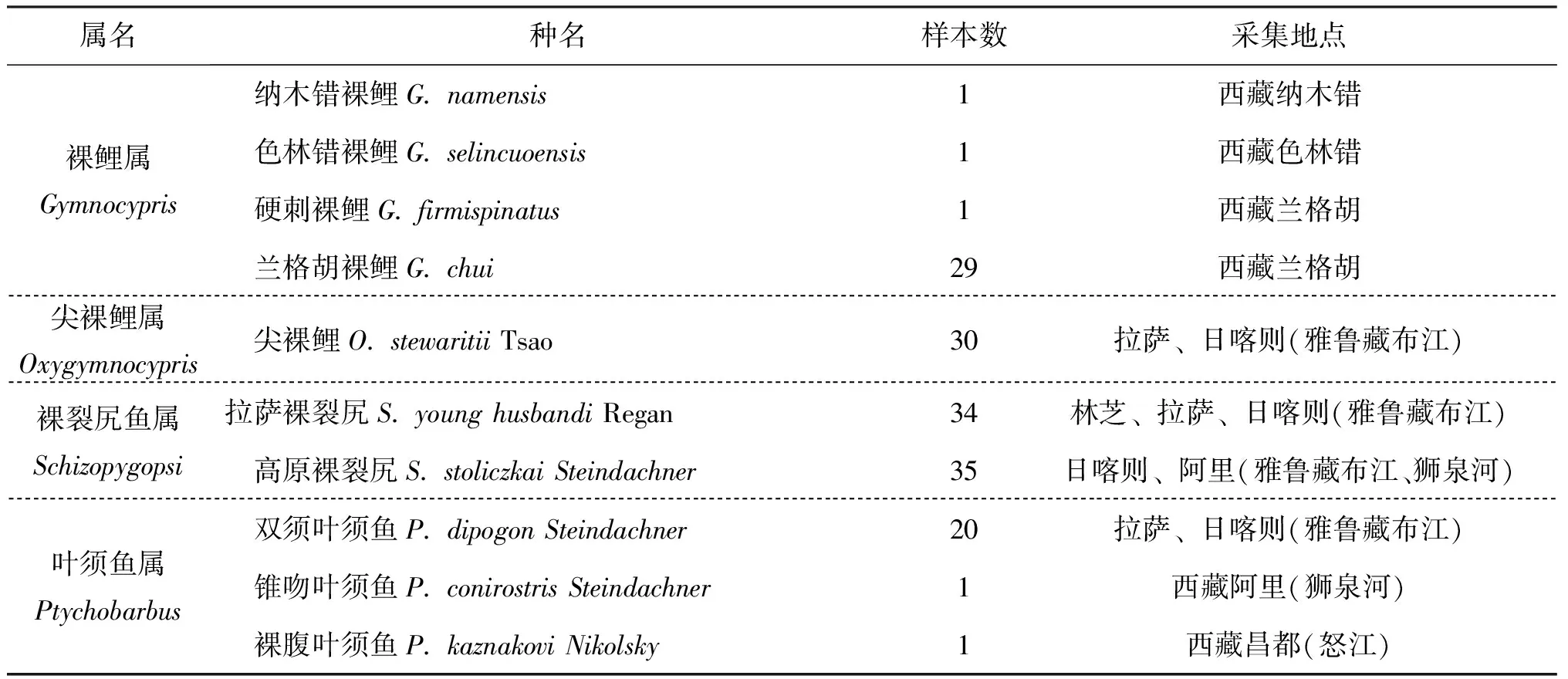
续表
2.2 COI基因序列特征
西藏水域裂腹鱼亚科鱼类COI基因片段经比对和人工校对后,序列长度为620 bp。包括外群所分析的262条序列620个位点中,有225个可变位点(无插入或缺失),其中161个为简约性位点,64个为单突变位点,信息位点最多的是第二位密码子(142个简约性信息位点),第三位密码子次之(18个简约性信息位点),而第一位密码子最少(1个简约性信息位点)。序列的碱基组成为A=25.74%、T=29.00%、C=27.44%、G=17.81%,平均转换/颠换值为8.754。裂腹鱼亚科内群260条序列620 bp位点中,有162个可变位点(无插入或缺失),其中143个为简约性位点,19个为单突变位点,序列的碱基组成为A=25.76%、T=28.98%、C=27.44%、G=17.82%,平均转换/颠换值为8.899。裂腹鱼亚科COI基因片段G+C含量见表2,碱基组成表现出反G偏倚的线粒体基因组的典型特征。
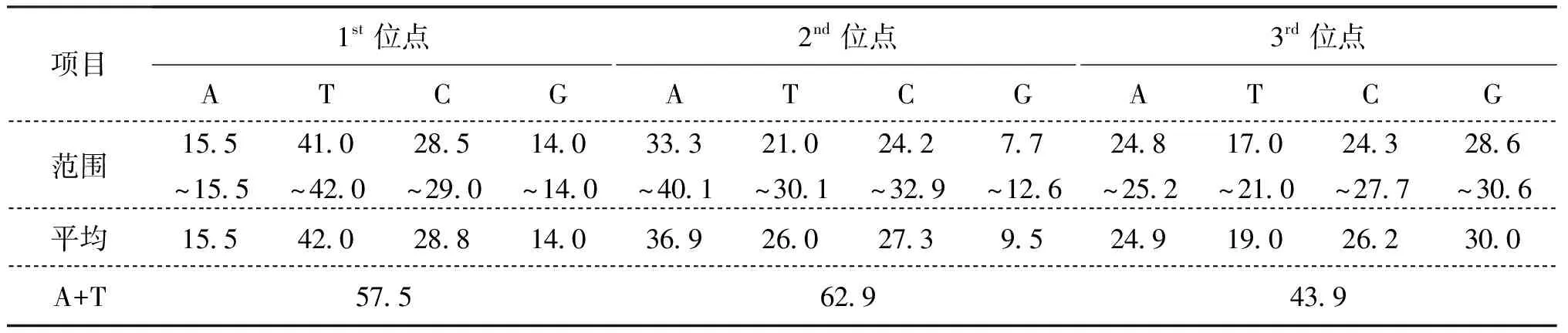
表2 裂腹鱼亚科鱼类不同密码子位碱基组成Tab.2 The comprises of base different codon position of Schizothroachinae %
2.3 遗传距离计算
基于形态学鉴定结果,本研究中构建了西藏水域裂腹鱼DNA条形码数据库,通过本地数据库和GenBank双重比对进行鉴定。计算260个样本种内及种间遗传距离,根据种内及种间遗传距离进行分组,分组原则是种内样本间遗传距离小于0.02,种间样本间遗传距离大于0.02,根据这一原则,260个个体共分为10组(表3)。对比形态学鉴定结果,Gp 1包含29尾拉萨裂腹鱼、38尾异齿裂腹鱼、29尾巨须裂腹鱼、1尾弧唇裂腹鱼和1尾长丝裂腹鱼;Gp2包含1尾纳木错裸鲤、1尾硬刺裸鲤、1尾色林错裸鲤、9尾兰格湖裸鲤、34尾拉萨裸裂尻、4尾尖裸鲤和2尾拉萨裂腹鱼;Gp 3包含35尾高原裸裂尻和20尾兰格湖裸鲤;Gp 4包含26尾尖裸鲤;Gp5包含20尾双须叶须鱼和3尾巨须裂腹鱼;Gp6包含1尾怒江裂腹鱼和1尾澜沧江裂腹鱼;Gp7包含1尾西藏裂腹鱼和1尾横口裂腹鱼;Gp8包含1尾锥吻叶须鱼;Gp9包含1尾裸腹叶须鱼;Gp10包含1尾墨脱裂腹鱼。10个组中每个种随机选取个体序列在NCBI中调用Blast程序进行比对以确定其种。
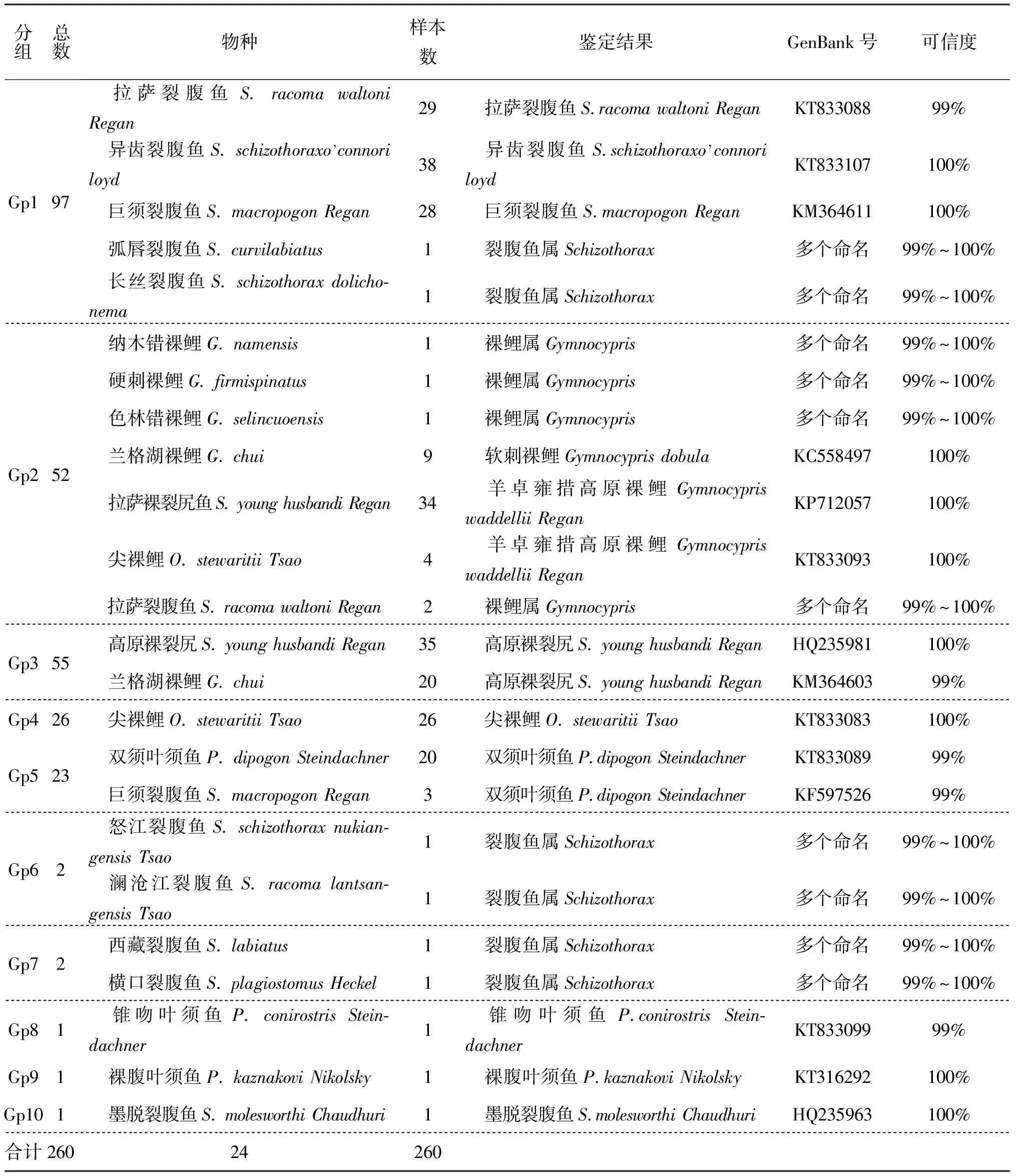
表3 260个样本根据遗传距离分组及鉴定结果Tab.3 The grouping and the identification results of 260 individuals based on genetic distance
通过Blast,Gp1中的弧唇裂腹鱼和长丝裂腹鱼,Gp2中的纳木错裸鲤、硬刺裸鲤、色林错裸鲤、拉萨裂腹鱼,Gp6中的怒江裂腹鱼、澜沧江裂腹鱼,Gp7中的西藏裂腹鱼、横口裂腹鱼由于数据缺失以及数据库中存在可信度较高的多个命名,只能鉴定到属水平。
考虑到各组中包含了由形态学鉴定结果的多个鱼种,并且有些鱼种在形态特征上差异比较明显,因此,对各组中个体数量大于1的鱼种按形态学鉴定结果再分组,计算组内及组间遗传距离用于进一步分析(表4)。

表4 组内及组间平均遗传距离Tab.4 The average genetic distance of with-in group and inter block
注:对应表3,Gp1-1为异齿裂腹鱼;Gp1-2为拉萨裂腹鱼;Gp1-3为巨须裂腹鱼;Gp2-1为拉萨裸裂尻;Gp2-2为兰格湖裸鲤;Gp2-3为尖裸鲤;Gp2-4为拉萨裂腹鱼;Gp3-1为高原裸裂尻;Gp3-2为兰格湖裸鲤;Gp5-1 为双须叶须鱼;Gp5-2为巨须裂腹鱼。
对各组进一步分析发现,Gp1中Gp1-1(异齿裂腹鱼)、Gp1-2(拉萨裂腹鱼)和Gp1-3(巨须裂腹鱼)之间形态特征具有明显的差异,再分组发现,组间遗传距离均大于0.01,组内遗传距离均小于0.01,其中组间遗传距离最小的是Gp1-1和Gp1-3,为0.011,组内平均遗传距离Gp1-1和Gp1-3均为0.001,组间遗传距离是组内遗传距离的11倍,Blast结果与再分组鉴定结果一致。Gp2中Gp2-1(拉萨裸裂尻)、 Gp2-2(兰格湖裸鲤)和Gp2-3(尖裸鲤)之间形态特征差异不明显,且遗传距离分析没有明显特征,Gp2-4为拉萨裂腹鱼,但拉萨裂腹鱼与该组中其他鱼种形态差异较大,由于样本时间收集跨度大,查证原始数据确定是样本分装时记录出错,且Blast结果证实该2个样本为裸鲤属,但存在多个命名。Gp2-1、Gp2-3经Blast比对,发现大部分命名为羊卓雍措高原裸鲤,Gp2-2为软刺裸鲤,均属裸鲤属,可信度均为100%。Gp3中Gp3-1(高原裸裂尻)和Gp3-2(兰格湖裸鲤)遗传距离分析没有明显特征,经Blast两种均在高原裸裂尻鱼。Gp 5中Gp 5-1(双须叶须鱼)和Gp 5-2数据记录为巨须裂腹鱼,但巨须裂腹鱼与该组中其他鱼种形态差异较大,由于样本时间收集跨度大,查证原始数据确定是样本分装时记录出错,Blast结果证实3个巨须裂腹鱼为双须叶须鱼,可信度为99%。
2.4 系统进化树构建
基于表3中的分组,每组中选取各物种代表性序列,总共24条,采用邻接法(Neighbor-Joining method,NJ)构建系统发育树,并通过Bootstrap检验聚类树各分支的置信度,系统发育树中的断线脂鲤和墨西哥丽脂鱼为外群,见图1。
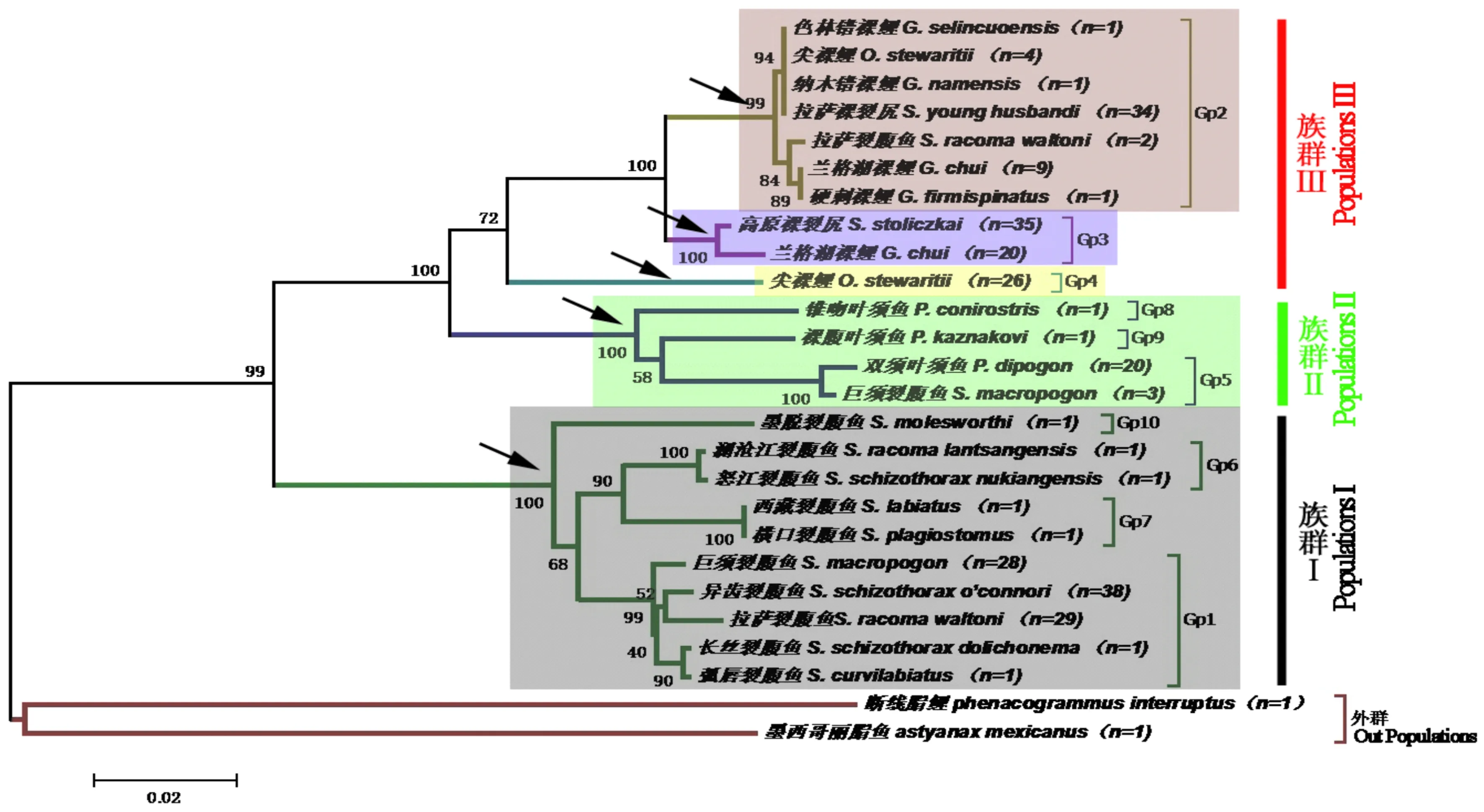
图1 裂腹鱼亚科系统进化树Fig.1 The phylogenetic tree of Schizothroacinae in Tibet water system
对应表3,图中不同的颜色区域代表不同的属,箭头所示为各个属分支Bootstrap检验支持率。
图1中,粉红色区域对应表3中的Gp2组,为裸鲤属,Bootstrap检验支持率为99%;淡蓝色区域对应表3中的Gp3组,为裸裂尻鱼属;支持率为100%,淡绿色区域对应表3中的Gp5、Gp8、Gp9组,为叶须鱼属,支持率为100%;浅灰色区域对应表3中的Gp1、Gp6、Gp7、Gp10组,为裂腹鱼属,支持率为100%;淡黄色区域对应表3中的Gp4组,为尖裸鲤属。断线脂鲤和墨西哥丽脂鱼为外群。裂腹鱼亚科鱼类形成一个支持率较高的单系群,Bootstrap检验支持率为99%。裂腹鱼亚科鱼类形成三个主要族群,族群I仅包括裂腹鱼属,Bootstrap检验支持率为100%,族群II仅包括叶须鱼属,Bootstrap检验支持率为100%,族群III包括裸鲤属、裸裂尻鱼属和尖裸鲤属,Bootstrap检验支持率为72%。系统进化树聚类方式与表3的分组方式及鉴定结果高度一致。
3 讨论
3.1 裂腹鱼亚科的形态学鉴定
本研究中的鱼类标本是在2014—2017年间所采集,采集到的鱼类以形态学鉴定共260尾,分属为5属20个种,均为裂腹鱼亚科,260尾个体种,249尾以分子生物学手段鉴定到了种水平,鉴出率为95.8%,179尾分子生物学鉴定结果与形态学鉴定结果一致,一致率为68.8%。在野外采样时,由于条件的限制,不能对所采集鱼类进行细致的形态学观察,尤其是裸鲤属的鱼类大多表观形态极为相似,不能准确鉴定到种,传统形态学在对物种的分类过程中耗时耗力,并且有些物种由于环境的差异使同一种类的物种呈现显著的形态学差异等[18],这都会给物种的鉴定带来一定的困难。在裂腹鱼亚科鱼类的分类过程中,也在不断修正,1962年,曹文宣等澄清了1个属名和8个种的同物异名[19],1964年又合并了20个同物异名[4],此后,张春霖等[20]、武云飞等[21]、陈毅峰等[5]均对西藏鱼类的分类进行了深入的研究,并修正了以往混乱的分类体系。由于物种间形态特征的模糊及分析样本量的限制,在裂腹鱼类个别种属的界定方面认为有些属并未形成单系群[11],说明在裂腹鱼亚科个别种属的界定方面仍存在较大争议,这与本研究中裸鲤属中种的界定比较混乱是一致的,而且,在形态特征相似物种的鉴定过程中,主观意志的存在也是不可避免的,本研究中拉萨裸裂尻鱼并未与高原裸裂尻归为一类,而归为裸鲤属,可能是形态学鉴定过程中的差异造成,若非形态学鉴定的错误,那么很有可能拉萨裸裂尻在分类学中本该归于裸鲤属,具体如何界定拉萨裸裂尻,在本研究中还需要补充裸鲤属其他种类的样本量。
3.2 DNA条形码在裂腹鱼亚科物种鉴定中的有效性
本研究中对所采集的260尾裂腹鱼亚科鱼类以组内遗传距离值小于0.02,组间遗传距离值大于0.02为原则,进行了分子生物学的鉴定,并与现场采集时形态特征鉴定的结果进行了互相印证。以遗传距离值为0.02为西藏水系裂腹鱼亚科物种鉴定的阈值在裂腹鱼属、裸鲤属的鉴定中具有一定的局限性。不同物种COI基因进化速率可能是不一致的,因此,使用DNA条形码进行物种的鉴定中,统一的遗传距离的阈值很难确定[22,23],本研究中,异齿裂腹鱼、拉萨裂腹鱼和巨须裂腹鱼之间以0.02作为遗传距离阈值,这3种鱼鉴定为一个种,事实上,它们在形态特征上具有比较明显的差异,而将其遗传距离阈值设定为0.01,则这3种鱼可以有效地得到区分。由于环境的影响,生存在不同水域的同一种鱼在形态特征上可能表现出较大的差异,而被认为是不同物种,但这些鱼类的基因序列在进化中并未表现出明显差异,Mohd Shamsudin等[24]研究表明龙鱼的体色变化与COI基因分化程度没有关系,陈信忠等[25]通过检索BOLD SYSTEMS和GenBank数据库中慈鲷、神仙鱼等观赏鱼的COI基因,检验DNA条形码在观赏鱼物种鉴定中的应用,表明这些观赏鱼由于频繁杂交,亲本的多样性等,COI基因相似性非常高,遗传距离小于0.01,使得DNA条形码难以鉴别这些鱼类。本研究中,裸鲤属种间遗传距离均小于0.01,最大为0.005,怒江裂腹鱼和澜沧江裂腹鱼种间平均遗传距离为0.002,西藏裂腹鱼和横口裂腹鱼种间平均遗传距离为0.000,弧唇裂腹鱼和长丝裂腹鱼与异齿裂腹鱼之间的遗传距离均小于0.001,因此,本研究中DNA条形码在这些鱼种间不能被有效区分。由于西藏水系纵横交错复杂,这些鱼类可能存在物种间的杂交种,或是同一物种在不同生存地理环境当中表现出的形态差异,比如兰格湖裸鲤和硬刺裸鲤均生活在兰格湖中,怒江和澜沧江均属于长江水系,西藏裂腹鱼和横口裂腹鱼均采自雅鲁藏布江上游水系,长丝裂腹鱼和弧唇裂腹鱼均采自雅鲁藏布江下游水系等生存环境和水系的影响可能使这些鱼类COI基因序列分化程度较低,也有同物异名存在的可能性。由于本研究中样本收集时间跨度较大,在样本整理过程中,存在样本分装、标记等人为失误,然而,通过COI基因片段可以将这部分样本有效区分。本研究中通过Blast程序比对后,裸鲤属中除羊卓雍措高原裸鲤外,其他均具多个命名,样本数只有1个的裂腹鱼属也具多个命名,事实上裸鲤属由于在形态上极为相似,在以往文献中的分类也是比较混乱的,提示裸鲤属内种间可能存在同种异名的情况,而样本数只有1个的裂腹鱼属中的几个种,在西藏水系中属非常见种,样本收集量本来就少,数据库中记录也很有限,且数据库中的数据也不一定是正确的。因此,本研究下一步将继续应用分子生物学鉴定手段,积累样本,着重分析上述问题。本研究中所构建的系统进化树直观地反映了以遗传距离阈值为0.02对裂腹鱼亚科鉴定分类结果,族群I作为原始族群置于发育树底部,而族群III以特化群置于发育树顶部。虽然线粒体DNA在物种鉴定中与形态学鉴定存在不一致性,但是能够很好地反映水系间的地理和历史联系,总体上相近的水系的物种具有更近的亲缘关系[26]。本研究中,采自长江水系的怒江裂腹鱼和澜沧江裂腹鱼以100%的支持率聚在一支,采自雅鲁藏布江上游水系的西藏裂腹鱼和横口裂腹鱼以100%的支持率聚在一支,采自雅鲁藏布江下游水系的长丝裂腹鱼和弧唇裂腹鱼以90%的支持率聚在一支。本研究中分析结果及推理仍受限于部分鱼种标本单一的事实,无法进行深入分析,这也是下一步需要开展的工作。
