刺参(Apostichopus japonicus)重要经济性状相关SNP标记的验证分析*
2019-01-17刘安然廖梅杰王印庚荣小军范瑞用陈贵平
刘安然 廖梅杰 李 彬 王印庚 荣小军 张 正 范瑞用 陈贵平
(1.上海海洋大学 上海 201306;2.农业农村部海洋渔业可持续发展重点实验室 中国水产科学研究院黄海水产研究所 青岛 266071;3.青岛海洋科学与技术试点国家实验室海洋渔业科学与食物产出过程功能实验室 青岛 266071;4.青岛瑞滋海珍品发展有限公司 青岛 266400)
刺参(Apostichopus japonicus)是我国北方重要的海珍品之一(廖玉林, 1997)。近年来,我国刺参养殖规模拓展迅猛,2016年海参养殖面积为218138 hm2,产量为204359 t,直接经济产值约300亿元(中国渔业年鉴, 2017),成为沿海渔业经济结构调整和渔民就业及增产增收的重要途径。但随着刺参产业规模的不断拓展,种质退化、生长缓慢、养殖周期长、抵御环境变化能力差、病害频发等一系列制约或潜在制约产业发展的瓶颈问题也日益凸显(王印庚等, 2014)。良种选育是解决上述问题的有效手段。在良种选育过程中,分子标记辅助选择育种(Marker-assisted selection, MAS)在选择效率和选择准确性上表现出良好的应用潜力(Würschumet al, 2014)。
在水产动物分子标记辅助育种方面,随着分子技术的发展,构建遗传连锁图谱,挖掘数量性状基因座(Quantitative trait locus, QTL),利用与目标性状紧密连锁的分子标记促进对性状的遗传改良是当今水产动物遗传育种的研究热点(Collardet al, 2005)。目前,已经构建遗传连锁图谱并发掘了候选 QTL标记的水产物种有鲤鱼(Cyprinus carpio)(Zhaoet al, 2013)、半滑舌鳎(Cynoglossus semilaevis)(Chuet al, 2014)、中华绒螯蟹(Eriocheir sinensis)(Qiuet al, 2016)、贻贝(Hyriopsis cumingii)(Baiet al, 2015)、九孔鲍鱼(Haliotis diversicolor)(Renet al, 2016)、大西洋鲑鱼(Salmo salar)(Gonenet al, 2015)等。随着刺参分子标记的开发,Tian等(2015)构建了高密度刺参遗传连锁图谱,并筛选出一个与生长关联的QTL;和飞(2016)发掘了刺参的 9个与生长和对灿烂弧菌(Vibrio splendidus)的抗病力相关的QTL。这些分子标记的发现为刺参的良种选育提供了宝贵资料。QTL的验证和鉴定是实施MAS的前提(Houet al, 2011),本研究在前期构建的刺参遗传连锁图谱和QTL分析的基础上,筛选与体重、体长、体宽、棘刺总数、抗病力5个性状相关的SNP位点,并在扩大繁育群体中进行验证,评估不同基因型与相关性状的相关性,以期获得真实可靠的 QTL结果,为刺参的分子标记辅助选育奠定基础。
1 材料与方法
1.1 实验材料
实验所用刺参苗种采集自青岛瑞滋海珍产品发展有限公司,为同一批次大规模育苗并同一养殖条件下培育的 8月龄苗种,该批苗种繁育用亲本数量为800头,苗种繁育与培育参照该公司《苗种繁育技术规范》进行。苗种采集后暂养于实验室玻璃钢桶(D=80 cm,h=60 cm)内,充气,水温(18.0±0.5)℃,每天换水1/2,每天投喂1次。暂养稳定后用于后续实验。
1.2 实验方法
1.2.1 刺参生长性状的测量 随机选择 200头刺参苗种进行体长、体宽、体重和棘刺总数的测定。体长、体重测量方法:用0.6 mol/L的MgSO4溶液浸泡参苗,待参苗麻醉后,用圆规标定参苗长度后,再用游标卡尺测量其体长、体宽,精确到0.01 cm;用电子天平测量其体重,精确到0.01 g,并记录每个刺参的棘刺总数(魏杰等, 2007; 廖梅杰等, 2010)。分别解剖分离其纵肌组织,于95%乙醇中,-20℃保存。
1.2.2 刺参抗病力性状的测量 随机选择 200头刺参苗种进行人工攻毒浸染实验,攻毒所用菌株为实验室分离保存的刺参腐皮综合征重要致病原-灿烂弧菌,人工浸染浓度为 1.0×107CFU/ml,攻毒实验为期30 d。攻毒期间,每3 d换水1次,换水后及时补充病原菌,每天观察刺参苗种化皮情况,对攻毒期间出现化皮症状的苗种及时捞出,记录其发病时间(记录为存活天数)并分离保存其纵肌组织。到攻毒实验结束时,仍未出现化皮症状的苗种记录为抗病苗种,其发病时间统一记录30 d。实验结束后分别解剖分离其纵肌组织置于95%乙醇中,-20℃保存(和飞等, 2017)。同时,设置未攻毒感染组作为对照组,以排除环境对刺参发病的影响。
1.2.3 基因组DNA的提取 使用Omega软体动物DNA提取试剂盒提取基因组DNA,相关操作参照试剂盒说明书。用1.0%的琼脂糖凝胶电泳检测DNA的完整性;用NanoDrop分光光度计测DNA的浓度,将提取的DNA统一稀释成50 ng/μl,-20℃保存备用。
1.2.4 SNP位点的筛选和引物设计 在前期构建的遗传连锁图谱中筛选到与体长、体宽、体重、棘刺总数及抗病力5个性状相关的9个QTL,这9个QTL包含81条SLAF标签、189个SNP位点。依据HRM分型的条件,挑选SNP位点两侧侧翼序列大于200 bp且无不确定碱基,并且 SNP的突变频率>1%的测序序列,利用软件Primer Premier5.0进行SNP扩增用引物设计,引物设计原则为长度在 20 bp左右,GC含量为45%~65%,产物链长度不超过120 bp,且利用Oligo7.0分析引物参数时不能有能值高的错配、二聚体和发卡结构。同时,合成HRM基因分型所需的标尺高低温内标及其反向互补序列(王婷等, 2014)。SNP引物和内标引物送生工生物工程(上海)技术服务有限公司进行引物合成。
1.2.5 PCR扩增及基因分型 自生长性状和抗病性状测定群体中各选取 96个个体,采用(HRM)法(Reedet al, 2007)进行SNP位点的PCR扩增和基因分型。10 µl PCR 反应体系:DNA 模板 1 μl (50 ng/μl),4.5 μl 2×ESTaqMaster Mix,上下游引物各 0.5 μl(10 μmol/L),3.5 μl ddH2O;PCR 反应程序:95℃预变性 5 min;94℃变性 30 s,退火 30 s(退火温度见表1),72℃延伸30 s,共35个循环;最后72℃延伸7 min,4℃保存。用2%琼脂糖凝胶电泳检测PCR扩增产物。向PCR产物中加入染料LC Green 1.0 μl,高低温内标的正负向引物各0.25 μl,95℃变性5 min后降温至4℃保存,利用Light Scanner进行基因分型,基因分型用程序为以0.1℃/s的速度从56℃快速升温到97℃,并以1次/s的密度采集荧光信号,得到熔解曲线,采集结束后用Light Scanner配套软件对熔解曲线进行分析(Tinget al, 2014)。
1.2.6 数据处理 统计各 SNP位点的基因分型结果,利用Popgene32软件处理统计各位点上的最小等位基因频率(Minor allele frequency, MAF)、观测杂合度(Observed heterozygosity,Ho)、期望杂合度(Expected heterozygosity,He)和固定指数(Fixation index,F),并进行Hardy-Weinberg平衡检测。用Cervus软件计算各位点多态信息含量(Polymorphism information content,PIC)等指标。
利用SPSS20对数据进行统计分析,用一般线性模型(GLM)对各 SNP位点以及二倍型与 5个性状的关联性进行最小二乘分析,采用Ducan’s多重比较分析 SNP位点各基因型、二倍型在生长及抗病性状差异的显著性,显著性水平为P<0.05。
2 结果与分析
2.1 引物设计结果
根据实验室前期高密度遗传连锁图谱和 QTL分析结果对所筛选的189个候选SNP位点的序列进行分析,筛选到可用于设计SNP扩增的引物序列26条,依据引物设计条件,成功设计SNP扩增引物14对,但鉴于SNP107位点引物与高温内标重合,最后用于SNP验证的扩增引物共13对,相关引物具体信息见表 1。其中,与体长性状相关的候选 SNP位点 4个(SNP6、SNP9、SNP40和SNP160),与体宽相关的候选位点6个(SNP6、SNP9、SNP40、SNP64、SNP140和 SNP160),与体重相关的候选位点 4个(SNP6、SP40、SNP64和SNP160),与棘刺总数相关的候选位点 5个(SNP6、SNP40、SNP64、SNP91和 SNP160),与抗病力相关的候选SNP位点11个(SNP6、SNP9、SNP64、SNP88、SNP91、SNP109、SNP112、SNP113、SNP126、SNP134和 SNP160)。由此可见,SNP6、SNP9、SNP64和 SNP160这 4个候选位点既与生长性状相关,也与抗病力相关。
2.2 SNP多态性分析
利用13对引物对所检测的个体进行基因分型,利用Popgen32对所获得的所有个体的基因型统计分析。结果显示,3个位点(SNP6、SNP64和 SNP91)在检测群体中没有多态性,为单态位点,其余10个位点为多态性位点。10个多态性位点的最小等位基因频率(MAF)介于 0.016(SNP9)~0.332(SNP160)之间,平均值为 0.173;各位点的观测杂合度(Ho)介于 0.031(SNP113)~0.818(SNP9)之间,平均值为0.433;期望杂合度(He)介于 0.031(SNP113)~0.834(SNP9)之间,平均值为0.402;多态信息含量(PIC)介于0.030(SNP113)~0.393(SNP160)之间,平均0.248(表2)。6个位点的观测杂合度(Ho)大于期望杂合度(He),固定指数(F)只有在 SNP160位点是正值,其他均为负值;Hardy-Weinberg平衡分析结果显示,有6个SNP位点显著偏离平衡(P<0.05)。
2.3 SNP位点与相关性状的关联性验证
对刺参体长、体重、体宽、棘刺总数和抗病力性状与10个多态性SNP标记的相关性分析结果见表3。经验证,SNP40、SNP88、SNP112、SNP126和SNP160位点与所关联的性状显著相关(P<0.05)。位点SNP40和SNP160与体长、体重、体宽、棘刺总数呈显著相关,其中,SNP40与体长、体宽、体重呈极显著相关(P<0.01);SNP160与体宽、体重呈极显著相关(P<0.01);SNP88、SNP126与存活天数显著相关(P<0.05);位点SNP112与存活天数极显著相关(P<0.01)。
2.4 SNP各位点不同基因型与生长性状的相关性分析
对与5个经济性状显著相关的10个多态性SNP位点进行不同基因型与生长性状多重比较分析,结果见表4。SNP40的CC基因型的个体在体长、体重、体宽、棘刺总数4个性状的平均值显著高于CT基因型的个体(P<0.05);SNP88、SNP112、SNP126 基因型分别为 CC、AA、TT的个体在灿烂弧菌的攻毒实验中的存活天数的平均值显著高于CT、AG、TG基因型(P<0.05);SNP160的AA基因型个体在体长、体重、体宽、棘刺总数的平均值显著高于基因型CC的个体。由此可见,与生长和棘刺相关的 SNP40和SNP160位点的优势基因型分别为CC和AA;抗病力相关的SNP88、SNP112和SNP126位点的优势基因型分别为CC、AA和TT。
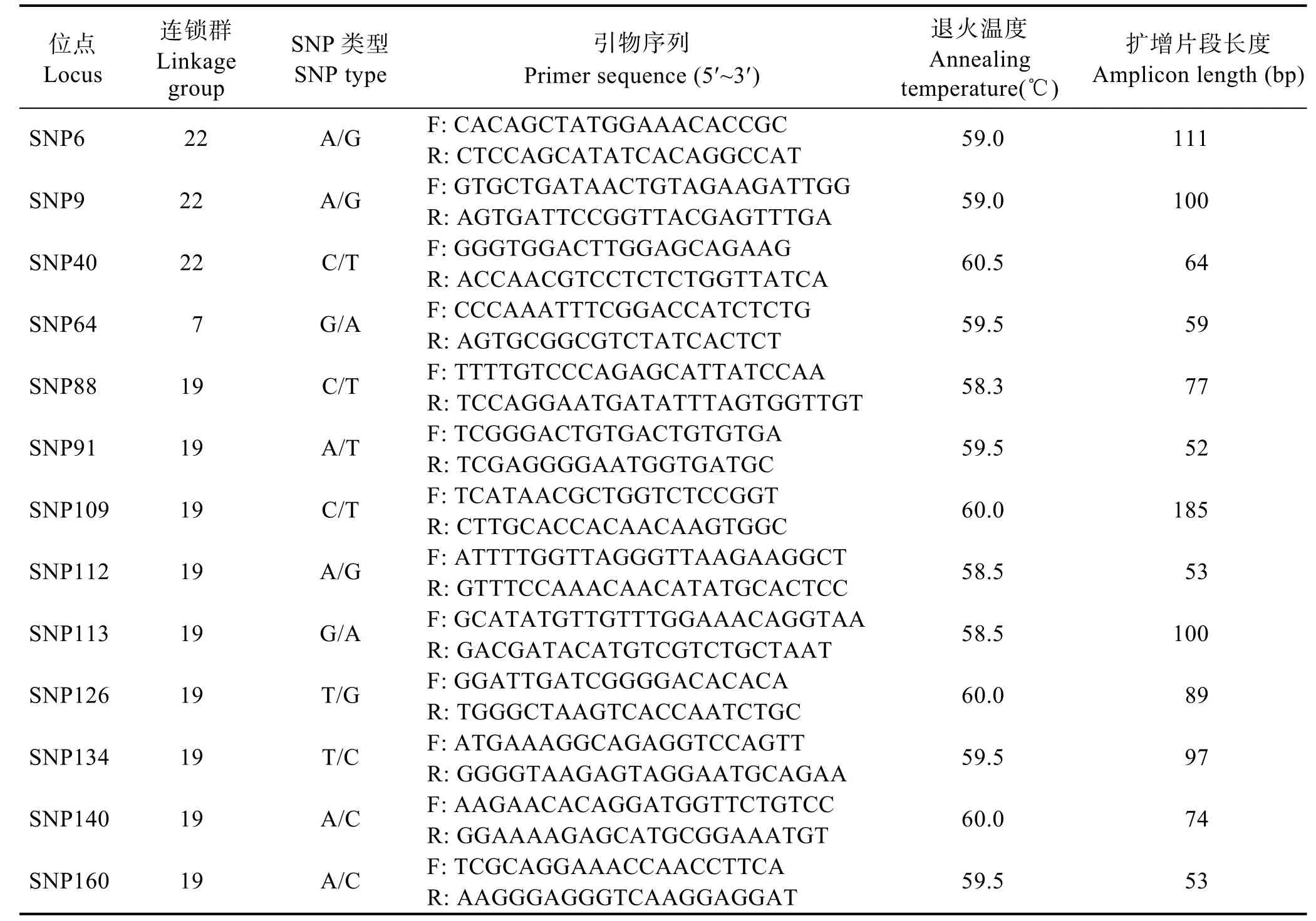
表1 刺参13个SNP位点引物信息Tab.1 Information of 13 SNPs of A.japonicus
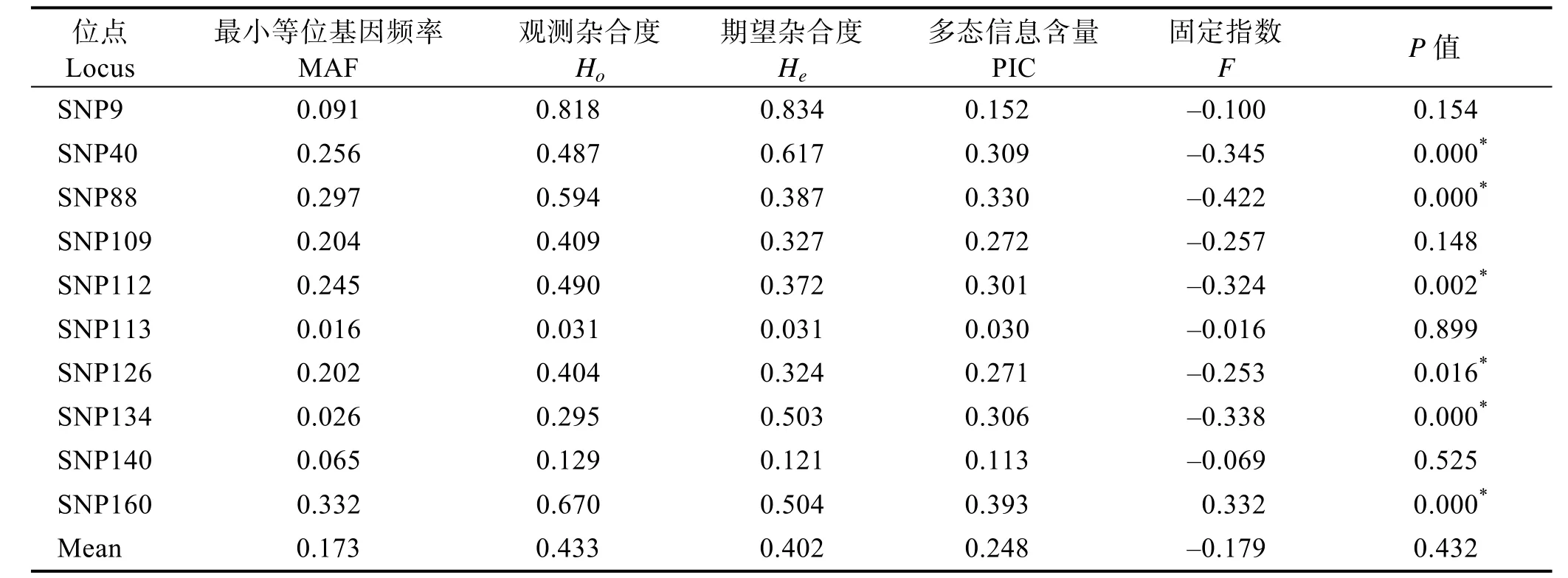
表2 10个多态性SNP位点的遗传参数Tab.2 Genetic diversity at 10 polymorphic SNPs of A.japonicus
2.5 SNP二倍型构建及其与性状的关联分析
分别对与抗病力相关和与生长性状相关的 SNP位点构建二倍型,比较二倍型关联性状之间的差异显著性,结果分别见表5、表6。利用SNP88、SNP112和SNP126这3个抗病力相关的SNP位点在检测群体中共检测到6种二倍型,其中,K1(CC AA TT)的存活天数最高,该基因型所有个体在整个攻毒期间均未发病,K2(CC AA TG)次之,但都显著高于其他二倍型;利用SNP40和SNP160这2个与生长和棘刺性状相关的SNP位点,在检测群体中共检测到6种二倍型,关联分析结果显示,S1(CC AA)和S3(CC AC)在生长性状(体长、体重、体宽)上显著优于其他二倍型。而棘刺总数性状关联分析结果显示,S5(CT AC)为劣势基因型,其他几种二倍型差异不大。

表3 SNP位点与体长、体重、体宽、棘刺总数、存活天数相关性分析结果Tab.3 Significant correlation analysis between SNP loci and body length, body weight, body breadth, pallet number and survival day
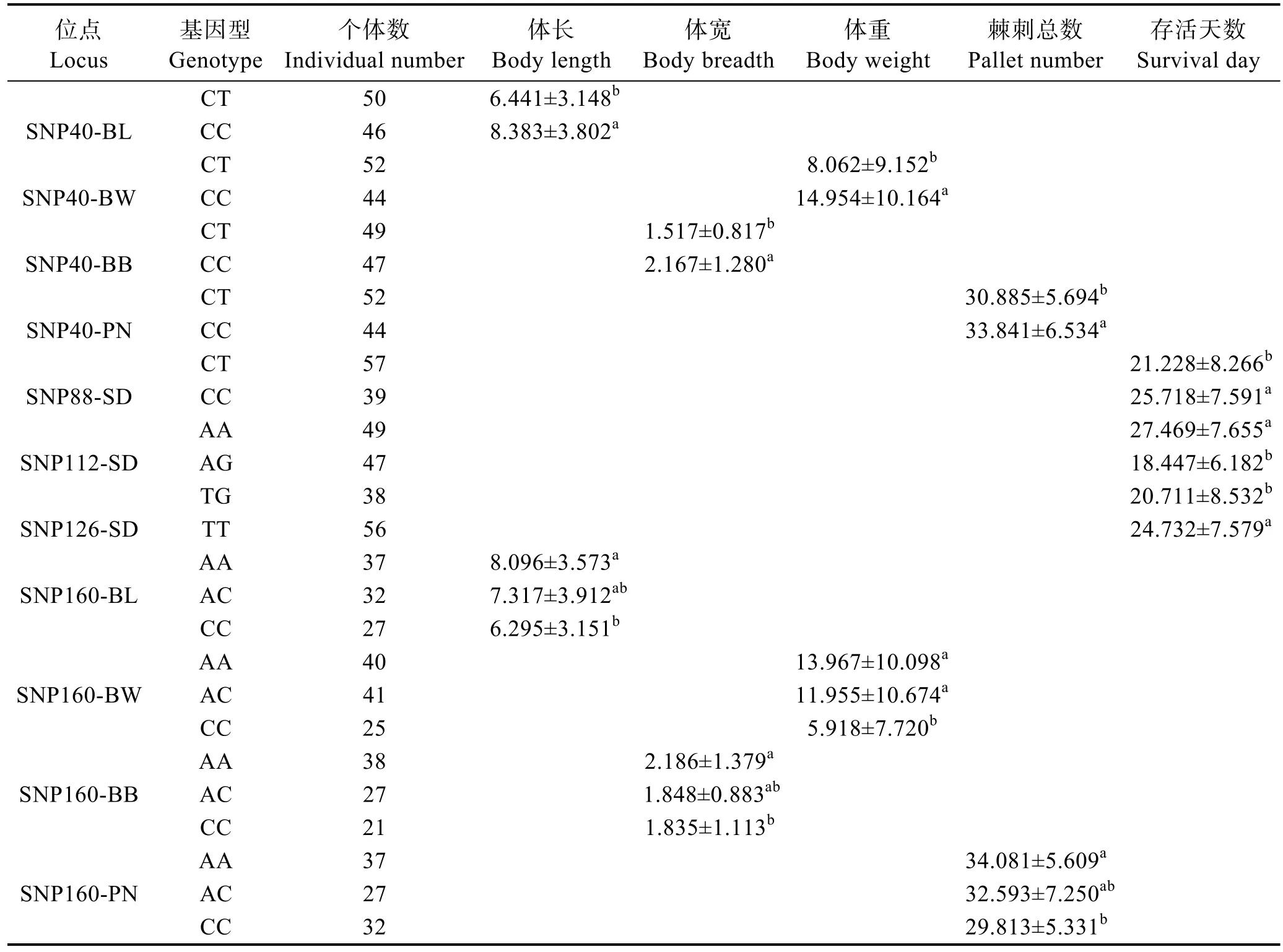
表4 SNP位点不同基因型在关联性状的多重比较分析Tab.4 Multiple comparisons of related traits with genotypes of SNP

表5 SNP二倍型与刺参抗病力的关联分析Tab.5 Association between diplotypes of SNPs and disease resistance
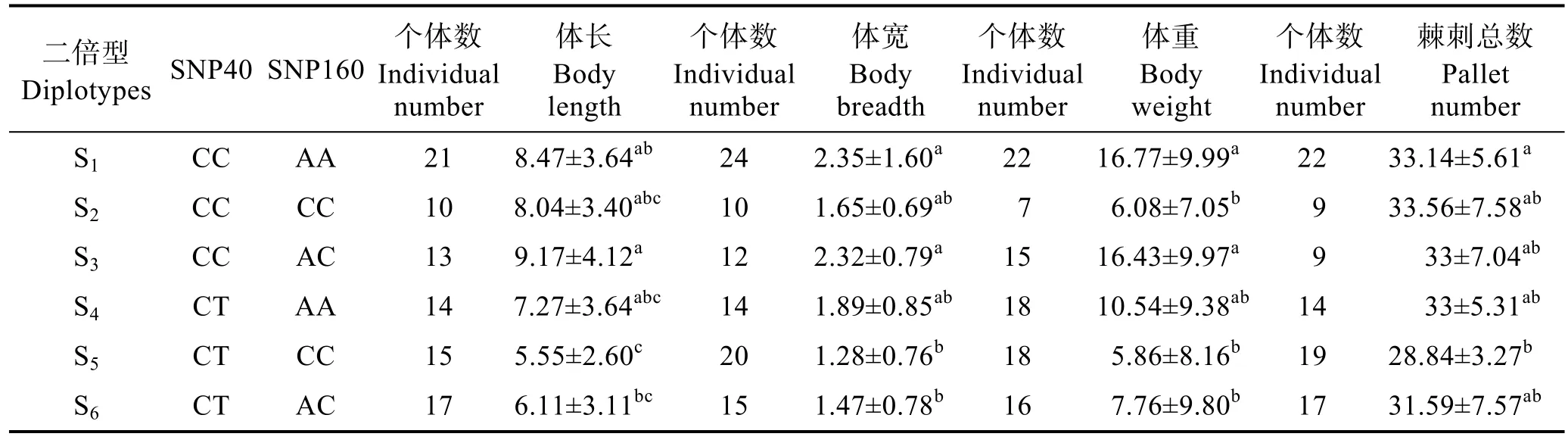
表6 SNP二倍型与刺参生长性状的关联分析Tab.6 Association between diplotypes of SNPs and growth trait
3 讨论
分子标记辅助水产动物育种是目前水产动物遗传育种的研究重点,但由于小样本作图 QTL区间过大,且数量性状受环境影响大,导致 QTL的检测和对QTL效应评估发生偏离(Würschumet al, 2014),在一个群体或者家系中定位到的 QTL可能不适用于另一个家系或者群体,因此,QTL的验证和鉴定是实施MAS的前提(Houet al, 2011)。目前,在水产物种中已进行了QTL验证的相关研究报道,Wang等(2014)对牙鲆(Paralichthys olivaceus)的抗病力关联的 QTL进行验证,韦国建等(2015)对马氏珠母贝(Pinctada martensii)的2个生长关联基因进行验证,发现优势基因型。随着刺参研究热度的增加(孙国华等, 2011),目前对刺参性状相关SNP发掘越来越多,Gao等(2013)开发了 26个与刺参的防御机制相关的 SNP位点;Dong等(2016)在对野生和人工养殖的刺参遗传多样性和种群结构的研究中,利用HRM发掘了51个相关SNP位点,解释了遗传多样性降低的原因;Li等(2016)发现了与刺参干重相关的3个SNP位点;董玉等(2016)在对SNP与生长的关联分析中发现了10个与生长性状显著相关的位点。但这些标记是否能直接用于遗传育种,需在扩大群体中进一步验证。本研究是在前期高密度遗传连锁图谱和 QTL分析获得的候选SNP位点的基础上,在扩大群体中对获得的SNP位点进一步验证,筛选出可用于遗传育种、与生长性状和抗病力相关的SNP位点,并确定其优势基因型,可为分子标记辅助育种在育种生产中直接应用提供技术支撑。
QTL验证分析的关键步骤是对SNP进行准确的基因分型,目前已有多种 SNP分型方法:半热不对称反向 PCR(Semi-thermal asymmetric reverse PCR)(Longet al, 2015);多光谱分析法(Mass spectrometry)(Parkaet al, 2017)、Fast-GBS(Fast Genotyping-bysequencing) (Torkamanehet al, 2017)。SNP的检测方法虽有很多种,但受设备与试剂昂贵、检测时间长、步骤繁琐的限制,不便大规模应用。近年来兴起的高分辨率熔解曲线分析技术(High Resolution Melting,HRM) (Liet al, 2012),因其具有灵敏度高、特异性好、操作简便、能实现真正的闭管操作、分型结果准确率高、费用成本较低的优势,已被广泛用于 SNP的检测和分型(Liewet al, 2004)。本研究对13个位点的多态性分析结果表明,10个SNP位点的多态信息含量在0.030~0.393之间,没有高等位多态性的位点,这与 SNP为二等位标记、基因型较少有关(Bmokes,1998)。检测结果中,位点SNP9、SNP40和SNP134所检测的个体中的期望杂合度大于观测杂合度,其他位点的观测杂合度大于期望杂合度,但相差不大,固定指数(F)除位点SNP160是正值,其他均为负值,说明普遍存在杂合子缺失现象,这可能是选育造成纯合度增加导致。
孙效文等(2010)建议,将 QTL结果应用育种之前,最好在大样本量的随机群体中进行验证,以增加QTL在育种群体中的利用价值。本研究对10个多态性的候选性状关联位点与性状进行相关性分析验证,筛选出5个性状相关位点,验证成功率为50%。由此可见,在扩大群体中对 QTL位点进行验证对提高标记的准确性具有重要意义。对所检测 SNP位点与相关性状的关联性验证表明,位点 SNP40和 SNP160与体长、体重、体宽、棘刺总数4个性状均呈显著相关,即出现了同一个位点关联多个性状的现象,说明这4个性状之间存在一定程度的关联,相关结果可为刺参不同性状间的间接良种选育提供参考。孙文静(2010)和董玉等(2016)相关研究结果也表明,刺参所检测的相关 SNP位点同时与体长、体宽、体重和体壁重4个性状显著相关,相关研究结果与本结果基本一致。此外,在抗病力相关SNP位点检测中,SNP88、SNP112和SNP126均与抗病力显著相关,出现一个性状有多个 SNP位点与之关联的现象,表明该性状受多个位点控制,符合数量遗传学相关理论(孙效文,2010)。抗病力相关SNP位点与生长相关SNP位点不存在重合现象,表明生长与抗病力2个性状相关性不大,后期选择育种过程中需作为2个性状进行独立选择。
对同一个 SNP位点不同基因型关联性状的多重比较分析结果显示,生长和刺型相关SNP位点SNP40和SNP160的优势基因型分别为CC和AA,抗病力相关 SNP位点 SNP88、SNP112、SNP126的优势基因型分别为 CC、AA、TT。由此可见,相应位点的纯合型会表现出更好的表型,在以后的选育工作中可对相应基因进行筛选与组合。
为了降低利用单个 SNP标记进行性状关联分析的局限性,本研究对性状相关的多个 SNP位点通过构建二倍性相关分析,以提高分析的准确性(De Bakkeret al, 2005)。利用SNP40、SNP160构建的与生长相关的二倍型,显示S1(CC AA)和S3(CC AC)在生长性状(体长、体重、体宽)上显著优于其他二倍型,均为与生长性状(体长、体重、体宽)相关的优势基因型组合,证明 SNP40位点对生长贡献率更大一些,鉴于 SNP40、SNP160分别位于LG22和LG19连锁群上,二者不存在连锁效应,在将来育种实践中对携带这 2个位点优势基因型的亲本进行组合构建优势二倍型个体。同样,利用SNP88、SNP112、SNP126这 3个位点构建的二倍型与性状分析结果表明,K1型(CC AA TT)个体的存活天数最高,也就是抗病力最强,鉴于SNP88、SNP112、SNP126均来自高密度遗传连锁图谱的LG19连锁群,相关位点是否存在互作效应有待下一步证实。
综上所述,本研究是在前期高密度遗传连锁图谱和QTL分析获得的候选SNP位点的基础上,在扩大群体中对获得的 SNP位点进一步验证,筛选出可用于遗传育种的、与生长性状和抗病力相关的 SNP位点,并确定其优势基因型,相关研究结果为实施刺参分子标记辅助育种在育种生产中应用有重要意义。
