药用植物冬凌草高通量转录组测序与分析△
2019-01-16陈延清胡志刚刘大会黄必胜刘迪
陈延清,胡志刚,刘大会,黄必胜,刘迪
(湖北中医药大学 药学院,湖北 武汉 430065)
大多数唇形科药用植物味甘苦,性微寒,中国民间习用于治疗失眠、消化不良、风湿病、肿瘤等[1-2]。现代化学和药理研究表明,唇形科植物主要的生物活性成分为二萜化合物,由于其复杂多样的碳骨架和具有多种抗肿瘤药理活性成分,二萜类化合物已经成为抗癌药物候选药物[3-4]。冬凌草Isodonrubescens(Hemsl.)H.Hara属于唇形科中药用价值和经济价值较为重要的药用植物,其中全草中含量较高二萜类化合物冬凌草甲素、冬凌草乙素具有良好杀菌消炎、抗肿瘤、抗氧化等作用。目前,冬凌草中的二萜类化合物的研究与市场开发成为了热点[5-6]。随着对冬凌草资源与品质的研究,其道地性和种质资源的遗传规律探索也逐渐深入[7]。采用现代化学分析技术,对冬凌草最佳采收期[8],以及对不同产地来源主要二萜活性成分冬凌草甲素和冬凌草乙素的含量差异进行了研究[9],并建立了不同产地冬凌草药材的HPLC-PDA指纹图谱特征图谱[10],阐述冬凌草药材的最佳采收时期和产地差异。利用随机引物标记RAPD和ISSR[11],以及特异标记简单序列重复SSR(simple sequence repeat,SSR)[12]等分子标记技术,探索了冬凌草遗传多样性规律,为筛选优质种质资源提供了丰富的遗传基础,探讨药材道地性的形成机制。
高通量测序(next generation sequencing,NGS)技术是对传统测序的革命性改变,转录组学研究方法转录组测序(RNA-seq)揭示药用植物遗传育种和基因进化规律,其具有通量高、成本低、不受基因组序列信息限制等优点[13]。对于非模式生物而言,例如冬凌草,转录组测序更偏重基因编码区域,与庞大的基因组信息相比,重复元件和富含GC的区域比较少,拼接相对容易[14],转录组测序针对物种转录本直接进行测序,为基因组序列信息有限的非模式生物提供可能。本研究利用 Illumina Hiseq 4000对药用植物冬凌草药用部位叶和茎进行了高通量转录组测序,建立冬凌草转录组数据库,为深入研究冬凌草中冬凌草甲素等有效成分的生物合成途径及其调控机制提供基础,为药用活性成分的合成代谢以及药材道地性品质的形成提供生物学依据。高效地发现非模式物种的新基因和SSR位点的信息分析,也为开展分子标记辅助育种、基因工程技术选育创制新的药用冬凌草优良品种奠定基础,对优质中药材的品种选育和规范化栽培生产具有重要的指导意义。
1 材料与方法
1.1 材料
本研究的冬凌草样品来自于道地产区河南济源[15],采于药材最佳采收期[8],在同一生境下,选取冬凌草地上部分叶和茎为研究材料,迅速用液氮研磨,用于 RNA样品提取。
1.2 总RNA的提取文库、构建和转录组测序
用 TRIzol©Reagent、试剂盒Plant RNA Purification Reaget试剂盒(Invitrogen,美国)提取、纯化叶与茎总RNA[16]。分别用Nanodrop 2000与 Agilent 2100 Bioanalyzer进行总RNA质量和纯度检测。为了获得尽可能丰富的冬凌草采收期转录组信息,特别地将同珠的叶和茎的RNA混合。富集mRNA使用 Dynabeads Oligo(dT)25的磁珠。在Thermomixer中加入fragmentation buffer将mRNA打断为短片段,添加0.1 mol·L-1Mg2+缓冲液到所得的 mRNA 中,高温下使其片断化后,然后将 mRNA为模板反转合成cDNA、然后通过磁珠纯化、末端修复、3’末端加碱基 A、加测序接头等步骤后选择片段大小,进行PCR扩增,adaptor构建冬凌草转录组测序文库,使用Illumina HiSeq 4000 进行测序及分析。
1.3 测序数据质量处理及组装
使用SeqPrep(https://github.com/jstjohn/SeqPrep)、Sickle(https://github.com/najoshi/sickle)软件进行过滤原始数据,从而去除含有Primer/Adaptor的序列、含N比率超过10%的reads和含接头的低质量reads,并且使Q30(碱基测序错误率低于0.1%)达到80%以上。从而获得去掉接头后的测序序列(cleanreads)进行后续分析。
由于缺乏冬凌草基因组的数据,因此利用Trinity软件对冬凌草叶与茎转录组重头组装(de novo assembly),测序序列之间的重叠(overlap)信息组装得到重叠群(contigs),依据两端信息(paired-end)和重叠群的相似性,然后使用TGICL软件进行聚类整合和延长得到非冗余的转录本(transcripts)。并从中选取最长的转录本作为非重复序列基因(unigenes)。Trinity 参数设置为K-mer=25,当序列延伸成重叠群时重复为 K-mer-1。拼接完毕后,将没有匹配结果的短读序的转录本删除。处理与注释冬凌草叶和茎的差异表达基因使用相同参考基因,得到同一时期冬凌草叶和茎所有Unigenes数据库使用TGIGL的聚类组装策略。
1.4 功能基因注释
通过Transdecoder version v 2.0.1对unigenes的CDS进行了预测。利用Trinotate软件包(v 2.0.0,https://trinotate.github.io/)的标准流程对All-unigenes进行了全面的注释,用 Swissprot[17]、Pfam[18]、eggNOG[19]、Gene Ontology[20]、SignalP[21]、Rnammer[22]和KEGG Ontology数据库注释。
我们使用Trinotate软件(版本:20140708)对预测出的编码序列进行功能注释。Trinotate软件整合了多个数据库,可以从不同角度对转录本进行功能注释,使用软件或数据库如下:使用diamond(版本:0.5.3)blastx,SwissProt、TrEMBL和kegg作为数据库,对转录本进行注释;同时,还使用blastp(版本:2.2.28+),SwissProt作为数据库,对转录本进行注释;使用hmmscan(HMMER)软件(版本:3.1),Pfam(Bateman et al.2002)作为数据库,对预测的蛋白序列的结构域进行注释;用SignalP(Petersen et al.2011)(版本:V4)软件,预测信号肽;使用tmHMM(版本:2.0c)预测跨膜结构;使用RNAmmer(版本:1.2)预测rRNA基因。
1.5 基因表达水平及表达差异量分析
基于Trinity拼接得到的转录本,我们进行表达丰度计算。最先利用bowtie(版本:1.0.1;参数:mismatch=2)将每个样品的reads分别比对到组装出来的转录本上,得到bam格式的比对文件,然后使用RSEM(版本:v1.2.17)(http://deweylab.biostat.wisc.edu/rsem/)计算比对到转录本上的reads的表达量。为了便于不同基因之间进行表达量的比较,我们使用FPKM对表达量进行标准化。FPKM(expected number of Fragments Per Kilobase of transcript sequence per Millions base pairs sequenced)[23]是每百万fragments中来自某一基因每千个碱基长度的fragments数目,其同时考虑了测序深度和基因长度对fragments计数的影响。
差异表达基因(differentially expressed genes,DEGs)是基于基因表达水平分析中得到的read count数据,根据筛选阈值筛选出来的表达差异的基因。参考edgeR进行差异基因分析,筛选阈值选择为False Discovery Rate(FDR)≤0.05,|log2FoldChange|≥1。其中FC(Fold Change)表示叶与茎间表达量的比值,其绝对值越大表明差异倍数越小,FDR值越大则为表达差异越不显著[24]。使用软件GOseq(版本:3.0)对样品间差异表达基因进行GO库注释的分类统计和功能富集分析,使用KOBAS(http://ko-bas.cbi.pku.edu.cn/home.do)进行KEGG pathway富集分析,使用Fisher精确检验法对差异表达基因KEGG通路富集的显著性进行评价。使用超几何检验来找到差异表达基因中显著富集的途径。并经过多次检测和校正后,定义P <0.05的通路途径为显著富集差异表达基因的途径。
1.6 SSR分析
以冬凌草转录组为研究对象,使用MISA(Microsatellite)软件进行SSR搜索。对Unigenes进行SSR检测,参数设置为“1~12、2~6、3~5、4~5、5~4、6~4”,1~12代表单碱基重复至少12次才算SSR,双碱基6次,三碱基5次,以此类推,重复单元最多有6个碱基。
2 结论与分析
2.1 总RNA质量检测和组装结果分析
经检测,样品总RNA的A260/A280均大于1.8,28 S/18 S在1.5~2.3之间,反应RNA完整性的RIN值在8.5~9.5之间为质量满足建立和基因功能注释、分类及样本间异同性分析。
通过组装,叶与茎冬凌草样品共获得3 7961个Unigene序列。All-Unigene总长为40366712 bp,平均长度为1063 bp,反应组装效果的N50值为1810。其中N50是评估组装质量最常用的指标之一,它反映了组装结果的连续性和完整性。所得Unigene的数目随长度的增加而减少,其Unigene大多集中在在200~3000 bp之间,平均Unigene为1063 bp。总的来说,Unigene的整体长度分布均匀(图1)。
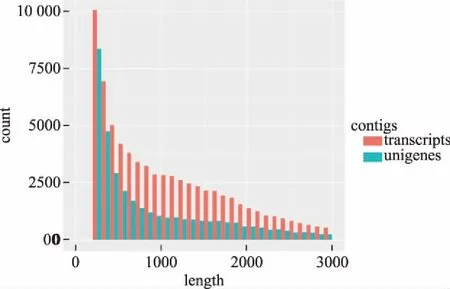
图1 All-Unigene长度分布统计
2.2 Unigene蛋白数据库比对结果
基于BLAST 算法对基因相似性分析,在37 961个Unigene 中,PFam库中17842Unigene 同源匹配得到信息,在SignalP数据库中有2394个,在eggNOG数据库中有8769个,在 GO 数据库中有18 776个,KEGG 数据库中有23 936个(表1)。通常情况下测序的难度与基因组、转录组复杂程度有关。此外,由于缺乏参考基因组信息,当前研究非模式生物的转录组仍具有一定挑战。即使目前国内对药用植物的从头转录组研究已有报道,但测序深度和基因检出数目仍有待完善和提高[25-26]。
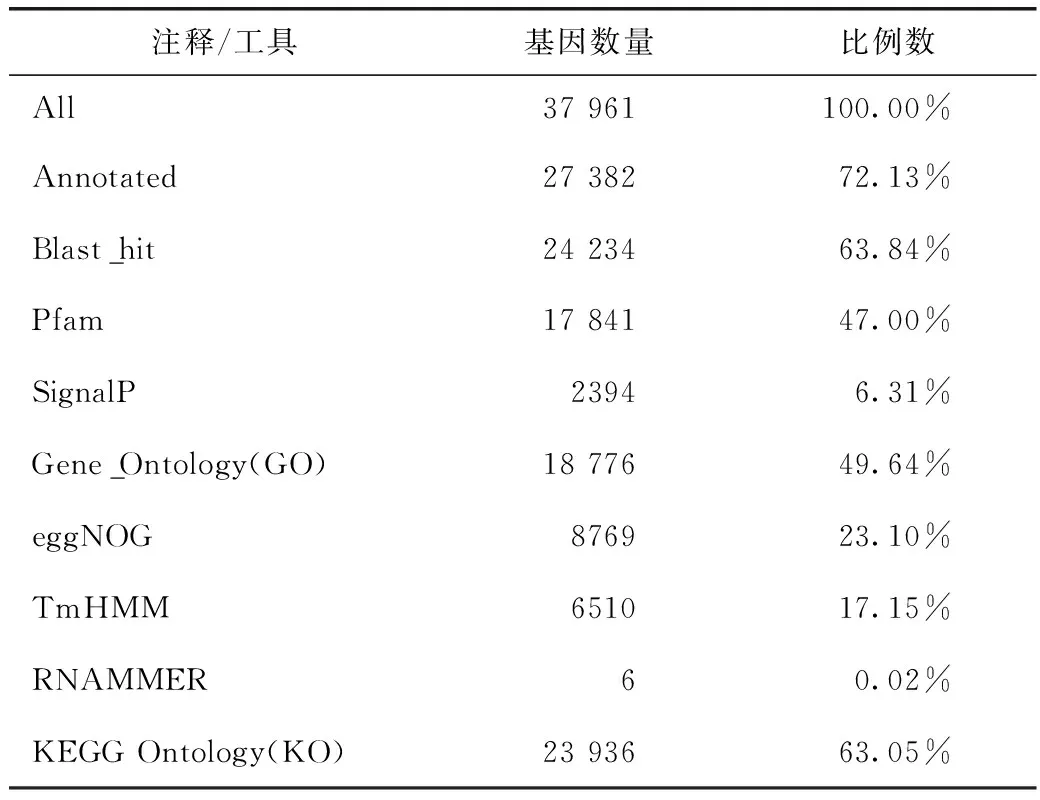
表1 All-Unigene蛋白数据库统计结果
2.3 GO注释和分类
GO注释和分类是转录组数据研究中的一种国际标准化的基因功能分类手段,它提供了一套动态更新的标准词汇表,用来全方面描述基因和基因产物的特性。GO功能分析由细胞组分、分子功能以及生物学过程三大部分组成。通过GO分析,37961Unigene中有18 775个获得了GO注释,被注释为“细胞组分”有15 639个;16 455个被注释为“分子功能”,1541515639被注释为“生物学过程”。我们对其中的level2的GO注释进行了统计(图2)。
2.4 KEGG 注释和分类
KEGG是全面分析生物体基因产物的代谢合成途径以及产物功能的参考数据库,根据生化途径,提供基因的功能注释和分类信息,对比分析表达基因在生物学上的系统行为。将6456个 Unigene 映射到 KEGG Pathway 数据库中,注释为代谢行为相关的序列共有4235个。其中涉及代谢(metabolism)、生物系统(organismal systems)环境信息处理(environmental information processing)、细胞进程(cellular processes)及遗传信息处理(genetic information processing)5个大类及102条代谢通路,包含 Unigene 最多的是Ribosome(核糖体)共有389个,占糖酵解/糖异生(glycolysis/gluconeogenesis)共有 226个,占6%;其次是Oxidative phosphorylation(氧化磷酸化)共有244个,占3.8%,这些转录水平上的数据变化充分说明了冬凌草药用部位在特定发育时期的新陈代谢变化(图3)。进一步研究还发现,在代谢途径中,与中药有效活性成分生物合成相关Unigenes 序列分别是苯丙素类合成(phenylpropanoid biosynthesis)101条、萜类化合物骨架合成(terpenoid backbone biosynthesis)60 条、各种萜类化合物合成(sesquiterpenoid,triterpenoid,monoterpenoid and diterpenoid biosynthesis)6条、(莨菪类、哌啶类、吡啶类)生物碱合成(tropane,piperidine andpyridine alkaloid biosynthesis)15条、黄酮类成分合成(flavonoid,flavone,flavonol and isoflavonoid biosynthesis)30条(图4)。

图2 Level-2的GO注释结果

图3 KEGG 注释和分类结果
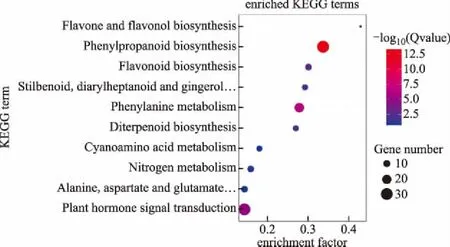
图4 KEGG富集分析图
2.5 二萜化合物合成途径相关基因
植物二萜化合物主要合成途径已经明确,分为三步:最先,活性异戊烯焦磷酸(IPP)及其异构体二甲基烯丙基焦磷酸(DMAPP)生成;之后为二萜化合物通用前体香叶基香叶基焦磷酸(GGPP)碳骨架的合成;最后在相关酶的作用下发生环化、羟基化等从而形成完整的二萜化合物分子[27]。利用本地化Blast的检索方式在冬凌草中确定了本数据中与二萜化合物生物合成相关的候选基因。发现二萜合成途径上的相关基因共有26个,其中与二萜骨架合成有关的基因有9个,其中与焦磷酸合成酶(copalyl diphosphate synthase)有关的 Unigene有6个,与贝壳杉烯合酶合成酶(KSL,kaurene synthase-like synthase)有关的unigene 有3个,具有细胞色素 P450(CYP450,cytochrome)功能的 Unigene共有5个。
2.6 差异表达基因的分析
通过冬凌草叶与茎两个不同组织样品的转录本/基因的表达量概率(FPKM scores)密度分布(图5),不同样品间差异基因在KEGG 通路富集具有特别显著性差异(P< 0.01)的通路描述,可以鉴定出一些在叶和茎两种组织在次生物质合成与代谢途径中的差异表达基因。对冬凌草叶与茎两种组织进行差异表达分析,得到差异表达基因4565个,其中在茎中表达较高的为1668个,在叶中表达较高的有2697个,与二萜类化合物合成相关的差异基因有15个。冬凌草叶与茎两种组织差异基因表达图如图6。

图5 冬凌草叶和茎基因表达量密度分布结果
2.7 SSR分析结果
简单重复序列又称微卫星序列(Simple Sequence Repeat,SSR)由于具有高度多态性,克服一般分子标记不确定、非特异的缺点,已在分子辅助育种、指纹数据库的构建、品种纯度鉴定及遗传多样性分析等方面得到广泛应用[28-29]。在本次所测得的冬凌草转录组中,共检测到8787条SSR序列,分布于7333条unigene中。SSR中二核苷酸重复的SSR标记有4016条,占比45.70%,是主要类型;其次是单核苷酸重复基序(2480条,占28.22%),接着是三核苷酸重复基序(1707条,占比19.43%)。

图6 冬凌草叶与茎差异表达基因的MA图和火山图
3 讨论
本文通过Illumina Hiseq4000 平台,利用RNA-seq 技术对道地产地冬凌草的叶与茎进行了转录组测序与功能分析,初步建立了它们的基因表达图谱,并通过比较药用部位和非药用部分合成途径差异酶基因的发掘初步阐述了不同组织冬凌草成分差异的分子机制。并分析了转录组中的SSR,揭示了冬凌草中遗传规律,也为药用冬凌草的遗传多样性研究和分子标记开发奠定了分子基础。
冬凌草叶和茎两个组织样品的高通量测序分析共获得3 7961个 Unigene序列。大量Unigene在多个数据库中不能匹配到已知基因,可能由于数据库中由于缺少冬凌草基因组信息;通过对转录组数据的差异表达分析发现与冬凌草合成途径有关的差异基因主要注释在苯丙素类化合物、萜类化合物骨架合成、生物碱类化合物以及黄酮类成分生物合成途径等通路中。冬凌草中的活性成分为二萜类物质[6],研究其生物合成途径下游关键基因尤为重要。基于本文转录组数据,陈[30]等人成功克隆并初步验证了下游IrCYP71功能基因,利用转录组数据挖掘药用植物的功能基因已经成为重要手段[31]。对药用植物的基因功能分化进行深入分析,并与活性成分种类、数量和积累变化相结合,进而从分子生药学角度揭示由于不同生长时间和生长环境基因表达的差异与药用部位中次生代谢成分差异的机制,才能为药材质量控制与提高提供理论依据[32]。高通量转录组测序技术的应用同时也加速了SSR 位点的挖掘,本研究中冬凌草的 All-Unigenes 转录本中 SSR 出现的频率特点为二核苷酸的重复单元类型最为丰富,这为后续冬凌草的种质资源和遗传多样性研究提供了丰富的分子标记数据。通过构建药用植物生长发育的不同时期、不同器官和不同产地的转录组文库,可以全方位地研究药用植物中活性成份生物合成的关键酶基因的表达水平,从而有效地揭示次生代谢产物生物合成的途径及其调控机制。
