藜麦β-香树酯醇合酶和鲨烯合酶基因的克隆与表达①
2019-01-03姜晓东李新凤郝艳平吕晋慧贾举庆张春来
姜晓东,李新凤,郝艳平,赵 靓,吕晋慧,贾举庆,张春来*
藜麦β-香树酯醇合酶和鲨烯合酶基因的克隆与表达①
姜晓东1,李新凤1,郝艳平2,赵 靓2,吕晋慧2,贾举庆1,张春来1*
(1 山西农业大学农学院,国家功能杂粮技术创新中心,山西省旱作栽培与作物生态重点实验室,山西省黄土高原特色作物高效生产协同创新中心,山西太谷 030801;2 山西农业大学林学院,山西太谷 030801)
为了探明藜麦皂苷生物合成的分子机制,试验以藜麦品种陇藜1号为材料,利用已有的转录数据,通过RT-PCR技术鉴定参与三萜皂苷生物合成关键酶基因,采用生物信息学分析方法进行了基因编码蛋白保守结构域、蛋白二级结构及系统发育树的构建,并利用半定量RT-PCR 方法进行了目的基因在籽粒不同发育时期和植株不同部位(根、茎、叶和籽粒)表达水平的研究。试验克隆得到三萜烯皂苷生物合成途径中关键酶编码基因、和的cDNA 全长序列。半定量RT-PCR 表达分析表明,和在叶片、籽粒中表达量最高,各基因在籽粒不同发育时期的表达量间无显著差异。本研究对藜麦三萜皂苷生物合成途径中关键酶基因表达模式的研究,将为进一步探明藜麦皂苷生物合成的分子机制及发掘影响藜麦皂苷含量的关键基因奠定基础。
藜麦;籽粒发育;关键酶基因;克隆;半定量RT-PCR
皂苷是由植物通过特定的代谢途径产生的具有生物活性、结构多样的次生代谢产物。其在制药、食品和化妆品等行业具有广泛应用。目前已基本明确了植物中三萜类皂苷物质生物合成途径的基本步骤。植物中皂苷物质主要是通过甲羟戊酸途径中产生的前体物质,用于三萜类皂苷化合物的生物合成。在这一过程中由两个异戊二烯焦磷酸(IPP)与一个烯丙基异构体——二甲基丙基焦磷酸(DMAPP)缩合生成焦磷酸烯丙基焦磷酸(FPP),之后两个FPP单元缩合生成三萜前体物质——角鲨烯。角鲨烯通过2,3-氧化鲨烯环化酶(OSC)的环氧化作用生成2,3-氧化鲨烯,这一环化产物成为植物中合成特定的三萜类化合物共同的前体物质。该前体物质通过特定合成酶的催化作用,产生不同的三萜化合物,如通过β-香树素合成酶对2,3氧鲨烯的环化作用,产生的β-香树酯醇是齐墩果烷型三萜皂苷的特定前体物质。之后,三萜前体通过一个或多个细胞色素P450依赖单加氧酶的氧化作用,形成具有三萜化合物结构的皂苷元,皂苷元经糖基转移酶的糖基化作用合成皂苷[1-5]。因此,三萜类皂苷物质的生物合成途径作为复杂的生物过程,生物合成途径的每一阶段均受到相应酶类的催化或修饰作用。
随着对甲羟戊酸途径中次生代谢产物生物合成途径研究的日趋深入,现已在多种植物中对代谢途径中关键酶基因进行了成功的克隆及分析。研究表明基因表达水平的变化,直接影响到次生代谢产物的生物合成,如Chen等[6]和Banyai等[7]通过对倍半萜内酯类化合物生物合成途径中基因作用机制的研究表明,法尼基焦磷酸合酶基因()的过表达,导致代谢产物青蒿素含量的显著增加。同样,植物三萜类皂苷生物合成分子调节机制研究的结果表明,皂苷生物合成途径中的相关酶基因的表达对三萜皂苷类物质的生物合成起着重要的调节作用[8-10]。藜麦中皂苷含量是其品质性状评价的重要指标,然而,鲜见有关藜麦皂苷生物合成途径中作用机制方面的研究报道。据此,本研究通过对藜麦三萜类皂苷生物合成途径中鲨烯合酶(squalene synthase,SS)、β-香树酯醇合成酶(β-amyrin synthase,bAS) 基因的克隆、组织特异性表达分析,旨在为探明藜麦中皂苷类物质生物合成的调节机制奠定一定的分子基础。
1 材料与方法
1.1 植物材料
试验以陇藜1号为试验材料,于2016 年6—10月分别选取花后15、20、25、30 d 5个时期的藜麦籽粒、叶片、根和茎组织,液氮处理后置于–80℃冰箱保藏备用。
1.2 RNA提取及cDNA一链合成(1.3 RNA 的提取和cDNA 的合成)
利用RNA提取试剂盒(TaKaRa公司)进行组织样品的总RNA提取,通过1% 琼脂糖凝胶电泳,并使用Nanodrop 分光光度计进行RNA质量和浓度检测。取1 μg总RNA为模板利用反转录试剂盒(TaKaRa公司),反转录合成一链cDNA。
1.3 cDNA 全长的获得
试验根据已有的转录组数据,以反转录cDNA一链为模板,进行藜麦鲨烯合酶基因(、)和藜麦β-香树酯醇合成酶基因()全长cDNA序列扩增。引物设计采用DNAMAN软件(version 6.0,Lynnon 公司)设计基因特异性引物,引物序列见表1。
反应体系为25 μl,包括1 µl 2.5 U/µl TransTaq® HiFi DNA Polymerase (TransGen Biotech),2.5 μl 10× TransTaq® HiFi BufferII,1.0 μl 2.5 mmol/L dNTPs,正向引物1 μl 10 mmol/L,反向引物1 μl 10 mmol/L,1.0 μl cDNA 模板和17.5 µl ddH2O。PCR 程序为:94℃预变性 5 min;35 个循环(94℃变性30 s;62℃()、53℃()、57℃()退火30 s;72℃延伸 1 min);72℃延伸10 min。PCR 扩增产物经胶回收纯化后,与pEASY-T1 克隆载体 (TransGen Biotech) 连接,连接产物转化到大肠杆菌DH5α中,随机挑选阳性克隆,经菌液PCR 验证后,送生工生物工程(上海) 有限公司测序。
1.4 半定量RT-PCR(Semi RT-PCR )分析
试验以陇藜1 号的根、茎、叶及花后15、20、25、30 d籽粒的cDNA为模板,以作为内参基因,利用半定量RT-PCR 对皂苷生物合成途径相关基因和的表达量进行分析,同时检测这3个基因在叶片、幼茎、根和籽粒中的表达水平。
采用Primer3软件(version4.0)进行基因的半定量表达引物设计,引物序列见(表1)。PCR 反应体系25 μl,其中引物各10 mmol,dNTP 各2 mmol,10×PCR 反应缓冲液2.5 μl,Taq 酶0.25 μl (5 U/μl),模版cDNA为50 ng。反应条件为:94℃预变性5 min;94℃变性30 s,60℃退火30 s,72℃延伸30 s,目的基因为28 个循环,为30 个循环;72℃延伸5 min。PCR产物经2% 琼脂糖凝胶电泳检测基因的表达量。

表1 基因克隆和半定量PCR引物设计
1.5 生物信息分析
利用DNAMAN软件进行基因开放阅读框(ORF)氨基酸序列推导;利用TRMHMM server v2.0 (http:// www.cbs.dtu.dk/services/TMHMM-2.0/)在线软件进行蛋白跨膜结构域预测;采用在线软件ProtParam (http://www.expasy.org)进行蛋白的分子量、理论等电点及保守结构域的预测;使用ExPASy 网站提供的在线分析软件SOPMA 预测蛋白质二级结构,利用Phyre2在线软件进行蛋白三级结构预测;采用Clustal W 软件进行氨基酸多序列比对;利用MEGA6.0 软件中的最大似然法(maximum likelihood,ML)构建系统发育树。
2 结果与分析
2.1 CqSS1和CqSS2基因克隆及序列分析
以花后25 d籽粒反转录cDNA为模板,扩增得到序列长度均为 1 242 bp 的两个鲨烯合酶基因,定名为和,其编码413个氨基酸。进一步对和基因编码蛋白的一级结构预测分析发现,CqSS1和CqSS2蛋白的分子量分别为 47.8、47.4 kDa,等电点分别为6.32、6.50。利用基于隐码模型的TMHMM 工具进行跨膜区分析表明,CqSS1 蛋白C端和C端邻近区有2个跨膜区,而CqSS2蛋白只有1个位于C端的跨膜区(图1)。
SOPMA 预测的CqSS 蛋白二级结构结果表明,CqSS1 和CqSS2编码的413个氨基酸残基中,主要为螺旋,分别占70.70% 和70.94%,β转角分别占3.87% 和3.63%,无规则卷曲分别占 21.79% 和21.07%,延伸链分别占3.63% 和4.36%,-螺旋和无规卷曲为该蛋白二级结构主要元件(图2A),这与Phyre2软件三级结构预测结果相符(图2B)。

(A: CqSS1的跨膜预测;B: CqSS2的跨膜预测)
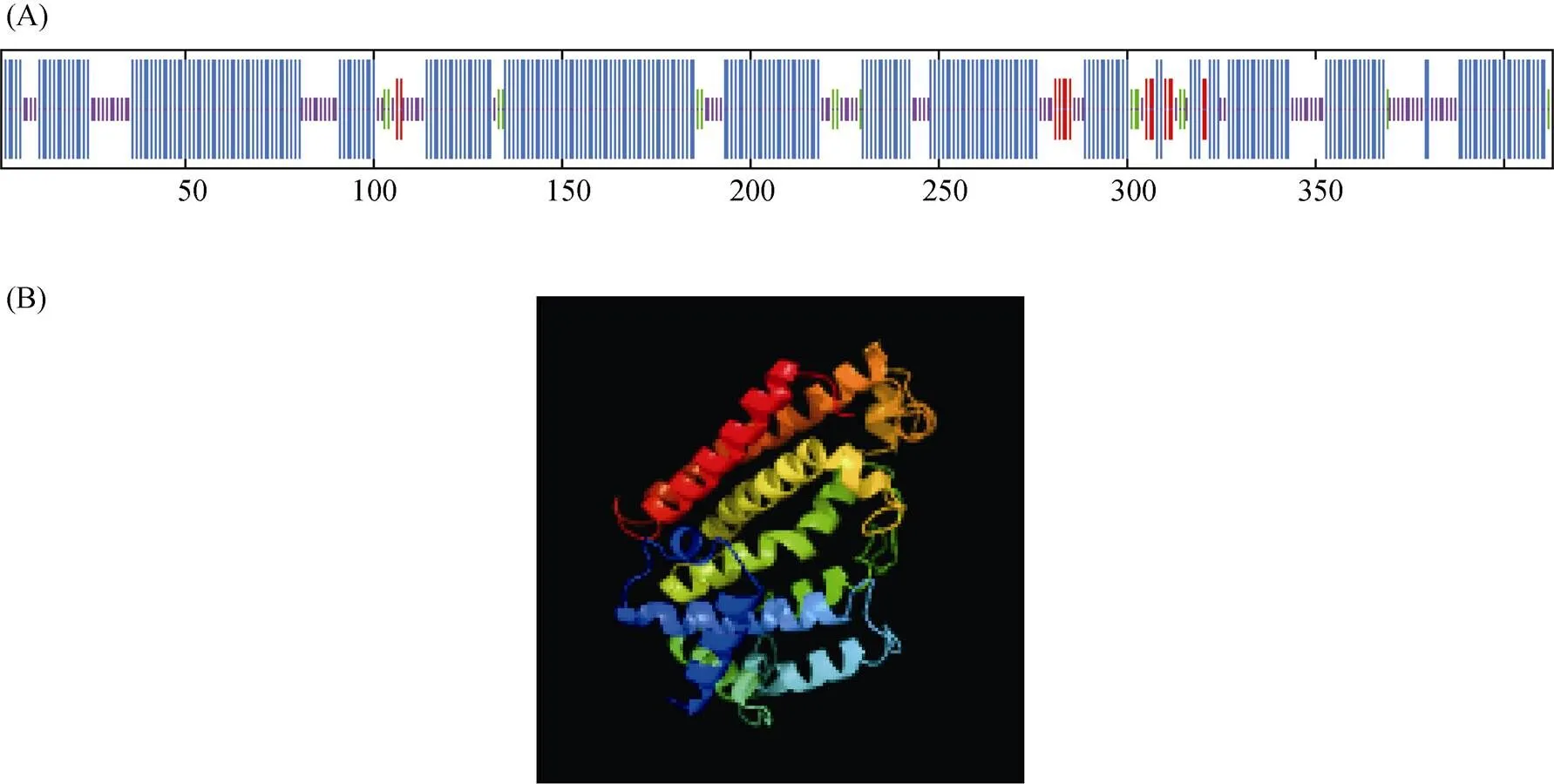
(A:CqSS1蛋白的二级结构,蓝色区域为α-螺旋,橙色区域为不规则卷曲,红色区域为延展延伸链;B:CqSS1蛋白的三级结构,蛋白红色部分为N端 α-螺旋,蓝色部分为C端α-螺旋)
利用MEGA6.0 软件中的最大似然法构建系统发育树(图3)。结果表明,藜麦中两个CqSS蛋白聚在同一分枝,但与玉米()、松属(a)、拟南芥()、青蒿素()、杨属()等物种进化关系较近,而与龙葵属()和茄属()亲缘关系相对较远。
2.2 CqbAS基因的全长cDNA克隆及序列分析
的cDNA全长 2 292 bp,编码763个氨基酸,蛋白的一级结构预测分析结果表明,蛋白预测理论分子量为87.9 kDa,等电点为5.73;SOPMA 预测的CqbAS 蛋白二级结构结果表明,CqbAS 蛋白主要由螺旋(44.17%)、无规则卷曲(35.39%)、延伸链(13.24%)和β-转角(7.21%)组成,可推测-螺旋和无规卷曲为CqbAS 蛋白二级结构主要的组成元件,该结果与蛋白三级结构预测结果相符(图4)。
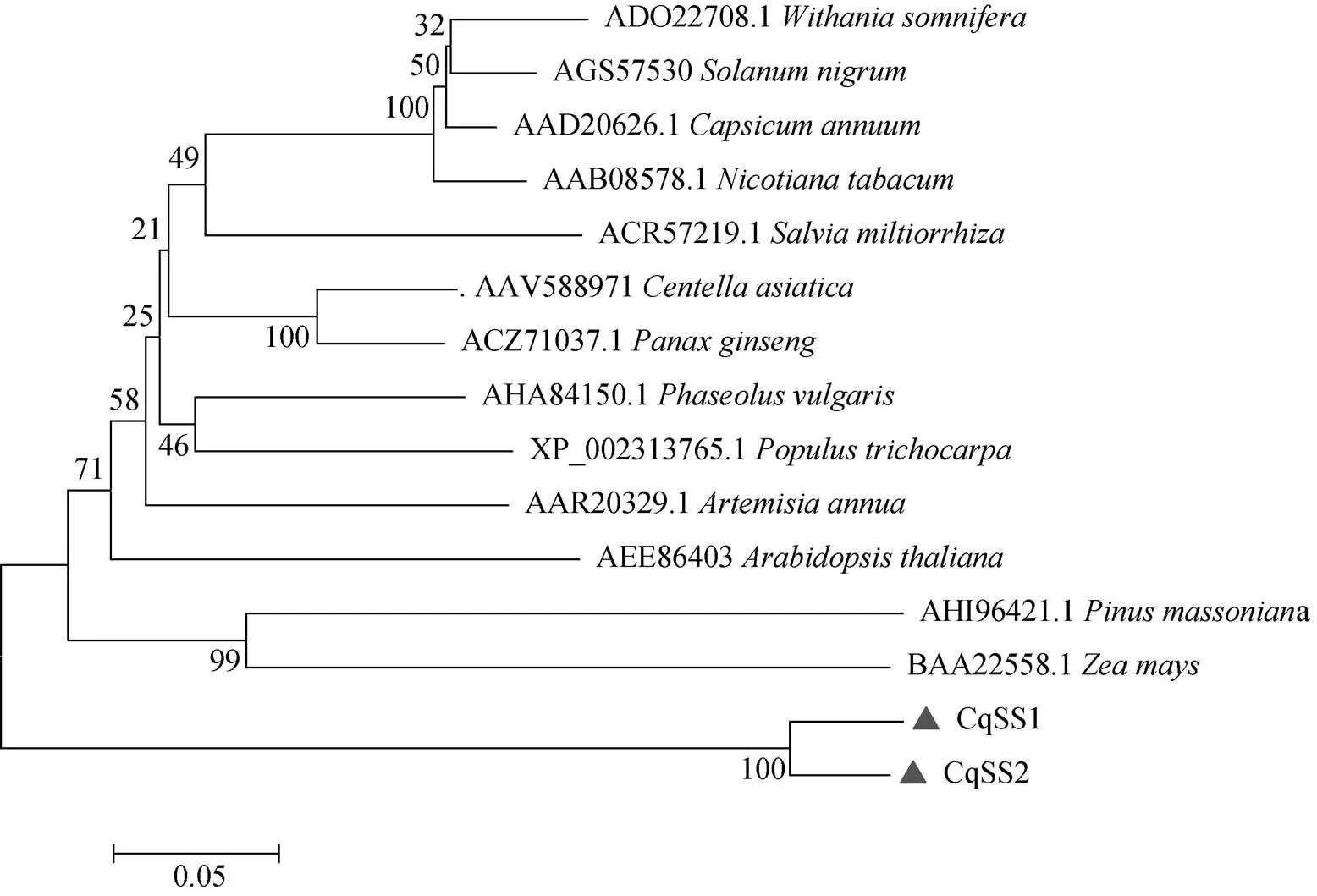
图3 藜麦CqSS 蛋白与植物中鲨烯合酶家族成员的进化关系分析
系统发育树(图5)分析结果表明,CqbAS蛋白与同为石竹科的药用植物麦蓝菜()亲缘关系最近,聚在同一分支,同时与其他植物如豌豆()、甘草()及大豆()的亲缘关系较近,被分在同一亚族,与灌木型植物紫菀属(Linn.)、杜茎山()属亲缘关系最远。
2.3 CqSS和CqbAS基因表达分析
2.3.1和基因组织特异性表达 试验结果表明(图6),基因在藜麦各组织中均有表达,但基因在不同组织中表达量存在明显差异,该基因在藜麦籽粒和叶片中的表达量相对较高,而在根和茎组织中的表达水平极低;基因在各组织中均有表达,但表达水平相对较低;与基因的表达趋势一致,在籽粒和叶片中高表达,而在根和茎组织呈现较低表达水平。

(A:CqSS1蛋白的二级结构,蓝色区域为α-螺旋,橙色区域为不规则卷曲,红色区域为延展延伸链;B:CqSS1蛋白的三级结构,蛋白红色部分为N端α-螺旋,蓝色部分为C端α-螺旋)

图5 藜麦CqbAS蛋白与植物β-香树酯醇合成酶家族成员的进化关系分析
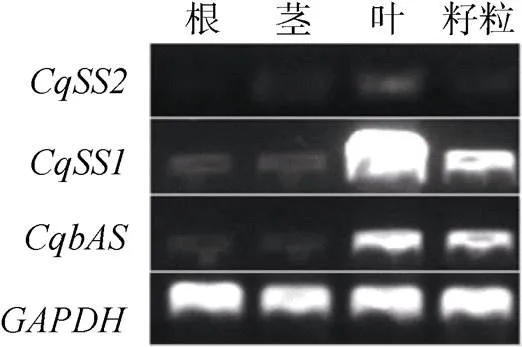
图6 CqSS和CqbAS的组织特异性表达
2.3.2和在籽粒发育过程中的表达 通过对花后15、20、25、30 d藜麦籽粒发育过程中和基因表达模式测定分析,结果表明两基因在花后不同时期的基因表达量无显著差异(图7)。

图7 藜麦籽粒发育时期种子中CqSS1和CqbAS表达水平的半定量RT-PCR
3 讨论
依赖于甲羟戊酸途径的三萜皂苷的生物合成途径,鲨烯合酶(SS)和β-香树酯醇合成酶(bAS)是催化齐墩果烷型三萜皂苷生物合成过程的两个关键酶。本研究对鲨烯合酶基因克隆,分别得到两个鲨烯合酶编码基因、,利用TMHMM 跨膜工具进行分析,结果显示CqSS1含有两个跨膜区,而CqSS2只有一个位于C-端的跨膜结构域。研究表明,拟南芥、烟草、酵母和人类的鲨烯合酶中只在蛋白C末端的疏水区域含有一个跨膜区[11-12]。而对于富含皂苷的物种,鲨烯合酶蛋白除在C末端含有一个跨膜区外,在靠近C-末端的邻近区域还含有额外的跨膜螺旋,如大豆、三七、西洋参以及甘草等富含皂苷物种,鲨烯合酶蛋白C端均含有两个跨膜螺旋[13-15]。研究认为额外的跨膜螺旋可能有利于促进鲨烯合酶在内质网膜(ER)的定位[16-18]。鲨烯合酶是甲羟戊酸途径中的第一个关键限制因子,鲨烯合酶在植物甾醇和三萜类皂苷物质合成具有重要的调节作用。本研究通过CqSS1蛋白氨基酸序列与其他物种参与皂苷生物合成的鲨烯合酶的序列比对结果表明,CqSS1含有植物中鲨烯合酶的特性序列,且根据基因的表达特性分析结果,进一步表明CqSS1参与藜麦三萜类皂苷的生物合成。甲羟戊酸合成途径中鲨烯合酶负责将法尼酰基二磷酸缩合生成由30个碳原子组成的三萜前体物质——角鲨烯,完成三萜类化合物生物合成途径中的前体物质的生物合成[10]。
在籽粒发育时期的表达特性表明,基因在籽粒发育各时期的表达水平间无显著差异,该结果表明,香叶基二磷酸合酶、法尼基二磷酸合酶、鲨烯合酶及鲨烯单加氧酶作为三萜皂苷生物合成途径中上游调控的关键酶,其在代谢途径中合成的各种次生代谢产物是植物维持生命活动所必需的,这些酶类编码基因的表达水平的稳定性表达,对植物维持正常的生命活动起着重要的调节作用。因此,基于基因表达特性的测定结果,推测藜麦中的皂苷物质生物合成途径下游关键酶的编码基因可能在皂苷的生物合成中起着重要的调节作用。
据此,本试验进一步对皂苷生物合成途径中的β-香树酯醇合成酶(bAS)基因的表达模式进行研究。β-香树酯醇合成酶作为三萜类皂苷生物合成的第一个限速酶[19-22],该酶在皂苷生物合成途径中负责催化三萜苷元骨架——β-香树酯醇的生物合成,因此,对皂苷物质的生物合成具有重要的调节作用。本研究对藜麦中β-香树酯醇合成酶基因的克隆和基因的组织特异性表达分析,结果显示该基因编码编码763个氨基酸。CqbAS蛋白序列分析和进化分析表明,CqbAS与其他植物中三萜皂苷的生物合成途径相关的β-香树酯醇合成酶蛋白的氨基酸序列具有高度的同源性。基因的组织特异性表达结果显示,该基因在籽粒和叶片中高表达,推测藜麦中皂苷物质的合成主要在籽粒和叶片中累积[23-26],但在籽粒发育过程中,基因的表达无显著性差异,可以推测影响藜麦皂苷生物合成的基因可能发生在三萜苷元骨架的生物合成过程之后。
三萜类皂苷化合物是来源于植物的一类重要的次生代谢产物,藜麦中的皂苷含量与藜麦加工品质关系密切。而作物品质性状不但取决于遗传因素,即品种基因型的影响,同时栽培方式同样对作物品质性状产生较大影响。众多研究表明,种植方式对作物的生长、病虫的发生情况,特别是对作物品质性状有重要的影响,邱立友等[27]对连作产生的障碍机理研究表明,包括萜类生物碱和苯丙烷类的次生代谢物所产生的新生植物自毒作用,是导致连作障碍发生的关键因素之一。因此,改变种植方式能够在较大程度上克服由于连作导致的障碍[28]。徐雪凤等[29]通过油葵与马铃薯的轮作研究,发现轮作可以通过提高土壤有机质、有效磷和碱解氮含量,而提高土壤酶活性、细菌数量,致使土壤环境得到明显改善,从而减轻病害的发生和自毒作用。这些研究结果为指导藜麦生产,以豆科作物作为轮作作物应用到藜麦种植中,不但克服了由于连作而产生的种植障碍,而且减轻了当今藜麦生产中病虫害发生严重的问题,在提高藜麦品质及产量方面将起到了重要作用。
本研究通过藜麦鲨烯合酶基因、β-香树酯醇合成酶基因的克隆和组织特异性表达分析,为进一步通过筛选三萜皂苷生物合成途径中下游酶编码的候选基因研究,及发掘影响藜麦皂苷生物合成的关键基因奠定了基础。
4 结论
本研究克隆得到藜麦皂苷生物合成途径中的两个编码鲨烯合酶基因(和)和β-香树酯醇合成酶基因(),和的cDNA长度为1 242 bp,编码413个氨基酸,CqSS1的 C端含有2个跨膜结构域,且CqSS1 具有植物鲨烯合酶的特征序列,而CqSS2的 C端含有1个跨膜结构域。的cDNA长度为2 292 bp,编码763个氨基酸。和基因在籽粒和叶片中高表达,而基因的表达量极低,基因和在种子不同发育时期的表达无显著差异,推测和基因参与了藜麦皂苷物质的生物合成,且推测三萜类皂苷生物合成途径中的下游基因对皂苷的生物合成具有重要的调节作用。
[1] Haralampidis K, Trojanowska M, Osbourn A E, et al. Biosynthesis of triterpenoid saponins in plants[M]. History and Trends in Bioprocessing and Biotransformation. Berlin Heidelberg: Springer, 2002 , 75: 31–49
[2] Zhao C L, Cui X M, Chen Y P, et al. Key enzymes of triterpenoid saponin biosynthesis and the induction of their activities and gene expressions in plants[J]. Natural Product Communincations, 2010 5(7): 1147–1158
[3] Suzuki H, Achnine L, Xu R, et al. A genomics approach to the early stages of triterpene saponin biosynthesis in[M]. The Plant Journal, 2002, 32(6): 1033–1048
[4] Seki H, Tamura K, Muranaka T. P450s and UGTs: Key players in the structural diversity of triterpenoid sapo activities and gene expressions in plants[J]. Plant & Cell Physiology, 2015, 56(8):1463–1471
[5] Kim Y S, Cho J H, Park S, et al. Gene regulation patterns in triterpene biosynthetic pathway driven by overexpression of squalene synthase and methyl jasmonate elicitation in[J]. Planta, 2011, 233(2): 343–355
[6] Chen Y F, Shen Q, Wang Y Y, et al. The stacked over-expression of FPS, CYP71AV1 and CPR genes leads to the increase of artemisinin level inL.[J]. Plant Biotechnology Reports, 2013, 7(3): 287–295
[7] Banyai W, Kirdmanee C, Mii M, et al. Overexpression of farnesyl pyrophosphate synthase (FPS) gene affected artemisinin content and growth ofL.[J]. Plant Cell Tissue & Organ Culture, 2010, 103(2): 255–265
[8] Lee M H, Jeong J H, Seo J W, et al. Enhanced triterpene and phytosterol biosynthesis inoverexpressing squalene synthase gene[J]. Plant & Cell Physiology, 2004, 45(8): 976–984
[9] Jiang D, Rong Q, Chen Y, et al. Molecular cloning and functional analysis of squalene synthase (SS) in[J]. International Journal of Biological Macromolecules, 2017, 95: 658
[10] Zhang F, Li X, Li Z, et al. UPLC/Q-TOF MS-based metabolomics and qRT-PCR in enzyme gene screening with key role in triterpenoid saponin biosynthesis of[J]. Plos One, 2014, 9(8): e105765
[11] Nguyen H T, Neelakadan A K, Quach T N, et al. Molecular characterization ofsqualene synthase genes in seed phytosterol biosynthesis[J]. Plant Physiology and Biochemistry, 2013, 73(41): 23–32
[12] Busquets A, Keim V, Closa M et al. Arabidopsis thaliana contains a single gene encoding sequalene systhase[J]. Plant molecular biology, 2008, 67(1-2): 25–36
[13] Filiz E, Ozyigit I I, Vatansever R. Comparative analyses of squalene synthase (SQS) proteins in poplar and pine by using bioinformatics tools[J]. Tree Genetics & Genomes, 2016, 12(2): 32
[14] Sanchita, Singh G, Sharma A. Erratum to: In silico, study of binding motifs in squalene synthase enzyme of secondary metabolic pathway of solanaceae[J]. Molecular Biology Reports, 2014, 41(12): 8333
[15] Inoue J, Sato R, Maeda M. Multiple DNA elements for sterol regulatory element-binding protein and NF-Y are responsible for sterol-regulated transcription of the genes for human 3-hydroxy-3-methylglutaryl coenzyme a synthase and squalene synthase[J]. Journal of Biochemistry, 1998, 123(6): 1191–1198
[16] Tansey T R, Shechter I. Structure and regulation of mammalian squalene synthase[J]. Biochimica et Biophysica Acta, 2000, 1529(1/2/3): 49
[17] Gold L M, Post D. Multiple sequence elements are involved in the transcriptional regulation of the human squalene synthase gene[J]. Journal of Biological Chemistry, 1997, 272(15): 10295–10302
[18] Tansey T R, Shechter I. Squalene synthase: structure and regulation[J]. Progress in Nucleic Acid Research & Molecular Biology. 2000, 65(65): 157–195
[19] Zhao C, Xu T, Liang Y, et al. Functional analysis of β-amyrin synthase gene in ginsenoside biosynthesis by RNA interference[J]. Plant Cell Reports, 2015, 34(8): 1307–1315
[20] Han J Y, Kim M J, Ban Y W, et al. The Involvement of β-Amyrin 28-Oxidase (CYP716A52v2) in oleanane-type ginsenoside biosynthesis in[J]. Plant & Cell Physiology. 2013, 54(12): 2034–2046
[21] Jo H J, Han J Y, Hwang H S, et al. β-Amyrin synthase (EsBAS) and β-amyrin 28-oxidase (CYP716A244) in oleanane-type triterpene saponin biosynthesis in[J]. Phytochemistry, 2017, 135: 53–63
[22] Han J Y, Kim H J, Kwon Y S, et al. The Cyt P450 enzyme CYP716A47 catalyzes the formation of protopanaxadiol from dammarenediol-II during ginsenoside biosynthesis in[J]. Plant & Cell Physiology, 2011, 52(12): 2062
[23] 龙月红, 李非非, 杨果, 等. 刺五加β-香树酯醇合成酶基因的克隆及其表达与皂苷量的相关性[J]. 中草药, 2015, 46(9): 1354–1359
[24] Kim T D, Han J Y, Huh G H, et al. Expression and functional characterization of three squale-ne synthase genesassociated with saponin biosynthesis in[J]. Plant Cell Physiology, 2011, 52(1): 125–137
[25] Fiallos-Jurado J, Pollier J, Moses T, et al. Saponin determination, expression analysis and functional characterization of saponin biosynthetic genes inleaves[J]. Plant Science, 2016, 250: 188–197
[26] Zhao H, Tang Q, Mo C, et al. Cloning and characterization of squalene synthase and cycloartenol synthase from[J]. Acta Pharmaceutica Sinica B, 2017, 7(2): 215–222
[27] 邱立友, 戚元成, 王明道, 等. 植物次生代谢物的自毒作用及其与连作障碍的关系[J]. 土壤, 2010, 42 (1): 1–7
[28] 康亚龙, 景峰, 孙文庆, 等. 加工番茄连作对土壤理化性质及微生物数量的影响[J]. 土壤学报, 2016, 53(2): 533–542
[29] 徐雪风, 李朝周, 张俊莲. 轮作油葵对马铃薯生长发育及抗性生理指标的影响[J]. 土壤, 2017, 49(1): 83–89
Gene Cloning and Express of Squalene Synthase and β-amyrin Synthase from
JIANG Xiaodong1, LI Xinfeng1, HAO Yanping2, ZHAO Jing2, LV Jinhui2, JIA Juqing1, ZHANG Chunlai1*
(1 College of Agronomy, Shanxi Agricultural University; National Innovation Centre for Functional Speciality Crops; Shanxi Key Laboratory for Arid Agriculture and Crop Ecology; Shanxi Collaboration and Innovation Centre for Efficient Production of Speciality Crops in Loess Plateau, Taigu, Shanxi 030801, China; 2 College of Forestry, Shanxi Agricultural University, Taigu, Shanxi 030801, China)
To explore the molecular mechanism of saponin biosynthesis from, varietycv. Longli 1 was used as materials, and experiment was carried out by using RT-PCR technology to identify the key enzyme which involved in triterpenes saponin biosynthesis. Then the conserved domain, secondary structure of protein and phylogenetic tree were analyzed by using bioinformatics methods, and organ-specific expression patterns of genes in seed, root stem and leaf were detected by semi-quantitative RT-PCR. The expression pattern of target gene were also investigated during seed developing period. Full-length cDNAs of,andwere cloned, and gene expression results showed that bothandwere highly expressed in leaf and seed than in other tissues, and express pattern remained in same level in different seed developing stage. This study is the first comprehensive analysis of the expression patterns of pivotal genes for triterpene saponin biosynthesis in, and provides a basis to further elucidate the molecular mechanism for the biosynthesis of saponin.
; Grain develepoment; Key enzyme gene; Cloning; Semi-quantitative RT-PCR
山西农谷建设科研专项项目(SXNGJSKYZX201704)和山西省重点研发计划项目(201703D221006-3)资助。
(3108827452@qq.com)
姜晓东(1965—),男,山东曹县人,博士,副教授,研究方向为杂粮分子育种。E-mail:caas08f2e1a@aliyun.com
10.13758/j.cnki.tr.2018.06.025
S-9
A
