Illumina MiSeq高通量测序分析不同品种桑树内生细菌多样性*
2018-12-18吴燕燕徐伟芳王玲莉刘祎杨
吴燕燕 徐伟芳 罗 琴 王玲莉 刘祎杨 陈 凯 谢 洁
(西南大学生物技术学院,重庆 400715)
桑树(MorusL.)是一种兼具经济与生态效益的树种,在中国蚕业发展和环境治理等方面均具重要作用。桑椹(Mulberry fruit)是桑树的果实,因其口感独特、滋味颇佳、营养丰富等特点而深受大众喜爱。桑椹中含有大量黄酮和多酚类活性物质,具有提高免疫力、抗氧化和抗衰老等功效[1-4],近年来果桑产业呈现出良好的发展势头。然而,随着果桑种植面积的不断扩大,桑椹菌核病对果桑产业的危害日趋严重,给果桑生产带来极大的经济损失。桑椹菌核病,又称白果病,是一种土传真菌性病害,可由桑实杯盘菌(Ciboriashiraiana)[5]、肉阜状杯盘菌(Ciboriacarunculoides)[6]、桑椹核地杖菌(Scleromitrulashiraiana)[7]以及核盘菌(Sclerotiniasclerotiorum)[8]等多种病原真菌引起。有关桑树抗病性的调查结果中显示,不同桑树品种对菌核病抗性差异较大。江西省蚕桑茶叶研究所果桑园内5个桑树品种中,台果46C019发病程度最轻,抗病性最强[9];黄传书等通过对重庆市蚕业科学技术研究院桑园内16个果桑品种的桑椹菌核病抗性能力的调查发现,川桑7637抗性最强,红果2号抗性最弱[10];魏晓军选育出的抗性品种台湾46C019和苏椹72号对防治桑椹菌核病的效果颇佳[11]。抗性育种是目前防治菌核病的有效方法之一,但该防治策略仍存在“选育时间长、人力投入大及部分抗性品种果质与经济性状欠佳”等一系列弊端。化学防治与农艺防治亦是生产上常用的防控手段,但农药滥用易威胁食品与环境安全,土地翻耕等农艺防控效果不佳[12]。因此,寻找绿色环保、安全有效的防治方法已迫在眉睫。
植物内生菌是指其生活史中的某一段时期生活在健康植物各组织器官的细胞间隙或者细胞内部,不引起宿主植物发生明显病害的一类微生物[13-14]。内生细菌作为宿主植物的重要组成部分,对调节宿主体内的微生态平衡以及促进宿主植物的健康生长均发挥重要作用。近些年来利用内生细菌防控植物病害的研究报道较多,但在实际生产中能够广泛应用的生防菌株却十分有限[15-16]。为寻找抑菌效果显著且稳定的拮抗菌株,了解宿主植物内生菌群落结构组成及多样性特征十分必要。目前关于内生菌多样性的研究方法主要有基于传统手段的组织分离培养法,以及基于宏基因组测序技术的非培养法,且至今所鉴定的内生菌大多数依靠培养法分离获得[17]。自然环境中存在大量的微生物,其数量高达106种,迄今依靠传统纯培养方法能够分离培养的微生物仅占总数的1.0%,而原核生物仅为0.1%[18],利用传统培养法研究植物内生菌的多样性存在明显局限。大量未培养微生物(即难以在实验室条件下生长的微生物)的存在严重制约了传统培养方法对植物内生菌资源的全面认识。因此,避开传统培养方法带来的弊端,依靠分子生物学手段的非培养法应运而生。有报道显示,植物内生菌的分布特征与其定殖宿主植物的种类或品种紧密相关[19-25]。论文研究基于不同桑树品种对菌核病菌抗性差异显著的特点,利用基于Illumina MiSeq的第二代高通量测序技术,探索不同品种桑树内生菌的种群分布特征及多样性,并探究内生菌的分布与宿主植物抗病性之间是否存在相关性,为利用内生菌改变宿主特性,开展抗病育种提供研究基础。
1 材料与方法
1.1 材料
1.1.1 供试桑树材料
红果2号、川桑7637、新伦教三种果桑品种样品于2015年4月上旬采自重庆市蚕业科学研究院桑园(29°50′39″N, 106°25′55″E),长果桑于同一时间段采自重庆市西南大学生物技术学院桑园“桑之源”(29°49′1″N, 106°24′57″E)。每个桑树品种采集3~5个长势一致的两年生健康枝条,4℃保存备用。
1.1.2 主要试剂和仪器
DNA marker、Taq酶、dNTP、10×PCR Buffer、Mg2+购自宝生物工程(大连)有限公司;QIAquick凝胶回收试剂盒购于QIAGEN公司;其余常规化学试剂均为国产分析纯;PCR仪(Bio-Rad,美国),Applied Biosystems®Gene Amp®PCR System 9700(Bio-Rad,美国)等。
1.2 方法
1.2.1 桑树表面消毒
按照文献[26-28]的方法进行桑树的表面消毒,具体操作方法为:将桑枝剪成5cm茎段,流水冲洗晾干后,完全浸润于75.0%乙醇中,取出后于酒精灯上燃尽茎段表面的乙醇,将其置于PDA培养基上滚动一周,使茎段表面均接触培养基,将该PDA平板置于22℃培养48h,确认表面消毒彻底。
1.2.2 内生菌富集与宏基因组DNA提取
内生菌富集[29-30]:将20g消毒的桑树树皮剪碎,加入160mL无菌水,置于组织捣碎机捣碎后,纱布过滤离心 (200×g,5min,4℃),取上清130mL,依次加入NaCl和SDS分别至终浓度为0.9%,0.063%,轻轻混匀后4℃静置1h,取上清离心(5000×g,10min,4℃),然后将所获沉淀重悬于160mL无菌水,再按上述浓度加SDS和NaCl,静置30min,4℃离心10min(5000×g),收集的沉淀即为富集的菌体,将其按30mg/ mL的比例加入TE缓冲液悬浮备用。
以富集的菌体为材料,参考文献[28]中的改良CTAB法提取宏基因组DNA,并以此为模板,利用引物799F (5’-AACAGGATTAGATACCCTG-3’)和1492R(5’-GGTTACCTTGTTACGACTT-3’)进行PCR扩增。PCR反应条件为:95℃预变性5min;94℃变性30s,50℃退火45s,72℃延伸1min,30个循环;72℃延伸10min。扩增产物用1%琼脂糖凝胶检测[31-32],将获得单一且明亮条带的扩增16S rRNA基因产物对应的总DNA样品视为合格样品,并将浓度≥50ng/uL的DNA样品送至成都罗宁生物科技有限公司进行上机测序与后续生物信息分析。
1.2.3 PCR扩增、Miseq文库构建及测序
以1.2.2确定的合格基因组DNA为模板,利用16S rDNA V3-V4区特异引物338F/806R,使用高效和高保真酶(TOYOBO KOD-Plus-Neo DNA Polymerase)进行PCR扩增。用2%琼脂糖凝胶电泳检测、QIAquick凝胶回收试剂盒(QIAGEN公司)回收扩增产物,并使用GE Nano Vue系统(GE Healthcare)进行荧光定量检测。利用Illumina MiSeq平台(Illumina,SanDiego,USA)标准操作规程将纯化后的扩增片段构建双末端读长(PE,Paired-end Reads)2×300的文库并测序。
1.2.4 生物信息分析流程
Miseq测序得到的双端序列数据,首先根据PE之间的重叠关系,利用FLASH软件拼接[33]、QIIME软件过滤[34]、UCHIME去除嵌合体后[35],得到有效优化序列。然后利用UPARSE算法对优化序列在97%水平上进行OTU聚类分析[36],挑选出OTU代表性序列,并利用Greengene和Silva等数据库进行物种注释[37-38],去除注释为叶绿体、线粒体及非细菌界的OTUs。最后利用Mother软件做稀释曲线分析[39],通过对OTU进行丰度和α-多样性分析,得到微生物细菌群落结构组成及多样性特征。
图中C、Q、X、H分别代表长果桑、川桑7637、新伦教、红果2号,M代表DNA marker 2000图1 富集菌体DNA的质量检测
2 结果与分析
2.1 内生细菌宏基因组DNA的提取
提取的桑树树皮组织总DNA片段较为完整,基因组片段大于15kb。通过细菌16S rDNA V6~V10区特异性PCR扩增,均获得大小为750bp左右的目的片段(图1)。经表面消毒的桑枝滚动的培养基在培养后,于22℃温度下培养48h,无菌落生长,说明实验中得到的结果为合格的桑树内生菌的基因组。
2.2 序列长度分布
从4个桑树品种中共测得128715条PE,经拼接和过滤处理后,获得92282条优化序列,除新伦教外,其余3个品种得到的优化序列均超过2万条(表1),且所有桑树品种的优化序列长度集中分布在420~429bp范围内(图2),约占总序列的90.02%。

表1 样品序列长度统计表

图2桑树内生细菌扩增序列长度分布情况
2.3 OTU数目统计
从4个果桑样品中共获得5858个细菌OTUs,不同品种桑树OTU数量存在差异(图3):抗性品种川桑7637的内生细菌OTU数最高,可达2228个,长果桑次之,含1907个OTUs,易感品种红果2号(1739)和新伦教(1713)则整体相对较低。4个品种共有内生细菌OTUs数为239个,远低于各品种果桑特有内生细菌OTUs数(川桑7637,1513个;长果桑,1209个;红果2号,1060个;新伦教,1034个)。

图3 (A)不同品种桑树内生菌OTU分布Venn图,(B)不同品种桑树内生细菌OTU数目统计;图中C、Q、X、H分别代表长果桑、川桑7637、新伦教、红果2号
2.4 样品多样性分析
基于OTU的稀释曲线结果显示,随着测序数量的增加,稀释曲线斜率逐渐降低,趋于平坦,且所有样品均已进入平台期,说明测序数量已经足够大,可以基本反映样品中绝大多数微生物信息(图4)。另外,基于Shannon和Chao1多样性指数曲线结果显示4个样本的多样性指数差异较大,川桑7637>长果桑>新伦教>红果2号,即桑椹菌核病抗性品种川桑7637内生细菌的种群丰富度最大,多样性最高,其次是长果桑,而易感品种新伦教和红果2号种群丰富度和多样性相对较低。

图4 不同桑树品种的稀释曲线和多样性指数曲线;C、X、Q、H分别代表长果桑、新伦教、川桑7637、红果2号
2.5 群落结构组成分析
在门的分类水平看(图5A),4个桑树样品中共检测出细菌21个门(相对丰度≥0.1%),不同品种桑树所测到的细菌种类相似,但在各品种中所占比例不同。整体而言,数量最多的5个门为变形菌门(Proteobacteria,36.10%)、厚壁菌门(Firmicutes,14.51%)、酸杆菌门(Acidobacteria,13.38%)、拟杆菌门(Bacteroidetes,11.85%)以及放线菌门(Actinobacteria,6.35%),尤以变形菌门的优势最为明显,其在长果桑、川桑7637、新伦教、红果2号各品种中所占比例分别为74.17%、68.87%、78.90%和79.97%。尽管浮霉状菌门(Planctomycetes,3.86%)、绿弯菌门(Chloroflexi,3.72%)、芽单胞菌门(Gemmatimonadetes,1.21%)和疣微菌门(Verrucomicrobia,1.18%)等常见的门类细菌含量较少,但在所有桑树样品中均有一定比例的分布。
在纲的分类水平看(图5B),4个桑树样品共检测出细菌18个纲(相对丰度≥0.5%)。γ-变形菌纲(Gammaproteobacteria,14.48%)、α-变形菌纲(Alphaproteobacteria,13.21%)、梭菌纲(Clostridia,11.93%)、拟杆菌纲(Bacteroidia,7.48%)4个纲所占比例较大。其中,γ-变形菌纲(Gammaproteobacteria)的优势尤为明显,其在长果桑、川桑7637、新伦教、红果2号各品种中所占比例分别为62.40%、48.38%、59.24%和50.34%;α-变形菌纲(Alphaproteobacteria)次之,其在红果2号中可达26.89%,在长果桑、新伦教中所占比例均大于8%。
在目的分类水平看(图5C),从长果桑、川桑7637、新伦教、红果2号4个桑树样品中共检测出梭菌目(Clostridiales,11.74%)、假单胞菌目(Pseudomonadales,8.59%)、根瘤菌目(Rhizobiales,8.04%)、(Bacteroidales,7.48%)等22个目(相对丰度≥0.5%)。假单胞菌目是所有品种桑树的优势菌目,其在长果桑、川桑7637、新伦教、红果2号中所占比例分别为57.91%、42.65%、54.30%和45.63%。鞘脂单胞菌目(Sphingomonadales)在不同品种桑树中所占的比例差异较大,其在长果桑、川桑7637、新伦教、红果2号各品种中所占比例分别为5.12%、2.69%、12.19%和23.96%。
在科的分类水平看(图5D),假单胞菌科(Pseudomonadaceae,8.35%)、鞘氨醇单胞菌科(Sphingomonadaceae,2.00%),肠杆菌科(Enterobacteriaceae,1.83%)、拟杆菌(Bacteroidaceae,1.23%)和甲基杆菌科(Methylobacteriaceae,0.92%)等25个科在4个桑树品种内生细菌中均有分布(相对丰度≥0.5%)。甲基杆菌科在川桑7637中所占比例较大(4.78%),而在红果2号内的比例却不足1%。
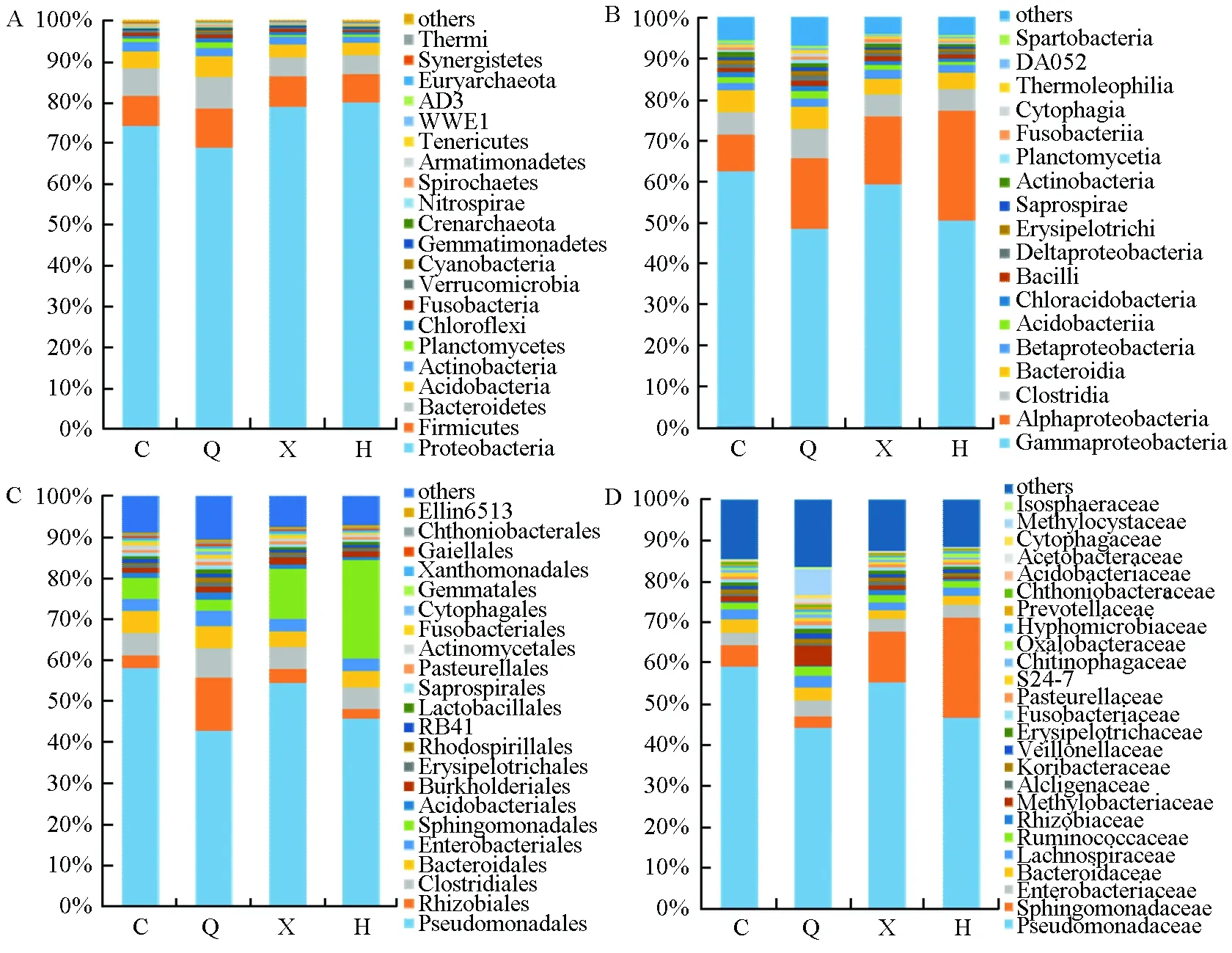
图5 不同样本在不同水平上的细菌群落组成。(A)门,(B)7纲,(C)目,(D)科;图中 C、X、Q、H分别代表长果桑、新伦教、川桑7637、红果2号
属水平上的细菌种群分布特征显示,4个桑树样品共有3857条序列被视为未知序列或尚未被分类到属的序列,这些无法归类的序列占总序列数的65.84%,说明还有很多潜在新菌种待发现。在可归类序列中,主要分布于假单胞菌属(Pseudomonas,8.01%)、鞘氨醇单胞菌属(Sphingomonas,1.78%)、拟杆菌属(Bacteroides,1.14%)、芽孢杆菌属(Bacillus,0.77%)以及甲基杆菌属(Methylobacterium,0.75%),且这些菌属在不同样品中所占比例有所差异。对丰度最高的前40个OTUs分析发现(表2),假单胞菌属为所有品种桑树中所占比例最大的菌属,其在长果桑、川桑7637、新伦教、红果2号中所占比例分别为57.89%、43.08%、54.47%、45.81%,是果桑优势菌属;甲基杆菌属(Methylobacterium)在川桑7637中所占比例较大(4.79%),而在其他三个品种果桑内的比例却不足1%;嗜血杆菌属(Haemophilus)、巨单胞菌属(Megamonas)等菌属亦在桑树中占有一定的比例。

表2 不同品种桑树内生细菌在属水平上的群落结构组成
表中“g__”表示在门或科水平上无法归类的属。
3 讨论与分析
植物内生菌种类繁多,广泛存在于植物的各种器官和组织中,是植物微生态系统的重要组成部分,对宿主植物生长发育发挥重要作用。已有报道显示,内生菌的多样性与植物种类、季节以及生长的地理环境等因素息息相关[40-41]。本研究调查了四个不同果桑品种内生菌多样性特征,结果发现,川桑7637、长果桑、新伦教和红果2号这4个桑树样品均富含大量内生细菌,各品种间的群落结构组成相似,但多样性特征存在较大差异,抗性品种(川桑7637及长果桑)的内生菌群多样性显著高于易感品种(红果2号及新伦教)。此现象表明桑树内生菌的种群分布亦受宿主基因型的影响,推测菌体对宿主的这种选择偏好性可能与宿主自身的生理结构、营养代谢途径以及其分泌的次生代谢产物有关。
本实验室前期利用培养法对桐乡青桑树品种内生菌群落结构组成的调查发现,变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)和厚壁菌门(Firmicutes)是主要的桑树内生菌资源[28]。然而,本研究利用第二代高通量测序技术对4个桑树品种内生菌多样性进行调查分析显示,四个桑树品种均含21个门类细菌,其中变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)、酸杆菌门(Acidobacteria)、拟杆菌门(Bacteroidetes)以及放线菌门(Actinobacteria)为优势菌群。通过传统的分离培养方法所发现的植物内生细菌种类及数量远远低于高通量测序得到的菌群数量,说明大量低丰度菌群及未培养微生物在培养法分离中被忽略,而未培养微生物中蕴藏着大量的未知功能基因和生物潜能,在生物能源开发利用和环境保护领域具有重要的应用潜力[42],Illumina MiSeq的第二代高通量测序技术具有测序通量高,操作简单快速,测序信息准确和成本低等特点,为低丰度及未培养微生物的研究带来了新的思路和方式。
本研究利用基于Illumina MiSeq的第二代高通量测序技术,比较了桑椹菌核病不同抗性桑树品种的内生细菌群落结构组成和多样性特征,解决了不可培养及低丰度菌种在传统方法中无法鉴别的局限。后续研究将在本研究基础上通过改良菌株的培养基成分及培养方法等手段,尽可能富集到更多的内生菌资源,并从多样的桑树内生菌资源库中分离筛选具高效稳定抑菌功效的拮抗菌株,为桑树病害的生物防治提供参考依据。
