AtNEK6在棉花旱盐胁迫响应中的功能分析
2018-11-30范鑫赵雷霖翟红红王远孟志刚梁成真张锐郭三堆孙国清
范鑫,赵雷霖,翟红红,王远,孟志刚,梁成真,张锐,郭三堆,孙国清
在棉花旱盐胁迫响应中的功能分析
范鑫,赵雷霖,翟红红,王远,孟志刚,梁成真,张锐,郭三堆,孙国清
(中国农业科学院生物技术研究所,北京 100081)
【目的】是在拟南芥中发现的一种NIMA相关激酶,在拟南芥中过表达能够促进植物的生长发育,提高植物的耐盐耐旱性。通过将转化棉花,研究其在抗逆中的分子机理,为培育耐旱耐盐碱棉花新品种提供理论基础和种质资源。【方法】采用农杆菌转化法,将来源于拟南芥的导入棉花,通过实时荧光定量PCR分析转基因株系中的表达量;通过观察转基因植株表型和扫描电镜观察细胞表皮细胞,分析转基因棉花的生长发育情况;利用甘露醇和NaCl模拟干旱处理和盐处理分析转基因棉花的耐盐耐旱能力,通过测定相关生理指标,鉴定对转基因棉花耐逆的贡献。【结果】利用卡那霉素筛选获得转基因幼苗,通过PCR鉴定获得10个不同的转基因株系;利用qRT-PCR分析筛选出表达量较高的L7、L17、L25株系。正常条件下,与野生型相比,转基因棉花的株高增高、叶面积增大,生长发育得到了促进,通过扫描电镜观察发现转基因棉花叶片的细胞表面积与野生型并无明显差异,细胞周期相关基因和及生长发育相关基因、、和的表达量均上调。通过耐盐耐旱性分析,正常条件下,与野生型相比,转基因株系的根长、鲜重和干重均无明显差异,仅侧根数增多;在250 mmol·L-1甘露醇处理条件下,转基因株系的根长、侧根数、鲜重和干重均显著高于野生型,表现出更好的生长状态;在200 mmol·L-1NaCl处理条件下,转基因株系的侧根数、鲜重和干重均显著高于野生型,相比于野生型生长状态更好。温室中正常生长1月龄的转基因棉花,在300 mmol·L-1甘露醇处理条件下,转基因植株的SOD活性比野生型提高了0.65倍、0.42倍和1.45倍,CAT活性提高了0.65倍、0.64倍和0.42倍,MDA含量降低了0.51倍、0.41倍和0.22倍;250 mmol·L-1NaCl处理结果与甘露醇类似,转基因棉花的耐盐生理指标均优于野生型。另外,相关胁迫响应基因、、和在转基因棉花中的表达量均显著高于野生型棉花。【结论】通过参与细胞周期和生长发育的调控过程促进棉花的生长发育,同时提高棉花在逆境中的耐盐耐旱性。
;转基因棉花;耐盐耐旱性;功能分析
0 引言
【研究意义】棉花是世界上重要的经济作物,在中国及世界经济发展中占有重要地位。干旱、盐碱是影响棉花产量的主要非生物胁迫因素。AtNEK6是在拟南芥中发现的一种NIMA相关激酶,在拟南芥中过表达能够促进植物的生长发育,提高植物的耐盐耐旱性。【前人研究进展】NIMA(never-in-mitosis- A-related kinase,NEKs)相关激酶被称为有丝分裂酶第三家族,属于丝氨酸/苏氨酸激酶家族,位于纺锤体极体,主要调控细胞周期G2到M期过渡,有调节细胞有丝分裂和减数分裂功能[1]。Oakley等[2]最早在细胞分裂周期突变体的遗传筛选中发现了构巢曲霉NIMA相关蛋白。研究表明,NIMA存在于大部分生物中,如,原生生物衣藻、疟原虫和四膜虫等,真核生物果蝇、非洲爪蟾、小鼠和人类等[3];当NIMA缺失后,致使G2期阻滞,NIMA过表达后,导致细胞较早进入有丝分裂[4-5]。目前,人类基因组已发现11个NIMA基因,命名为—[3],更重要的是,从真菌到人类,许多NEKs都在各自的微管组织中心找到[6-9]。进一步研究发现,NEK1在DDR(DNA damage response)和纤毛功能中起重要作用[10];NEK2、NEK6、NEK7和NEK9主要参与细胞从间期转变为有丝分裂过程中中心体分离和有丝分裂纺锤体装配以及染色质的浓缩,核孔复合体(nuclear pore complex,NPC)的分解和核膜破裂等结构变化[11-12];NEK3不仅参与催乳素介导的信号途径,而且可以调节神经元中微管的乙酰化状态[13-14];NEK11通过磷酸化CDC25A蛋白促进其在DNA损伤诱导的细胞周期检查点中的降解,且该基因突变诱导癌症发生[15];NEK4主要参与DNA损伤反应、纤毛维持、微管稳定、细胞凋亡信号、RNA剪接及应激反应[16]。目前,在植物中已经发现了很多NEK基因。Zhang等[17]在金鱼草中发现了第一个NIMA类基因。随后,拟南芥、番茄、水稻等植物中也发现了NIMA基因家族。在番茄中的NIMA蛋白与SP蛋白相互作用,调控茎的发育和开花[18]。在拟南芥中过表达杨树导致植株的花和果荚异常发育,表明可能参与了花器官发育的调控[19]。Fujii等[20]发现在水稻中,OsNEK3被线粒体磷酸酶2C DCW11调控,控制花粉的萌发。目前在植物中研究的比较系统的是拟南芥中的AtNEK6,张博[21]等报道过表达促进莲座叶生长、侧根形成、结种数以及胁迫耐受能力。突变体的叶子和果荚都很短,侧根数也减少,同时对盐胁迫和渗透胁迫过敏感。此外,过表达上调周期蛋白基因和的表达,而突变体下调细胞周期蛋白的表达,但突变体表现出乙烯相关基因表达上调和乙烯含量的积累。结果表明,AtNEK6通过细胞周期蛋白基因的激活和乙烯的抑制来调节植物的生长和胁迫应答过程。大豆中,与亲缘关系较近,将其转入拟南芥后,提高了植株的耐盐耐寒性[22]。【本研究切入点】过表达促进了拟南芥的生长发育并提高了其耐逆性。然而,将转入棉花等经济作物后是否同样可以促进棉花植株的生长发育并提高其耐逆性并未见报道。【拟解决的关键问题】本研究鉴于在拟南芥模式植物中过表达赋予受体植株较优良的抗逆性状,通过将转化棉花,并分析其在棉花中的功能,以期获得优良抗逆种质,为培育抗逆广适的棉花新品种提供支持。
1 材料与方法
1.1 试验材料
由中国科学院遗传与发育研究所陈受宜研究员惠赠。陆地棉(L.)品种R18、农杆菌()菌株GV3101为生物技术研究所作物分子育种实验室保存,植物表达载体pBI121-由生物技术研究所作物分子育种实验室构建并保存。植物DNA提取试剂盒购于天根生化科技公司,植物总RNA提取试剂盒购于原平皓生物技术公司,反转录试剂盒购于全式金生物技术公司,实时荧光定量PCR(qRT-PCR)试剂盒购于东洋纺生物科技公司,检测植物生理指标的试剂盒购于北京索莱宝科技公司,其余常规试剂购于拜尔迪生物技术公司,引物(电子附表1)由上海生工生物工程公司合成。
1.2 转化棉花及阳性植株的鉴定
通过电击法将pBI121-质粒转化GV3101农杆菌感受态,通过农杆菌介导法转化棉花,使用诱导筛选培养基培养2—3个月后,转到诱导分化培养基上,4个月后分化出胚状体,然后转移到胚萌发培养基上,2个月后获得转基因棉花。将种子种植于花盆中,待棉花长出真叶,提取棉花基因组DNA,PCR鉴定阳性转基因植株[23]。
通过卡那霉素和PCR鉴定筛选阳性植株,温室培养收获T1代种子,将T1代植株在花盆中于温室培养,通过筛选鉴定阳性植株,获得T2代种子。
1.3 实时荧光定量PCR(qPCR)分析
用RNA提取试剂盒提取棉花样品总RNA,反转录获得cDNA,按照KOD SYBR qPCR MIX(QKD-201)的说明书配置荧光定量PCR反应体系,使用仪器ABI公司的FAST-7500进行反应和分析,其中以棉花作为内参基因,利用Primer3设计内参基因和目的基因的引物(电子附表1),按照2-ΔΔCt法计算目的基因的相对表达量。
1.4 转基因棉花表型分析
对1月龄棉花进行株高及叶面积(第三叶至第十叶)测量,参考Tao等[24]方法进行植株表皮细胞测量,取自下而上第五叶片进行扫描电子显微镜(scanning electron microscope,SEM)分析,每个材料进行4次重复。
1.5 转基因棉花的耐盐耐旱性分析
将消毒后的转基因棉花种子和野生型棉花种子种于含250 mmol·L-1甘露醇和200 mmol·L-1NaCl的1/2MS培养基上,待种子萌发培养1周后,每个株系选取6株进行根长、侧根数、鲜重和干重的测定。
在温室中,将不同株系的种子种植于含1﹕1(v/v)蛭石和珍珠盐的小盆(直径12 cm)中,用Hogland营养液培养1个月,每隔3 d浇灌100 mL/次,待棉花长至1个月,进行胁迫处理,盐处理组每隔3 d浇灌100 mL含250 mmol·L-1NaCl营养液;干旱处理组每隔3 d浇灌100 mL含300 mmol·L-1甘露醇营养液;对照组每隔3 d浇100 mL营养液。处理24 h时取棉花植株叶片材料,检测相关胁迫因子的表达量,并测定SOD、CAT活性和MDA含量,每个株系取4个重复。胁迫处理2周后,观察表型并拍照。
2 结果
2.1 转基因棉花阳性植株鉴定
通过农杆菌介导法将植物表达载体pBI121-转化棉花,共获得10个不同的转基因株系。根据qPCR的检测结果,选择表达量较高的3个不同的转基因株系L7、L17、L25进行下一步试验。(图1-A—图1-D)在转基因植株中均有表达,野生型植株未检测到表达(图1-E)。
2.2 在棉花中过表达AtNEK6促进植物生长
待棉花长至1个月,正常生长条件下进行表型观测,转基因株系株高、叶片大小和叶面积均高于野生型(图2-A—图2-D),通过扫描电镜观察转基因株系和野生型叶片表皮细胞的变化,结果表明,转基因株系的叶片表皮细胞和野生型植株的叶片表皮细胞大小相似(图2-E和图2-F)。
为了解促进棉花生长发育的分子机制,利用qPCR技术分别检测野生型植株和转基因株系棉花中细胞周期蛋白基因、和生长发育相关基因[24]、[25]、[26]、[27]在幼苗期的表达情况。结果表明,与野生型相比,转基因植株中、、、、和均上调表达(图2-G)。
2.3 转基因棉花的耐盐耐旱性分析
为了研究转棉花的耐旱性和耐盐性,分别将转基因棉花和野生型棉花种子种植于含甘露醇和NaCl的1/2MS培养基中进行胁迫处理(图3-A)。结果表明,正常条件下,转基因株系的根长、鲜重和干重与野生型相比均无明显差异(图3-A—图3-B和图3-D—图3-E),但转基因株系侧根数均高于野生型(图3-C),平均侧根数分别提高了11.49%、22.41%和22.41%;甘露醇胁迫处理7 d后,与野生型植株相比,转基因植株根系更加发达,且平均根长分别提高了19.89%、22.04%和29.56%,平均侧根数分别提高了86.84%、71.05%和90.78%,平均鲜重分别增加了1.23倍、1.07倍和1倍,平均干重分别增加了1倍、0.72倍和1倍;NaCl胁迫处理7 d后,转基因株系和野生型植株根长没有明显差异,但转基因株系平均侧根数分别提高了1.31倍、1.10倍和1.23倍,平均鲜重增加了0.75倍、0.90倍和0.62倍,平均干重增加了0.73倍、1.04倍和0.82倍。
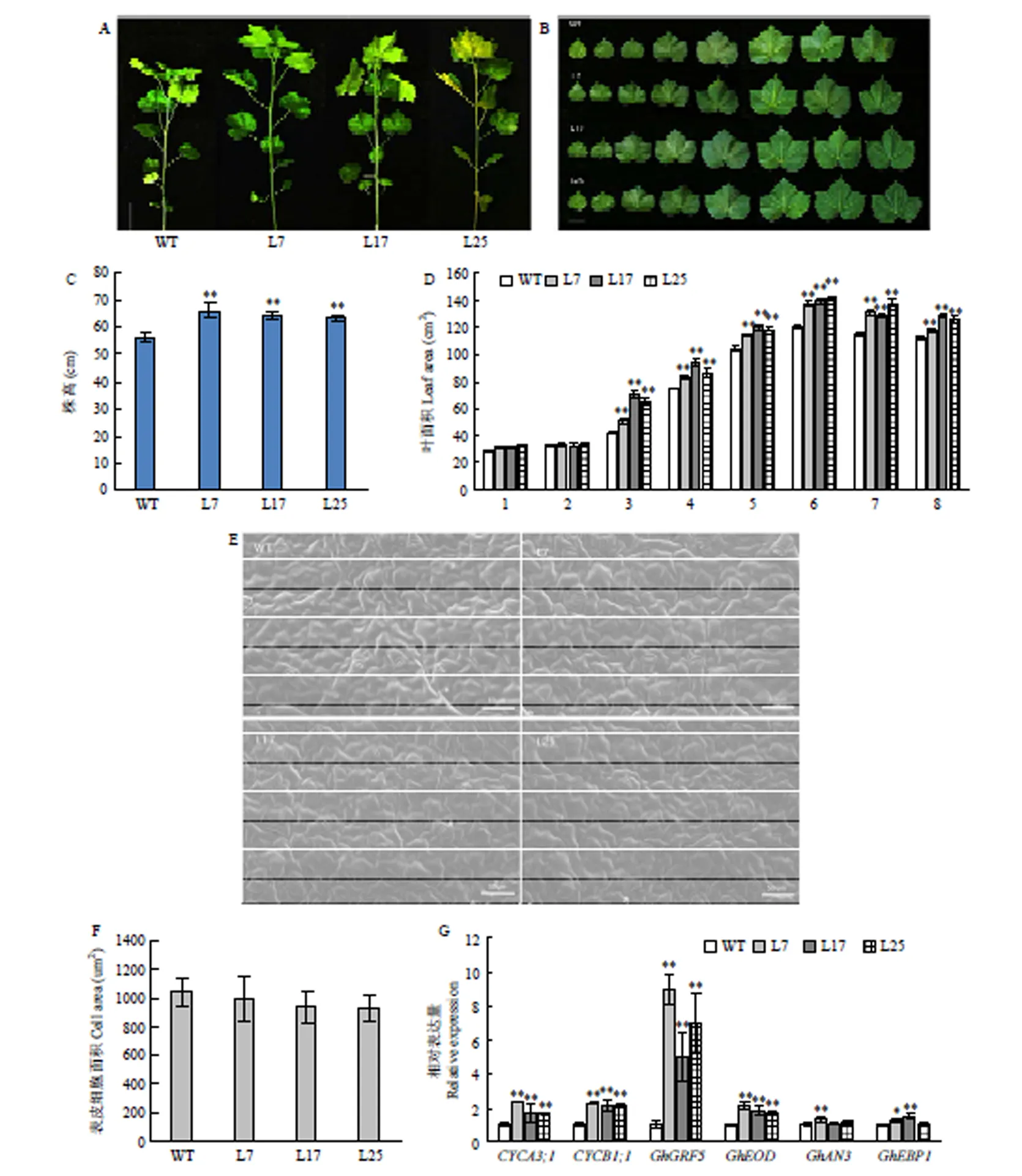
A:野生型植株和转基因植株的表型,Bar=10 cm;B:野生型植株和转基因植株的第三叶至第十叶,Bar=5 cm;C:野生型植株和转基因植株的株高;D:野生型植株和转基因植株的叶面积,1—8:第一片真叶到第八片真叶;E:扫面电镜观察野生型植株和转基因植株的叶表皮细胞,Bar=50 μm;F:野生型植株和转基因植株的表皮细胞面积;G:细胞周期和生长发育相关基因的表达量检测。*表示在P<0.05水平差异显著。下同
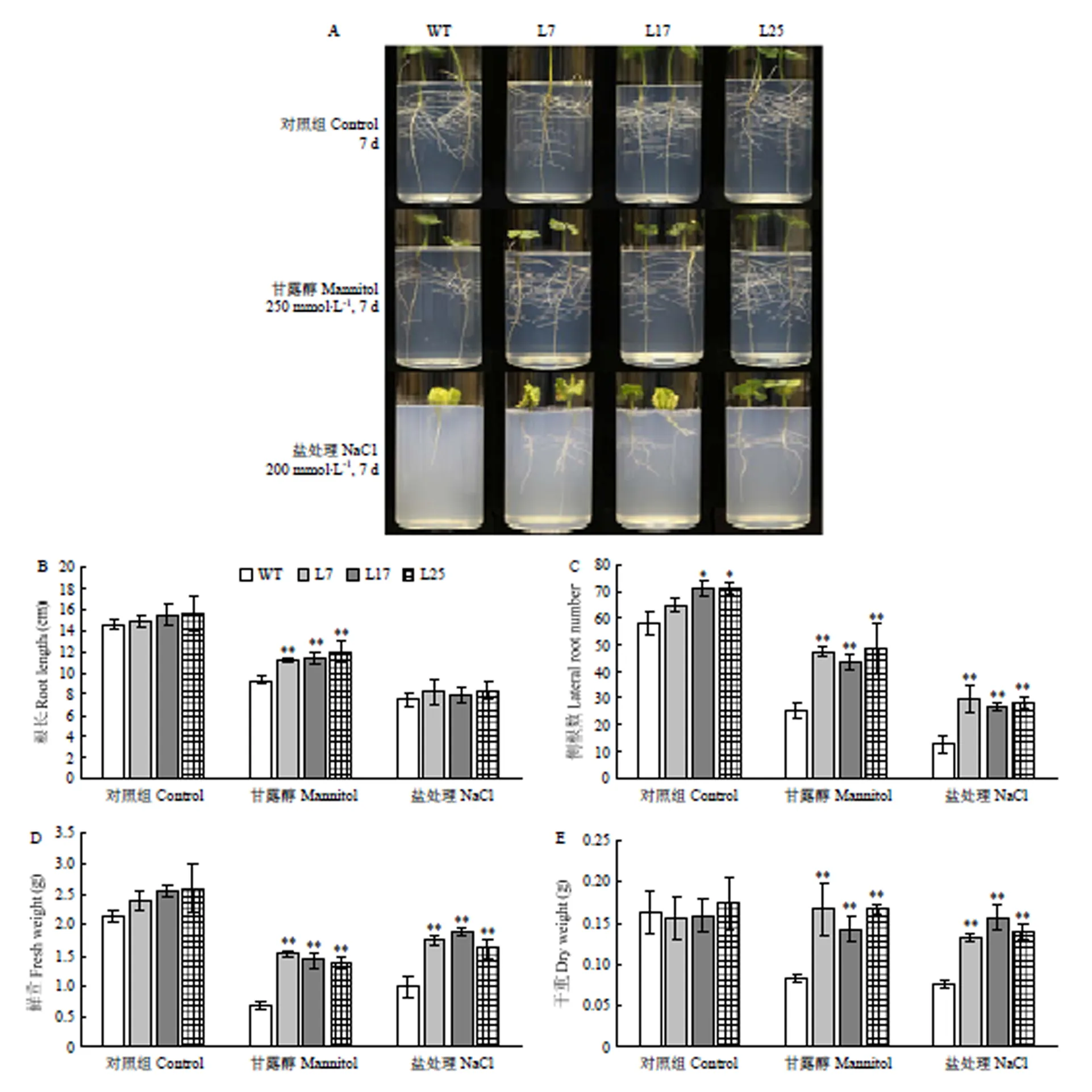
A:在含250 mmol·L-1甘露醇和200 mmol·L-1 NaCl的1/2MS培养基上生长7 d的野生型植株和转基因植株;B:根长;C:侧根数;D:鲜重;E:干重
室温条件下,对正常培养1月龄的转基因植株和野生型植株进行干旱和盐胁迫处理,胁迫处理前,转基因株系和野生型植株均正常生长,但转基因株系株高均高于野生型植株(图4-A);甘露醇处理后,野生型植株萎蔫程度较转基因株系严重,盐胁迫处理后,野生型植株和转基因株系均未出现萎蔫(图4-A)。
SOD和CAT的活性以及MDA含量是植物在抗逆性方面的重要生理指标。正常条件下,野生型植株与转基因植株的CAT活性、SOD活性和MDA含量并无明显差异。甘露醇处理组中,野生型棉花的SOD酶活为89.71 U·g-1,转基因植株L7、L17和L25的SOD酶活分别为148.12、127.39和219.91 U·g-1,比野生型提高了0.65倍、0.42倍和1.45倍(图4-B);野生型棉花的CAT酶活为311.88 U·g-1,转基因植株L7、L17和L25的CAT酶活分别为517.54、513.02和442.96 U·g-1,比野生型提高了0.65倍、0.64倍和0.42倍(图4-C);野生型棉花的MDA含量为39.35 nmol·g-1,转基因植株L7、L17和L25的MDA含量分别为19.11、23.05和30.50 nmol·g-1,比野生型棉花降低了0.51倍、0.41倍和0.22倍(图4-D)。盐处理组中,野生型棉花的SOD酶活为141.30 U·g-1,转基因植株L7、L17和L25的SOD酶活分别为197.12、206.88和205.72 U·g-1,比野生型提高了0.40倍、0.46倍和0.46倍;野生型棉花的CAT酶活为216.96 U·g-1,转基因植株L7、L17和L25的CAT酶活分别为345.78、334.48和409.06 U·g-1,比野生型提高了0.59倍、0.54倍和0.89倍;野生型棉花的MDA含量为38.61 nmol·g-1,转基因植株L7、L17和L25的MDA含量分别为31.07、32.12和33.02 nmol·g-1,比野生型棉花降低了0.2倍、0.17倍和0.14倍。
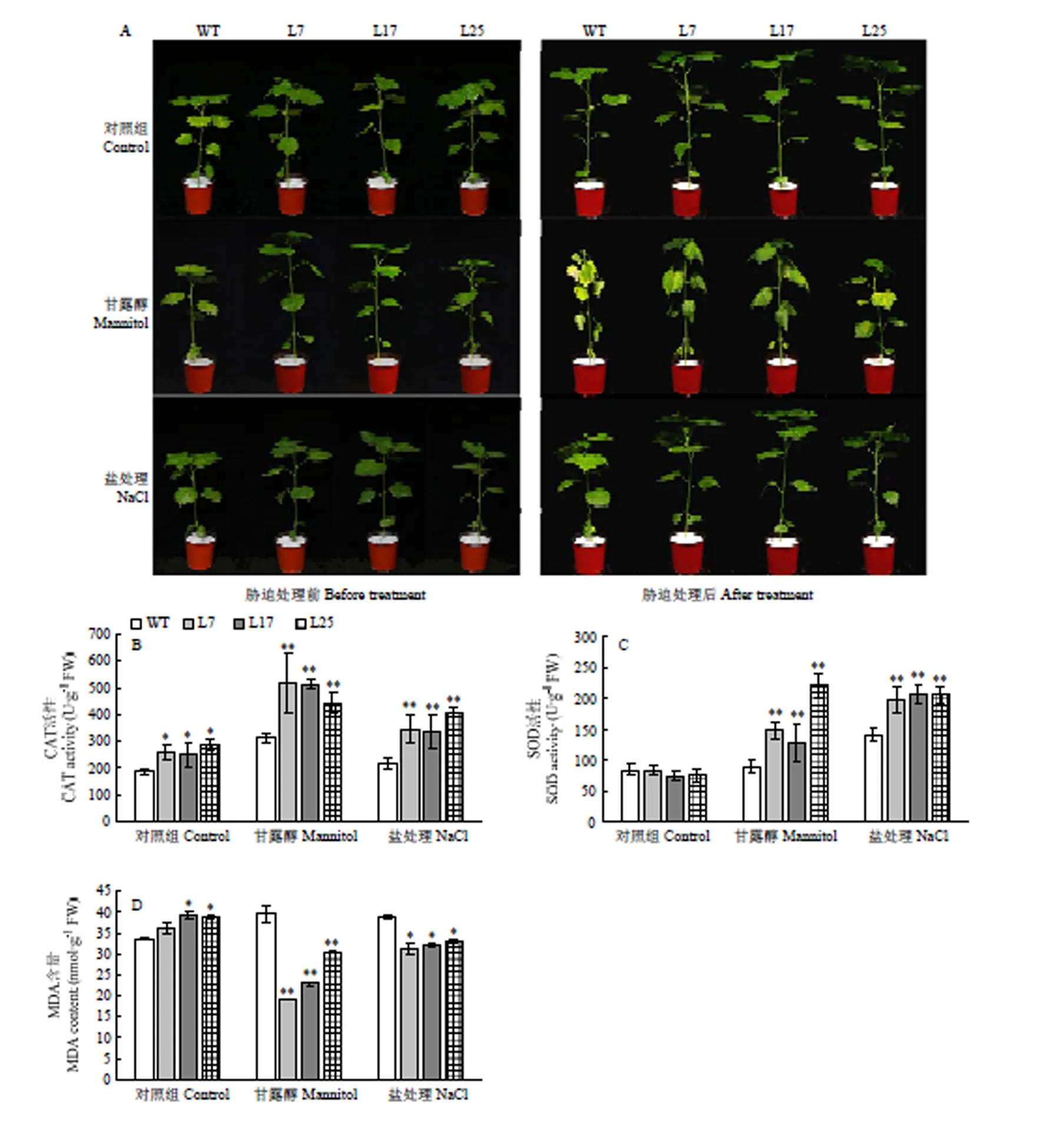
A:胁迫处理后野生型植株和转基因植株的表型;B:CAT活性;C:SOD活性;D:MDA含量
2.4 逆境胁迫相关基因在转基因棉花中的表达分析
通过实时荧光定量(qRT-PCR)分析表明,在甘露醇和NaCl的处理下,逆境胁迫相关基因(ABA-responsive element binding)、(dehydration-responsive element binding)、(9-cis-epoxycarotenoid dioxygenase)、(late embryogenesis-abundant protein)均上调表达(图6)。在正常条件下,在野生型和转基因植株中的表达量没有明显差异,而在盐和甘露醇胁迫后,转基因植株中的表达量相比野生型约提高1倍。正常条件下,、和在转基因植株中的表达量显著高于野生型,进行胁迫处理后,这些基因在野生型中的表达量无明显变化,而胁迫处理后的转基因植株中这三个基因的表达量均显著提高。
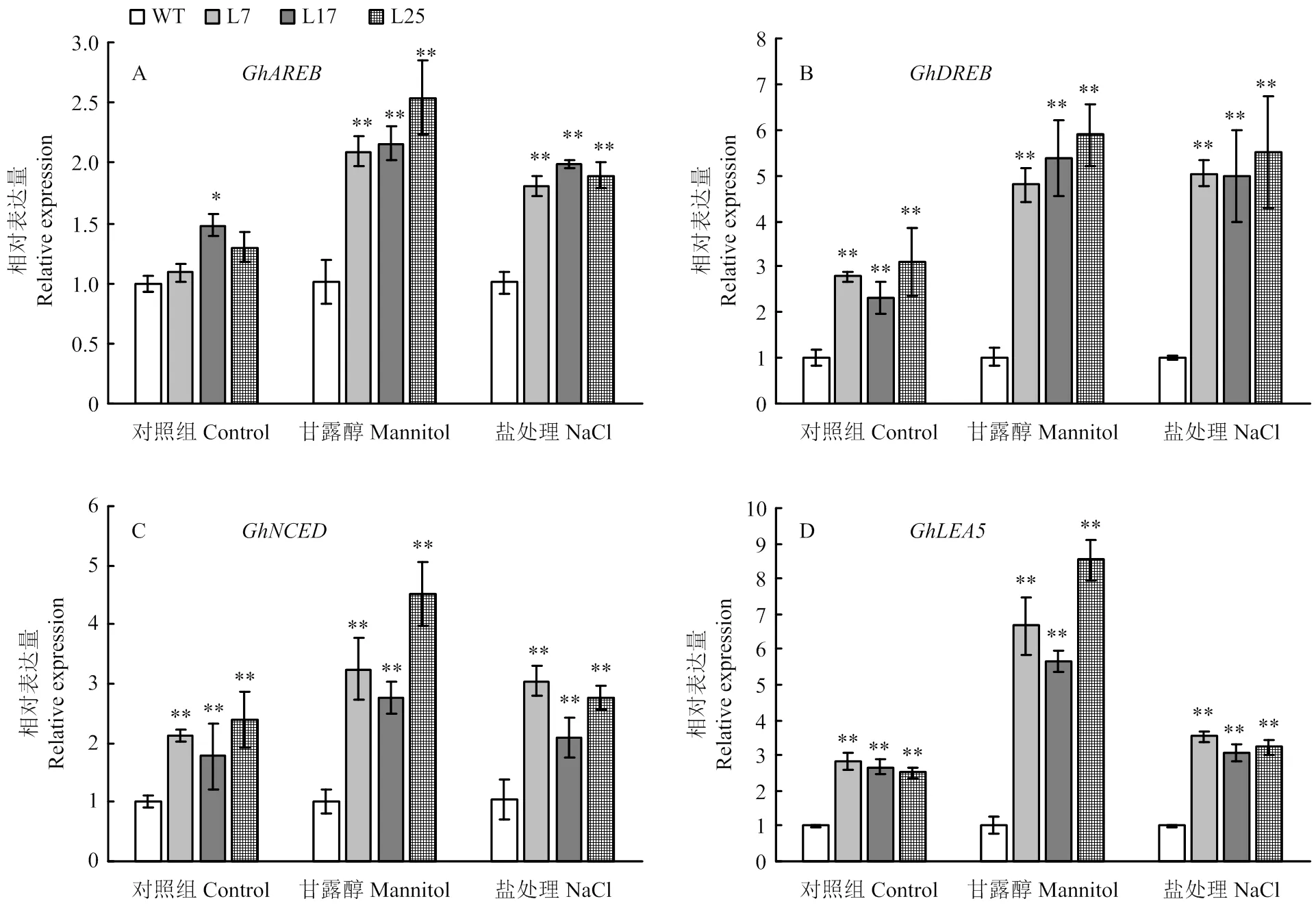
图5 胁迫处理下转AtNEK6棉花中逆境胁迫相关基因的表达分析
3 讨论
3.1 在棉花中过表达AtNEK6对植物生长发育的影响
植物在不良环境中自身会产生一系列的生理代谢反应,如代谢和生长的可逆性抑制,严重时甚至引起不可逆的伤害,导致整个植株死亡。本研究在棉花中过表达,在提高植物耐盐耐旱性的同时,也促进了植物的生长发育。通过观察发现,转基因株系株高、叶片大小和叶面积均高于野生型,这与在拟南芥的研究结果一致[21],进一步通过扫描电镜观察棉花叶片表皮细胞,发现野生型和转基因株系的表皮细胞面积并无明显差异,说明是通过促进细胞增殖影响了植株的生长发育。在过表达拟南芥中,转基因株系的细胞周期相关基因和的表达量均上调,本研究对转棉花中的细胞周期基因以及些重要的生长发育相关基因、、和的棉花同源基因进行表达量分析,亦得到相似结果,因此推测在棉花中可能是通过上调这些基因的表达促进细胞增殖,进而促进了叶片的生长。此外有研究发现,一些细胞周期调控基因是应激诱导型的,可以提高植物的耐受能力[28-29],因此,很有可能是通过调节细胞周期依赖性蛋白,促进了植物的生长发育进而提高了其耐受能力。
3.2 AtNEK6对转基因棉花幼苗的影响
通过在无菌的1/2MS培养基上播种棉花种子,分析在甘露醇和盐的胁迫下,转基因棉花种子的萌发情况以及根的发育情况。发现转基因植株的根系不论在盐胁迫下还是甘露醇胁迫下,相对于野生型都要更加发达,根长更长、侧根数更多;进一步称量并统计了鲜重和干重,转基因植株的鲜重和干重相较于野生型均显著提高,说明在盐和甘露醇的胁迫下,AtNEK6通过维持植株的根系发育,提高了转基因植株在萌发期的耐盐耐旱能力。
3.3 AtNEK6对棉花植株的抗氧化能力的影响
在逆境胁迫下,植物体内会产生大量的超氧化物阴离子和过氧化氢,SOD可以清除过量的阴离子,CAT可以清除过量的过氧化氢,并且它们的活性与非生物胁迫下的氧化损伤相关[30]。本研究转基因植株中的SOD和CAT的活性显著高于野生型,表明通过促进植株的活性氧清除能力,使棉花避免氧化损伤。MDA是细胞膜脂过氧化的产物之一,反应膜脂过氧化程度,MDA含量越低代表植物受损伤程度越低,转基因植株中的MDA含量显著低于野生型,因此,MDA含量的降低可能是转基因植株耐盐耐旱能力增强的一个重要因素。
3.4 AtNEK6对棉花相关耐逆基因的表达影响
耐逆基因在一定程度上反应了植物的耐逆能力,通过直接上调的表达提高了过表达拟南芥的耐盐耐寒能力[22]。本研究检测了逆境胁迫类的重要基因(ABA反应元件)、(脱水反应元件)、(9-顺式-环氧加氧酶)、(水通道蛋白),这些基因表达的升高也表明可能是通过影响多条途径来调控植株的胁迫反应过程,进而提高了植物的耐旱性和耐盐性。
众所周知,各种胁迫导致微管重组和解聚,这对于抗逆性至关重要[31]。在膜蛋白和膜相关蛋白和脂质的调节下,微管在响应植物激素和胁迫中起重要作用[32-33];另外,微管对于植物细胞壁的延伸性至关重要,与细胞生长相关[34]。先前研究表明,拟南芥中NEK6、NEK4和NEK5彼此相互作用以调节表皮细胞扩增期间的定向细胞的生长和微管组织[35],所以植物中的NEK很可能通过控制细胞形态发生和扩张来调节细胞生长和胁迫应答,而这个过程主要由β-微管蛋白磷酸化和微管去稳定化介导[36]。基于这些结果,除了通过调节逆境胁迫相关基因的表达来实现功能,猜测也可能通过自身的激酶活性与微管相关作用,参与微管的重组,从而使植物在不利环境下适应性生长。
总之,作为丝氨酸/苏氨酸激酶家族成员基因,在棉花过表达可能通过多种途径促进了植株的生长发育,并提高了其耐盐耐旱性。该转基因棉花种质资源的获得,为今后培育品质优良、抗逆性高的棉花材料提供了试验依据。
4 结论
转棉花的耐盐耐旱能力得到提高,表现为萌发期根系发育更好、清除活性氧能力增强和不同逆境胁迫相关基因的表达提高。通过影响生长发育的相关基因促进棉花植株的生长发育,通过多条胁迫响应途径调控棉花植株的耐盐耐旱性。
致谢:感谢中国科学院遗传发育研究所陈受宜研究员提供的和相关资料,以及在试验中给予的指导和帮助。
[1] Osmani S A, May G S, Morris N R. Regulation of the mRNA levels of nimA, a gene required for the G2-M transition in., 1987, 104(6): 1495.
[2] Oakley B R, Morris N R. A Mutation inthat Blocks the Transition from I nterphase to Prophase., 1983, 96(4): 1155-1158.
[3] Fry A M, O'Regan L, Sabir S R, Bayliss R. Cell cycle regulation by the NEK family of protein kinases., 2012, 125(19): 4423-4433.
[4] Osmani A H, Mcguire S L, Osmani S A. Parallel activation of the NIMA and p34cdc2cell cycle-regulated protein kinases is required to initiate mitosis in., 1991, 67(2): 283.
[5] Osmani S A, Pu R T, Morris N R. Mitotic induction and maintenance by overexpression of a G2-specific gene that encodes a potential protein kinase., 1988, 53(2): 237-244.
[6] Krien M J, West R R, John U P, Koniaras K, Mcintosh J R, O'Connell M J. The fission yeast NIMA kinase Fin1p is required for spindle function and nuclear envelope integrity., 2002, 21(7): 1713-1722.
[7] Mahjoub M R, Montpetit B, Zhao L, Finst R J, Goh B, Kim A C, Quarmby L M. Thegene ofencodes a NIMA family kinase with roles in cell cycle progression and microtubule severing during deflagellation., 2002, 115(8): 1759-1768.
[8] Prigent C, Glover D M, Giet R.Nek2 protein kinase knockdown leads to centrosome maturation defects while overexpression causes centrosome fragmentation and cytokinesis failure., 2005, 303(1): 1-13.
[9] Wloga D, Camba A, Rogowski K, Manning G. Members of the NIMA-related kinase family promote disassembly of cilia by multiple mechanisms., 2006, 17(6): 2799-2810.
[10] Spalluto C, Wilson D I, Hearn T. Nek2 localises to the distal portion of the mother centriole/basal body and is required for timely cilium disassembly at the G2/M transition., 2012, 91(9): 675-686.
[11] O'Regan L, Blot J, Fry A M. Mitotic regulation by NIMA-related kinases., 2007, 2(1): 25.
[12] Sdelci S, Bertran M T, Roig J. Nek9, nek6, nek7 and the separation of centrosomes., 2011, 10(22): 3816-3817.
[13] Chang J, Baloh R H, Milbrandt J. The NIMA-family kinase Nek3 regulates microtubule acetylation in neurons., 2009, 122(13): 2274.
[14] Miller S L, Antico G, Raghunath P N, Tomaszewski J E, Clevenger C V. Nek3 kinase regulates prolactin-mediated cytoskeletal reorganization and motility of breast cancer cells., 2007, 26(32): 4668.
[15] Melixetian M, Klein D K, Sørensen C S, Helin K. NEK11 regulates CDC25A degradation and the IR-induced G2/M checkpoint., 2009, 11(10): 1247-1253.
[16] Basei F L, Meirelles G V, Righetto G L, Migueleti D L D S, Smetana J H C, Kobarg J. New interaction partners for Nek4.1 and Nek4.2 isoforms: from the DNA damage response to RNA splicing., 2015, 13(1): 1-13.
[17] Zhang H, Scofield G, Fobert P, Doonan J H. A nimA-like protein kinase transcript is highlyexpressed in meristems of, 1996, 181(2): 186-194.
[18] Pnueli L, Gutfinger T, Hareven D, Ben-Naim O, Ron N, Adir N, Lifschitz E. Tomato SP-interacting proteins define a conserved signaling system that regulates shoot architecture and flowering., 2001, 13(12): 2687-2702.
[19] Cloutier M, Vigneault F, Lachance D, Séguin A. Characterization of a poplar NIMA-related kinase PNek 1 and its potential role in meristematic activity., 2005, 579(21): 4659.
[20] Fujii S, Yamada M, Toriyama K. Cytoplasmic male sterility- related protein kinase,, is regulated downstream of mitochondrial protein phosphatase 2C, DCW11., 2009, 50(4): 828-837.
[21] Zhang B, Chen H W, Mu R L, Zhang W K, Zhao M Y, Wei W, Wang F, Yu H, Lei G, Zhou H F, Ma B, Chen S Y, Zhang J S. NIMA-related kinase NEK6 affects plant growth and stress response in., 2011, 68(5): 830-843.
[22] Pan W J, Tao J J, Cheng T, Shen M, Ma J B, Zhang W K, Lin Q, Ma B, Chen S Y, Zhang J S. Soybean NIMA-related kinase1 promotes plant growth and improves salt and cold tolerance., 2017, 58(7): 1268.
[23] 岳建雄, 孟钊红, 张炼辉, 韩少杰, 林亲铁, 王巍. 以甘露糖作为筛选剂的棉花遗传转化. 棉花学报, 2005, 17(1): 3-7.
YUE J X, Meng Z H, Zhang L H, Han S J, Lin Q T, Wang W. Cotton transformation with mannose as selective agent., 2005, 17(1): 3-7. (in Chinese)
[24] Tao J J, Cao Y R, Chen H W, Wei W, Li Q T, Ma B, Zhang W K, Chen S Y, Zhang J S. Tobacco translationally controlled tumor protein interacts with ethylene receptor tobacco histidine kinase1 and enhances plant growth through promotion of cell proliferation., 2015, 169(1): 96-114.
[25] Horiguchi G, Gyung-Tae K, Tsukaya H. The transcription factor AtGRF5 and the transcription coactivator AN3 regulate cell proliferation in leaf primordia of., 2005, 43(1): 68-78.
[26] Li Y, Zheng L, Corke F, Smith C, Bevan M W. Control of final seed and organ size by thegene family in., 2011, 138(20): 4545-4554.
[27] Horváth B M, Magyar Z, Zhang Y, Hamburger A W, Bakó L, Visser R G, Bachem C W, Bögre L. EBP1 regulates organ size through cell growth and proliferation in plants., 2006, 25(20): 4909-4920.
[28] Burssens S, Himanen K, Van d C B, Beeckman T, Van M M, Inzé,D, Verbruggen N. Expression of cell cycle regulatory genes and morphological alterations in response to salt stress in., 2000, 211(5): 632-640.
[29] West G, Inzé D, Beemster G T. Cell cycle modulation in the response of the primary root ofto salt stress., 2004, 135(2): 1050-1058.
[30] Lin A, Wang Y, Tang J, Xue P, Li C, Liu L, Hu B, Yang F, Loake G J, Chu C. Nitric oxide and protein S-nitrosylation are integral to hydrogen peroxide-induced leaf cell death in rice., 2012, 158(1): 451-464.
[31] Khatri N, Mudgil Y. Hypothesis: NDL proteins function in stress responses by regulating microtubule organization., 2015, 6(437): 1-6.
[32] Shi L, Wang B, Gong W, Zhang Y, Zhu L, Yang X. Actin filaments and microtubules ofsuspension cells show different responses to changing turgor pressure., 2011, 405(4): 632.
[33] Zhang Q, Zhang W. Regulation of developmental and environmentalsignaling by interaction between microtubules and membranes in plant cells., 2016, 7(2): 81-88.
[34] Cosgrove D J. Plant cell wall extensibility: connecting plant cell growth with cell wall structure, mechanics, and the action of wall-modifying enzymes., 2016, 67(2): 463.
[35] Motose H, Hamada T, Yoshimoto K, Murata T, Hasebe M, Watanabe Y, Hashimoto T, Sakai T, Takahashi T. NIMA-related kinases 6, 4, and 5 interact with each other to regulate microtubule organization during epidermal cell expansion in., 2011, 67(6): 993-1005.
[36] Takatani S, Otani K, Kanazawa M, Takahashi T, Motose H. Structure, function, and evolution of plant nima-related kinases: implication for phosphorylation-dependent microtubule regulation., 2015, 128(6): 875-891.
(责任编辑 李莉)
Functional Characterization ofoverexpression in Cotton under Drought and Salt Stress
FAN Xin, ZHAO LeiLin, ZHAI HongHong, WANG Yuan, MENG ZhiGang, LIANG ChengZhen, ZHANG Rui, GUO SanDui, SUN GuoQing
(Biotechnology Research Institute, Chinese Academy of Agricultural Sciences, Beijing 100081)
【Objective】is a NIMA related kinase in. Overexpression ofcan promote plant growth, and improve the salt tolerance and drought tolerance of plants. By transforminginto cotton, the molecular mechanism of its resistance to stress was studied, so as to provide theoretical basis and germplasm resources for breeding new cotton varieties with drought tolerance and salinity tolerance.【Method】Thegene was introduced into cotton by thetransformation method, and the expression level ofto the stress tolerance of transgenic cotton was identified by measuring related physiological indexes.【Result】The transgenic seedlings were screened by Kanamycin, and 10 different transgenic lines were identified by PCR. qRT-PCR analysis was used to select L7, L17 and L25 with higher expression levels. Under normal conditions, the transgenic lines were exhibited higher height and larger leaf than wild-type plants. But the cell surface area of transgenic cotton leaves was not significantly different from that of wild type by scanning electron microscope. The expressions of cell cycle related genesandand growth related genes,,andwere upregulated in the transgenic lines. Salt and drought tolerance of transgenic cotton was analyzed. On the normal 1/2MS medium, the root length, fresh weight and dry weight of transgenic lines were not significantly different from those of wild type, and the number of lateral roots increased. However, in medium containing 250 mmol·L-1mannitol, the root length, the lateral root number, fresh weight and dry weight of transgenic lines were significantly higher than those of wild type, showing a better growth state. In medium containing 200 mmol·L-1NaCl,the number of lateral roots, fresh weight and dry weight of the transgenic lines were significantly higher than those of the wild type. Cotton seedlings of 30 days normal growth in the greenhouse were treated with 300 mmol·L-1mannitol, the SOD activity of the transgenic plants was increased by 0.65 times, 0.42 times, 1.45 times compared with the wild type, and the CAT activity was increased by 0.65 times, 0.64 times, 0.42 times, and the MDA content was decreased. 0.51 times, 0.41 times, 0.22 times. Similarly, the changes of physiological indexes in transgenic lines in 250 mmol·L-1NaCl treatment were higher than the ones in WT. In addition, the expression levels of related stress responsive genes,,和【Conclusion】promotes the growth of cotton by participating in the regulation of cell cycle and growth. At the same time, it improved the salt tolerance and drought tolerance of cotton in adversity.
; transgenic cotton; salt and drought tolerance; functional analysis
2018-06-08;
2018-07-23
国家重点研发计划“棉花杂种优势利用技术与强优势杂交种创制(2016YFD0101419)”、国家转基因重大专项资助项目(2016ZX08005-004)
范鑫,Tel:010-82106128;E-mail:fanxin_1102@qq.com。
孙国清,Tel:010-82106128;E-mail:sunguoqing02@caas.cn。通信作者郭三堆,E-mail:guosandui@caas.cn
10.3864/j.issn.0578-1752.2018.22.002