基于ITS和ISSR的羊肚菌种质资源遗传多样性分析
2018-11-30张小平毋校辉罗金洲
杨 燕,田 鸿*,张小平,毋校辉,罗金洲
(1.四川农业大学 资源学院,四川 成都 611130;2.四川菌之味农业科技有限公司,四川 成都 611130)
【研究意义】羊肚菌属于子囊菌门(Ascomycota),盘菌纲(Pezizomycetes),盘菌目(Pezizales),羊肚菌科(Morchellaceae),羊肚菌属(Morchella),是一种珍惜名贵的食用菌,具有较高的营养价值,被称之为“菌中之王”,备受人们喜爱[1]。长久以来,市场主要以野生羊肚菌采集为主,随着消费和认知水平的扩大,野生羊肚菌已经无法满足市场需求,国内不少地区已出现野生羊肚菌产量明显下降趋势。可喜的是,在全球蘑菇爱好者和我国菇民朋友的不懈努力下,羊肚菌人工栽培取得了长足进展,特别是近年来发源自我国川渝地区的羊肚菌大田栽培技术,逐渐成熟并推广至全国各地,实现了羊肚菌的商业化栽培[2]。羊肚菌驯化栽培道路艰辛而漫长的一个主要原因是可栽培菌株的确定。美国的工厂化栽培菌株为变红羊肚菌,而适应于我国当前栽培模式的菌株主要是黑色羊肚菌支系种类[3],主要为梯棱羊肚菌(Morchellaimportuna),六妹羊肚菌(Morchellasextelata)和七妹羊肚菌(Morchellaseptimelata)[4]。种质资源研究匮乏、种性退化严重、同物异名和异物同名的现象[5],这是制约我国羊肚菌稳步发展的主要原因。【前人研究进展】随着分子生物学的发展,羊肚菌多基因序列模标数据库MLST[6](Multilocus sequence typing)的开发,众多分子标记技术广泛应用于羊肚菌的分子鉴定。刘丛文利用核糖rDNA内部转录间隔区(Internal Transcribed Spacer,ITS)序列指导鉴定羊肚菌所属物种[7]。Kerry O’Donnell等[8]人利用28S rDNA-RPB1-RPB2-EF-1α多基因序列联合分析羊肚菌系统发育,最后将羊肚菌分为三大支系:黑色羊肚菌支系(Elata,Clade)、黄色羊肚菌支系(Esculenta Clade)和变红羊肚菌支系(Rufobrunnea Clade), 杜习慧等[9]通过多基因序列研究中国羊肚菌,同样将其分为以上三大支系,目前最具权威的分类体系。杜习慧等[10]利用羊肚菌交配型基因(Mating-Type locus,MAT)构建系统发育树,能很好区分近缘物种,分析其亲缘关系,对指导种质鉴定具有重要意义。目前商业化栽培的羊肚菌大部分是黑色羊肚菌支系,而黄色羊肚菌支系和变红羊肚菌支系的羊肚菌未有成功商业化栽培的报道。学者们从各地采集野生羊肚菌,驯化人工栽培,鉴定羊肚菌所属种类,这有利于选育适合人工栽培的菌株。 国内关于羊肚菌遗传多样性研究的报道较少,刘文丛[11]利用ISSR(Inter-Simple Sequence Repeat)分子标记方法对滇西北地区56株羊肚菌子实体进行了遗传多样性及系统发育分析,结果表明,羊肚菌居群间的遗传分化程度较大,而居群内的遗传分化程度相对较小。羊肚菌物种丰富,杜习慧等[12]第一次进行了同区域2种羊肚菌种群间遗传多样性研究,结合分子标记手段表明两者有明显的变异性,表明两个同源物种具有不同的潜在进化史。【本研究切入点】本研究以18株羊肚菌菌株(其中人工栽培菌株14株,菌株4株)为供试材料,结合形态学特征和ITS序列对供试菌株进行鉴定,并构建了供试菌株的系统发育关系。结合ISSR分子标记技术对其进行聚类分析,系统的对供试菌株间的种质资源进行分析。【拟解决的关键问题】本研究有助于羊肚菌遗传育种工作的开展。
1 材料与方法
1.1 供试菌株
供试菌株共计18株,具体信息见表1。
1.2 试剂与仪器
PDA培养基:马铃薯200 g、葡萄糖20 g、琼脂20 g、水1000 mL。主要试剂:葡萄糖、琼脂粉、琼脂糖(北京奥博星生物技术有限责任公司);真菌基因组DNA抽提试剂盒(Ezup)、PCR MIX、DNA凝胶回收试剂盒及DNA Marker DL 2000 bp均购自上海生工生物工程有限公司;ITS1、ITS4和ISSR引物,均由北京擎科新业生物技术有限公司合成。主要仪器:研钵、恒温培养箱、涡旋振荡器、冷冻离心机、PCR仪、电泳仪、凝胶成像仪等。

表1 供试菌株
1.3 菌种活化及DNA的提取
将羊肚菌菌丝块接种至新鲜PDA平板上,于22 ℃恒温培养,待菌丝长满后备用。
取已经准备的羊肚菌菌丝体为材料,液氮研磨,按照真菌基因组DNA提取试剂盒上操作步骤提取供试菌株DNA。采用紫外分光光度计检测DNA的浓度和纯度,-20 ℃冰箱保持备用。
1.4 ITS基因PCR扩增
ITS1:TCCGTAGGTGAACCTGCGG; ITS4:TCCTCCGCTTATTGATATGC。PCR反应体系(50 μl)如下:2×TaqMix 25 μl,真菌通用引物(10 μM)ITS1和ITS4各1 μl,模板DNA加1 μl,加入22 μl ddH2O补齐体系。PCR程序:94 ℃预变性5 min;94 ℃变性1 min,61 ℃退火1 min,72 ℃延伸1 min,共计35个循环;循环结束后再72 ℃延伸10 min,反应结束。PCR产物于1.5 %的凝胶上电泳,电压120 V,电泳20 min。用凝胶成像系统拍照并保存,用DNA凝胶回收试剂盒回收纯化的片段,将其于擎科生物技术有限公司(成都)完成测序。
1.5 ISSR-PCR扩增
参考刘文丛的文章[11],经过筛选出条带清晰、重复性和稳定性好且多肽条带相对较多的引物用于羊肚菌遗传多样性分析,其引物ISSR1:AGAGAGAGAGAGAGAGC。ISSR-PCR扩增反应体系(30 μl):2×TaqMix 15 μl,ISSR引物(10 μM)1.5 μl,模板DNA 3 μl,加入10.5 μl ddH2O补足体系。PCR程序:95 ℃预变性2 min;94 ℃变性1 min,48 ℃退火1 min,65 ℃延伸8 min,30个循环;循环结束后再65 ℃延伸16 min,反应结束。取5 μl PCR产物于2 %的凝胶上电泳,电压80 V,电泳1.5 h。将凝胶移至凝胶成像仪下观察并照相保存。
1.6 数据分析
登录NCBI数据库,将所测得的羊肚菌ITS序列进行BLAST比对,下载相似度高的序列,用MEGA6.0软件进行系统发育分析,根据邻近遗传距离法(NJ法)构建系统发育树[13],对结果进行分类,结合形态特征分析,鉴定羊肚菌菌株。
ISSR标记的电泳图中每1个条带代表引物的1个特异结合位点人工读取电泳条带,有扩增条带的为“1”,同一位置无条带的为“0”,在EXCEL表中建立数据矩阵。采用NTSYS-pc2.1软件计算样品之间的相似系数,并进行UPGMA(非加权算术平均聚类)进行聚类分析[14]。
2 结果与分析
2.1 系统发育分析
我国目前的羊肚菌人工栽培菌株主要是梯棱羊肚菌和六妹羊肚菌[16],其中梯棱羊肚菌的推广应用面积最大,野生梯棱羊肚菌主要集中在四川、云南、湖北、贵州等地。梯棱羊肚菌、六妹羊肚菌和高羊肚菌子实体形状如图1~3所示。
供试菌株的系统发育树如图4所示。9株来自不同地区的9株人工栽培菌株(YD9a、YD13、YD7、YD2、YD1、cang1、YD16、YD17、YD9b)和野生菌株GS(采自陕西)为梯棱羊肚菌。2株人工栽培菌株(12wen、1212M)为六妹羊肚菌;野生羊肚菌菌株1431B(采自成都大邑)、WL3(采自重庆武隆)和2株人工栽培菌株(cang2、YD6)为高羊肚菌(M.elata)。以上菌株均属于黑色羊肚菌支系。而野生菌株LF(采自湖北恩施)和YD15(采自绵阳平武)为粗柄羊肚菌(M.crassipes)[15],属于黄色羊肚菌支系。

图1 梯棱羊肚菌Fig.1 Morchella importuna

图2 六妹羊肚菌Fig.2 Morchella sextelata
2.2 ISSR-PCR聚类分析
利用已经优化的羊肚菌ISSR-PCR扩增体系,对引物ISSR1进行扩增(图5),不同的菌株扩增出的条带具有明显的差异,且多态性丰富,重复性强。
2.3 供试菌株ISSR聚类分析
采用UPGMA法构建了羊肚菌菌株间聚类图,如图6所示,在相似系数0.54处可将18株羊肚菌分为2大类,即M1、M2、M9、M7、M12、M11、M8、M18、M10、M13、M6、M16、M5为1类;M3、M14、M4、M15、M17为1类。其中M7和M12相似系数高达99 %,两者亲缘关系最近;M1和M2相似系数高达95 %,M8和M18相似系数高达95 %,M6和M16相似系数高达95 %,他们之间遗传关系比较近,而M1和M17遗传关系最远。
3 讨 论
羊肚菌是一种名贵的食用菌,具有较高的营养价值。其鲜品可以直接进入消费市场,也可以做成干品,便于运输和储藏,其干品有种特殊的菌香味,深得消费者喜爱[17]。目前羊肚菌已经实现了人工栽培,不会再因为野生羊肚菌的稀缺而无法满足消费者的需求,谭方河[18]阐述了羊肚菌栽培历史,经过不断探索,栽培技术已有了很大的进步。羊肚菌属于子囊菌,遗传特性不稳定,在栽培过程中,学者们开始意识到菌种在羊肚菌驯化及栽培中的重要性,羊肚菌菌株退化后,栽培将面临绝收的可能,造成巨大的经济损失[19-21]。目前解决羊肚菌菌种稳定性,主要从品种选育及驯化栽培,筛选出优良性状的菌株,同时研究羊肚菌种质资源,生活史,菌种老化与退化,遗传育种等基础理论[22]。

图3 高羊肚菌Fig.3 Morchella elata

加粗为供试菌株图4 供试菌株的系统发育树Fig.4 Phylogeny tree constructed by the tested strains

图5 18株羊肚菌扩增ISSR图谱分析Fig.5 ISSR profile of 18 Morchella strains
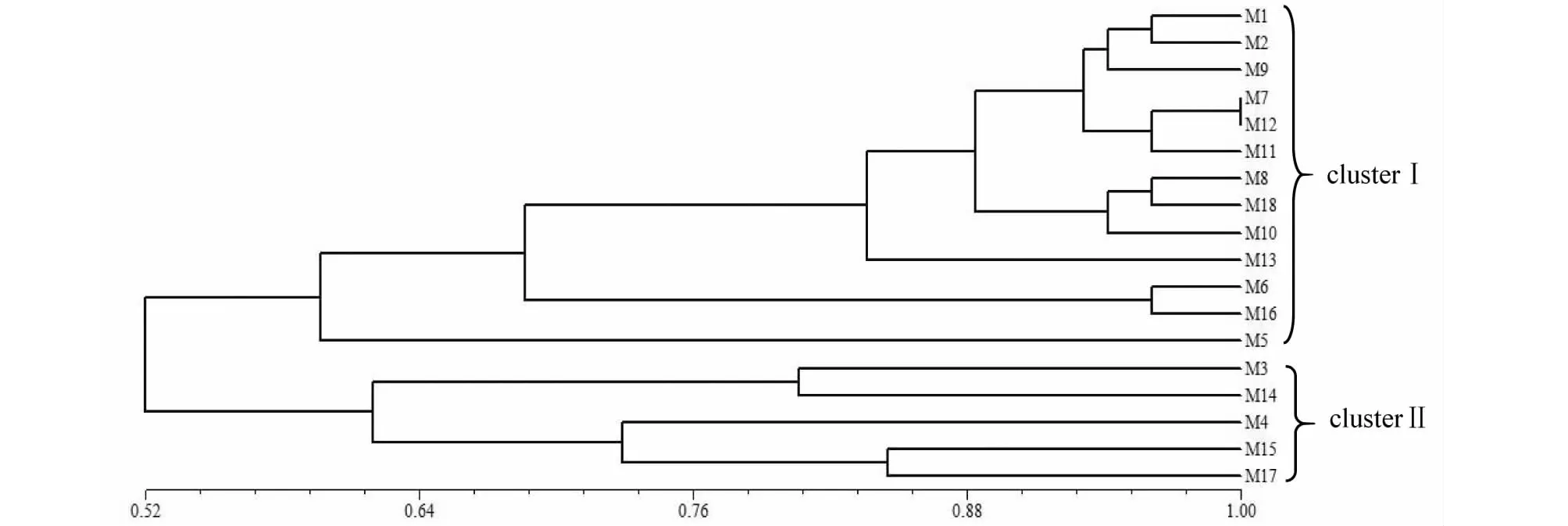
图6 供试菌株ISSR聚类分析Fig.6 Cluster analysis of tested strains based on ISSR
羊肚菌的生长易受到环境的影响,造成形态的多样性,因此传统的形态学分类法不能有效区分,分子生物学的发展,推动了分子标记法在菌株鉴定上的应用。闫晓雪等[23]基于ITS序列,对西南地区部分野生羊肚菌鉴定,将供试菌株分为7个种类。本项研究采用ITS分子标记手段结合形态特征对羊肚菌进行鉴定,以确定分类地位。结果表明,8个人工栽培菌株和1个野生菌株鉴定为梯棱羊肚菌,2株人工菌株鉴定为六妹羊肚菌,2株人工栽培菌株和2株野生菌株鉴定为高羊肚菌,2株野生菌株鉴定为粗柄羊肚菌。目前的栽培技术,梯棱羊肚菌和六妹羊肚菌产量较高,已经商业化推广,因此,在收集野生菌株的时候,可以有目的的选择梯棱羊肚菌和六妹羊肚菌进行驯化,这样选育出商业化品种成功的可能性更大,丰富栽培品种,解决羊肚菌退化及老化问题,促使羊肚菌行业稳定发展。
近年来,由于全球气候变化太大,加之人们过渡采集及不合理采集野生羊肚菌,其产量正逐年下降,为使羊肚菌资源得到可持续发展,需充分了解其物种遗传多样性。物种多样性越丰富,对环境变化的适应能力越强,生存能力和进化潜力也就越大,进而也就不容易成为濒危物种[24]。ISSR分子标记技术广泛用于食用菌遗传多样性,不同的羊肚菌菌株的DNA序列存在差异,在ISSR-PCR 扩增过程中,DNA序列与引物结合,不同的菌株出现不同的ISSR条带,能反映出菌株间的遗传多样性。魏巍等[14]选择了16个羊肚菌菌株,应用ISSR-PCR技术讨论了遗传多样性。本文对18株羊肚菌采用ISSR分子标记方法,其供试菌株的电泳图谱具有明显的差异性,多态性丰富,重复性高。采用UPGMA法对羊肚菌聚类分析,结果表明相似系数小于0.52时,供试菌株聚为一类,相似系数为0.54时,聚为两大类,说明18株羊肚菌具有明显的遗传多样性,在9个人工栽培梯棱羊肚菌菌株中,其中M1和M17遗传差异最大,可能是由于它们生长条件、生长方式和生长阶段的不同导致,推测环境的影响对羊肚菌遗传的变化有很大关系。
4 小 结
综上所述,本文采用ITS和ISSR分子技术对18株羊肚菌进行系统发育和遗传多样性分析,结果表明,供试菌株鉴定为梯棱羊肚菌、六妹羊肚菌、高羊肚菌、粗柄羊肚菌,18个菌株具有明显的遗传多样性。我国蕴含丰富的羊肚菌种质资源,可为人工驯化栽培和菌种选育提供大量素材,有利于羊肚菌产业长远发展。
