南海虾酱中可培养细菌的遗传多样性分析
2018-11-15陈永敢李珍邢增葵张来军黄海
陈永敢,李珍,邢增葵,张来军,黄海*
(1.海南热带海洋学院 生命科学与生态学院,海南 三亚 572022;2.海南省热带海洋渔业资源保护与利用重点实验室,海南 三亚 572022)
传统虾酱发酵将小虾经简单挑选并清洗后,加入适量食盐,拌匀浸没缸中,随后置于自然环境中日晒夜露,利用虾体内源或环境菌群的蛋白酶、糖化酶等催化,1个月左右可完成发酵[1]。发酵后的虾酱微红、细腻。由于传统虾酱发酵一般只将虾体进行清洗,一定程度上去除了部分菌群,但多数虾体上的微生物并没有去除[2,3]。此外,整个发酵过程并无特别除菌步骤,与外界环境密切接触,在一定程度上不可避免地使虾酱中有微生物生长。因此,环境差异造成了参与发酵菌群的差异,这些微生物生长必然产生大量代谢产物,从而影响风味[4]。然而以我国南海小型虾制作的虾酱中存在何种可培养的细菌至今鲜有报道。本研究收集我国南海虾酱,分离可培养细菌并纯化,提取基因组DNA,扩增16S rDNA片段,构建系统发育树,分析不同菌株间的遗传关系,为了解我国南海虾酱中可培养微生物的类群提供了参考信息。
1 材料与方法
1.1 材料与试剂
乙醇、氯化钠等:均为国产分析纯。2016年2月~2018年2月收集了6份虾酱样品,分别来自海南乐东(HL1801,YGH1601)、海南三亚(SY1601)、海南定安(DA1601)、海南文昌(WC1601)、广东台山(GD1601)。
1.2 主要仪器
电热恒温鼓风干燥箱 宁波江南仪器厂;无菌超净工作台 苏州安泰空气技术有限公司。
1.3 菌株的分离及纯化保藏
虾酱做系列稀释,取0.1 mL涂布于牛肉膏蛋白胨培养基,菌落计数。每个样品设3个平行,以平均值±标准差表示,SPSS 20.0软件用Duncan检验判定组间差异,以p<0.05为差异有统计学意义。菌落划线纯化,重复3次。
1.4 基因组DNA提取、PCR扩增及测序
基因组DNA的提取及目的片段扩增参照Chen等的方法[5]。采用引物A8-27f和B1523-1504r扩增16S rDNA片段[6],PCR产物委托北京华大基因科技股份有限公司测序。
1.5 系统发育学分析
根据菌株16S rDNA的测序结果,进行Blast比对,搜索同源序列。选取不同种的代表性菌株进行分析,采用ClustalX 1.83将序列对齐,利用MEGA 5.05软件进行系统发育分析,并以自展法(Bootstrap)进行检测,循环1000次,采用邻接法、最大简约法构建系统发育树[7,8]。
2 结果与分析
2.1 南海虾酱中可培养细菌的分离与纯化
6种虾酱以淡紫色或淡黄色为主,呈稠糊状,含细颗粒。利用这6种虾酱进行稀释涂布,获得每种虾酱中可培养细菌的含菌量,其中样品HL1801含菌量最多,而样品WC1601,GD1601含菌量最少(见表1)。此外,从每种虾酱中都分离并纯化得到了5株可培养细菌,菌落主要呈白色、乳白色、黄色、红色,透明或不透明(见图1)。这些结果都反映了不同地区虾酱发酵工艺有别,加之发酵时间长短不同,最终造成了虾酱中所含的细菌数量及形态存在差异。

表1 虾酱的形态、可培养细菌的分离及纯化Table 1 The morphological characteristics, isolation and purification of culturable bacteria of shrimp paste

图1 部分虾酱的形态、可培养细菌的分离及纯化Fig.1 The morphological characteristics, isolation and purification of culturable bacteria of shrimp paste
注:A为HL1801;B为HL1801的菌株分离;C为菌株HL1801B5的纯化;D为DA1601;E为DA1601的菌株分离;F为菌株DA1601B4的纯化。
2.2 分离菌株的遗传多样性分析
随机选取18株分离菌株,提取基因组DNA,扩增16S rDNA片段并测序。将序列在NCBI数据库中进行比对,对测得菌株的序列在GenBank数据库中进行Blast搜索同源序列,所得结果表明本研究中测得的序列均与Bacillus属菌株序列相似度最高,挑取部分代表性菌株序列用于构建系统发育树。由邻接树可以看出,18株菌株在聚类上分成3个类群。分离自海南三亚虾酱样品的3株菌株SY1601B1,SY1601B4,SY1601B5与B.amyloliquefaciens,B.siamensis及B.methylotrophicus这3个种亲缘关系较近,自展值为86(见图2中A)。来自海南文昌和海南莺歌海的虾酱样品,虽然在产地上位于海南岛的东北部和西南部,相距较远,但从这2个样品中分离到的菌株WC1601B2,YGH1601B4与B.megaterium,B.aryabhattai聚类,这反映了虾酱中微生物的种类可能不仅与发酵所处的环境相关,发酵工艺也是影响其组成的重要因素。其他13株来自海南定安、广东台山、海南三亚、海南黄流、海南莺歌海、海南文昌的菌株则与B.toyonensis,B.thuringiensis,B.anthracis及B.cereus聚为一类。事实上,这些菌株来自6个不同地区,在发酵过程中虾酱接触的环境差别较大,但分离到的菌株亲缘关系较近,再次反映了相似的发酵工艺可能造成虾酱中存在亲缘关系较近的菌群。
根据16S rDNA序列构建的最大简约树与邻接树结构相似,18株菌株聚为3个不同的类群(见图2中B)。Han等[9]研究表明虾酱中存在丰富的微生物菌群,其中以Staphylococcusequorum,Halanaerobiumsaccharolyticum,Salimicrobiumluteum及Halomonasjeotgali为主要类群。Jung等[10]在另一项研究中发现,发酵初期虾酱中优势菌群为变形菌门,然而很快就被Pseudoalteromonas,Staphylococcus,Salimicrobium和Alkalibacillus等厚壁菌门取代。然而本研究及系列相关工作中并未分离到这些菌株,这不仅体现了同一虾酱产品中存在多种微生物菌群,也反映了不同来源的虾酱,其微生物菌群差异显著,可能是造成最终风味差异的重要原因之一。
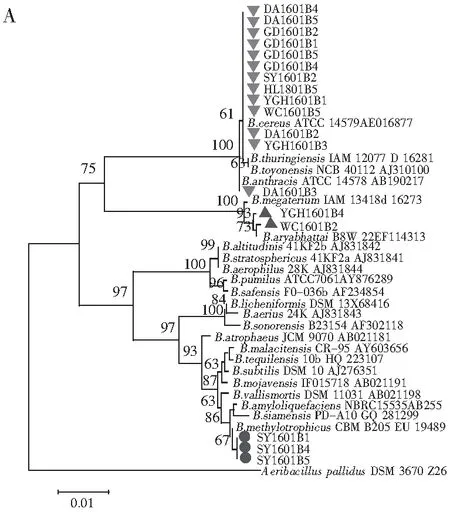
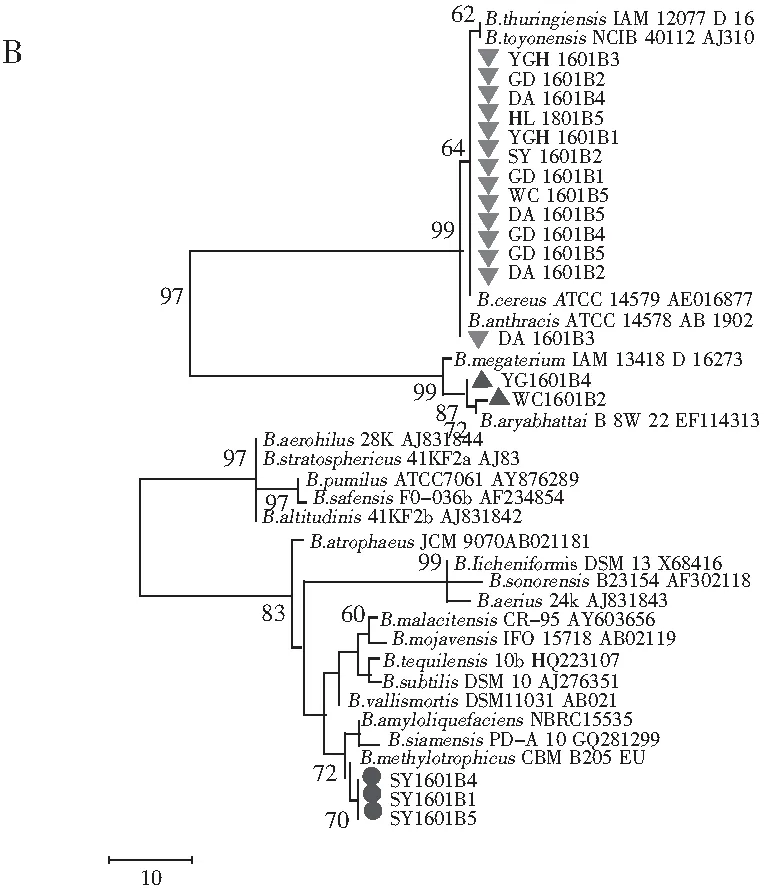
图2 根据16S rDNA序列构建的系统发育树Fig.2 Phylogenetic trees based on 16S rDNA
注:1000次重复,自展值(Bootstrap)>60%;A为邻接树;B为最大简约树,步长=181;一致性指数(CI)=0.769;总留存指数(RI)=0.970。
3 结论
本研究对6种南海虾酱中可培养的细菌进行分离,通过测定16S rDNA序列并构建系统发育树,结果表明虾酱中可培养细菌分为3个不同的类群,反映了我国南海虾酱中可培养细菌遗传多样性丰富。由于制作工艺差异、发酵环境差异造成参与发酵的微生物类群存在多样性,最终导致产品品质不同。
