浙江及周边地区猪流行性腹泻病毒S基因分子特征分析
2018-11-10单颖刘子琦施杏芬李国炜陈聪罗浩刘亚杰方维焕李肖梁
单颖,刘子琦,施杏芬,李国炜,陈聪,罗浩,刘亚杰,方维焕,李肖梁*
(1.浙江大学动物科学学院,浙江省动物预防医学重点实验室,杭州310058;2.浙江省畜产品质量安全检测中心,杭州310020)
猪流行性腹泻(porcine epidemic diarrhea,PED)是由猪流行性腹泻病毒(porcine epidemic diarrhea virus,PEDV)引起的高度接触性肠道传染病,以呕吐、腹泻、脱水等为主要特征,多发于秋冬季节,主要感染哺乳仔猪,其中7日龄以下的哺乳仔猪死亡率最高[1]。1978年,英国、比利时首次发现该病[2]。近年来,PEDV在包括中国、韩国、日本等国家在内的亚洲地区广泛流行[3-5],给养猪业带来巨大经济损失。PEDV在浙江省的临床检出率由2010年的42.86%提高到了2013年的70%以上[6]。
PEDV是套式病毒目(Nidovirales)冠状病毒科(Coronaviridae)冠状病毒属(Coronavirus)的成员,为有囊膜的、不分节段的、单股正链RNA病毒,其基因组全长约28 kb,包括5′端非编码区、3′端非编码区及至少7个开放阅读框(open reading frame,ORF)[7-8]。其中5个开放阅读框编码主要的结构蛋白,包括纤突(spike,S)蛋白、ORF3、小膜(envelope,E)蛋白、膜(membrane,M)蛋白、核衣壳(nucleocapsid,N)蛋白[9]。PEDV的S蛋白是主要的结构蛋白,属于Ⅰ型膜蛋白,含有主要的抗原决定簇,约由1 383个氨基酸组成,与其他冠状病毒属成员的S蛋白结构相似,包含3个结构域:较长的胞外结构域、跨膜结构域及较短的胞内结构域。根据其他冠状病毒S蛋白保守的基序,可以将PEDV的S蛋白分为S1区(第1~789位氨基酸)和S2区(第790~1 383位氨基酸)[10]。研究表明,PEDV的S蛋白对被感染宿主中和抗体的产生、特异性受体的结合及细胞膜融合方面具有重要的生物学意义,S1可识别受体,S2负责病毒囊膜与宿主细胞膜融合[11]。S蛋白中的4个中和表位区(499~638、748~755、764~771和1 368~1 374)均已被鉴别[12-14]。由于受到宿主免疫选择的压力,S蛋白易发生变异,因此,S蛋白对于了解PEDV的流行现状、分子多样性、基因突变的联系及疫苗的研发也有很重要的作用。
为了解现阶段PEDV流行毒株的分子演化特征,本研究采集了2013年4月到2017年4月间浙江省及周边地区仔猪腹泻样品,对引起腹泻的主要病原进行了检测分析,同时对其中16株临床分离PEDV毒株的S基因进行了克隆和分子特征分析。研究结果丰富了PEDV分子流行病学资料,同时为疫病准确诊断和有效防控提供了基础数据。
1 材料与方法
1.1 病料采集和处理
采集浙江省及周边24个地区共282份病料,对其进行PEDV等病原的检测。在小肠组织及粪便样品中加入适量灭菌的0.01 mol/L磷酸盐缓冲液(pH 7.4)并研磨,置于-80℃冰箱中反复冻融2次,4℃、5 000 r/min离心10 min,取上清液。将上清液经0.45 μm滤膜过滤后,保存于-80℃冰箱中,备用。
1.2 主要试剂
总RNA提取试剂盒购自天根生化科技(北京)有限公司;MMLV购自美国Promega公司;pMD18-T载体、RNA酶抑制剂(HPRI)、dNTP、随机引物、限制性内切酶、DNA分子质量标志物(DL2000、DL5000)等均购自日本TaKaRa公司;琼脂糖购自美国Sigma公司;胶回收试剂盒购自杭州新景生物试剂开发公司。
1.3 聚合酶链式反应(polymerase chain reaction,PCR)引物设计
参考GenBank中公布的PEDV、猪传染性胃肠炎病毒(porcine transmissible gastroenteritis virus,TGEV)、猪δ冠状病毒(porcine deltacoronavirus,PDCoV)和轮状病毒A型(porcine group A rotavirus,GARV)全基因序列,利用Geneious 7.1.7软件设计病原检测和S基因克隆所用引物(表1),其中用3个重叠套式PCR扩增S基因全长。引物均由上海华大基因科技股份有限公司合成。
1.4 病毒RNA的抽提和反转录
取-80℃保存备用的病料上清液,按总RNA提取试剂盒的操作说明书提取病毒总RNA,核酸经NanoDrop 1000分光光度计检测,质量浓度为1 800~2 500 ng/μL,D(260 nm)∶D(280 nm)=1.95~2.05,保存于-80℃冰箱中。
反转录步骤简述如下:1 μg总RNA与1 μL随机引物混匀,用去核酸酶水定容至10 μL,混匀后适当离心,65℃温浴5 min后,立即放入冰水中冰浴5 min,适当离心后,加入8 μL 5×RT缓冲液、4 μL 2.5 mmol/L dNTP、0.5 μL RNA 酶抑制剂、0.5 μL MMLV反转录酶,用去核酸酶水定容至40 μL,充分混匀后,在42℃条件下作用1 h,95℃灭活10 min,获得的cDNA保存于-20℃冰箱中,备用。
1.5 检测病原的反应体系和条件
对PEDV、PDCoV和TGEV均采用一步法TaqMan探针检测,按 LightCycler®multiplex RNA virus master试剂盒(购自上海罗氏有限公司)说明书进行反应。反应体系为:RT-PCR反应混合物4.0 μL,PEDV-RTF/RTR或TGEV-F/T或PDCoV-RTF/RTR 各 0.4 μ L,PEDV-RTP 或 TGEV-Probe或PDCoV-RTP 0.1 μL,反转录酶缓冲液0.1 μL,RNA模板5.0 μL,ddH2O 5.0 μL。反应条件为:50 ℃反转录10 min;95 ℃变性30 s;95 ℃扩增5 s,60 ℃扩增40 s,30个循环。扩增循环阈值(CT)小于30为阳性,30~35为可疑,大于35为阴性。
对GARV进行RT-PCR检测,反应体系简述如下:2×Easy Taq mix酶10.0 μL,GARV-VP6-F1/R1各1.0 μL,cDNA1.0 μL,ddH2O 7.0 μL。反应条件为:95 ℃变性5min;95℃扩增30s,55℃扩增30s,72℃扩增40s,30个循环;72℃延伸1 min。PCR产物用1%琼脂糖凝胶电泳观察,预期片段大小为309bp。
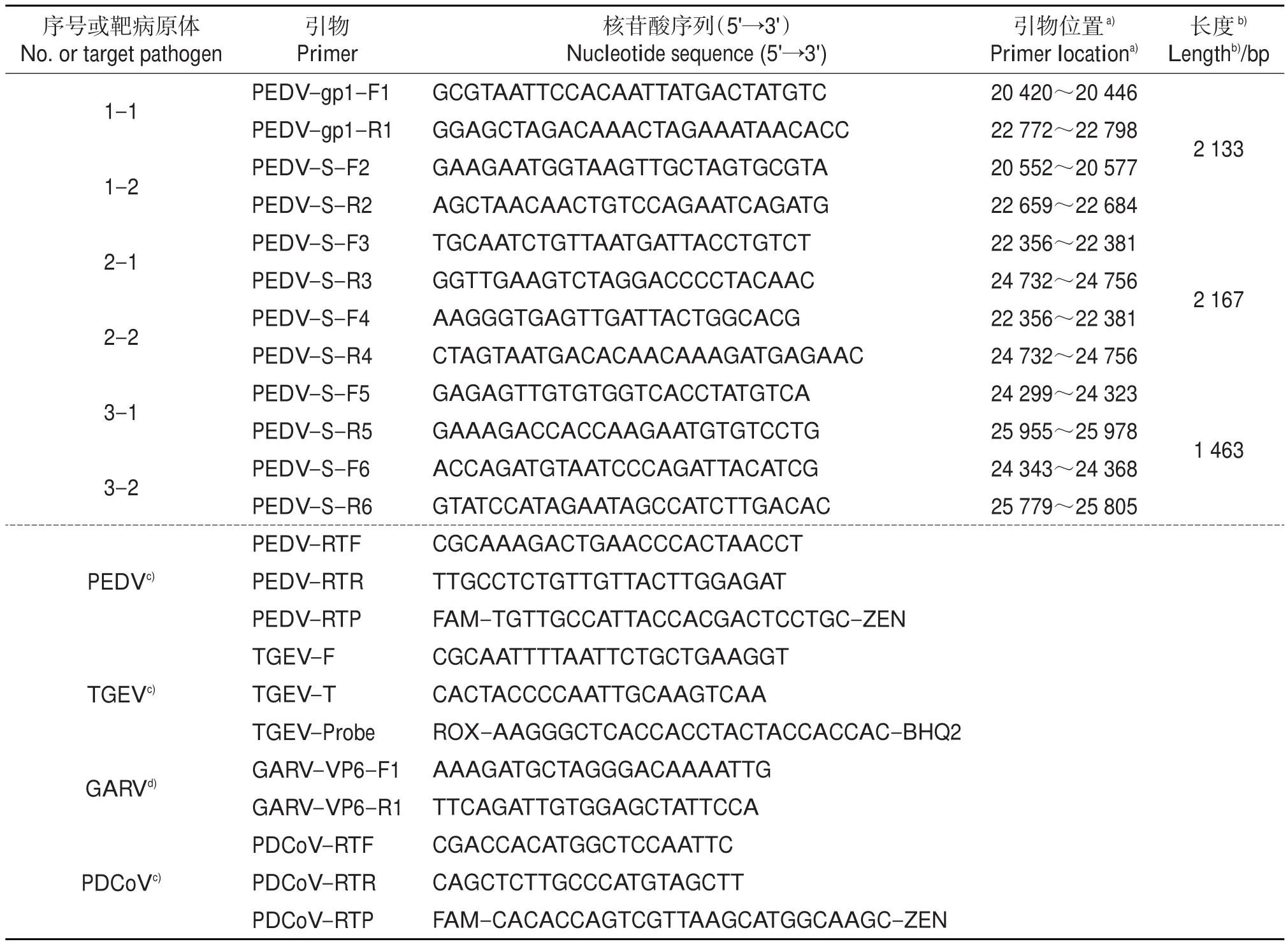
表1 病原检测和序列克隆所用引物Table 1 Primers for pathogens detecting and sequences cloning
1.6 PEDV的S基因PCR扩增
第1轮PCR扩增体系:cDNA模板2.0 μL,10×缓 冲 液 5.0 μL,10 μmol/L PEDV-gp1-F1/R1 或PEDV-S-F3/R3或PEDV-S-F5/R5上下游引物各1.0 μL(表1),10 mmol/L dNTP 2.0 μL,PrimerSTAR DNA聚合酶0.5 μL,ddH2O 39.5 μL。第2轮PCR扩增体系:模板(第1轮PCR产物)2.0 μL,10 μmol/L PEDV-gp1-F2/R2或PEDV-S-F4/R4或PEDV-SF6/R6上下游引物(表1)各1.0 μL,其他试剂与剂量同第1轮PCR反应。
2轮反应条件均为:98℃变性2 min;98℃扩增10 s,58 ℃扩增50 s,72 ℃扩增3 min,30个循环;72℃延伸10 min。
1.7 PEDV的S基因克隆、测序和拼接
对PCR产物进行1%琼脂糖凝胶电泳,回收获得目的片段,将纯化产物直接送至上海华大基因科技股份有限公司测序。采用DNAStar软件中的SeqMan进行序列拼接。
1.8 序列分析
采用DNAStar软件中的MegAlign及DNAman软件对获得的16份PEDV阳性样品和我国其他地区的分离株、国外分离株及PEDV疫苗株CV777等(附表1,http://www.zjujournals.com/agr/CN/10.3785/j.issn.1008-9209.2017.06.231)的S基因进行核苷酸及氨基酸序列同源性分析;采用MEGA 5.0中的邻接法(neighbor-joining,NJ)进行系统发育分析[15]。使用SignalP 4.0软件对PEDV的S蛋白信号肽进行预测[16]。使用在线软件(http://www.cbs.dtu.dk/services/NetNGlyc/)进行糖基化位点分析,采用DNAStar软件中的Protean分析抗原指数。
2 结果与分析
2.1 浙江省及周边地区生猪腹泻物中主要病原检测
对浙江省及周边24个地区的282份病料进行PEDV、GARV、TGEV及PDCoV等病原的检测,结果(表2)显示:PEDV、GARV及PDCoV的检出率分别为64.89%(183/282)、0.35%(1/282)和4.25%(12/282),而未检出TGEV;PEDV在11月1日至次年4月1日的检出率为74%,其余时间段的检出率为26%(数据未列出)。

表2 浙江省及周边地区生猪腹泻物中主要病原检测Table 2 Investigation on main pathogens of pig diarrhea in Zhejiang Province and surrounding areas
2.2 PEDV的S基因克隆和序列分析
选取其中16份PEDV阳性样品(包括2013年的4份、2014年的4份、2015年的3份、2016年的3份和2017年的2份),进行S基因克隆和测序。除ZJ13SX1101和YN170101样本扩增获得的S基因全长为4 158 bp,编码1 386个氨基酸外,其他14株S基因的全长均为4 161 bp,编码1 387个氨基酸(图1)。与经典毒株CV777(JN599150.1)S蛋白相比,16份PEDV阳性样品的S蛋白存在突变,分别为27QSTI30→SANT、56MN57→GE、62S→N、64S→T、68GGIETD74→AGQHPT、84Y→H、86DS87→RG、89Q→H、120I→T、131N→I、120I→T、150S→F、159D→S、161KNI163→EHS、178A→S、178A→S、185I→F、196R→K、210T→E、227Y→S、229E→Q、237S→I、246DS247→EP、287W→L、328F→S、367VTE369→GAT、382K→N、442V→I、637Q→E、728N→S、768L→S、778M→T、810V→A、977H→Y、1048S→A、1055I→V、1171D→A、1177GD1178→DE、1204L→F、1302R→Q、1363G→C;插入58KQGV61、140N(除ZJ13SX1101外);缺失160G;这些突变、插入或缺失主要集中在S1蛋白片段(21~790 aa)。在PEDV疫苗株CV777的S蛋白已鉴别的4个中和表位(COE:499~638;SS2:748-755;SS6:764~771和2C10:1 368~1 374)中,所获阳性样品在其中有2个中和表位(SS2:748~755和2C10:1 368~1 374)与疫苗株序列完全一致。具体见附图1,http://www.zjujournals.com/agr/CN/10.3785/j.issn.1008-9209.2017.06.231。

图1 16份PEDV临床阳性样品的部分S基因同源性比对结果Fig.1 Homology comparison result of partial S gene sequences from 16 clinical PEDV positive samples
2.3 PEDV的S蛋白分子演化分析
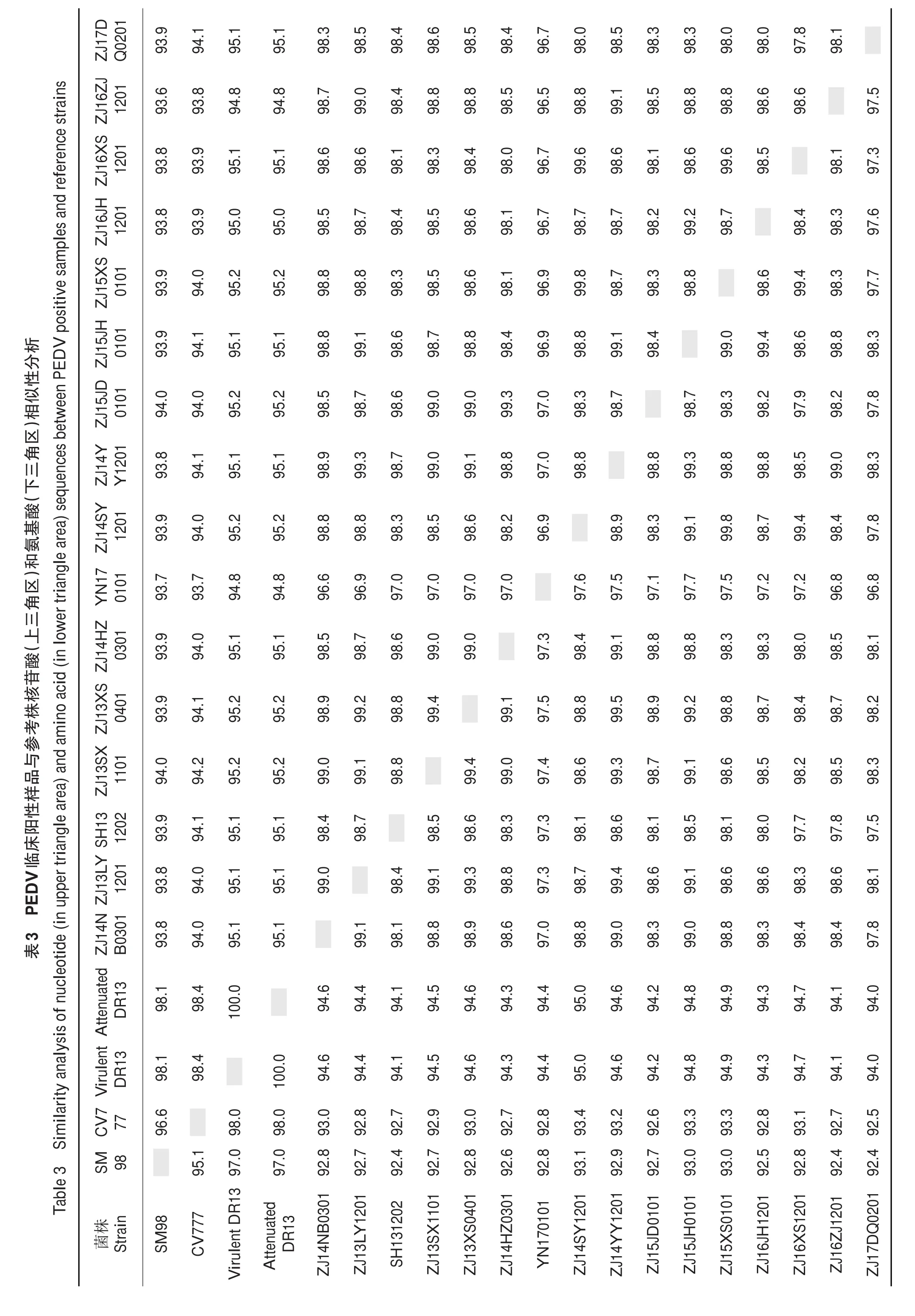
与疫苗毒株CV777、SM98和DR13相比,16份阳性样品的核苷酸同源性为93.6%~95.2%,氨基酸的同源性为92.4%~94.9%(表3)。以PEDV的S蛋白进行分子演化分析表明:PEDV可分成3个群,疫苗株(包括CV777、SM98和DR13)均位于Ⅱa亚群,2012年后的大部分PEDV临床分离株均位于Ⅲ群,但其中分别有1株2014年韩国分离株(KM403158.1)、1株2014年越南分离株(KP455320)和1株2015年比利时分离株(KR003452)位于Ⅱb亚群;分离株也不存在地理分布的差异性(图2)。
2.4 PEDV的S蛋白糖基化位点分析
16份PEDV阳性样品、CV777(JN599150.1,1994,中国)和CV777(AF353511.1,2001,瑞士)的S基因潜在的“Asn-X-Ser/Thr”(X为除Pro外的任何氨基酸)和Asn的N-连接糖基化位点分析如图3所示。结果表明:在这18个序列中,糖基化位点的数量分别为28个或29个,其中,ZJ13LY1201、ZJ13XS0401、ZJ14NB0301、ZJ14YY1201、ZJ14SY1201、ZJ15JH0101、ZJ15XS0101、ZJ16XS1201、ZJ16ZJ1201的阳性样品及疫苗株CV777(JN599150.1,1994,中国)含有28个潜在的N-糖基化位点,但是流行株与疫苗株潜在糖基化位点的位置存在一定的差异;其余阳性样品及疫苗株CV777(AF353511.1,2001,瑞士)均存在29个潜在的N-糖基化位点,且存在的潜在N-糖基化位点都一样。16份阳性样品与疫苗株CV777(JN599150.1,1994,中国)相比,形成了3个新的N-糖基化位点,分别为第58位的S→N、第116位的I→T、第1193位的T→N;另外,第232位的S→I氨基酸突变则破坏了1个疫苗株原有的N-糖基化位点。
3 讨论
本研究收集了浙江省及周边24个地区共282份病料,并对其进行了PEDV、GARV、PDCoV和TGEV等病原的检测,其中PEDV的检出率达到64.89%(183/282),感染率较高,但大部分猪场都使用了TGEV-PEDV二联疫苗(基于CV777),说明疫苗不能为现有的流行毒株提供有效的免疫保护。因此,对PEDV流行毒株的主要抗原基因的分子流行动态进行监测,在预防和控制猪流行性腹泻中具有重要的意义。
与猪传染性胃肠炎流行的特征类似,猪流行性腹泻的发生也呈现一定的季节性。浙江省及周边地区PEDV检出率在11月到次年4月期间比其余时间段高出48%。这可能是由于浙江省及周边地区在这一时间段内天气比较寒冷、昼夜温差较大。研究表明,冠状病毒在低温条件下比暴露于高热或阳光条件下更稳定[17],因此,寒冷的天气可能有利于PEDV的存活。
?

图2 用邻接法构建的基于S蛋白的PEDV分子进化树Fig.2 PEDV molecular evolutionary trees constructed by neighbor-joining method based on S protein
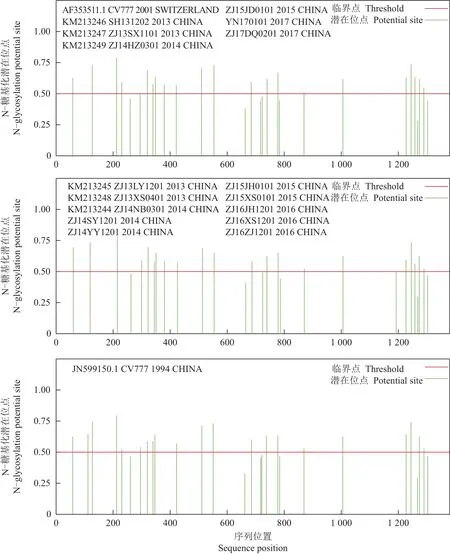
图3 PEDV的S蛋白序列中Asn-X-Ser/Thr(X为除Pro外的任何氨基酸)和Asn的N-糖基化位点预测Fig.3 Prediction ofAsn-X-Ser/Thr(X is any amino acid except Pro)andAsn N-glycosylation sites within PEDV S protein
猪流行性腹泻病毒的S蛋白被认为具有较高的遗传变异性[18],不仅免疫原性良好[19-20],而且在感染宿主体内介导中和抗体产生的过程中起着重要作用[12,21]。针对S基因全长进行的遗传变异分析,可将PEDV分为3个群[5]。对临床分离毒株及疫苗株的S基因进行分析发现,疫苗株均位于Ⅱa亚群,而2012年以后的大部分PEDV分离株均位于Ⅲ群,包括本次获得的16份PEDV阳性样品,这些浙江及周边地区的阳性样品与中国常用疫苗株CV777、2010年韩国疫苗株SM98、2009和2011年韩国疫苗株强弱毒株DR13亲缘关系较远。PEDV新的变异株也无明显的地理分布差异,这也部分解释了基于CV777或SM98等经典毒株的现有疫苗不能很好地控制当前PEDV变异株的流行。至于S基因的变异是否能导致PEDV毒力增强,还有待于进一步研究。S蛋白为Ⅰ型跨膜糖蛋白,富含N-糖基化修饰位点。尽管流行株与中国常用的PEDV CV777疫苗株大部分预测的糖基化位点一致,但流行株由于突变产生了3个新的N-糖基化位点并破坏了1个糖基化位点。流行毒株与CV777(JN599150.1,1994,中国)疫苗毒株之间N-糖基化位点的变化是否会影响病毒本身的致病性及抗原性,值得进一步探究。
4 结论
综上所述,PEDV是当前引起仔猪腹泻的主要病原,且在冬季的发生率远远高于其他季节,呈现一定的季节性。虽然迄今为止PEDV只有1个血清型,但是不同毒株之间基因差异较大[22-24]。对浙江省及周边地区的16份PEDV阳性样品及参考株S基因的分析表明,目前在我国广泛流行的PEDV毒株与以往的毒株相比已经发生了较为明显的变异,这可能也是免疫失败的重要原因。因此,防控猪流行性腹泻需要注意季节的波动,也需要加快流行株疫苗的开发。
参考文献(References):
[1] JUNG K,SAIF L J.Porcine epidemic diarrhea virus infection:Etiology,epidemiology,pathogenesis and immunoprophylaxis.The Veterinary Journal,2015,204(2):134-143.
[2] PENSAERT M B,DE BOUCK P.A new coronavirus-like particle associated with diarrhea in swine.Archives of Virology,1978,58(3):243-247.
[3]KUSANAGI K I,KUWAHARA H,KATOH T,et al.Isolation and serialpropagation ofporcine epidemic diarrhea virus in cell cultures and partial characterization of the isolate.Journal of Veterinary Medical Science,1992,54(2):313-318.
[4] PARK S J,KIM H K,MOON H J,et al.Molecular detection of porcine kobuviruses in pigs in Korea and their association with diarrhea.Archives of Virology,2010,155(11):1803-1811.
[5]PURANAVEJA S,POOLPERM P,LERTWATCHA RASA RAKUL P,et al.Chinese-like strain of porcine epidemic diarrhea virus,Thailand.Emerging Infectious Diseases,2009,15(7):1112.
[6] 杜晓莉,王一成,吴润,等.2010—2013年浙江省猪流行性腹泻病毒临床检测及PEDV-S基因型分析.浙江农业学报,2014,26(3):581-587.DU X L,WANG Y C,WU R,et al.Genotyping and clinical detection of porcine epidemic diarrhea virus during 2010—2013 in Zhejiang Province.Acta Agriculturae Zhejiangensis,2014,26(3):581-587.(in Chinese with English Abstract)
[7]BRIDGEN A,DUARTE M,TOBLER K,et al.Sequence determination of the nucleocapsid protein gene of the porcine epidemic diarrhoea virus confirms that this virus is a coronavirus related to human coronavirus 229E and porcine transmissible gastroenteritisvirus.Journalof General Virology,1993,74(9):1795-1804.
[8] SONG D,PARK B.Porcine epidemic diarrhoea virus:A comprehensive review of molecular epidemiology,diagnosis,and vaccines.Virus Genes,2012,44(2):167-175.
[9] LEE C.Porcine epidemic diarrhea virus:An emerging and re-emerging epizootic swine virus.Virology Journal,2015,12(1):193.
[10]SUN D B,FENG L,SHI H Y,et al.Spike protein region(aa 636789)of porcine epidemic diarrhea virus is essential for induction of neutralizing antibodies.Acta Virologica,2007,51(3):149-156.
[11]DUARTE M,LAUDE H.Sequence of the spike protein of the porcine epidemic diarrhoea virus.Journal of General Virology,1994,75(5):1195-1200.
[12]CHANG S H,BAE J L,KANG T J,et al.Identification of the epitope region capable of inducing neutralizing antibodies against the porcine epidemic diarrhea virus.Molecules and Cells,2002,14(2):295-299.
[13]SUN D B,FENG L,SHI H Y,et al.Identification of two novel B cell epitopes on porcine epidemic diarrhea virus spike protein.Veterinary Microbiology,2008,131(1/2):73-81.
[14]GODET M,GROSCLAUDE J,DELMAS B,et al.Major receptor-binding and neutralization determinants are located within the same domain of the transmissible gastroenteritis virus(coronavirus)spike protein.Journal of Virology,1994,68(12):8008-8016.
[15]TAMURA K,PETERSON D,PETERSON N,et al.MEGA 5:Molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods.Molecular Biology and Evolution,2011,28(10):2731-2739.
[16]PETERSEN T N,BRUNAK S,VON HEIJNE G,et al.SignalP 4.0:Discriminating signal peptides from transmembrane regions.Nature Methods,2011,8(10):785-786.
[17]CASANOVAL M,JEON S,RUTALAWA,et al.Effects of air temperature and relative humidity on coronavirus survival on surfaces.Applied and Environmental Microbiology,2010,76(9):2712-2717.
[18]CAVANAGH D,DAVIS P J,COOK J K A,et al.Location of the amino acid differences in the S1 spike glycoprotein subunit of closely related serotypes of infectious bronchitis virus.Avian Pathology,1992,21(1):33-43.
[19]OH J,LEE K W,CHOI H W,et al.Immunogenicity and protective efficacy of recombinant S1 domain of the porcine epidemic diarrhea virus spike protein.Archives of Virology,2014,159(11):2977-2987.
[20]HUY N X,KIM S H,YANG M S,et al.Immunogenicity of a neutralizing epitope from porcine epidemic diarrhea virus:M cell targeting ligand fusion protein expressed in transgenic rice calli.Plant Cell Reports,2012,31(10):1933-1942.
[21]CRUZ D J M,KIM C J,SHIN H J.The GPRLQPY motif located at the carboxy-terminal of the spike protein induces antibodies that neutralize porcine epidemic diarrhea virus.Virus Research,2008,132(1/2):192-196.
[22]KUBOTA S,SASAKI O,AMIMOTO K,et al.Detection of porcine epidemic diarrhea virus using polymerase chain reaction and comparison of the nucleocapsid protein genes among strains of the virus.Journal of Veterinary Medical Science,1999,61(7):827-830.
[23]BRIDGEN A,KOCHERHANS R,TOBLER K,et al.Furtheranalysisofthe genomeofporcineepidemic diarrhoea virus//Coronaviruses and Arteriviruses.Boston,USA:Springer,1998:781-786.
[24]YEO S G,HERNANDEZ M,KRELL P J,et al.Cloning and sequence analysis of the spike gene of porcine epidemic diarrhea virus Chinju99.Virus Genes,2003,26(3):239-246.
