发卡型万能传导在基于核酸分子线路的基因诊断中的应用和性能优化
2018-11-01唐艺丹刘一辰吕佰阳郭路路李冰凌
唐艺丹 刘一辰 吕佰阳 郭路路 李冰凌


摘 要 核酸等温扩增技术作为核酸体外扩增技术,其反应过程始终维持在恒定温度下。与聚合酶链反应相比,核酸等温扩增反应具有优异的便携型,因而被视为最有望实现基因快检甚至即时快检的体外基因扩增方法。然而,由于反应过程中假阳性扩增频发、反应后对产物的检测方法缺乏特异性和灵敏度等缺点,限制了其在实际分析检测中的应用。通过构建发卡型结构万能中转探针,成功地将恒温扩增产物转到一套性能良好的已知核酸分子线路上;借助核酸分子线路的百倍放大性能和序列特异性,实现对上游基因序列信息的精准识别和放大信号输出。针对不同的待测序列,仅需改变发卡型中转探针的序列,即可实现对不同序列目标物的检测。基于中转探针的重要性,本研究对中转探针的设计原理和方法进行了重点阐述,提出并验证了一套行之有效的普适性设计规律,确保中转探针良好的中转效率(信噪比)。利用这一规律获得的中转探针,与核酸分子线路偶联,可成功为低至近单分子(20个拷贝)的模型基因提供显著荧光和电化学信号输出。
关键词 等温扩增; 万能中转; 发卡型中转探针; 核酸分子线路; 催化发卡型结构自组装; 荧光; 电化学
1 引 言
基因诊断是应用分子生物学技术对病患特定基因片段进行定量或定性分析的检测方法。由于病原体基因含量通常极低,因此對基因片段进行高效扩增,是对其进行识别和分析的首要步骤。目前,以聚合酶链反应(PCR)为代表的热循环核酸扩增反应和一系列恒温扩增反应,被认为是功能最为强大的核酸扩增体系。恒温扩增反应包括滚环放大(RCA)[1]、自主序列复制扩增(3SR)[2]、依赖于核酸序列扩增(NASBA)[3]、链替换扩增(SDA)[4]、环介导等温扩增(LAMP)[5]、解旋酶替换扩增(HAD)[6]、重组酶复制扩增(RPA)[7]等。在这一系列等温扩增技术中,日本Eiken Chemical公司于2000年发明了LAMP技术[5](图1,STEP I),使用一种具有链置换特性的Bst DNA聚合酶和4条特征性引物,可在恒定温度((65±5)℃)下实现对双链DNA、单链DNA或RNA模板序列的109~1010倍扩增。因此,该技术自发明以来一直被视为PCR最有力的竞争者和最具即时诊断(POC)兼容性的恒温扩增技术。尽管如此,LAMP的实际应用依然受到“可靠性低”的严重制约[8~13]。原因是在恒温条件下,核酸扩增反应失败率(假阴性)和非特异性(假阳性)激增;而目前普遍使用的表征方法(如凝胶电泳、荧光染色、共沉淀、双引物标记比色法等)仅能识别碱基对的增加,并不能有效辨别正确的扩增序列,易发生误诊和漏诊。这也是目前阻碍各种恒温扩增技术广泛应用的主要因素。
无酶核酸分子线路是通过控制核酸链交换反应的动力学性质而建立的核酸分子工程新方向[14~18],该理论证明单个或多个单链DNA输入存在下可引发一系列并联或串联的链交换反应,实现线性或指数循环放大效果,且整个过程不需要任何蛋白质辅助。最具代表性的无酶分子线路包括杂交链反应(Hybridization chain reaction,HCR)[19,20]。 熵驱动反应(Entropy driven reaction)[21]和催化发卡型结构自组装(Catalytic hairpin assembly,CHA)反应[22,23]。在CHA反应中,一条线性DNA输入(C1)通过两次核酸链交换依次打开并组装两条序列互补、但杂交受限的发卡型DNA(H1和H2);每完成一次组装,C1可自发脱离组装体(H1-H2),作为催化剂再次参与另一次组装过程[23]。相比于一步链交换,CHA明显的优势在于具有50~100倍放大功能;信噪比高,可程序化;对错配响应更加灵敏和特异;产物简单且带有单链,可灵活、简便地偶联到下游荧光、电化学和比色等常规检测器中[23]。基于上述独特优势,CHA已作为新一代的信号放大和传感元件被用于多种分析应用中[24~27]。
在前期工作中,本课题组已证明将CHA作为LAMP扩增反应与下游信号输出间的智能传导元件,能够克服上述LAMP假性信号频发的缺点和不足[12,28]。LAMP扩增后的环状单链产物一般含有25~35个碱基,可将其直接作为催化剂C1设计CHA的反应组分H1、H2和F-Q报告探针。CHA的特征性传导可以精准地识别待测序列,在完全排除假阳性误诊的前提下进一步放大信号幅度,降低假阴性检出率。然而,该方法在检测新的待测基因时需重新设计CHA组分和反应,过程繁琐复杂。由于大多数基因片段含有自折叠二级结构,使得CHA反应的高效性也难以保证。本研究以提高方法普遍适用性为目的,提出基于“发卡结构探针”的“万能传导”策略[12]。其主旨是将一套高效CHA反应的C1封闭在发卡型中转探针的茎部,使其无法诱导CHA反应;只有当中转探针被LAMP环状产物打开时,C1才被游离出来,开启有效的CHA反应。利用这种策略,检测方式由直接检测LAMP产物转变为检测一套高效CHA的C1;因此针对不同的待测序列,只需更换发卡型中转探针即可实现检测。虽然此方法的可行性已在前期工作中得到验证,但后续研究表明,发卡型探针的设计过程复杂且失败率高; 打开率低或封闭效率低的发卡探针会严重影响后续CHA对LAMP产物的检测结果,信噪比无法达到预期水平。因此,本研究将对发卡型探针的设计和优化方法进行详细讨论,总结出一套行之有效的设计规律,从而实现CHA对基因的超灵敏、超特异和超普适分析。经过优化,实现了对模型基因(大肠杆菌的malB基因片段)的超特异检测,检测限低至20个拷贝,完全满足实际需求。
实验中所使用的缓冲液包括:1 × TNaK 缓冲液:20 mmol/L Tris-HCl溶液(pH 7.5),含140 mmol/L NaCl、5 mmol/L KCl; 1 × 等温扩增缓冲液:20 mmol/L Tris-HCl溶液(pH 8.8),含10 mmol/L (NH4)2SO4、50 mmol/L KCl、2 mmol/L MgSO4、0.1% Tween 20;电解质缓冲溶液I-Buffer(pH 7.4):10 mmol/L Tris-HCl溶液、1 mmol/L EDTA、0.1 mol/L NaCl;实验用水为超纯水(四川沃特尔水处理设备有限公司超纯水机制备)。
2.2 实验方法
2.2.1 发卡型中转探针-CHA反应检测 不同浓度模拟LAMP产物(Target和Target 2)配制体系为20 mmol/L Tris-HCl(pH 8.8)、10 mmol/L (NH4)2SO4、50 mmol/L KCl、20 mmol/L MgSO4、0.1% Tween 20、甜菜碱终浓度为1 mol/L。取12 μL Target或Target 2加入22.2 nmol/L 中转探针(Transducer),退火5 min(即95℃孵育5℃后,以0.1℃/min 降温至37℃)。以1∶1∶1∶1的体积比加入Target-Transducer混合物、200 nmol/L H1、800 nmol/L H2、200 nmol/L报告基因(包括200 nmol/L F和400 nmol/L Q)。反应液中Mg2+终浓度为5 mmol/L。
2.2.2 LAMP等温扩增反应 以带有目标基因的质粒为模板进行扩增,25 μL LAMP反应体系为20 mmol/L Tris-HCl(pH 8.8)、10 mmol/L (NH4)2SO4、50 mmol/L KCl、4 mmol/L MgSO4、0.1% Tween 20,其中内引物FIP/BIP浓度为0.8 μmol/L,外引物F3/B3濃度为0.2 μmol/L,甜菜碱浓度为1 mol/L,dNTPs终浓度为0.4 mmol/L,将24 μL混合液退火5 min,最后加入1 μL(120 U)Bst 2.0 DNA聚合酶。反应温度为65℃,反应时间为90 min,最后80℃热处理20 min 终止反应。
2.2.3 LAMP-发卡型中转探针-CHA反应检测 在12 μL LAMP反应物中加入终浓度为20 nmol/L Transducer 3,95℃退火5 min,以1∶1∶1∶1的体积比加入LAMP扩增子-Transducer 3混合物、200 nmol/L H1、800 nmol/L H2、200 nmol/L报告基因(包括200 nmol/L F和400 nmol/L Q)。反应液中Mg2+终浓度为5 mmol/L。
2.2.4 电化学检测 电化学检测实验中所用电极为新型PEG(Plastic-gold electrode)芯片电极[29],三电极均为金电极的体系。电极以0.1 mol/L H2SO4清洗后,修饰HS-P巯基探针,并用MCH进行封闭。在12 μL LAMP反应物中加入终浓度为20 nmol/L Transducer 3,95℃退火5 min,以1∶1∶1∶1的体积比加入LAMP扩增子-Transducer 3混合物、800 nmol/L H1-MB、800 nmol/L H2和缓冲溶液。在37℃反应2 h后,将20 μL溶液滴在HS-P/MCH修饰的金电极表面,于37℃反应1 h后,用电解质缓冲溶液I-Buffer清洗电极,并在I-Buffer溶液中进行SWV电化学测量。
3 结果与讨论
3.1 CHA反应历程
如图1(STEP Ⅲ)所示,CHA反应基本组分为两个序列部分互补的发卡型DNA(简称H1和H2)。由于互补序列均被发卡结构封闭,因此二者反应动力学严重受阻,无法自发杂交形成双链结构。在加入引发剂C1(仅3-2-1部分)后,C1的区域1片段会作为立足点,识别H1的粘性末端1*,随即开启链迁移反应,打开H1,形成C1-H1杂交体。C1-H1中暴露出的区域3会作为立足点识别H2的粘性末端区域3*,进而引发第二次链迁移反应,形成C1-H1-H2中间产物。在该中间产物中,C1与H1只有8个碱基互补,因此无法稳定杂交[23,30],会自发从H1上脱落,生成反应终产物H1-H2杂交体。而脱落的C1则会再次识别未反应的H1,参与下一轮H1-H2自组装反应。为表征反应进程,H1-H2杂交体中暴露的区域2会与区域5-6一起再次开启另一次基于立足点的链迁移反应,将标记有淬灭集团的探针Q从标有荧光集团的F上置换掉,最终产生可以检测到的荧光增强信号。由反应历程可见,C1能否与H1作用的关键在于区域1是否处于非封闭的自由状态。
3.2 LAMP-发卡型中转探针-CHA的检测原理
通过发卡型中转探针将LAMP产物传导至上述CHA检测的原理如图1,可分解为3个步骤: 在55~60℃的恒温条件下对待测基因进行LAMP扩增(图1,STEP I); LAMP扩增产物与发卡型中转探针作用,打开发卡型中转探针的茎部,释放出原本被发卡型探针封闭的CHA催化剂C1(图1,STEP Ⅱ); C1开启CHA反应(图1,STEP Ⅲ)。
整个原理的核心部分是发卡型中转探针对LAMP产物和下游CHA的中转效率。由上述CHA工作原理可知,C1能否与H1作用的关键在于区域1是否处于非封闭的自由状态。因此,发卡型中转探针设计的核心思想即是将C1的区域1包埋(封闭)在发卡型探针的茎部,使其无法再行使立足点的功能。而如果将发卡型中转探针的环部设计成LAMP某一环状产物的互补序列,则可利用环状产物与探针环部的杂交作用强制性打开中转探针的茎部,从而游离出C1的区域1,进而开始CHA反应。
3.3 发卡型探针的设计和优化
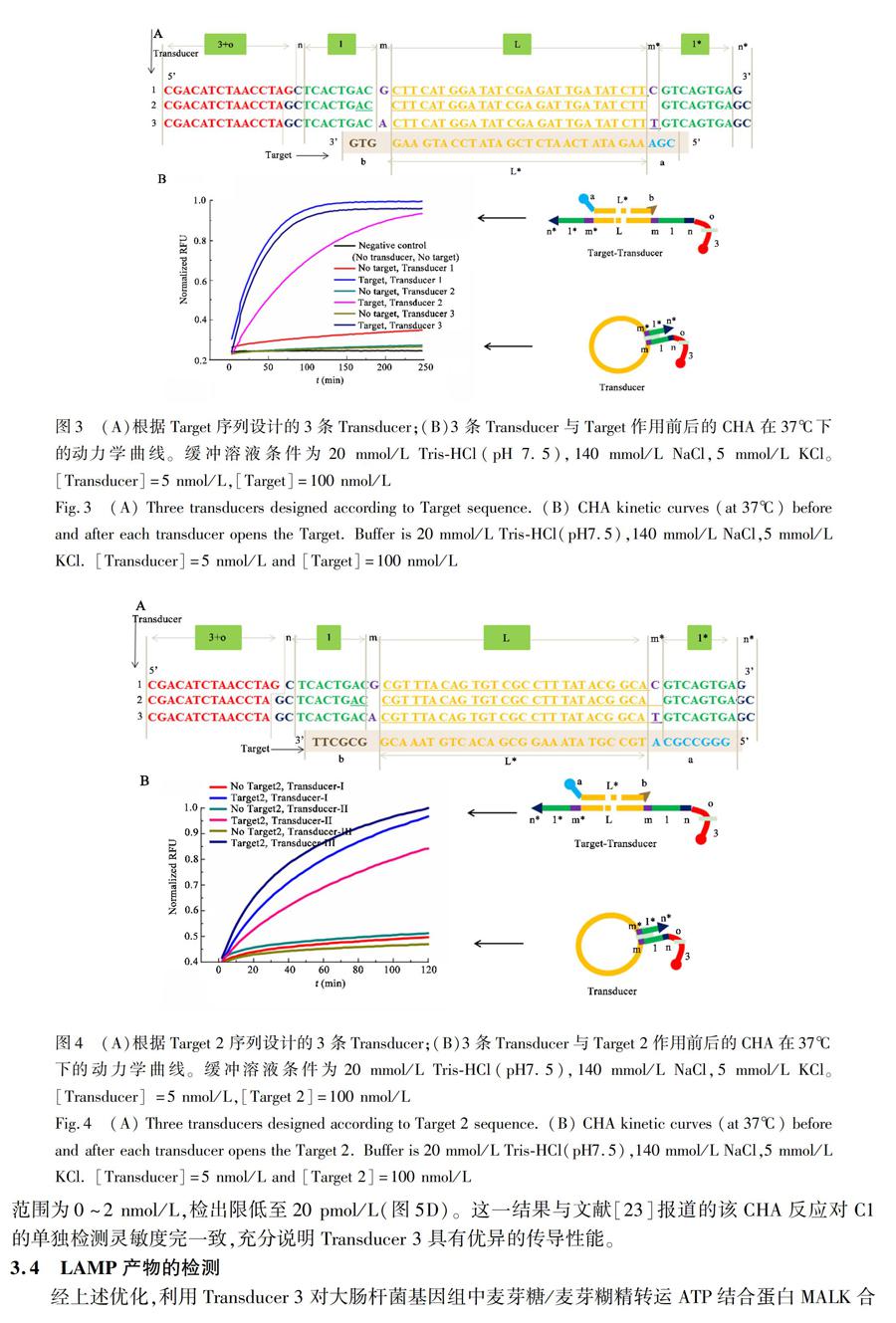
鉴于上述检测原理和设计思想,中转探针的中转效率主要取决于两个因素:封闭效率和打开效率。前者是发卡探针是否能有效封闭C1的区域1,决定检测背景噪音;后者则是LAMP是否能高效打开中转探针,决定检测信号强度。为方便优化,本研究首先以一条线性单链DNA模拟LAMP单链环状产物,并将其命名为Target,序列从5'端到3'端划分为3个区域(a、L*、b)。相应地,发卡型中转探针从5'端到3'端划分为9个区域(3、o、n、1、m、L、m*、1*、n*),命名为Transducer。其中区域o和n合并为区域2;区域3,2和1合并构成C1序列。区域L与Target的L*互补,是待测物识别位点。m是随机加入的缓冲序列。在进行检测时,预期历程如图2所示:Target的L*部分与Transducer的L部分杂交,强制性打开Transducer的茎部,游离出C1的区域1。然而,反应还可能存在如图2所示的其它多种副反应和非预期的情况,降低封闭效率或打开效率。
3.4 LAMP产物的检测
经上述优化,利用Transducer 3对大肠杆菌基因组中麦芽糖/麦芽糊精转运ATP结合蛋白MALK合成基因(简称malB)的LAMP产物进行中转和CHA检测(LAMP引物和灵敏度参见文献[31])。如圖 6所示,当LAMP模板浓度低至20拷贝时,仍可被CHA有效检出,信噪比良好。但即使在大量LAMP产物存在下,CHA的检测曲线也无法如图5C(如5 nmol/L Target)所示在短时间内达到反应终点(平台)。原因是在LAMP产物中,L*序列被限制在环状结构中,与Transducer为环对环杂交,这种情况下杂交效率会降低很多。
除荧光法之外,本研究还利用电化学方法对上述LAMP产物的传导检测体系进行了验证。方法原理如图7A所示,将H1的区域6去掉,并在其3'端修饰上亚甲基蓝(MB)探针,称作H1-MB。将巯基修饰的区域5*-2*序列(简称HS-P探针)通过金-硫键固定于金电极表面,并用巯基己醇(MCH)进行封闭,形成HS-P/MCH修饰电极。当CHA反应未发生前,H1-MB的3'端只有区域5游离,其只有8个碱基,并不能与HS-P的5*稳定杂交。而当LAMP产物经过Transducer 3中转,并引发H1-MB和H2的CHA组装后,H1释放出游离的区域2,区域2和区域5组成的区域2~5长达16个碱基,可与HS-P的区域5*-2*杂交成稳定的双链结构,进而将MB探针带至电极表面,引发可以检测到的MB氧化还原电流。如图7B所示,采用电化学方法,同样达到了如荧光方法相近的信噪比,证明此中转方法的普适性。
4 结 论
本研究提供了一套设计发卡型中转探针的详细规律和思路,使其能作为恒温扩增反应和下游CHA核酸分子线路间的有效桥梁,将基因序列信息准确、特异地传导至荧光和电化学信号。在这样一个恒温扩增-中转探针-CHA检测构成的检测流程中,恒温扩增是灵敏度的主要来源,中转探针和CHA是特异性的主要来源,也是信号幅度的主要来源。而中转探针的引入,也使得基于CHA的基因检测更为方便,并更具普适性。针对不同的基因序列,仅仅通过简单地设计一套中转探针,即可将基因序列传导至一套性能良好的CHA反应,无需重新设计整个CHA反应。中转探针的设计方法并非仅限于发卡型一种,本课题组也报道过三通、四通结构的中转方法[31~33]。这些不同的中转方法各具优势,可以全面满足各种类型的检测需求,充分显现出核酸分子线路在实际应用中的巨大潜力和广阔前景。
References
1 Liu D Y, Daubendiek S L, Zillman M A, Ryan K, Kool E T. J. Am. Chem. Soc., 1996, 118(7): 1587-1594
2 Guatelli J C, Whitfield K M, Kwoh D Y, Barringer K J, Richman D D, Gingeras T R. Proc. Natl. Acad. Sci. USA, 1990, 87(5): 1874-1878
3 Compton J.Nature, 1991, 350(6313): 91-92
4 Walker G T, Fraiser M S, Schram J L, Little M C, Nadeau J G, Malinowski D P. Nucleic Acids Res., 1992, 20(7): 1691-1696
5 Notomi T, Okayama H, Masubuchi H, Yonekawa T, Watanabe K, Amino N, Hase T. Nucleic Acids Res., 2000, 28(12): e63
6 Vincent M, Xu Y, Kong H M. EMBO Rep., 2004, 5(8): 795-800
7 Piepenburg O, Williams C H, Stemple D L, Armes N A. PLOS Biol., 2006, 4(7): e204
8 Craw P, Balachandran W. Lab Chip, 2012, 12(14): 2469-2486
9 Gill P, Ghaemi A. Nucleosides Nucleotides Nucleic Acids, 2008, 27(3): 224-243
10 Zhang X Z, Lowe S B, Gooding J J. Biosens. Bioelectron., 2014, 61: 491-499
11 Li J, Macdonald J. Biosens. Bioelectron., 2015, 64: 196-211
12 Li B L, Chen X, Ellington A D. Anal. Chem., 2012, 84(19): 8371-8377
13 Zhao Y X, Chen F, Li Q, Wang L H, Fan C H. Chem. Rev., 2015, 115(22): 12491-12545
14 Stojanovic M N, Stefanovic D, Rudchenko S. Acc. Chem. Res., 2014, 47(6): 1845-1852
15 Wang F A, Lu C H, Willner I. Chem. Rev., 2014, 114(5): 2881-2941
16 Jung C, Ellington A D. Acc. Chem. Res., 2014, 47(6): 1825-1835
17 Bi S, Yue S Z, Zhang S S. Chem. Soc. Rev., 2017, 46(14): 4281-4298
18 Qu X M, Zhu D, Yao G B, Su S, Chao J, Liu H J, Zuo X L, Wang L H, Shi J Y, Wang L H, Huang W, Pei H, Fan C H. Angew. Chem. Int. Edit., 2017, 56(7): 1855-1858
19 Dirks R M, Pierce N A. Proc. Natl. Acad. Sci. USA, 2004, 101(43): 15275-15278
20 Choi H M T, Chang J Y, Trinh L A, Padilla J E, Fraser S E, Pierce N A. Nat. Biotechnol., 2010, 28(11): 1208-1212
21 Zhang D Y, Turberfield A J, Yurke B, Winfree E. Science, 2007, 318(5853): 1121-1125
22 Yin P, Choi H M T, Calvert C R, Pierce N A. Nature, 2008, 451(7176): 318-322
23 Li B L, Ellington A D, Chen X. Nucleic Acids Res., 2011, 39(16): e110
24 Wu Z K, Fan H H, Satyavolu N S R, Wang W J, Lake R, Jiang J H, Lu Y. Angew. Chem. Int. Edit., 2017, 56(30): 8721-8725
25 Li F, Zhang H Q, Wang Z X, Li X K, Li X F, Le X C. J. Am. Chem. Soc., 2013, 135(7): 2443-2446
26 Wu C C, Cansiz S, Zhang L Q, Teng I T, Qiu L P, Li J, Liu Y , Zhou C S, Hu R, Zhang T, Cui C, Cui L, Tan W H. J. Am. Chem. Soc., 2015, 137(15): 4900-4903
27 Ma C P, Wang W S, Li Z X, Cao J J, Wang Q Y. Anal. Biochem., 2012, 429(2): 99-102
28 Jiang Y, Li B L, Milligan J N, Bhadra S, Ellington A D. J. Am. Chem. Soc., 2013, 135(20): 7430-7433
29 Yang Z L, Liu Y C , Lu W, Yuan Q P, Wang W, Pu Q S, Yao B. Talanta, 2016, 152: 301-307
30 Qu X M, Wang S P, Ge Z L, Wang J B, Yao G B, Li J, Zuo X L, Shi J Y, Song S P, Wang L H, Li L, Pei H, Fan C H. J. Am. Chem. Soc., 2017, 139(30): 10176-10179
31 Tang Y D, Lu B Y, Zhu Z T, Li B L. Chem. Sci., 2018, 9(3): 760-769
32 Li B L, Jiang Y, Chen X, Ellington A D. J. Am. Chem. Soc., 2012, 134(34): 13918-13921
33 Tang Y D, Zhu Z T, Lu B Y, Li B L. Chem. Commun., 2016, 52(88): 13043-13046
Abstract Isothermal nucleic acid amplifications, as powerful as polymerase chain reaction but functioning at a constant temperature, are considered to be very promising technique in achieving point-of-care gene diagnostics. However, until now, their practical applications are still seriously lagged by the bad reliability resulting from the problems such as false positive amplification and low signal amplitude. In this work, a universal transduction method in which any sequence (including loop-mediated isothermal amplification products) could be transduced via a hairpin transducer into a catalyst of a well-engineered circuit (catalytic hairpin assembly, CHA) was established. Because CHA circuit could amplify tens to hundreds fold with especially high sequence specificity, it could provide both accuracy and high amplitude for sequence detection. And for a new targeting sequence, the only sequence needed to be changed was the hairpin transducer. Due to the importance of the transducer, we provided and verified a universal designing rule-set to guarantee the transducing efficiency (signal to background ratio) of the transducer. Transducers designed following this rule set were then proved to be very efficient in detecting pathogen gene targets. As less as near single molecule (20 copies) of pathogen genes could be detected with significant fluorescent and electrochemical signals.
Keywords Isothermal amplification; Universal transduction; Hairpin transducer; Nucleic acid circuit; Catalytic hairpin assembly; Fluorescence; Electrochemistry.
(Received 24 January 2018; accepted 10 April 2018)
