我国沿海典型水产养殖水域底泥中抗生素抗性细菌的多样性分析
2018-10-26李云莉高权新齐占会岳彦峰施兆鸿彭士明
李云莉,高权新,齐占会,岳彦峰,施兆鸿,彭士明,高 阳
(1.中国水产科学研究院东海水产研究所,上海 200090;2.中国水产科学研究院南海水产研究所,广州 510300;3.浙江海洋大学水产学院,舟山 316022)
我国拥有辽阔的海洋面积,沿海滩涂分布十分广泛,是全球最大的海水养殖国家,水产养殖产业迅速发展,养殖规模逐年扩大,品种与产量日益增加,2015年海水养殖产量达到1 875.6×104t,约占世界海水养殖总产量的70%以上,是世界上唯一养殖产量超过捕捞产量的国家[1-2]。近年来,随着我国沿海滩涂总量的增加,沿海滩涂成为了发展现代水产养殖业的最佳区域[3]。然而,随着我国水产养殖事业的蓬勃发展,水产养殖规模的不断扩大,养殖业已经给环境造成了较大的影响,产生了一系列的环境问题。造成养殖污染的原因主要有:1)抗生素的使用,2)投入水中的饵料,3)养殖生物的代谢物。其中,抗生素的使用不仅对水体环境造成污染,而且长期使用抗生素会诱导致病菌产生抗药性,从而导致无法有效治疗致病菌引发的疾病。
在水产养殖中使用抗生素能有效的预防爆发性疾病的发生、促进水生动物的生长以及降低一些营养成分的需求量从而节约营养成分,但长期使用抗生素会诱导动物体内产生携带抗性基因的菌株,特别是会产生具有多重耐药性的菌株[4]。抗生素一旦进入水生环境中,整个水体中的菌落结构将可能发生改变,这种改变会使水生菌产生抗生素抗性基因,从而对抗生素具有耐受性。抗生素抗性基因可以在各种环境介质(比如土壤、河水、地下水)中进行迁移、转化,继而整合到质粒、转座子、整合子等可移动基因元件[5],之后再进入到微生物环境中,在细菌之间利用基因的横向转移进行传播,使原本没有抗生素抗性的细菌获得耐药性,整个过程伴随着某些菌的减少,甚至消失[6];另外,具有耐受性的细菌就会增加,成为优势菌,通过食物链进入动物体内或人体内,严重威胁人类健康甚至生命安全。因此,对养殖密集型水域抗生素抗性细菌的多样性进行即时有效地研究分析是十分必要的。
我国养殖密集水域的底泥是抗生素及其抗性基因的“蓄积池”,其中滋生大量的抗生素抗性细菌,所以本研究将底泥作为研究对象,分别在我国沿海11个典型养殖密集水域采样,利用DNA提取试剂盒提取底泥样品中的DNA并将其纯化,之后进行高通量测序分析,通过比较不同养殖区域底泥在不同的抗生素作用下微生物群落的多样性特征变化,以期掌握当前我国东部沿海养殖密集水域抗生素抗性菌的生物多样性情况,为后续有效开展养殖水域的生态保护及可持续利用提供参考依据。
1 材料和方法
1.1 样品采集
本实验于2015年9-10月,从中国沿海养殖密集水域采集底泥样品,从北至南分别是大连、唐山、蓬莱、连云港、启东、象山、宁德、东山、湛江、陵水和美济礁等11个地点。各个水域的海水养殖品种不尽相同,山东以北主要养殖半滑舌鳎(Cynoglossus robustus)、大菱鲆(Scophthalmus maximus)、对虾以及海蜇(Rhopilema esculentum)、刺参等;江浙的养殖品种主要是文蛤(Meretrix meretrix)、梭子蟹、对虾以及大黄鱼(Larimichthys crocea)、黑鲷(Acanthopagrus schlegeli)等;福建以南主要养殖鲍鱼、中国花鲈(Lateolabrax maculates)以及珍珠龙胆、点篮子鱼(Siganus guttatas)等。每片水域随机收集10个底泥样品。底泥样品用密封袋封好,-80℃保存备用[7]。
1.2 抗生素抗性菌的筛选
将各地采集到的底泥样品分别充分混匀后,每份样品称取3 g进行平板涂布实验。实验采用的培养基为LB培养基,其配方为:胰化蛋白胨10 g,酵母提取物 5 g,NaCl 10 g,琼脂 15~20 g,水1000 mL,最终pH为7.4。高温灭菌后,添加到培养基中的抗生素的浓度分别为:盐酸四环素10 μg·mL-1,盐酸土霉素20μg·mL-1,环丙沙星2 μg·mL-1,恩诺沙星 2μg·mL-1,磺胺嘧啶 50 μg·mL-1,磺胺甲恶唑 100μg·mL-1。涂布后,36℃培养72 h,每组5个平行。挑选单菌落,分别放入无菌EP管中,并混合。
1.3 抗生素抗性菌的多样性分析
将经抗生素筛选后的细菌样品送至上海派森诺生物科技有限公司,在IlluminaMiSeq平台上对抗生素抗性菌的多样性进行分析。
1.4 生物信息学分析
测序得到的原始数据存在一定的干扰数据,为了使信息分析的结果更加准确、可靠,先对原始数据进行拼接、过滤,得到有效数据[8]。然后对有效数据在97%水平上进行OTU聚类,利用Greengene数据可对物种进行注释[9]。通过对OTUs进行丰度、Alpha多样性以及物种在各分类水平上的群落结果统计分析,可以得到微生物群落组成[10],再通过Beta多样性分析,研究海水养殖水域底泥样本间的微生物群落结构差异[11]。
1.5 数据分析
利用 Uparse(version 7.1 http://qiime.org)软件平台进行OTU聚类,采用 RDP classifier贝叶斯算法对97%相似水平的OUT代表序列进行分类学分析;利用Mothur软件进行稀释曲线分析;利用Shannon指数图和Chao指数图进行细菌群落多样性分析;利用Qiime生成不同分类水平上的多样品物种分布图;利用R语言制作热图。
2 结果与分析
2.1 多样性分析
多样性指数是反映丰富度和均匀度的综合指标,与种类的数目(即丰富度)和种类中个体分配上的均匀性有关。Chao指数可以反映微生物群落的丰富度,而Shannon指数可以反映微生物群落的多样性[12]。通过Chao指数和Shannon指数等数据进行多样性分析可知,不同养殖区域中抗生素抗性菌株的丰富度和多样性存在着较大的差异,见图1。由Chao指数图可知(图1-a),大部分海水养殖区域抗磺胺甲恶唑的细菌菌群丰富度最高,抗恩诺沙星和环丙沙星的细菌群落丰富度较低;总体来看,北方海水养殖区域的细菌菌群比南方海水养殖区域的细菌菌群丰富度高。由Shannon指数图可知(图1-b),大部分海水养殖区域中抗四环素类和磺胺类抗生素的底泥细菌群落多样性高于抗喹诺酮类抗生素的底泥细菌群落多样性,其中尤以抗磺胺嘧啶的细菌群落的多样性最高。
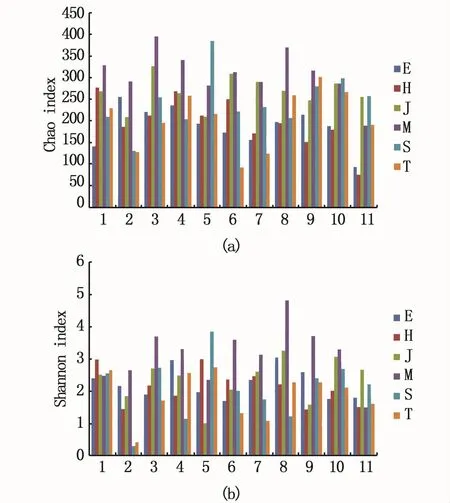
图1 不同抗生素作用下各养殖区域底泥细菌微生物的丰富度Fig.1 Species richness of bacteria in sediments from different culture areas under different antibiotics
2.2 群落结构分析
由图2可见,海水养殖的底泥中的主要细菌菌群包括酸杆菌门(Acidobacteria)、放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)、衣原体门(Chiamydiae)、绿弯菌门(Chioroflexi)、厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)、疣微菌门(Verrycomicrobia)以及分类地位不明确和一些稀有的细菌菌群。其中,在图2-a中,大部分水域变形菌门占主要优势,其次是厚壁菌门;变形菌门在南方海水养殖海域所占的优势大于北方海水养殖水域所占的优势;采样点11(美济礁)的底泥细菌菌群几乎都是变形菌门。在图2-b中,大部分海水养殖区域的底泥中,厚壁菌门的细菌群落占主要优势,其次是变形菌门;采样点11(美济礁)在磺胺甲恶唑的作用下,变形菌门占主要优势,而在磺胺嘧啶的作用下,厚壁菌门占主要优势。在图2-c中,大部分海水养殖区域的底泥中,厚壁菌门的细菌群落占主要优势,其次是变形菌门;采样点11(美济礁)在盐酸四环素和盐酸土霉素的作用下,底泥细菌菌群几乎都是厚壁菌门。
Heatmap图通过颜色变化可以直接反映二维矩阵或者表格中的数据信息,以数值的大小来定义颜色的深浅,并将高峰度和低丰度的物种分块聚类,通过颜色梯度及相似程度来反映多个样本在分类水平上的群落组成的相似性和差异性[13]。由图3可见,不同养殖区域中,各类细菌菌群的丰度在不同抗生素的作用下具有较大的差异,可见养殖区域中使用抗生素会显著影响菌落结构。,由图 3-a可知,在 E8、E4、E9(东山、连云港、湛江水域恩诺沙星抗性菌)组皆具有明显的优势菌,且优势菌的种类不同,优势菌的种类依次减少;而其它组优势菌的种类和数量较少;由图3-b可知,M8(东山水域磺胺甲恶唑抗性菌)、J11(美济礁水域磺胺嘧啶抗性菌)、J3(蓬莱水域磺胺嘧啶抗性菌)、M3(蓬莱水域磺胺甲恶唑抗性菌)组皆具有明显的优势菌,且优势菌的种类不同,并依次减少,而其它组则优势菌的种类和数量皆较少;由图3-c可知,在T1(大连水域抗盐酸土霉素)、T4(连云港水域盐酸土霉素)、S3(蓬莱水域盐酸四环素)、S1(大连水域盐酸四环素)组具有种类和数量较多优势菌,且优势菌的种类皆不同,而其它组优势菌的数量和种类则较少。
3 讨论
抗生素的广泛使用导致了自然界中抗性菌株的数量剧增,尤其是抗生素抗性致病菌的出现和传播,严重危及人和动物健康。有报道指出,在美国每年有200万的病例是由抗生素抗性致病菌引发的,同时导致14 000人死亡[14]。越来越多的研究发现[15-16],大量的抗生素抗性菌株源自水产养殖密集水域。海水养殖密集水域的底泥成了抗性基因的蓄积地,并不断的通过水环境中的细菌载体,向外周环境散播[17]。我国是世界第一水产养殖大国,抗生素的使用量非常大,尤其是在密集型水域养殖,所以这些水域的菌群结构必然发生改变,抗生素抗性细菌的比例也会随之升高[12,18]。众所周知[19],水生细菌可以通过移动基因元件的水平移动而获得耐药基因,同时细菌的结构组成也在抗生素的自然选择下发生动态变化。然而有关我国沿海典型养殖水域抗生素抗性菌株的文献报道较少。
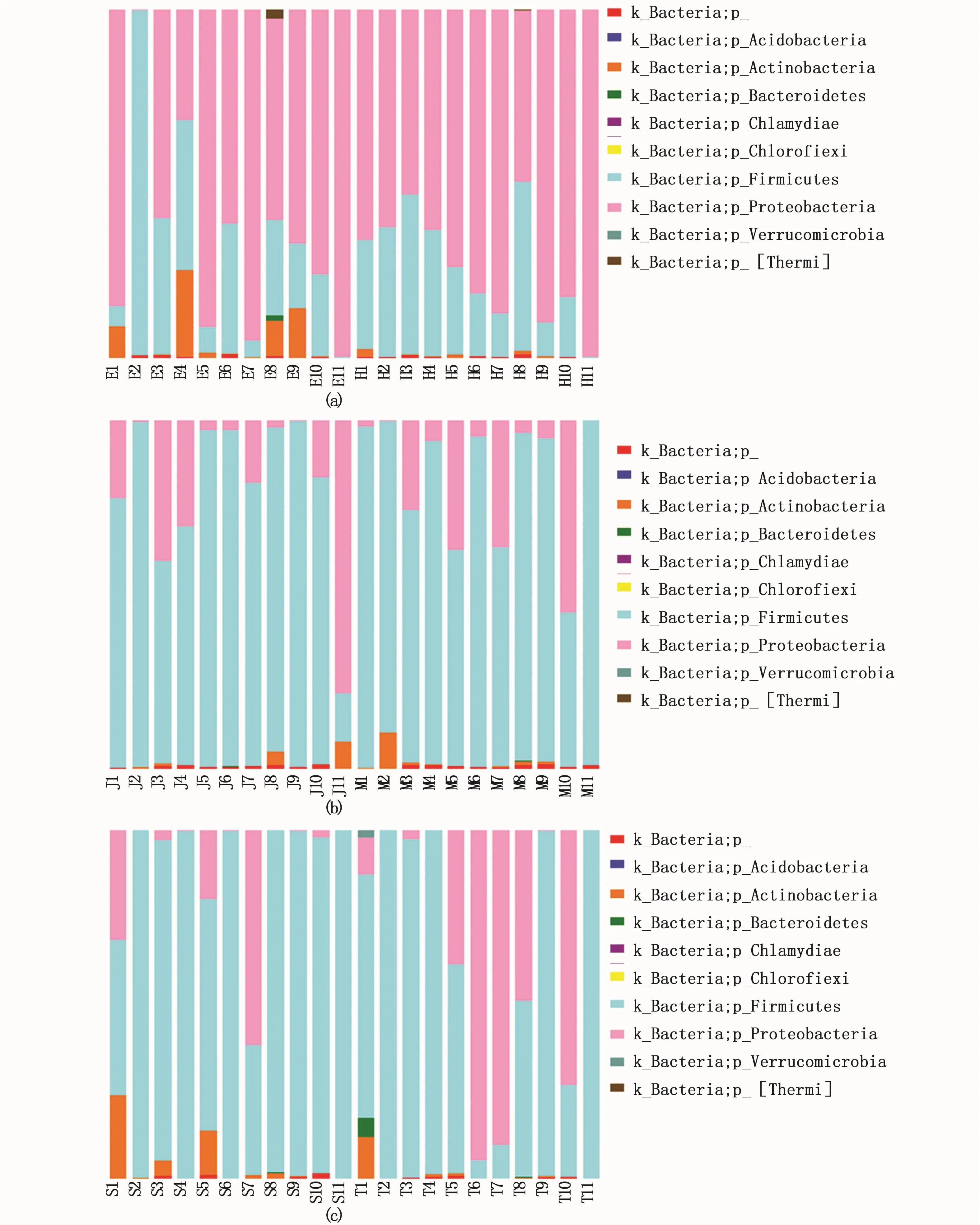
图2 不同抗生素处理底泥细菌在门水平的相对丰度Fig.2 Relative abundance of bacteria in the level of phylum in seawater treated w ith different antibiotics

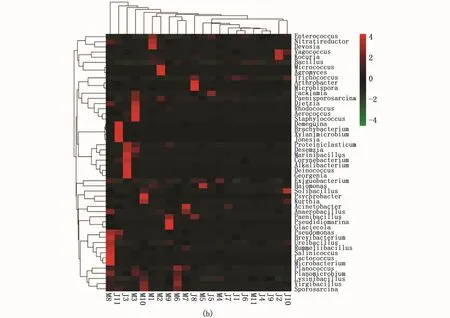
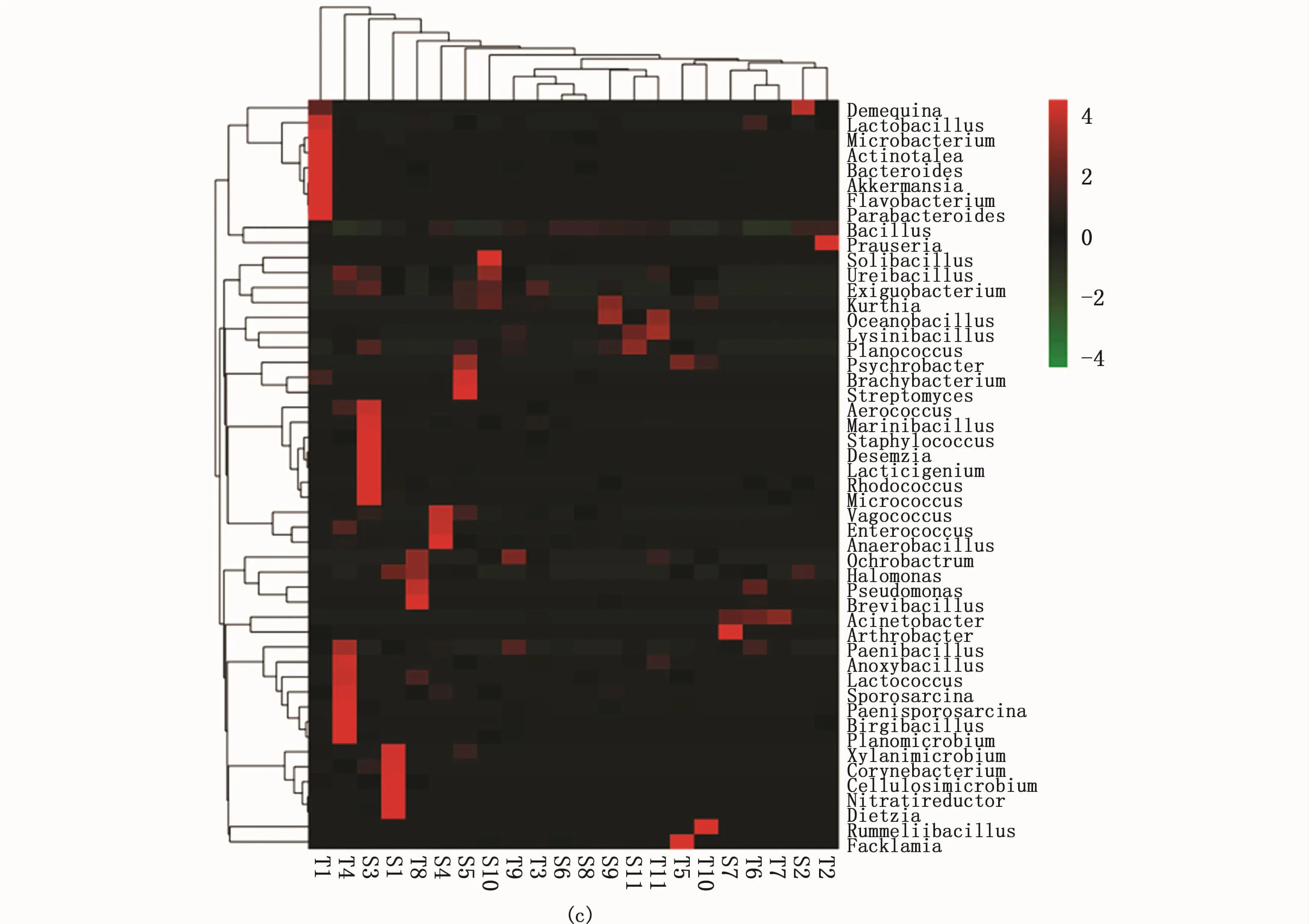
图3 不同抗生素处理海水底泥细菌属水平菌群分析Fig.3 Bacteria analysis of seawater sediment treated by different antibiotics at the genus level
近年来,随着抗生素在水产养殖中使用量的不断增加,抗生素的过度使用和处理不当引起了人们的广泛关注[20]。滥用抗生素导致药物的生态释放,从而使环境中的细菌组成向着适应抗生素的方向转化[21-22],抗生素抗性细菌随之成为优势菌群。早期的研究报道显示[23-24],在大海、沿海水域以及海水沉积物中都发现了抗生素抗性细菌。在自然条件下,水生环境是抗生素抗性细菌的一个巨大的集聚地[25-26],越来越多的抗生素抗性菌株在各种水生环境中被发现和鉴定[27-30]。尤其是在养殖密集水域,抗生素通常被混入鱼类饲料或是直接加入到养殖水体中,用于预防和治疗水产动物的细菌性疾病,从而加速了抗性基因的传播和抗性菌株的增殖[17]。所以,随着养殖水域的富营养化、生物耗氧的增加以及抗生素的大量使用,细菌的多样性必将发生显著变化。然而大海的巨大流动系统也会减缓水产养殖的人为影响。因此,对比分析不同养殖水域细菌多样性,可初步了解水产养殖活动尤其是抗生素的大量使用对海洋生物群落的影响。在本研究中,通过Chao和Shannon指数图分析可知,在不同的养殖区域中使用同一种抗生素,底泥的细菌群落多样性存在差异;在同一养殖区域中使用不同的抗生素,底泥的细菌群落的多样性也不相同;这说明抗生素的使用显著改变了细菌的群落组成,这与SØRUM等[31]的研究结果一致。
本研究发现,喹诺酮类、磺胺类、四环素类抗生素抗性菌在我国沿海典型水域中均有分布,而且这些抗生素抗性菌的数量较大,种类较多,在部分水域优势菌明显。研究结果说明这3大类抗生素在我国海水养殖过程中使用普遍,这与实地调查结果一致。此外,实验结果进一步说明抗生素对水生细菌群落结构具有较大的影响,可诱导产生大量的抗性菌株。喹诺酮类、磺胺类、四环素类等抗生素都是水产养殖中常用的抗生素,而且有报道指出这些抗生素的使用可显著影响水生细菌的多样性[32-33]。SHAH等[34]对智利鲑鱼养殖水域底泥进行了基因分析,发现海水养殖活动对抗生素抗性菌株的多样性影响显著。大量研究证实[34-35],抗生素在养殖水体和淤泥中的残留量和时间与抗性基因的含量和抗生素抗性菌株的数量呈显著相关;这些文献直接或间接指出抗生素对水生细菌的多样性影响较大,本研究的结果与之相似。
通过细菌群落门水平分析发现,本研究选择的密集养殖水域中变形菌门和厚壁菌门的相对丰度优势明显。以往的研究指出[36-37],变形菌门和厚壁菌门是河水、海水以及海洋底泥中的主要类群。在我国渤海和黄海区域的海水养殖池塘的沉积物中,也发现主要细菌群落为变形菌门和厚壁菌门[38]。此外亦有研究指出,海水循环水养殖系统中优势细菌种群中有变形菌门和厚壁菌门[39]。由此可以看出,海水养殖活动对我国密集养殖水域细菌在门水平上的影响较小。然而在属分类水平上,通过对各样本进行聚类分析发现,添加不同的抗生素会显著改变细菌群落的多样性和丰富度;添加同一种抗生素,相邻水域细菌群落的相似性不高,南、北养殖水域底泥中细菌群落的组成结构存在着较大的差异。究其原因,除了在海水养殖过程中抗生素的使用种类和使用量不同外,可能还由不同海水养殖水域在地区天气、养殖模式、温度变化、水产动物种类等方面的差异造成[40-41]。
综上所述,喹诺酮类、磺胺类、四环素类抗生素抗性菌我国沿海典型水域分布广泛;在不同的养殖区域中使用同一种抗生素,底泥的细菌群落多样性存在差异;在同一养殖区域中使用不同的抗生素,底泥的细菌群落的多样性也不相同;变形菌门和厚壁菌门是养殖水域的主要门类。研究结果表明,抗生素的使用对抗生素抗性菌的丰度具有显著的影响,所以规范抗生素的使用和开展相关的生态风险评估是今后海洋生态管理的重点。
