滨麦叶片转录组分析
2018-10-09李红婷魏露阳李中虎朱路英柏新富
李红婷,魏露阳,李中虎,朱路英,柏新富
(1.鲁东大学农学院,山东烟台 264025; 2.鲁东大学生命科学学院,山东烟台 264025)
滨麦(LeymusmollisL.)属于禾本科小麦族赖草属,是一种分布于沿海海滩及贫瘠土地上的野生植物。作为小麦的野生近源物种,滨麦具有耐盐碱、耐干旱、抗病虫害、秸秆粗壮和大穗多花等优良性状,被认为是小麦遗传改良的重要资源[1-2]。同时,滨麦还是一种典型的盐生植物,在沿海沙地生长旺盛。因此,滨麦也是研究植物耐盐机理的良好材料。
将野生近源物种的优良基因引入小麦从而培育性状优良的小麦品种,已成为多年来小麦育种工作的重要任务之一。近年来,人们先后将滨麦与普通小麦进行杂交,以期获得具有滨麦优良性状的普通小麦种质。傅 杰等[3-4]、陆文辉等[5]先后利用野生滨麦与普通小麦杂交的方法选育出了表现滨麦优良特性的附加系、代换系和易位系。Forsstrom等[6]将波斯小麦与滨麦的杂交种再与普通小麦进行杂交,在后代中鉴定出6个抗白粉病的纯合系。王金平等[7]将普通小麦与八倍体小滨麦杂交,在杂交后代中鉴定出了兼具抗白粉病和条锈病的种质材料山农6343。Pang等[8]在普通小麦和滨麦的杂交后代中筛选出了抗叶锈病的代换系,利用分子细胞遗传学进行鉴定,证实普通小麦和滨麦的杂交后代中含有滨麦的部分染色体;和普通小麦亲本相比,杂交后代的部分农艺性状得到改善[9-11]。这些研究成果积极推进着小麦的遗传育种工作进程。尽管滨麦在丰富小麦遗传基因资源和提高小麦的抗病性、抗逆性等方面具有巨大的利用价值,但人们对滨麦的基因信息和相关功能了解却非常少,滨麦响应多种生物胁迫和非生物胁迫的生理生化反应的遗传基础还不清楚。
转录组是指某一条件下或某一时刻,生物体细胞内所有转录产物的总和,包括mRNA和非编码RNA。转录组测序是将总RNA中的mRNA反转录成cDNA,通过高通量测序技术,快速全面地获得某个特定样本中几乎所有cDNA的序列信息及含量[12]。通过转录组测序得到的信息能够对基因组功能元件、剪切方式、转录后修饰、转录产物分类以及胁迫条件的基因差异表达进行研究。因此,转录组测序已成为研究基因表达的重要手段。目前,植物、动物和微生物的转录组研究均有报道[13-15]。本研究利用Illumina HiSeqTM2000 高通量测序技术,首次对生长在海滨沙地的滨麦叶片进行转录组测序,对测序组装的转录本进行分类统计、功能注释和Pathway分析,并对Na+转运相关蛋白基因和氧化胁迫响应基因进行挖掘,以期为进一步研究滨麦的基因信息和耐盐机理提供重要参考。
1 材料与方法
1.1 材 料
供试材料滨麦叶片采集于山东省烟台市烟台大学东邻的海滨沙地(东经121°46′,北纬37°47′),采集时间在5月下旬,此为滨麦的旺盛生长期。选取20株生长健壮的野生滨麦植株的中部叶片为试验材料,采摘后立即置于液氮中保存备用。
1.2 方 法
1.2.1 RNA提取及cDNA文库的构建
将滨麦叶片在液氮里研磨成细粉状,用Trizol试剂(Invitrogen,美国)提取总RNA。分别通过琼脂糖凝胶电泳和微量核酸定量仪检测提取RNA的完整性和浓度,将符合转录组RNA的检测标准(浓度≥400 ng·μL-1,OD260/280=1.8~2.1,rRNA Ratio (28S:18S)≥2.0,RIN≥7)的样品用于后续试验。用Illumina Gene Expression Sample Prep Kit(Illumina,美国)中的Oligo(dT)磁珠富集mRNA,随后将mRNA片段化处理,再用随机引物和SuperscriptⅡ反转录酶(Invitrogen,美国)合成cDNA第一链,然后采用Super Script II Reverse 试剂盒(Invitrogen,美国)合成cDNA第二链。经过QiaQuick PCR试剂盒(Qiagen,德国)纯化后做末端修复和3′端加poly(A)并连接到测序接头,然后用琼脂糖凝胶电泳选择200 bp的片段,用高通量测序平台Illumina Hiseq 2000进行PE100测序。上述测序工作在深圳华大基因股份有限公司完成。
1.2.2 数据拼接
测序得到的原始图像数据首先经base calling转化为序列数据获得raw reads,通过FastQC检查raw reads的质量,去除含有带接头的、重复的以及测序质量低(N的比例大于5%、质量值Q≤10的碱基数占整个read的20%以上)的reads后,获得clean reads。使用短reads 组装软件Trinity进行从头组装[16]。首先将具有一定长度重叠的reads拼接成Contig,然后利用paired-end reads确定同一转录本的不同Contig及其距离,将这些Contig连接,获得两端不能延长的序列Unigene,即转录本。
1.2.3 功能注释
通过BlastX软件将获得的转录本序列分别比对到NCBI-NR(ftp://ftp.ncbi.nih.gov/blast/db/nr/)、Swiss-Prot (http://www.gpmaw.com/html/swiss-prot.html)、KEGG (http://www.kegg.jp/kegg/pathway.html)和COG(https://www.ncbi.nlm.nih.gov/COG/),得到与给定转录本具有最高相似度的蛋白(E-value<10-5),利用 InterProScan(http://www.ebi.ac.uk/Tools/pfa/iprscan/)对蛋白功能进行注释,从而获取该转录本的蛋白功能注释信息。根据NR的注释信息,使用Blast2GO软件得到转录本的GO (http://www.geneontology.org/)注释信息,再使用WEGO将所有转录本做GO功能分类统计。
1.2.4 荧光定量PCR验证
为了验证转录组测序结果的准确性,根据转录组测序结果,随机选取了12个基因(包括2个转录因子基因NAC和DREB,3个Na+转运蛋白相关基因 NHX2、 SOS1和H+-ATPaseI,2个氧化胁迫响应基因 POD21和TRX,1个热胁迫响应基因HSP,1个光合作用相关基因RcaB,1个信号转导相关基因PP,1个脱水素基因Dehydrin和1个水通道蛋白基因TIP)进行实时荧光定量反转录PCR(qRT-PCR)验证。根据转录组测序得到的基因序列,采用Premier Premier 5.0软件设计特异性的qRT-PCR引物(表1)。利用PrimeScript RT reagent Kit with gDNA Eraser(Perfect Real Time)(TaKaRa,日本)将提取的总RNA反转录为cDNA,以之为模板,按照 SYBR Premix ExTaqTM Kit(TaKaRa,日本)说明书在ABI 7300 荧光PCR仪(ABI,美国)进行qRT-PCR。以β-actin基因为内参,用2-ΔΔCt法分析滨海野生滨麦叶片各基因的相对表达水平,每个反应进行3次生物学重复。

表1 qPCR所用引物序列Table 1 Primer sequences for qPCR
2 结果与分析
2.1 测序序列De novo组装
对滨麦叶片样品进行转录组测序,总计获得46 406 704条clean reads,Q20 比值为96.96%,GC含量为55.29%,说明测序结果准确度较高,可用于后续分析。采用Trinity将clean reads进行组装,得到228 637条Contigs,N50长度为438 bp;再将Contig进一步组装,得到112 846条Unigene,其中属于聚类的Unigene 45 563条,独立的Unigene 67 283条,N50长度为1 319 bp(表2)。Unigene的长度分布情况见图1,超过1 kb的Unigene有25 878 条,占22.93%;超过2 kb的有8 031条,占7.12%。测得的转录组数据已提交至NCBI-SRA(https://www.ncbi.nlm.nih.gov/sra/)(登录号为SRP109343)。
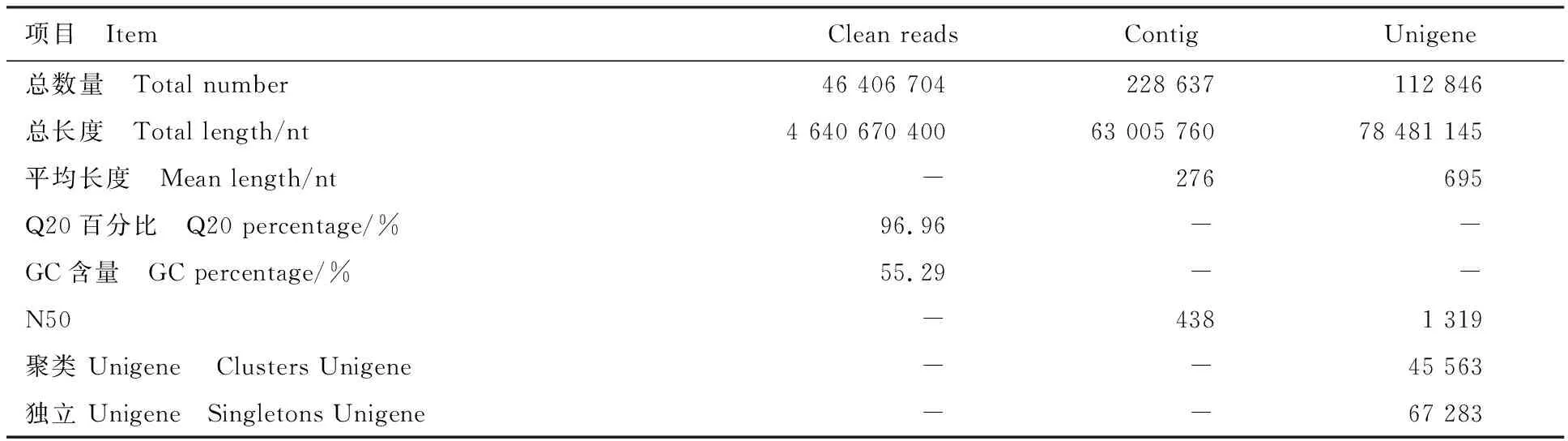
表2 测序产量和组装质量统计Table 2 Statistics of sequencing and assemble quality

图1 组装的Unigene长度分布
2.2 Unigene功能注释
将上述Unigene分别在NCBI-NR、Swiss-Prot、KEGG、COG和GO进行注释,成功注释的Unigene数目依次为41 358、26 184、18 550、15 786和20 350。去除重复注释后的Unigene共计59 380条,占Unigene总数的52.62%。因为滨麦及滨麦所属的赖草属植物基因组信息比较缺乏,因此,滨麦Unigene在与NCBI-NR进行比对时,所注释的基因信息主要来自同为小麦族的大麦(40.69%)、二穗短柄草(23.88%)和小麦(20.05%)。
2.3 转录组Unigene的COG分类
将滨麦叶片转录组Unigene与COG进行比对,得到15 786条注释结果,归属于25个大类(图2)。其中,归属于“一般功能预测(General function prediction only)”类的Unigene数量最多,达7 644条,占48.42%;其次是“功能未知(Function unknown)”类,有6 444条;“翻译、核糖体结构和生物起源(Translation,ribosomal structure and biogenesis)”类有5 689条;“转录(Transcription)”类有5 467条;“复制、重组和修复(Replication,recombination and repair)”类4 755条;“细胞周期控制、细胞分裂和染色体分区(Cell cycle control,cell division,chromosome partitioning)”类4 267条。Unigene数量较少的组类分别是“核结构(Nuclear structure)”(14条)、“胞外结构(Extracellular structure)”(32条)、“RNA加工与修饰(RNA processing and modification)”(149条)和“核染色质结构与动力学(Chromatin structure and dynamics)”(187条)。
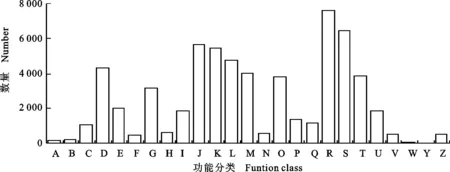
A:RNA加工和修饰;B:核染色质结构与动力学;C:能量产生和转化;D:细胞周期控制,细胞分裂和染色体分隔;E:氨基酸转运和代谢;F:核酸转运和代谢;G:碳水化合物转运和代谢;H:辅酶转运和代谢;I:脂类转运和代谢;J:翻译,核糖体结构和生物起源;K:转录;L:复制,重组和修复;M:细胞壁/膜/包膜生物合成;N:细胞迁移;O:翻译后修饰,蛋白翻转和分子伴侣;P:无机离子转运和代谢;Q:次生代谢物合成,转运和代谢;R:一般功能预测;S:功能未知;T:信号转导机制;U:胞内运输,分泌和膜泡运输;V:防御机制;W:胞外结构;Y:核结构;Z:细胞骨架。A:RNA processing and modification; B:Chromatin structure and dynamics; C:Energy production and conversion; D:Cell cycle control,cell division,chromosome partitioning; E:Amino acid transport and metabolism; F:Nucleotide transport and metabolism; G:Carbohydrate transport and metabolism; H:Coenzyme transport and metabolism; I:Lipid transport and metabolism; J:Translation,ribosomal structure and biogenesis; K:Transcription; L:Replication,recombination and repair; M:Cell wall/membrane/envelope biogenesis; N:Cell motility; O:Post-translation modification,protein turnover,chaperones; P:Inorganic ion transport and metabolism; Q:Secondary metabolites biosynthesis,transport and catabolism; R:General function prediction only; S:Function unknown; T:Signal transduction mechanisms; U:Intracellular trafficking,secretion and vesicular transport; V:Defense mechanisms; W:Extracellular structures; Y:Nuclear structure; Z:Cytoskeleton.
2.4 转录组Unigene的GO分类
滨麦叶片转录组Unigene注释到GO数据库,共有20 350条Unigene被注释分类到“生物学过程(Biological process)”、“细胞组件(Cellular component)”和“分子功能(Molecular function)”三个大类中(图3)。其中,归属“生物学过程”的Unigene数量最多,占总数的43.56%。在“生物学过程”大类中,参与“代谢过程(Metabolic process)”和“细胞过程(Cellular process)”的Unigene数量最多,分别为11 070条和10 922条;其次是“单有机体过程(Single organism process)”(6 153条)、“刺激响应(Response to stimulus)”(4 838条)和“生物调控(Biological regulation)”(3 399条)。在“细胞组件”大类中,归属于“细胞(Cell)”(14 589条)、“细胞区域(Cell part)”(14 589条)以及“细胞器(Organelle)”(12 630条)的Unigene数量较多;在“分子功能”大类中,Unigene归属较多的类别是“结合活性(Binding)”(10 230条)和“催化活性(Catalitic activity)”(10 024条)。

B:生物学过程大类;C:细胞组件大类;M:分子功能大类;B1:生物附着;B2:生物调控;B3:细胞成分组织或发生;B4:细胞过程;B5:发育过程;B6:定位建立;B7:生长过程;B8:免疫系统过程;B9:定位;B10:代谢过程;B11:多有机体过程;B12:多细胞有机体过程;B13:生物过程的负调控;B14:生物过程的正调控;B15:生物过程调控;B16:再生;B17:再生过程;B18:刺激响应;B19:节律性过程;B20:信号;B21:单有机体过程;C1:细胞;C2:细胞结合;C3:细胞区域;C4:胞外区域;C5:大分子复合体;C6:生物膜;C7:膜部分;C8:包膜腔;C9:类核;C10:细胞器;C11:细胞器部分;C12:共质体;M1:抗氧化活性;M2:结合活性;M3:催化活性;M4:电子转运体活性;M5:酶调节活性;M6:分子传感器活性;M7:核酸结合转录因子活性;M8:营养库活性;M9:蛋白质结合转录因子活性;M10:受体活性;M11:结构分子活性;M12:运输活性。B:Biological process category; C:Cellular component category; M:Molecular function category; B1:Biological adhesion; B2:Biological regulation; B3:Cellular component organization or biogenesis; B4:Cellular process; B5:Developmental process; B6:Establishment of localization; B7:Growth;B8:Immune system process; B9:Localization; B10:Metabolic process; B11:Multi-organism process; B12:Multicellular organism process; B13:Negative regulation of biological process; B14:Positive regulation of biological process; B15:Regulation of biological process; B16:Reproduction; B17:Reproductive process; B18:Response to stimulus; B19:Rhythmic process; B20:Signaling; B21:Single-organism process; C1:Cell; C2:Cell junction; C3:Cell part; C4:Extracellular region; C5:Macromolecular complex; C6:Membrane; C7:Membrane part; C8:Membrane-enclosed lumen; C9:Nucleoid; C10:Organelle; C11:Organelle part; C12:Symplast; M1:Antioxidant activity; M2:Binding activity; M3:Catalytic activity; M4:Electron carrier activity; M5:Enzyme regulator activity; M6:Molecular transducer activity; M7:Nucleic acid binding transcription factor activity; M8:Nutrient reservoir activity; M9:Protein binding transcription factor activity; M10:Receptor activity; M11:Structural molecular activity; M12:Transporter activity.
2.5 转录组Unigene的代谢途径分析
将滨麦叶片转录组所有的Unigene比对到KEGG,有18 550条Unigene得到注释,共涉及到128条代谢途径。其中,Unigene数量归属最多的代谢途径是“代谢通路(Metabolic pathways)”,涉及到的Unigene占总数的27.86%;其次是“RNA运输(RNA transport)”、“mRNA监控通路(mRNA surveillance pathway)”、“次生代谢物的合成(Biosynthesis of secondary metabolites)”、“甘油磷脂代谢(Glycerophospholipid metabolic)”和“内吞作用(Endocytosis)”途径,其涉及到的Unigene数量分别占总数的17.48%~10.11%。另外,“植物病原互作(Plant-pathogen interaction)”和“植物激素信号转导(Plant hormone signal transduction)”途径涉及的Unigene数量也较多,分别为1 367条和861条(表4)。这些结果暗示,以上途径在滨麦的生理代谢中处于比较活跃的状态。
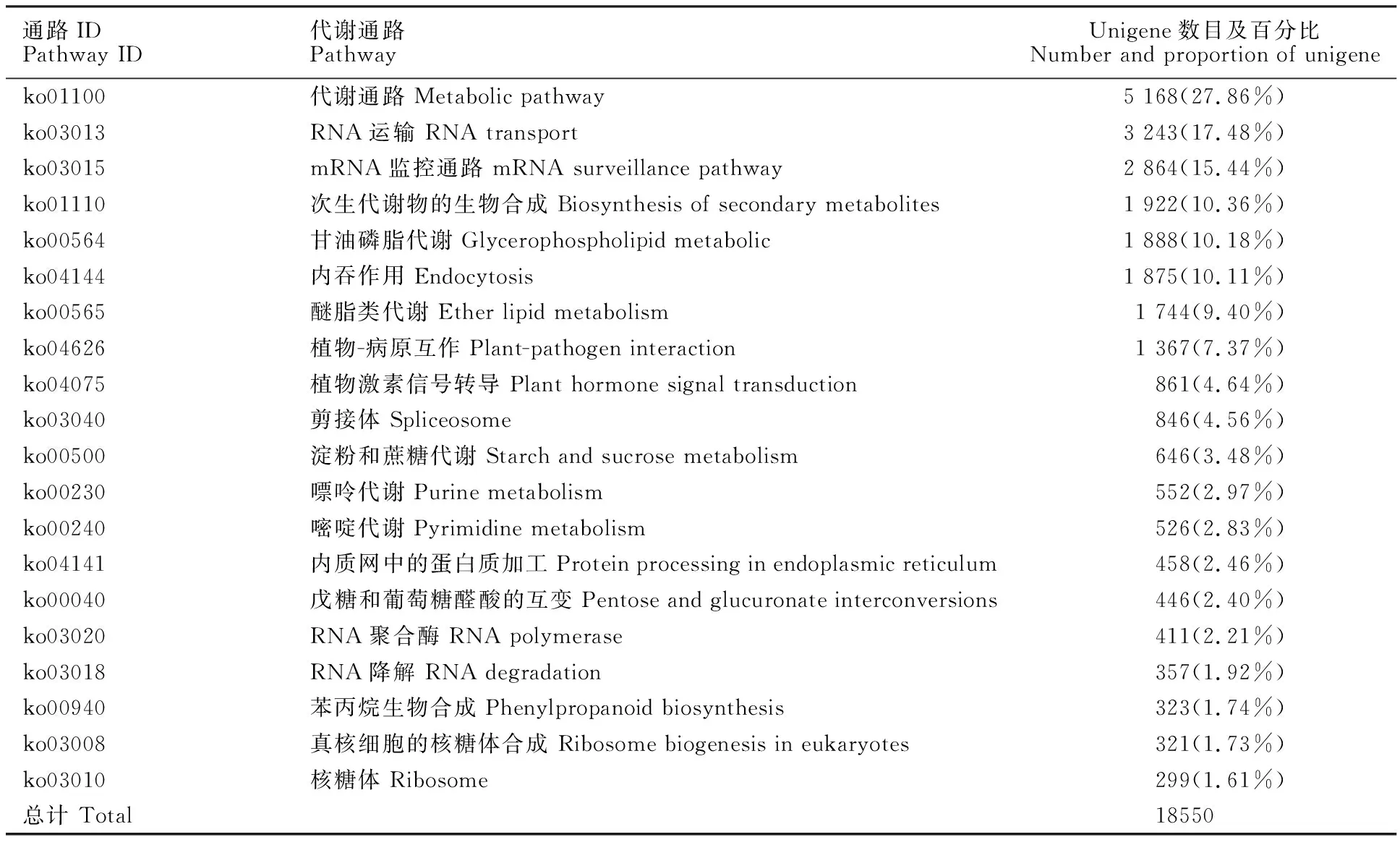
表4 滨麦叶片转录组主要的KEGG通路分析Table 4 Summary of KEGG pathway for Leymus mollis leaf transcriptome
2.6 Na+转运蛋白相关基因和氧化胁迫响应基因的挖掘
基于本研究的材料取自滨海沙地这种高Na+的盐渍环境,故对滨麦叶片转录谱进行了与Na+转运相关的几种蛋白基因的分析,包括液泡膜Na+/H+反向转运蛋白基因(NHX)、质膜Na+/H+反向转运蛋白基因( SOS1)、以及为这两种蛋白转运离子提供能量的液泡膜及质膜上的H+-ATP酶基因(H+-ATPase)或H+-焦磷酸化酶基因(H+-PPase)。根据转录组Unigene比对几个数据库的注释结果,最终检索到与其他物种NHX序列具有高度相似性的Unigene有15条(表5),其中,以拟南芥做参照注释为 NHX1、 NHX2、 NHX6、 NHX7(即 SOS1)和 NHX8( SOS1类似蛋白基因)的Unigene分别有2条、4条、2条、5条和1条。另外,还有1条Unigene与短芒大麦草(Hordeumbrevisubulatum)的 NHX1序列有高度的相似性。检索到注释为液泡膜H+-ATP酶B亚基基因的Unigene 3条、液泡膜H+-PPase的Unigene 4条、质膜H+-PPase8条。这些结果暗示,滨麦叶片细胞中向液泡内转运Na+以及细胞外排Na+的行为较为活跃,这可能是滨麦耐盐的一种策略。
盐渍环境中,植物根际周围过多的Na+会导致植物产生渗透胁迫和离子胁迫,进而引发过氧化胁迫[17-18],因此,本研究对滨麦叶片转录组中的氧化胁迫相关基因进行了分析,共搜索到了175条响应氧化胁迫的Unigene。其中,注释为过氧化物酶(EC:1.11.1.7)的Unigene 数量最多,为50条;其次是谷胱甘肽巯基转移酶(EC:1.8.1.7)和硫氧还蛋白,分别为40条和37条;注释到谷胱甘肽过氧化物酶(EC:1.11.1.9)12条,L-抗坏血酸过氧化物酶(EC:1.11.1.11)11条,超氧化物岐化酶(EC:1.15.1.1)10条,过氧化物还原酶(EC:1.11.1.15)8条,过氧化氢酶(EC:1.11.1.6) 7条(表5)。这说明,滨麦叶片细胞中过氧化物酶、谷胱甘肽巯基转移酶和硫氧还蛋白在抗氧化过程中发挥着重要作用。

表5 滨麦叶片转录组中耐盐相关基因的挖掘Table 5 Analysis of salt-tolerance genes in Leymus mollis leaf transcriptome
2.7 荧光定量PCR结果分析
为了验证转录组测序结果的准确性,本研究随机挑选了12个基因进行qRT-PCR验证。结果表明,这12个基因的相对表达趋势与转录组测序的结果基本一致。其中,位于液泡膜上的水通道蛋白基因TIP表达量最高,转录因子NAC基因和质膜H+-ATPase Ⅰ基因的表达量次之,而转录因子DREB基因和过氧化物酶 POD21基因的表达量相对较低(图4)。
3 讨 论
近几年发展起来的转录组测序(RNA-Seq)技术,因其具有高通量、高灵敏度等优点已成为大规模转录组学研究的一种全新且更为有效的方法。目前,RNA-Seq不仅在基因转录水平和SNP标记物开发等领域广泛应用,而且在完善基因组结构信息和挖掘新基因方面也发挥着重要作用[19-21]。本研究中滨麦叶片经Illumina HiSeq 2000高通量测序后,共获得了46 406 704条过滤后的测序序列。碱基质量及组成分析显示,Q20比值为96.96%,GC含量为55.29%,说明文库构建成功且测序质量良好。经组装和拼接后,共获得了112 846条非冗余Unigene,其中52.62%的Unigene在NR、GO和Swiss-Prot等数据库中获得功能注释。
对滨麦叶片转录组Unigene进行GO功能注释,在“生物学过程”大类中共涉及21个分类,其中归属于“代谢过程”类的Unigene所占比例最多,其次为“细胞过程”类和“单有机体过程”类,这些均为常规的生物学过程。此外,“生物学过程”大类中还包含“刺激响应”和“生物调节”这两个与环境适应性相关的类别,且涉及到的Unigene数量比例也较大,共有8 237条。这个结果与本研究中供试材料的生长环境滨海沙地相符合。在“细胞组件”大类中共涉及12个分类,除了“细胞”和“细胞区域”类所占比例最多外,“细胞器”和“膜”类别也占有重要比例。说明参与滨麦叶片生理活动的分子组分主要集中在细胞的细胞器和膜系统上,这暗示滨麦叶片的膜组分在环境适应性方面发挥着重要作用。在“分子功能”大类共涉及 12个分类,其中“结合”类别和“催化活性”类别所涉及的Unigene数量最多,这说明这两类活动在滨麦叶片的生理活动中比较活跃。KEGG分析表明,滨麦叶片表达基因共涉及128个通路,其中所含Unigene最多的是“代谢通路”和“RNA运输”这两个通路,Unigene数量分别为5 168条(27.86%)和3 243条(17.48%)。另外,“次生代谢物的生物合成”、一些有机分子的代谢途径(包括甘油磷脂代谢、醚脂代谢、淀粉与蔗糖代谢和苯丙烷生物合成等)、“植物病原互作”和“植物激素信号转导”等途径在滨麦叶片细胞的生理活动中占有重要地位,所涉及到的Unigene数量也较多,这些次生代谢途径及信号途径均为植物体内重要的抗逆途径,能减轻或消除因干旱、盐碱和病虫害等逆境引起的活性氧伤害[13]。

NAC:NAC转录因子基因;DREB:DREB转录因子基因; NHX2:液泡膜Na+/H+反向转运蛋白基; SOS1:质膜Na+/H+反向转运蛋白基因;H+-ATPase I:质膜H+-ATP酶I亚基基因; POD21:过氧化物酶基因;TRX:硫氧还蛋白基因;HSP:热激蛋白基因;RcaB:二磷酸核酮糖羧化酶活化酶B基因;PP:蛋白磷酸化酶基因;Dehydrin:脱水素基因;TIP:液泡膜水通道蛋白基因。NAC:NAC transcription factor gene; DREB:Dehydration responsive element binding protein gene; NHX2:Vacuolar Na+/H+ antiporter 2 gene; SOS1:Plasma membrane Na+/H+ antiporter gene; H+-ATPase I:Plasma membrane H+-ATPase submit I gene; POD21:Peroxidase gene; TRX:Thioredoxin gene; HSP:17 kDa class I small heat shock protein gene; RcaB:Ribulose bisphosphate carboxylase activase B gene; PP:Protein phosphatase 2C gene; Dehydrin:Dehydrin gene; TIP:Tonoplast intrinsic protein.
滨海沙地盐渍化程度很高,其中Na+是主要的有害离子[22]。过多Na+的吸收会降低植物细胞中K+/Na+比例而引发离子胁迫[18,23]。降低根部对Na+的吸收、减少对地上部Na+的运输以及将地上部细胞中的Na+区隔化至液泡是植物常见的耐盐策略[24,25]。叶片液泡膜Na+/H+反向转运蛋白(NHX)能将细胞质基质中过多的Na+区隔化至液泡中,从而降低细胞质中Na+的浓度。这个过程由位于液泡膜上的H+-ATP酶或H+-焦磷酸化酶提供能量[23]。位于质膜上的Na+/H+反向转运蛋白(SOS1)依靠质膜上的H+-ATP酶水解ATP产生的能量将细胞基质中的Na+外排至胞外[26],减少Na+在胞质中的积累。在滨麦叶片转录谱中检索到注释为液泡膜Na+/H+反向转运蛋白的Unigene 9条、质膜Na+/H+反向转运蛋白Unigene 6条,暗示滨麦可能通过区隔化Na+至液泡及外排Na+至胞外这两种途径降低叶片细胞基质中Na+的含量,减少Na+的毒害。
盐渍环境中,离子胁迫和渗透胁迫会进一步引发氧化胁迫[18,23]。抗氧化酶(或蛋白)是植物体内消除活性氧、应对氧化胁迫的一类重要的抗氧化剂。本研究在滨麦叶片转录组中发现了175条响应氧化胁迫的Unigene,暗示滨麦叶片中的过氧化作用和抗氧化作用都比较活跃。其中,注释为过氧化物酶和谷胱甘肽巯基转移酶的Unigene数量多达50条和40条,硫氧还蛋白达37条,说明滨麦叶片细胞中过氧化物酶、谷胱甘肽巯基转移酶和硫氧还蛋白在抗氧化系统中发挥着重要作用。
为了验证转录组测序结果的准确性,本研究选择了12个基因进行qRT-PCR验证。结果显示,12个基因在滨麦叶片中均有表达,其中,TIP基因、转录因子NAC基因和质膜H+-ATPaseⅠ基因的表达水平相对较高,而转录因子DREB基因和 POD21基因的表达水平相对较低,该结果与转录组测序的结果基本一致,证明了转录组测序结果的准确性。
综上所述,本研究采用Illumina HiSeq 2000 高通量测序技术,首次对生长于滨海沙地的耐盐植物滨麦叶片进行了转录组分析,在无参考基因组的情况下,采用De novo测序策略构建了滨麦叶片转录组数据库,获得了大量的转录组信息。通过与已知的核酸、蛋白质数据库比对,得到功能注释成功的Unigene有59 380条,其中发现了大量参与Na+转运和抗氧化作用的基因。本研究获得的滨麦叶片转录组数据为进一步研究滨麦基因信息和功能、分析滨麦的耐盐机理提供了重要参考。
