内蒙古鄂尔多斯地区酸粥真菌多样性研究
2018-09-07王玉荣折米娜刘康玲张振东
王玉荣,折米娜,刘康玲,张振东,双 全*
(1.内蒙古农业大学 食品科学与工程学院,内蒙古 呼和浩特 010018;2.湖北文理学院 食品科学技术学院,鄂西北传统发酵食品研究所,湖北 襄阳 441053)
谷物发酵食品是以谷物为主要原料经微生物代谢活动以及酶与谷物各成分相互作用形成的一类特色食品,通过发酵,谷物的营养组分和产品特性可得到一定改善[1-2]。酸粥作为一种传统的谷物发酵类食品,在我国陕西、山西和内蒙古等地区有着悠久的制作和食用历史,其主要以糜米、小米和大米等谷物为原料经自然发酵制作而成[3]。目前对酸粥的研究多集中于制作工艺探究和采用传统微生物学手段对其所含乳酸菌和酵母菌进行分离鉴定,有关全面解析酸粥中真菌多样性的研究尚少。薛建岗等[4]对内蒙古西部地区酸粥样品微生物组成进行分析时发现该地区自然发酵酸粥细菌总数(7.67±1.23)lgCFU/mL、乳酸菌数(7.51±1.23)lgCFU/mL和酵母菌数(5.55±0.68)lgCFU/mL;李文亚等[5]对不同发酵时段的晋西北酸粥中酵母菌进行分离鉴定,发现东方伊萨酵母(Issatchenkia orientalis)、库德毕赤酵母(Pichia kudriavzevii)和酿酒酵母(Saccharomyces cerevisiae)存在于整个发酵过程中;GOBBETTI M等[6]利用合成培养基对乳酸菌和酵母菌进行共同培养时发现,发酵过程中酵母菌和乳酸菌存在共代谢关系;张春林[7]采用PCR-DGGE技术研究内蒙古传统发酵酸粥中微生物多样性,结果表明乳杆菌、一些未培养菌以及酵母菌为样品中主要的优势菌。
被称为下一代测序(next generation sequencing,NGS)的高通量测序技术(high-throughput sequencing),是从第一代测序技术发展而来的最新一代的测序方法,具有长读长、测序时间短以及数据准确率高等优点,克服了基于纯培养的传统微生物学手段操作繁琐、检出率低等缺陷,能够客观全面地反应微生物多样性[8-11]。目前该技术已被广泛应用于饮用水中微生物多样性分析[12-13]、发酵食品微生物多样性研究[14-15]以及环境微生物监测[16]等领域。然而,有关应用高通量测序技术评价酸粥中真菌多样性的研究少见报道。
因此,本研究采用Illumina Miseq高通量测序技术与传统微生物学手段相结合的方法对内蒙古鄂尔多斯地区酸粥样品中真菌多样性进行研究,利用多元统计学方法对研究结果进行解析,以期为酸粥中微生物多样性研究以及品质改良提供一定的理论依据。
1 材料与方法
1.1 材料与试剂
1.1.1 材料
酸粥样品:于2017年9月分别采集自内蒙古鄂尔多斯市、准格尔旗和达拉特旗,依次编号为A1、A2、A3,采集的样品置于加有冰盒的采样箱中运回实验室。
1.1.2 菌株
大肠杆菌(Escherichia coli)Top10:由民族特色食品研究室提供。
1.1.3 培养基
马铃薯葡萄糖琼脂(potato dextrose agar,PDA)培养基和酵母浸出粉胨葡萄糖(yeast extract peptone dextrose,YPD)培养基:青岛海博生物技术有限公司。
1.1.4 主要试剂
氯化钠、三氯甲烷、氢氧化钠、Tris碱、浓盐酸、乙二胺四乙酸二钠(ethylenediaminetetraaceticacid-2Na,EDTA-2Na)、三羟甲基氨基甲烷(Tris(hydroxymethyl)methylaminomethane,THAM)、乙酸钙、十二烷基硫酸钠(sodium dodecyl sulfate,SDS)、Tris饱和酚、氯仿、异戊醇、醋酸钠和乙醇(均为分析纯):国药集团化学试剂有限公司;5×TransStartTM、蛋白酶K(30 U/mg)、FastPfu Buffer、脱氧核糖核苷三磷酸(deoxyribonucleoside triphosphate,dNTP)Mix、FastPfu Fly 脱氧核糖核酸(deoxyribonucleic acid,DNA)Polymerase(5 U/μL)、PMD18-T Vector、Solution I和2×TaqMaster Mix:宝生物工程(大连)有限公司;6×Loadingbuffer和DL2000DNAMarker:宝日医生物技术(北京)有限公司;DNeasymericon Food Kit试剂盒:德国QIAGEN公司;Axygen清洁试剂盒:康宁生命科学(吴江)有限公司;引物SSU0817F/SSU1196R、NS1/NL4和M13F(-47)/M13R(-48):均由天一辉远生物科技有限公司合成。
1.2 仪器与设备
ND-2000C微量紫外分光光度计:美国NanoDrop公司;Veriti96孔梯度聚合酶链式反应(polymerasechainreaction,PCR)仪:赛默飞世尔科技(中国)有限公司;DYY-12电泳仪:北京市六一仪器厂;FC2化学发光凝胶成像系统:美国ProteinSimple公司;Miseq高通量测序平台:美国Illumina公司;HR40-IIB2生物安全柜:海尔集团电子商务有限公司;LRH-1000F生化培养箱:上海一恒科学仪器有限公司;PowerLyzerR24强力珠磨式破碎仪:美国MOBIO公司;CT15RE离心机:株式会社日立制作所;ECLIPSE Ci生物显微镜:日本Nikon公司。
1.3 方法
1.3.1 酸粥样品宏基因组的提取
参照QIAGEN DNeasymericon Food Kit试剂盒中的方法提取酸粥样品的总DNA,使用微量紫外分光光度计检测DNA的浓度及纯度,OD260nm/OD280nm值在1.8~2.0之间的视为合格,将合格DNA置于-20℃备用。
1.3.2 真菌18S rRNA V4-V5区PCR扩增及检测
引物:采用文献[17]中的引物对SSU0817F/SSU1196R。扩增时在正向引物中加入7个核苷酸通用标签用于区分不同样品:A1的标签为5'-TAGTGTG-3',A2的标签为5'-TCGTCAT-3',A3的标签为5'-TCATACA-3'。
PCR扩增体系:4 μL 5×PCR Buffer,2 μL dNTP Mix(2.5mmol/L),0.8μLSSU0817F(5μmol/L),0.8μLSSU1196R(5 μmol/L),0.4 μL rTaq(5 U/μL),模板DNA 10 ng,用无菌超纯水将体系补充至20 μL[18]。
PCR扩增条件:参照曹国君等[19]的方法调整为95℃预变性5 min;95℃变性1 min,55℃退火45 s,72℃延伸90 s,30个循环;72℃再延伸10 min。
琼脂糖凝胶电泳:扩增产物用1.5%的琼脂糖凝胶进行电泳(120 V,30 min),检测是否扩增出目的条带。
1.3.3 扩增产物纯化及定量
用PCR清洁试剂盒纯化扩增产物,并用无菌超纯水将产物浓度稀释至100 nmol/L后寄至上海美吉生物医药科技有限公司进行高通量测序。
1.3.4 生物信息学分析
参照CAPORASO J G等[20-22]的方法利用QIIME平台对序列进行拼接、质量控质及序列划分,建立分类操作单元(operational taxonomic units,OTU);然后利用RDP[23]和Greengenes数据库[24]对OTU进行分类学分析。
1.3.5 核酸登录号
本研究中所有序列数据已提交至MG-RAST数据库,ID号为mgp84785
1.3.6 酵母菌的分离纯化与鉴定
(1)酵母菌的分离纯化
采用稀释平板法将倍比稀释后的样液取10-1、10-2和10-3三个梯度涂布于PDA琼脂培养基上,28℃培养3~5 d后进行菌株纯化,并用30%的甘油将纯种菌株冻存于-80℃超低温保存箱中[25-26]。
(2)酵母菌的分子生物学鉴定
DNA提取:采用玻璃珠破碎法对纯化菌株进行DNA提取,每1 mL菌悬液中添加0.3 g直径为0.5 mm的玻璃珠,组织破碎仪的运行条件为每隔20s运行60 s,循环5次,频率为70 Hz[27]。
PCR扩增:引物为NS1(5'-GCATATCAATAAGCGGAGGAAAAG-3')和NL4(5'-GGTCCGTGTTTCAAGACGG-3')。PCR扩增体系为25μL,反应程序为94℃、4min;95℃、1min,50 ℃、45 s,72 ℃、1 min,30个循环;72 ℃、10 min[28]。
PCR扩增产物的验证、纯化与克隆:对PCR扩增产物进行验证、纯化,然后与载体TMD18-T连接,转化至大肠杆菌Top10中,将阳性克隆送至天一辉远生物科技有限公司进行测序。
系统发育树的构建:利用BioEdit7.1.3和DNAMAN7.0软件对返回的序列进行处理,并将去除引物的序列传至美国国立生物技术信息中心(national center of biotechnology information,NCBI)(https://blast.ncbi.nlm.nih.gov/Blast.cgi)上进行同源性比对,应用分离株和模式株序列构建系统发育树。
1.3.7 数据分析
采用α多样性分析法对测序深度进行评价,使用柱形图和热图展示不同分类水平下各门和属的相对含量,热图由Matlab2010b软件绘制;使用维恩(Venn)图展示不同样品中特有或共有的OTU数和序列数,Venn图由Venny2.1.0(http://bioinfogp.cnb.csic.es/tools/venny/index.html)在线绘制;系统发育树选用Mega7.0中的邻近(neighbor-joining,NJ)法进行1000次Bootstrap检验后构建;其他分析图使用Origin8.5和SAS9.0软件绘制。
2 结果与分析
2.1 序列丰富度和多样性分析
超1(Chao1)指数和香农(Shannon)指数是衡量群落物种总数和多样性的常用指数[29-30]。本研究首先依次在界、门、纲、目、科和属分类水平以及物种多样性指数上统计各样品中的真菌群落结构组成,结果如表1所示。

表1 3种酸粥样品的测序结果及各分类水平数量Table 1 Results of sequencing and numbers of different taxonomical levels about three kinds of acidic-gruel samples
由表1可知,3个酸粥样品共检测出98133条序列,平均每个样品测得32 711条序列。采用两步UCLUST法对其进行划分:根据100%相似度可得到15 521条序列,再经97%相似度分析可将这些序列划分到74个OTU中。值得注意的是,在测序深度为36 510条序列时,样品A2和A3的Chao1指数和Shannon指数均比A1样品大,说明A2和A3样品中真菌丰富度和多样性要比A1样品中的高。进一步采用香农指数曲线和稀疏曲线对样本进行分析,结果如图1所示。
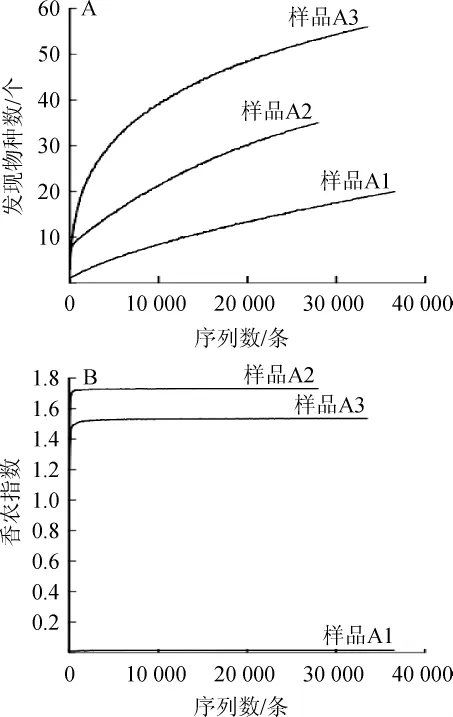
图1 3种酸粥样品的稀疏曲线(A)和香农指数曲线(B)Fig.1 Rarefaction curves(A)and Shannon index curves(B)of three kinds of acidic-gruel samples
由图1可知,在一定序列条数范围内,物种数(A)和香农指数(B)均随序列数的增加而增加,测序深度达到27000条序列左右时,稀疏曲线仍有上升趋势,而香农指数曲线早已进入平台期,说明平均每个样品32 711条序列的测序深度合适,满足后续分析要求。
2.2 基于各分类地位酸粥中真菌相对含量分析
经质控合格后,3种酸粥样品中测得的98 133条序列被鉴定为4个门、12个纲、11个目、14个科和18个属,仅有0.26%的序列不能鉴定到属水平。内蒙古鄂尔多斯地区酸粥样品中鉴定的4个真菌门分别为子囊菌门(Ascomycota)、担子菌门(Basidiomycota)、壶菌门(Chytridiomycota)和球囊菌门(Glomeromycota),平均相对含量>1.0%的真菌门和属构成如图2所示。
由图2(A)可知,酸粥样品中平均相对含量>1.0%的核心优势真菌门为Basidiomycota和Ascomycota,其中Basidiomycota的平均相对含量高达96.46%。由图2(B)可知,在属水平上,酸粥样品中平均相对含量>1.0%真菌属主要由隶属于Ascomycota的假丝酵母属(Candida)和耐碱酵母属(Galactomyces)以及隶属于Basidiomycota的毛孢子菌属(Trichosporon)构成,其平均相对含量分别为84.3%、11.86%和3.45%,由此可见,鄂尔多斯地区酸粥中的优势真菌主要为Candida、Galactomyces和Trichosporon。白梅等[31]研究结果与本研究相似,其采用传统微生物学手段从内蒙古地区28份酸粥样品中分离纯化出40株酵母菌,经分子生物学手段鉴定后发现在样品中出现频次较高的菌属及其频率依次为伊萨酵母属(Issatchenkia)75.00%、假丝酵母属(Candida)17.85%和毛孢子菌属(Trichosporon)10.71%。未分离出Galactomyces的菌株,这可能与制作酸粥的方法、原料以及菌株的特殊生长需求等因素有关。

图2 3种酸粥样品中核心门(A)和属(B)的相对含量Fig.2 Relative contents of the core phylum(A)and genus(B)in three kinds of acidic-gruel samples
在对97%的相似度下划分的74个OTU分析时发现,OTU68、OTU71、OTU43、OTU42、OTU2、OTU44、OTU61 和OTU73是本研究分析的酸粥样品中共有的操作分类单元,即核心OTU,其结果如图3所示。
由图3可知,这8个OTU无明显的聚类趋势,经Green genes和RDP数据库比对后,这些OTU依次被鉴定为未分类子囊菌纲(unclassified Sordariomycetes)、青霉属(Penicillium)、枝孢菌属(Cladosporium)、丝孢菌属(Tetracladium)、假丝酵母属(Candida)、假丝酵母属(Candida)、毛孢子菌属(Trichosporon)和未分类银耳纲(unclassified Tremellomycetes)。Candida在3种酸粥样品中的平均相对含量明显比其他属高,这与图2分析结果一致。
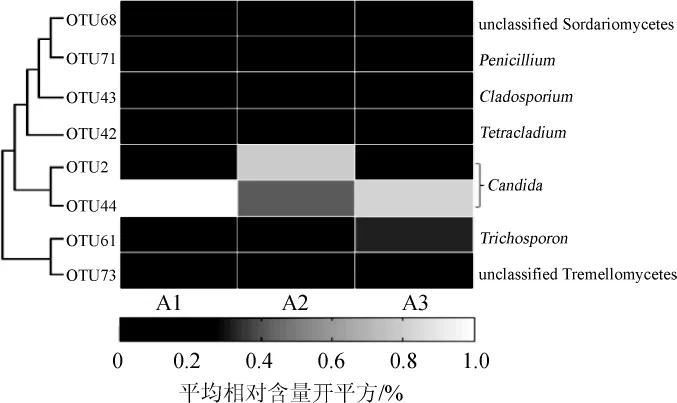
图3 3种酸粥样品中核心OTU相对含量的比较分析Fig.3 Comparative analysis on relative contents of core OTU in three kinds of acidic-gruel samples
OTU在3种酸粥样品中的出现次数统计结果如图4所示。

图4 OTU在3种酸粥样品中出现次数的统计Fig.4 Occurrence frequency statistics of OTU in 3 kinds of acidic-gruel samples
由图4可知,74个操作分类单元中仅出现一次的OTU有45个,占OTU总数的比例为60.81%,其包含序列数为897条;出现两次的OTU有21个,占OTU总数的28.38%,包含12 013条序列;出现三次的OTU仅有8个,占OTU总数的10.81%,包含85 223条序列。说明鄂尔多斯地区酸粥样品中存在着大量共有真菌菌群,同时各样本间微生物群落结构也存在较大差异。
采用Venn图对各样本中所含OTU及序列数进行分析,结果如图5所示。
由图5可知,3种酸粥样品A1、A2和A3各自特有的OTU数分别为5、12和28个,其所包含的序列数及占总序列数的比例分别为5条(0.0051%)、720条(0.73%)和172条(0.18%);A1和A2共有的OTU数为1个,序列数为634条,占总序列数的0.65%;A2和A3共有的OTU数为14个,序总列数为11 184条,占总序列数11.40%;A1和A3共有的OTU数为6个,序列数为195条,占总序列数的0.20%;A1、A2和A3共有的OTU数为8个,序列数为85223条,占总序列数的86.84%。由这些数据可直观的看出,各个样品都有自己独特的真菌菌落,但彼此间又有共有微生物,虽然3种酸粥样品共有的OTU较少,但其所含序列占总序列数的比例却极高,说明酸粥中核心真菌种类较多且基本一致。

图5 3种酸粥样品基于OTU水平的Venn图Fig.5 Venn diagram of three kinds of acidic-gruel samples based on OUT level
2.3 酸粥中酵母菌的分离及分子生物学鉴定
采用传统微生物学手段从3种酸粥样品中共分离纯化出9株酵母菌,编号分别为JA1-1、JA1-2、JA1-3、JA2-1、JA2-2、JA2-3、JA2-4、JA3-1和JA3-2,对其28S rRNA序列进行分析,构建系统发育树,结果如图6所示。

图6 基于28S rRNA序列分析的酵母菌的系统发育树Fig.6 Phylogenic tree of yeasts based on 28S rRNA sequence analysis
由图6可知,各菌株与其近源种间的自展值均为100,说明二者亲缘关系可靠,故判定菌株JA1-1和JA1-3为近平滑假丝酵母(Candida parapsilosis),菌株JA3-2为茶叶籽酵母(Meyerozyma caribbica),菌株JA1-2为索拉尼假丝酵母(Candida solani),菌株JA2-2和JA3-1为库德毕赤酵母(Pichia kudriavzevii),菌株JA2-1为白地霉(Galactomycescandidum),菌株JA2-4为解脂耶氏酵母(Yarrowia lipolytica),菌株JA2-3为隐球酵母(Cryptococcus albidus)。分离出的菌株中有2株隶属于Candida,1株隶属于Galactomyces,但未分离出Miseq高通量测序技术检测出的隶属于Trichosporon的菌株,这可能与该菌属特殊生长需求以及本研究所采用的培养方式和条件有关。
3 结论
本研究采用高通测序技术研究鄂尔多斯地区酸粥中真菌多样性,发现3种酸粥样品中核心优势真菌门为担子菌门(Basidiomycota)和子囊菌门(Ascomycota),优势真菌属分别为假丝酵母属(Candida)、耐碱酵母属(Galactomyces)和毛孢子菌属(Trichosporon),其平均相对含量分别为84.3%、11.86%、3.45%;在对各样品共有或特有的OTU进行分析时发现各个样品都有自己独特的真菌菌落,但彼此间又有共有微生物,这些核心真菌种类较多且基本一致。采用传统微生物学方法从3种酸粥样品中共分离出9株酵母菌,经分子生物学鉴定,近平滑假丝酵母(C.parapsilosis)2株,茶叶籽酵母(M.caribbica)、索拉尼假丝酵母(C.solani)、库德毕赤酵母(P.kudriavzevii)、为白地霉(G.candidum)、解脂耶氏酵母(Y.lipolytica)和隐球酵母(C.albidus)各1株。为酸粥中微生物群落结构多样性研究以及发酵剂的研发提供一定参考。
