海带hsp70基因的克隆、分析及转录水平定量研究
2018-08-31袁艳敏刘福利梁洲瑞汪文俊孙修涛王飞久
袁艳敏 刘福利 梁洲瑞 汪文俊 孙修涛 王飞久
海带基因的克隆、分析及转录水平定量研究
袁艳敏1,2刘福利2,3①梁洲瑞2汪文俊2孙修涛2王飞久2
(1. 上海海洋大学水产与生命学院 上海 201306;2. 农业农村部海洋渔业可持续发展重点实验室 中国水产科学研究院黄海水产研究所 青岛 266071;3. 青岛海洋科学与技术试点国家实验室 海洋渔业科学与食物产出过程功能实验室 青岛 266071)
本研究以海带()‘海天1号’为材料,通过RACE-PCR方法获得海带()基因全长cDNA序列。序列分析表明,全长cDNA长度为3778 bp,含有1个2892 bp的开放阅读框,5ʹ和3ʹ非编码区的长度分别为101、785 bp。蛋白质氨基酸含量和理化性质分析显示,该蛋白为稳定蛋白。应用BLASTP程序对不同物种HSP70蛋白比较分析表明,海带与长囊水云()的HSP70蛋白具有较高的同源性,用HSP70蛋白构建的进化树显示,海带与长囊水云聚为一类。二级结构预测该蛋白由963个氨基酸组成,包含35个α螺旋和25个β折叠。以酿酒酵母()HSP110蛋白三维结构3C7N为模板,在I-TASSER软件上预测海带HSP70蛋白的三维结构,预测的蛋白结构与3C7N的A链在折叠和二级结构方面非常类似。对基因在温度胁迫下表达变化分析表明,高温对的表达量具有显著影响,在25℃、24 h胁迫条件时,表达量达到最大值。本研究为阐明海带HSP70蛋白特性及其应对环境胁迫的机理提供理论依据。
海带‘海天1号’;HSP70;RACE;生物信息学分析;荧光定量PCR
热休克蛋白(Heat shock proteins, HSPs)是由暴露于应激条件下的细胞产生的,结构上高度保守,在原核和真核细胞中广泛存在的一类蛋白。HSPs首次应用于描述果蝇()的热休克反应,从此,HSPs开始受到广泛的关注和研究(Wang, 2004; 栗振义等, 2016)。HSP在正常和应激情况下均发挥重要的生理功能,但在胁迫条件包括热激、物理、化学和生物等胁迫因子下表达量显著增加(齐妍等, 2013)。按照分子量的不同,HSPs分为HSP100、HSP90、HSP70、HSP60以及小分子热休克蛋白(Waters, 1996)。其中,HSP70是研究较多、较深入的蛋白,在逆境环境下,该蛋白具有分子伴侣的功能,参与新生肽的成熟及蛋白质变性后的复性、降解,维持细胞内环境的稳定,进而对细胞具有保护作用(刘伟等, 2012)。近年来,已从多种藻类中克隆获得了序列,对多种藻类基因的结构和功能、调控机理等进行了研究,Muller等(1992)在研究单细胞藻莱茵衣藻()基因的结构时发现,其不同于高等植物基因的结构,说明在不同的生物体中,基因具有序列的多样性。
海带()是一种冷温性海藻,是海洋生态系统中的重要组成成分,也是我国最早开展全人工养殖和育苗的经济海藻,为我国水产养殖发挥重要贡献(王飞久等, 2008)。海带‘海天1号’是南方耐高温品系与北方高产品系经过杂交后,再通过连续自交几代后,选育获得的海带新品系,其具有产量高、耐高温、成熟稍早等特点(姚海芹等, 2016)。本研究以海带‘海天1号’为材料,通过RACE方法获得全长cDNA序列,根据序列信息研究其蛋白质结构和性质,并通过荧光定量PCR分析温度对基因表达量的影响,以期为海带耐高温的分子机制研究提供理论依据。
1 材料与方法
1.1 材料处理
2015年11月12日,海带‘海天1号’采自山东荣成青鱼滩养殖海域,挑选大小均匀、健康无损伤的个体,长度约为5~6 cm。将取回的海带幼苗在温度为13℃、光强为30~50 μmol photons/(m2·s)、光周期为12L : 12D条件下,培养1 d,培养液为含有NaNO3和KH2PO4溶液的灭菌海水,N浓度为3 mg/L,N : P= 10 : 1,充气悬浮培养。在高温(25℃)分别处理0、3、6、12、18、24、30、36 h,以0为参考,比较基因在高温胁迫不同时间的表达情况。在不同温度(2℃, 6℃, 10℃, 14℃, 18℃, 22℃, 25℃)时,处理3 h,以10℃为参考,比较基因在不同温度的表达情况。
1.2 总RNA提取
采用RNeasy Plant Mini Kit(QIAGEN)进行海带总RNA的制备和提取,方法参照试剂盒说明书。制备后的RNA经过琼脂糖(1.2%)凝胶电泳检测,检查其完整性,并在A260: A280条件下,检测总RNA的纯度。
1.3 RACE法克隆Sjhsp70 cDNA全长序列
5ʹ-RACE和3ʹ-RACE模板采用Gene RacerTMKit (Invitrogen)进行合成。根据本实验室转录组测序数据库中的海带基因序列,采用Primer Premier 6.0软件分别设计5ʹ-RACE引物HSP70-R1、HSP70-R2和3ʹ-RACE引物HSP70-F1、HSP70-F2(表1),由生工生物工程(上海)股份有限公司进行合成。所用的其他引物序列见表1。根据RACE试剂盒说明,分别使用PCR扩增得到5ʹ末端和3ʹ末端,采用琼脂糖凝胶(1.5 %)电泳对PCR产物进行分析。确定目的条带后切胶回收,连接于pUCm载体,进行转化感受态细胞DH5α,挑选阳性克隆,送往生工生物工程(上海)股份有限公司进行测序,根据测序序列拼接获得全长cDNA。
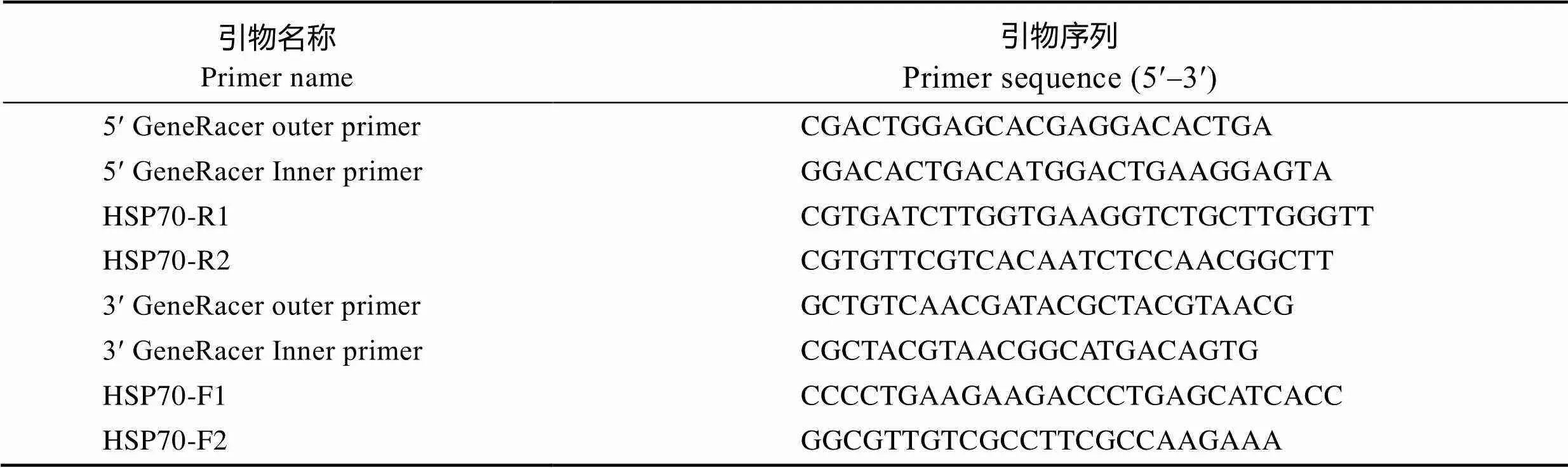
表1 引物名称及序列
1.4 生物信息分析
开放阅读框(ORF)识别:利用NCBI中的ORF Finder (http:www.ncbi.nlm.nih.gov/gorf/gorf.htm1)寻找基因序列完整的ORF。
蛋白质组分分析:利用ProtParam (http://web. expasy.org/protparam/)在线工具分析HSP70蛋白性质的基本参数。用NetPhos 2.0 (http://www.cbs.dtu.dk/ services/NetPhos/)在线工具预测蛋白磷酸化位点。
多序列比对和进化树构建:用BLASTP程序寻找海带基因的同源序列,再用CLC Genomics Workbench 8软件对获得的同源序列进行多序列比对。根据多序列比对结果,用MEGA5.2软件中的Neighbor- Joining法进行进化树构建。
蛋白质二级结构预测:利用CLC Genomics Workbench 8软件进行蛋白质二级结构分析来预测其二级结构及序列中含有的蛋白激酶C磷酸化位点、N端糖基化位点,并在二级结构图中标注。
蛋白三维结构的预测:目标蛋白三维结构基于同源建模的I-TASSER在线软件(http://zhanglab.ccmb.m ed.umich.edu/I-TASSER/)进行预测。
1.5 定量PCR分析
采用qPCR技术,分析在不同温度及不同高温诱导下mRNA的转录差异,选择α-微管蛋白(α-Tubulin)为内参基因,与内参基因参照引物序列见表2。荧光qPCR反应体系为2 × SYBR Green Mix,25 μl;PCR正向引物,1 μl;PCR反向引物,1 μl;模板cDNA,5 μl;ddH2O,18 μl,总反应体系为50 μl。针对每个模板的每对引物设置3个重复以及1个阴性对照。荧光定量PCR反应程序: 95℃预变性10 min后,按照95℃,30 s;53℃,30 s;72℃,1 min的反应程序进行40个循环。融解曲线反应程序:由60℃递进升温到95℃步进0.5℃,恒温10 s。用2–DD法计算的相对表达量。
2 结果与分析
2.1 Sjhsp70全长cDNA克隆及分析
利用RNeasy Plant Mini Kit提取海带总RNA,A260: A280比值为1.92,表明所制备的海带RNA纯度较高。琼脂糖凝胶电泳检测28 s和18 s 2条主要条带清晰明显,表明获得的海带总RNA完整性较好(图1A)。用特异性引物与Gene Racer引物分别进行5ʹ-RACE和3ʹ-RACE扩增,1.5%琼脂糖凝胶电泳检测表明,5ʹ-RACE PCR特异性条带大小为250 bp左右(图1B),3ʹ-RACE PCR特异性条带大小为2000 bp左右(图1C),分别测序并拼接后获得全长cDNA序列,含有一个2892 bp长的开放阅读框,5ʹ和3ʹ非编码区的长度分别为101、785 bp,Poly A尾巴信号肽区域位于3765~3770 bp处。将序列提交至NCBI数据库,序列号为KY986415。
2.2 SjHSP70理化性质分析
使用ProtParam对编码的SjHSP70蛋白进行分析,SjHSP70含有963个氨基酸,其分子量为103290.11 Da,理论等电点为4.83。该蛋白含有163个带负电荷的氨基酸残基和118个带正电荷的氨基酸残基,它的N端以甲硫氨酸起始。该蛋白脂肪族指数为79.00,疏水性指数为-0.369,不稳定性指数为33.12,可归类为稳定性蛋白。
2.3 SjHSP70序列分析
对SjHSP70的氨基酸序列进行BLAST分析。结果显示,SjHSP70与其他物种的HSP蛋白具有较高的同源性。其中,与长囊水云的HSP70蛋白的相似度达到了76.69%,与单胞藻()的HSP70蛋白相似度为44%,说明本研究获得的基因序列确为热休克蛋白家族的编码基因。对SjHSP70序列进一步分析发现,SjHSP70包括N末端核酸结合区、底物结合区和C末端,N末端核酸结合区位于第31~418个氨基酸之间,底物结合区位于第426~598个氨基酸之间。HSP70蛋白N末端氨基酸序列高度保守,具有结合并水解ATP的活性中心。应用SignalP 4.1软件分析发现,SjHSP70的N端具有一段28个氨基酸的信号肽,且SjHSP70的C末端氨基酸序列为EDEL,据此判断SjHSP70可能位于内质网内。
本研究得到的SjHSP70与之前报道的海带HSP70 (Fu, 2009)相似度约为30%,其中,2个蛋白的前600个氨基酸序列相似度约为40%,说明海带中HSP70的N端的保守性更强。为了比较SjHSP70与其他在大型海藻中报道的HSP70,从NCBI数据库中下载了裙带菜()、坛紫菜()、浒苔(sp)等12种海藻的HSP70序列(图2),对其进行多序列比对分析发现, SjHSP70与长囊水云的HSP70蛋白的相似度达到了76.69%,与其他大型海藻的HSP70相似度在30%~ 40%之间,进化树显示,SjHSP70与长囊水云HSP70聚为单独的一枝,与其他海藻中的HSP70的距离较远。
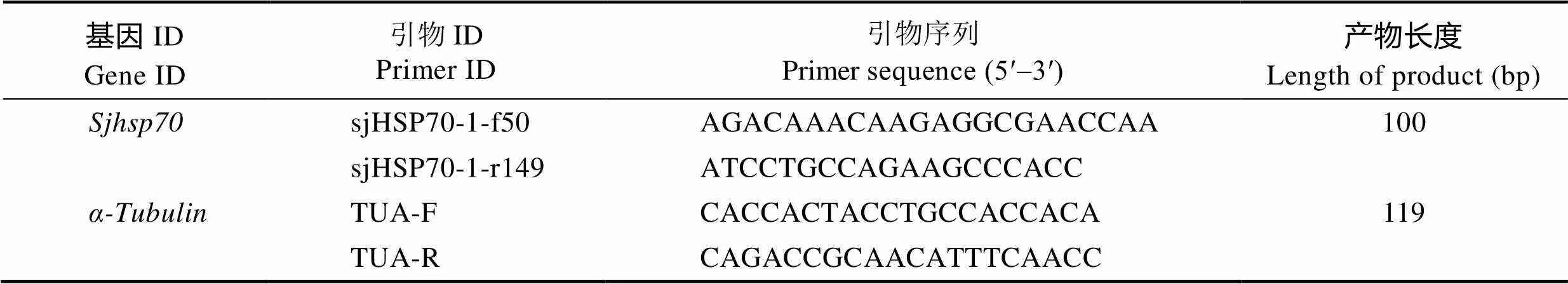
表2 Sjhsp70定量PCR引物与内参基因引物序列
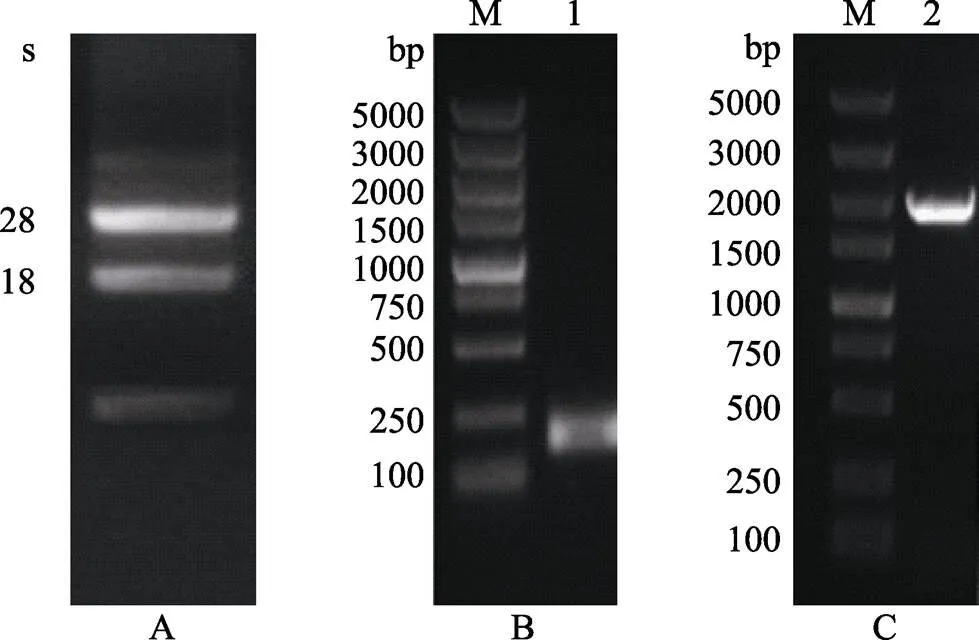
图1 Sjhsp70总RNA和RACE电泳
A:海带总RNA电泳;B:5ʹ-RACE PCR电泳; C:3ʹ-RACE PCR电泳;M:DNA分子量标准;泳道1:5ʹ-RACE PCR电泳结果;泳道2:3ʹ-RACE PCR电泳结果
A: Total RNA electrophoresis patterns; B: 5ʹ-RACE PCR electrophoresis patterns; C: 3ʹ-RACE PCR electrophoresis patterns; Lane M:DL5000 Marker; Lane 1:70 5ʹ-RACE PCR results; Lane 2:3ʹ-RACE PCR results
2.4 SjHSP70蛋白结构分析
应用CLC Genomics Workbench软件进行SjHSP70蛋白的二级结构预测,该蛋白由963个氨基酸组成,包含35个α螺旋和25个β折叠。氨基酸序列中的蛋白激酶C磷酸化位点和糖基化位点见图3。从图3可以看出,应用NetPhos工具预测蛋白质中丝氨酸、苏氨酸和络氨酸的磷酸化位点,在HSP70蛋白质中共预测出23个丝氨酸磷酸化位点,12个苏氨酸磷酸化位点和8个络氨酸磷酸化位点。
海带SjHSP70和酿酒酵母() HSP110分别有963和650个氨基酸,酿酒酵母HSP110蛋白在PDB数据库里的ID为3C7N。海带SjHSP70的三维结构(图4)是以3C7N为模板在I-TASSER软件上预测的,该蛋白从红色的碳端到蓝色的氮端,蛋白结构C值为–2.28,表示其与3C7N的A链在折叠和二级结构方面非常类似。目标蛋白SjHSP70和模板蛋白酿酒酵母HSP110结构相似性的TM值和RMSD分别为0.662和0.96Å。
2.5 温度诱导下Sjhsp70基因的转录分析
用荧光定量PCR对基因受高温(25℃)胁迫在不同时间和不同温度时,处理3 h的转录水平进行分析,结果见图5和图6。从图5可以看出,高温(25℃)胁迫时,24 h内随着胁迫时间的延长,的表达量逐渐提高,在24 h时达到表达量的最大值,12 h前增加速度较快,12~24 h间增加速度缓慢,24 h后表达量逐渐降低,原因可能是的表达具有时效性,在温度胁迫短期时间内大量表达,长时间胁迫会超出海带自身的调控能力,从而导致表达量降低。
从图6可以看出,不同温度处理海带幼孢子体,转录受到明显的影响,以海带生长的适宜温度10℃为参考,温度小于10℃和大于10℃都会使得转录上调,小于10℃时,上调不显著,大于10℃时,温度越高表达量越高,25℃时表达量最高,高温胁迫比低温胁迫对表达的影响更显著。

图2 基于HSP70氨基酸序列构建的NJ系统进化树
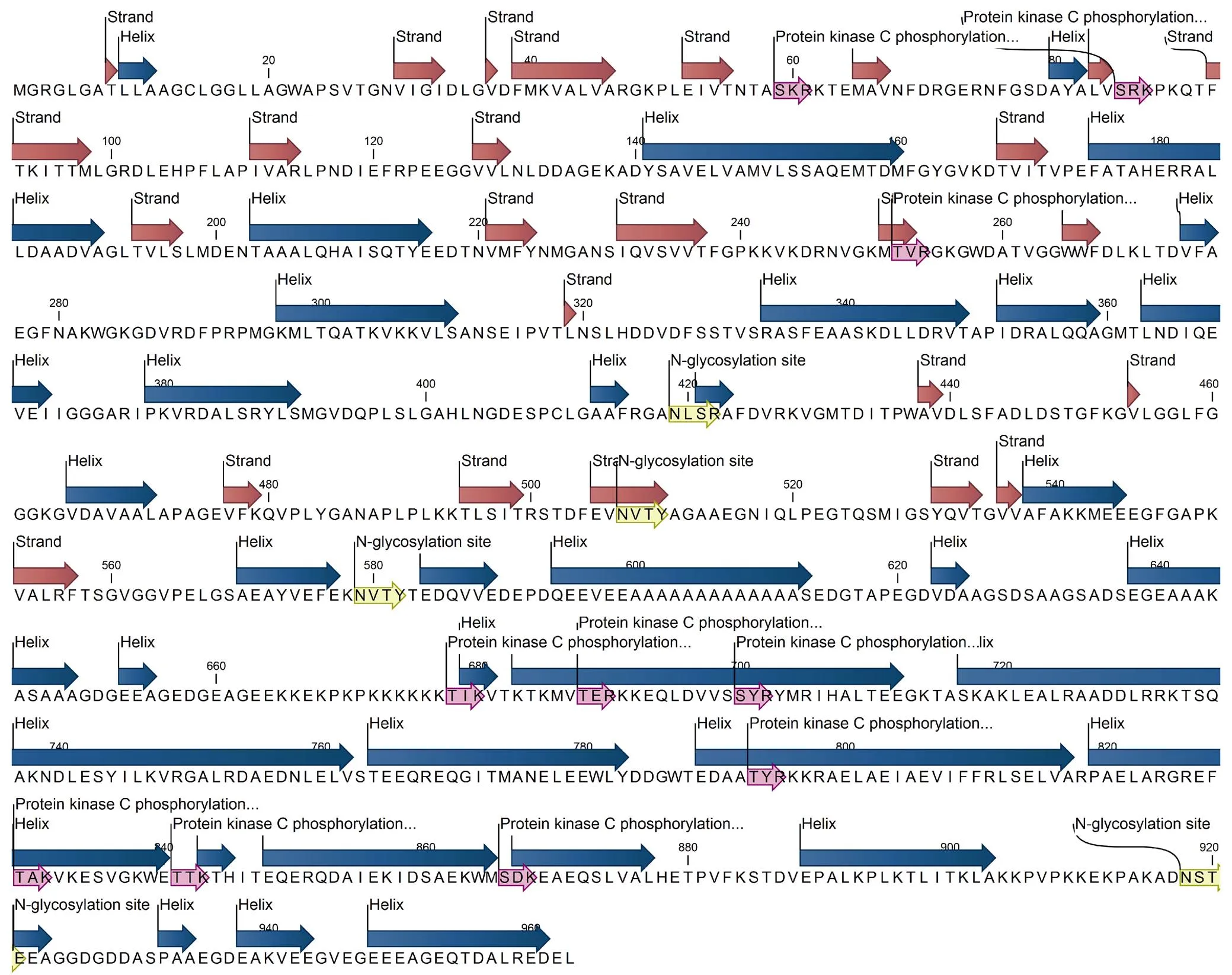
图3 SjHSP70蛋白质二级结构预测及功能位点注释
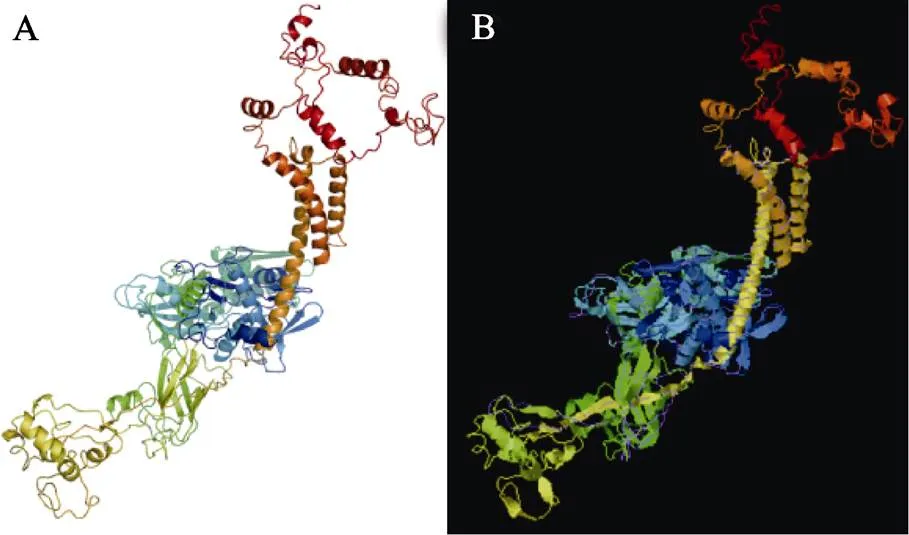
图4 SjHSP70蛋白的预测三维结构图及其与模板碳主链的重叠
A:SjHSP70的三维结构;B:SjHSP70结构和酿酒酵母HSP110蛋白碳主链的重叠。彩色结构的是海带HSP70;紫色线是酿酒酵母HSP110碳主链
A: Predicted 3D structure model of SjHSP70; B: Superimposed prediction model and native cartoon structures of SjHSP70 and HSP110 of. Rainbow structure was represen ted of the SjHSP70. Purple line is displayed using the alpha carbon backbone trace ofHSP110
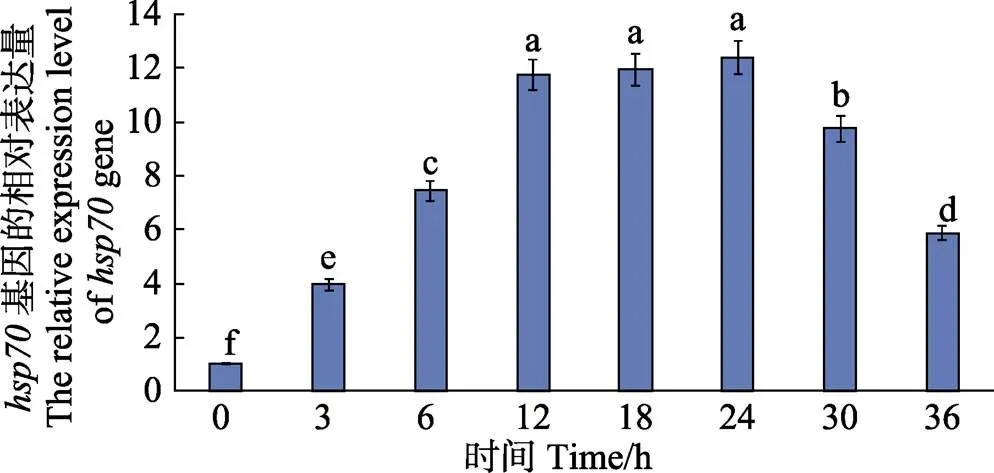
图5 Sjhsp70在高温(25℃)诱导下的转录水平变化
注:不同字母表示差异显著(<0.05)
Note: Different letters represent significant difference (<0.05)
3 讨论
大量研究表明,基因在调控植物抗逆过程中发挥重要作用,植物在受到不利条件,如高温逆境胁迫时,会上调的表达量,增强对高温胁迫的抵抗力,其机制可能是HSP70 蛋白可以参与新生蛋白折叠、稳定蛋白正常构象以及协助变性蛋白降解等(Wang, 2004; 王明强等, 2015)。Fu等(2009)研究表明,海带中基因表达受温度调控。本实验室在之前的海带转录谱研究发现,多个基因在高温胁迫下表达上调(Liu, 2014)。本研究克隆了其中的1个基因,该基因编码的SjHSP70蛋白含有963个氨基酸。与之前报道的海带中HSP70蛋白(Fu, 2009)相比,多出306个氨基酸。二者都具有HSP70蛋白特有的核酸结合区、铰链区、底物结合区以及C末端区,其中,在N端尤其是ATP结合位点位区,二者相似度高,在铰链区、底物结合区和C末端,二者的相似度较低。本研究获得SjHSP70初步判定位于内质网内,而之前报道的海带HSP70 (Fu, 2009)是胞质蛋白,二者可能具有不同的生理功能,作用的底物可能不同,故二者底物结合区的相似性较低。
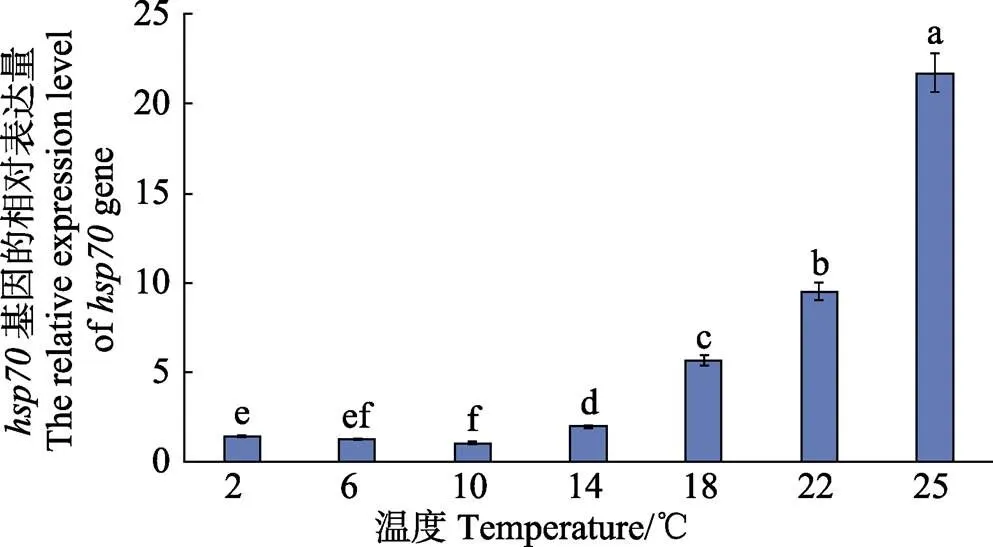
图6 Sjhsp70在不同温度下的转录水平
根据HSP70的表达类型,可分为组成型 HSP70和诱导型HSP70,前者在正常条件下就有本底表达,以维持细胞正常生命活动的需要,而后者在受环境因素胁迫时诱导表达(Swindell, 2007)。本研究发现,基因在正常生长条件下有微量表达,受到高温和低温诱导时,都会表达上调,尤其是受到高温胁迫时,表达量会大幅增加,25℃时的表达量是10℃的21倍之多。在受到高温胁迫(25℃)时,24 h后表达量达到顶峰,超过24 h后,表达量又开始降低。之前报道青岛养殖的海带受到热激处理时,其在25℃处理7 h后,表达量即可达到最大值(Fu, 2009)。二者差异的原因,可能是‘海天1号’的高温耐受性比青岛地区养殖海带的高温耐受性强,可以在高温胁迫下,持续高表达热休克蛋白,以提高自身的耐受性。
本研究克隆获得了1个海带基因,定量了该基因在海带高温胁迫时的表达特征,并探讨了其对调控海带高温耐受性的作用。下一步工作将继续研究在具有不同高温耐受性海带材料中的序列多样性,同时,分析序列多样性与高温耐受性之间的相关性,相关结果将为海带耐高温机制的研究提供重要的理论依据,也为海带耐高温新品种的培育打下基础。
Fu WD, Yao JT, Wang XL,. Molecular cloning and expressi on analysis of a cytosolic Hsp70 gene from(Laminariaceae, Phaeophyta). Marine Biotechnoloy, 2009, 11 (6): 738–747
Li ZY, Long RC, Zhang TJ,. Research progress on plant heat shock protein. Biotechnology Bulletin, 2016, 32 (2): 7–13 [栗振义, 龙瑞才, 张铁军, 等. 植物热激蛋白研究进展. 生物技术通报, 2016, 32 (2): 7–13]
Liu FL, Wang WJ, Sun XT,. RNA-Seq revealed complex response to heat stress on transcriptomic level in(Laminariales, Phaeophyta). Journal of Applied Phycology, 2014, 26(3): 1585–1596
Liu W, Yang R, Xu LN,. Cloning and expression offor. Journal of Ningbo University (Natural Science and Engineering Edition), 2012, 25(2): 17–25 [刘伟, 杨锐, 徐丽宁, 等. 坛紫菜基因克隆与表达. 宁波大学学报(理工版), 2012, 25 (2): 17–25]
Muller FW, Igloi GL, Beck CF. Structure of a gene encoding heat-shock protein Hsp70 from the unicellular alga. Gene, 1992, 111 (2): 165–173
Qi Y, Xu ZS, Li PS,. Research progress on molecular mechanism and application of Hsp70 in plants. Journal of Plant Genetic Resources, 2013, 14 (3): 507–511 [齐妍, 徐兆师, 李盼松, 等. 植物热激蛋白70的分子作用机理及其利用研究进展. 植物遗传资源学报, 2013, 14 (3): 507–511]
Swindell WR, Huebner M, Weber AP. Transcriptional profiling of Arabidopsis heat shock proteins and transcription factors reveals extensive overlap between heat and non-heat stress response pathways. BMC Genomics, 2007, 8 (1): 125
Wang FJ, Sun XT, Gao CR. Cultivation ofcultivars and non-polluted culture: One. Scientific Fish Farming, 2008, 85 (7): 14–16 [王飞久, 孙修涛, 高淳仁. 海带苗种培育及无公害养殖技术(一). 科学养鱼, 2008, 85 (7): 14–16]
Wang MQ, Zhang DY. Research advance of heat shock protein 70 gene family and its biological functions in plant. Genomics and Applied Biology, 2015, 34 (2): 421–428 [王明强, 张道远. 植物热激蛋白70基因家族及其生物学功能研究进展. 基因组学与应用生物学, 2015, 34 (2): 421–428]
Wang WX, Vinocur B, Shoseyov O,. Role of plant heat-shock proteins and molecular chaperones in the abiotic stress response. Trends in Plant Science, 2004, 9(5): 244– 252
Waters ER, Lee GJ, Vierling E. Evolution, structure and function of the small heat shock proteins in plants. Journal of Experimental Botany, 1996, 47 (3): 325–338
Yao HQ, Liang ZR, Liu FL,. Preliminary studies on the photosynthetic and respiration rate of young sporophyte of a newvariety “Haitian No. 1” using liquid-phase oxygen measurement system. Progress in Fishery Sciences, 2016, 37 (1): 140–147 [姚海芹, 梁洲瑞, 刘福利, 等. 利用液相氧电极技术研究“海天1号” 海带幼孢子体光合及呼吸速率. 渔业科学进展, 2016, 37 (1): 140–147]
(编辑 陈严)
Cloning, Bioinformatics and Quantitative Expression Analysis ofin
YUAN Yanmin1,2, LIU Fuli2,3①, LIANG Zhourui2, WANG Wenjun2, SUN Xiutao2, WANG Feijiu2
(1. College of Fisheries and Life Science, Shanghai Ocean University, Shanghai 201306; 2. Key Laboratory of Sustainable Development of Marine Fisheries, Ministry of Agriculture and Rural Affairs, Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Qingdao 266071;3. Laboratory for Marine Fisheries Science and Food Production Processes, Pilot National Laboratory for Marine Science and Technology(Qingdao), Qingdao 266071)
HSP70s are heat shock proteins that exist widely in prokaryotes and eukaryotes, and play important roles in biological resistance.The full cDNA sequence of agene in() of 3778 bp was obtained by RACE technology.consisted of an open reading frame (ORF) of 2892 bp, a 5ʹ untranslated region (UTR) of 101 bp, and a 3ʹ UTR of 785 bp. Analysis of amino acid content and physicochemical properties showed that SjHSP70 is a stable protein. Comparison between the amino acid sequences of SjHSP70 and the well-characterized orthologous HSP70 proteins from various species indicated that SjHSP70 shares the highest similarity with the orthologous protein from. The phylogenetic tree of the examined proteins indicated that SjHSP70 formed a group with a HSP70 from. Secondary structure analysis of SjHSP70 showed thatit is composed of 35 α-helices and 25 β-strands. Three-dimensional homology structure modeling of SjHSP70 was predicted by the I-TASSER server according to the 3C7N template. Overall, the folding and secondary structures of SjHSP70 were highly similar to those of 3C7N.To study the correlation between SjHSP70 and the resistance to high-temperature stress in, real-time variation in the expression ofunder different temperatures and different stress times was determined. The results showed that high temperature had a significant effect on the expression of. The expression ofcontinued to increase with increase in temperature, with the maximumexpression at 25℃ after 24 h of high-temperature stress. This result indicates thatis probably related to high-temperature resistance in. This study provides a foundation for the characterization of SjHSP70 and for the study of SjHSP70 function in the high-temperature stress response in.
‘Haitian No.1’; HSP70; RACE;Bioinformatics analysis;Quantitative real-time PCR
LIU Fuli, E-mail: liufl@ysfri.ac.cn
10.19663/j.issn2095-9869.20170407001
Q75; S917
A
2095-9869(2018)04-0152-07
* 中国水产科学研究院基本科研业务费专项(2016PT03)、现代农业产业技术体系藻类体系离岸式养殖岗位专项(CARS-50)和青岛市民生科技计划项目(17-3-3-65-nsh)共同资助[This work was supported by the Central Public-Interest Scientific Institution Basal Research Fund, CAFS(2016PT03), China Agricultural Research System-Algae Offshore Aquaculture (CARS-50), and Qingdao City Science and Technology Project (17-3-3-65-nsh)]. 袁艳敏,E-mail: yuanym2991@163.com
刘福利,副研究员,E-mail: liufl@ysfri.ac.cn
2017-04-07,
2017-05-08
袁艳敏, 刘福利, 梁洲瑞, 汪文俊, 孙修涛, 王飞久. 海带基因的克隆、分析及转录水平定量研究. 渔业科学进展, 2018, 39(4): 152–158
Yuan YM, Liu FL, Liang ZR, Wang WJ, Sun XT, Wang FJ. Cloning, bioinformatics and quantitative expression analysis ofin. Progress in Fishery Sciences, 2018, 39(4): 152–158
