人端粒酶RNA CR4/5结构域的X射线小角散射分析
2018-08-15杨卓茹张敏敏蔡汝洁张含笑管俊豪AltafAhmedSimair陆昌瑞
杨卓茹, 张敏敏, 蔡汝洁, 张含笑, 管俊豪, Altaf Ahmed Simair, 陆昌瑞
(东华大学 化学化工与生物工程学院, 上海 201620)
端粒酶是一类独特的核糖核蛋白复合体,主要负责保护染色体末端端粒的完整性和稳定性[1-6]。端粒酶包含两个主要部分:蛋白催化亚基端粒酶逆转录酶(TERT)和RNA亚基(TR)。不同物种的端粒酶TR在大小和序列上均有差异[7],根据野生型和突变型TR的二级结构系统发育比较揭示了已发现的所有物种中都存在两个催化功能区域:template/pseudo-knot (t/PK,core 结构域)结构域、CR4/5结构域[8]。t/PK结构域包含一个模板和一个封闭的、带有保守嘧啶三重螺旋的假结结构[9-11],CR4/5结构域主要与TERT的RNA结合结构域(TRBD)连接,刺激端粒酶催化活性[12]。在已发现的物种中,CR4/5结构域通常含有催化作用的一个三通结构,由茎P5,P6、茎环P6.1和内部环组成。而hTR除了这部分结构以外,还有一个由P6额外的螺旋P6b扩展形成的线性P6a-P6b结构[13],但P6b不是端粒酶催化活性所必须的[10],所以本实验在设计序列时,只保留了对端粒酶有催化活性的部分。然而,现阶段RNA的功能研究很少,运用结构生物学来进行研究RNA的功能将显得尤为重要。目前只有四膜虫和青鳉[14]的CR4/5结构域已经被解析出来,而hTR CR4/5的结构研究遇到了瓶颈,至今三维结构仍没有解析出来。
随着同步辐射光源的发展,X射线小角散射(SAXS)技术应用越来越广泛。相比于X射线晶体衍射,SAXS的样品为溶液,不需要结晶且时间短,因此利用SAXS技术解析生物大分子结构变得非常有意义。SAXS[15-17]是目前发展最快的一种探究生物大分子三维结构的技术,其原理是当X射线照射到样品上时,如果样品内部存在纳米尺度的电子密度不均匀区,则会在入射光束周围的小角范围出现散射X射线的现象。SAXS不仅能够提供生物大分子的低分辨率三维结构信息,而且还可以用于动态学方面的研究。因此,作者采用SAXS技术并成功解析了hTR CR4/5在溶液中的构象。
1 材料和方法
1.1 实验材料
hTR CR4/5重组质粒由上海生工生物公司合成。NTP,dNTP,甲酰胺购于生工生物;Taq酶及T7酶由本实验室自己制备;spermine购于sigma生物公司;其他试剂均为国产分析纯。
主要仪器:离心机,凝胶成像仪(BIO-RAD),电泳仪(BIO-RAD)等。
1.2 实验方法
1.2.1 hTR CR4/5原始序列的基因设计
在设计hTR CR4/5序列时保留了P5、J5/6、J5/6.1、P6.1、P6a,去掉了P6b,在第266个和第291个核苷酸之间用GAAA序列连接起来形成一个loop环。修改后的hTR CR4/5重组质粒由上海生工生物公司合成。
1.2.2 hTR CR4/5体外转录及RNA的纯化
首先,目的基因通过PCR扩增获得足量的PCR产物,然后进行体外转录(TCX)实验获得RNA产物,最后用12%的变性聚丙烯酰胺凝胶以及后续的离心浓缩来获得目的RNA。
1.2.3 hTR CR4/5的SAXS方法
利用上海蛋白质同步辐射光源(五站六线)BL19U2站线进行散射,制备不同浓度的hTR CR4/5样品和相应的缓冲液放入仪器中,使用 RAW完成从2D图像到1D数据的转换工作,Primusqt对1D数据的处理,最后利用Dammin进行3D建模。
2 结果
2.1 hTR CR4/5的PCR结果
图1为50 μL体系RCR实验结果。从结果来看,没有拖尾、没有非特异性及其他杂带,在进行PCR条件优化后就可用于体外转录实验。
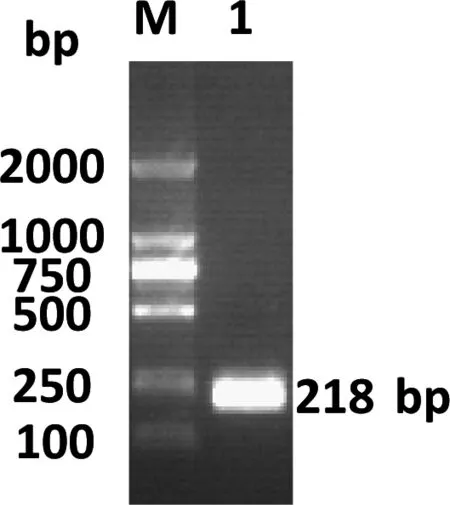
图1 hTR CR4/5的 PCR实验结果(1%琼脂糖凝胶)
M:DNA marker;泳带1:hTR CR4/5质粒的PCR产物
2.2 hTR CR4/5的体外转录及纯化结果
运用文献[18]的体外转录体系后,发现目的基因的浓度较低。所以后续实验对体外转录体系进行了优化,并发现增大NTP和Mg2+含量能有效地提高目的基因的浓度。根据体外转录,hTR CR4/5条带能够被HDV核酶完全剪切,并且hTR CR4/5的浓度也较高。切下的目的基因条带经离心浓缩后得到目的RNA。从图2可以看出,在hTR CR4/5稀释100倍后条带依然清晰,无降解,测定RNA浓度为2.6 mg/mL。
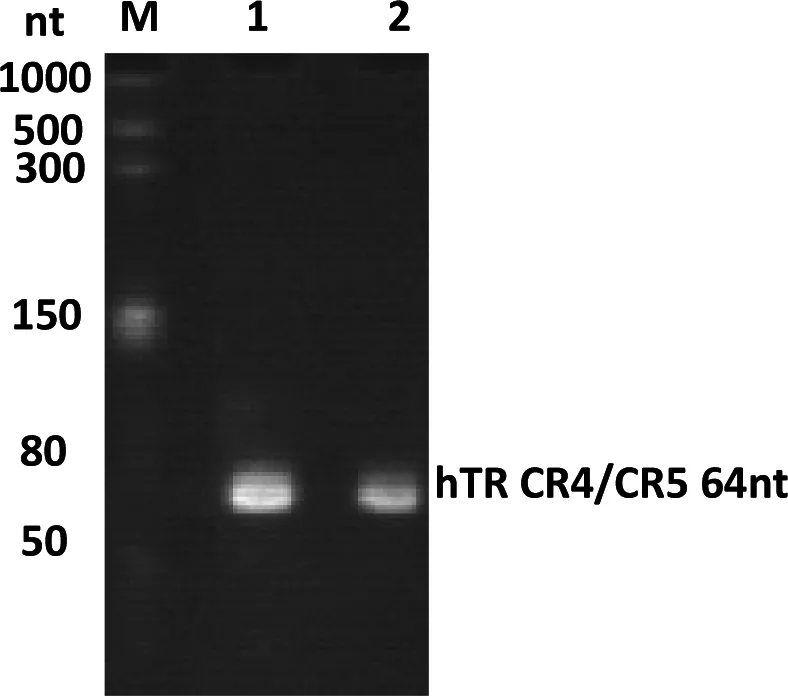
图2 hTR CR4/5纯化结果(12%变性聚丙烯酰胺凝胶)
M:RNA marker, 泳带1:纯化后的hTR CR4/5(稀释 50倍);泳带2:纯化后的hTR CR4/5(稀释100倍)
2.3 hTR CR4/5 的SAXS结果
从不同浓度(3.1、4.3、6.8和10.5 mg/mL)的hTR CR4/5和相应缓冲液的SAXS结果(图3-A)可以看出,有一个样品(紫红色曲线)与其他3个样品曲线不同,造成这种结果的原因可能是样品中有气泡或者样品浓度太高。之后我们进行强化归一,从扣除缓冲液后的散射结果(图3-B)可以看出棕色曲线的头部波动较大。出现这种结果的原因是由于样品的浓度太高,于是在后面的数据处理时,放弃了样品浓度最高的实验数据。对其他3组不同浓度的样品进行散射后作Guinier图,结果(图3-C)显示当hTR CR4/5浓度为4.3 mg/mL时散射曲线和理论值可以完全重合。之后再进行Dammif分析并调整不同的Dmax值,结果(图 3-D)显示实验值与理论值叠加后具有很好的吻合度。同时,可以看出计算得到的P(r)值(图3-E)在大r区域与横坐标结合处较平缓,这说明得到的数据具有很好的可靠性。最后利用晶体结构拟合hTR CR4/5的SAXS数据。
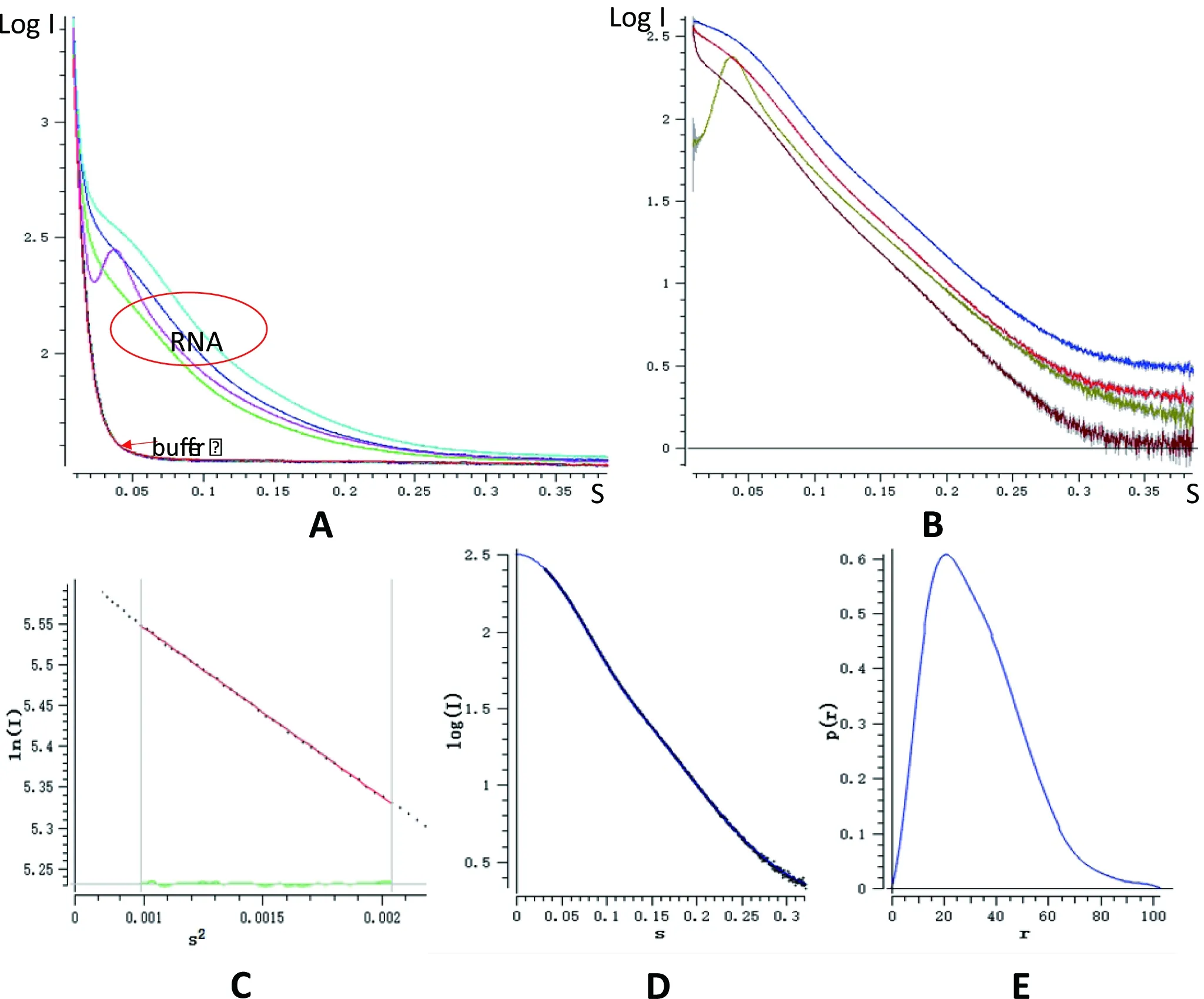
图3 hTR CR4/5的SAXS数据处理
A: 对不同浓度的hTR CR4/5和缓冲液进行数据平均的结果。B:扣除缓冲液后不同浓度的hTR CR4/5的散射结果。C:Guinier 分析,从第 40 到第 110 个点进行分析,并计算Rg值为24.86。D:Dammif值分析,调整 Dmax值,使散射曲线与理论曲线重合。E:GNOM分别从残差、系统误有效性稳定性等进行评估得到的P(r)值
2.4 端粒酶RNA(TR) CR4/5基因序列比对及hTR CR4/5的溶液构象模型
由于SAXS具有一定的局限性,为进一步验证实验结果,我们将人、青鳉、小鼠、四膜虫的TR CR4/5基因序列进行了比对。结果(图4-A)发现人和小鼠基因序列相似度达到99%,其次是青鳉,黑色字体碱基是4种物种共同具有的序列。通过构建进化树(图4-B),推测人、小鼠、青鳉应具有相似的三级结构及生化功能。
通过SAXS模拟了hTR CR4/5在溶液中的构象(图5-A),得到形状似“L”型的模型。经同源序列比对得知,人和小鼠的TR CR4/5同源性最高,但小鼠TR CR4/5结构域的三维结构至今未解析出来。所以我们采用了青鳉TR CR4/5的晶体结构(图5-B)与hTR CR4/5的SAXS模型进行拟合,发现青鳉TR CR4/5可以拟合到hTR CR4/5的模型中(图5-C)。从拟合的模型中看出人与青鳉TR CR4/5结构的最大区别是hTR CR4/5的内部环(CUGGAGGCGUCAGCC)要比青鳉(GCGUU)的大很多,相当于图5-C中红色圆内的那部分结构。而其他区域(P6.1/P5/P6)的位置大致相同。这能够为hTR CR4/5结构域和hTR晶体结构的解析提供相关的理论数据。
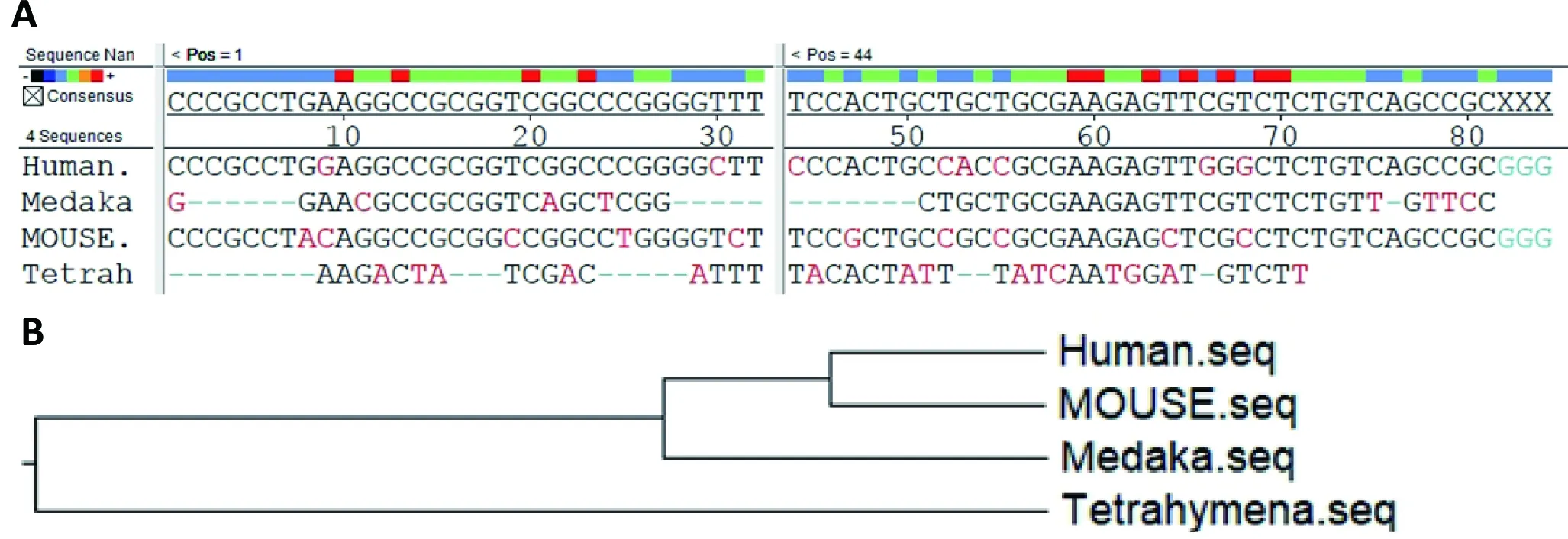
图4 不同物种TR CR4/5的基因序列比对及进化树分析结果
A:分别对人、青鳉、小鼠、四膜虫进行序列比对,黑色碱基为4种物种有相同的碱基,红色为不同碱基,人和小鼠的蓝色碱基相同;B: 系统进化树

图5 hTR CR4/5的SAXS结构模型
A: hTR CR4/5的SAXS结构模型;B: 青鳉的TR CR4/5三维结构;C: 青鳉的TR CR4/5 P6区域与hTR CR4/5 P6a区域的拟合三维结构模型,其中两物种最大的区别是内部环的大小即图中红色圆区域
3 结论
SAXS是目前发展最快的一种解析生物大分子结构的技术。本实验选取hTR CR4/5为实验对象,通过体外转录和RNA的纯化过程,探究了用于SAXS的最佳RNA浓度,并发现当hTR CR4/5浓度为4.3 mg/mL时,所得SAXS数据最优。为了进一步验证SAXS结果的准确性,本实验通过各物种间TR CR4/5的同源序列对比发现人和小鼠的CR4/5同源性最高,但由于人和小鼠的CR4/5结构都未解析出来,所以挑选了具有较高同源性的青鳉TR CR4/5与hTR CR4/5结构域进行了拟合,发现青鳉TR CR4/5可以嵌入到hTR CR4/CR5的模型中。最后分析预测了hTR CR4/5结构域各区域的大体位置,推测hTR CR4/5和青鳉TR CR4/5在功能上具有相似性。本结果将为hTR CR4/5和hTR晶体结构解析提供宝贵的实验数据。
