菊叶香藜psbA基因生物信息学分析*
2018-07-14雷鸣任海龙
雷鸣任海龙
(1.西藏大学理学院;2.西藏大学医学院,西藏 拉萨 850000)
菊叶香藜是藜科刺藜属植物[1],其具有刺激性气味,具有抑菌、昆虫活力抑制等作用[2,3],中药用于治疗哮喘、支气管炎和偏头痛[4],据藏药志记载有抗肿瘤活性[5]。菊叶香藜也为中等饲用植物[6],通常情况下为亚优势种或伴生种[7]。研究发现在群落演替中重要值在明显增加,同时也可作为固定沙地的指示植物[8]。许新勇等[9]研究发现,在西藏“一江两河”流域地区,伴随着植被发育和群落演替的进程,在半固定沙丘和固定沙丘,菊叶香藜逐渐成为优势种,说明菊叶香藜本身生理生态特征非常适应“一江两河”流域气候环境。2003年,朱格鳞等在Flora of China中,基于形态学特征将菊叶香藜等植物归类到刺藜属植物[1],并未对菊叶香藜的进行分子方面的鉴定。另外,psbA基因是叶绿体基因,序列相对保守,编码产物D1蛋白,是PSⅡ反应中心2个核心蛋白之一,介导光合作用过程中的电子传递[10],在植物属以上水平的分子系统发育的比较中具有显著作用[11]。因此,在本研究中,通过菊叶香藜psbA序列生物信息学分析和系统发生树构建,为菊叶香藜的分类学提供分子依据。
1 方法
1.1 基于转录组测序对菊叶香藜psbA基因全序列的获取
通过转录组测序,将注释到psbA基因的unigenes在NCBI进行比对,然后再进行拼接。
1.2 菊叶香藜psbA基因编码序列生物信息学分析
通过NCBI在线分析软件ORF Finder对核苷酸序列进行分析,预测出可能的氨基酸序列;通过蛋白质二级结构在线预测软件PredictProtein,预测出氨基酸序列的二级结构、相对亲水表面、氨基酸组分和核苷酸组分;用CondonW和CUSP对菊叶香藜psba基因编码序列进行密码子偏好性分析,基于中性绘图和ENC-plot探索影响psba基因密码子偏好性的因素;psba基因编码蛋白的理化性质采用ExPasy-ProtParam分析软件及BioEdit软件预测;疏水性/亲水性采用Ex-Pasy-ProtScale预测;跨膜区域通过软件TMHMM进行分析;结构功能域及功能分类采用ProtFun预测;通过Swiss-Model在线预测软件预测出三级结构;通过BioEdit进行序列差异分析;用MEGA6.05计算菊叶香藜与其近缘物种的遗传距离,并构建系统进化树。
1.3 菊叶香藜psbA基因系统发生树构建
从NCBI数据库中下载其它物种psbA基因完整序列,共9个物种(表1)。
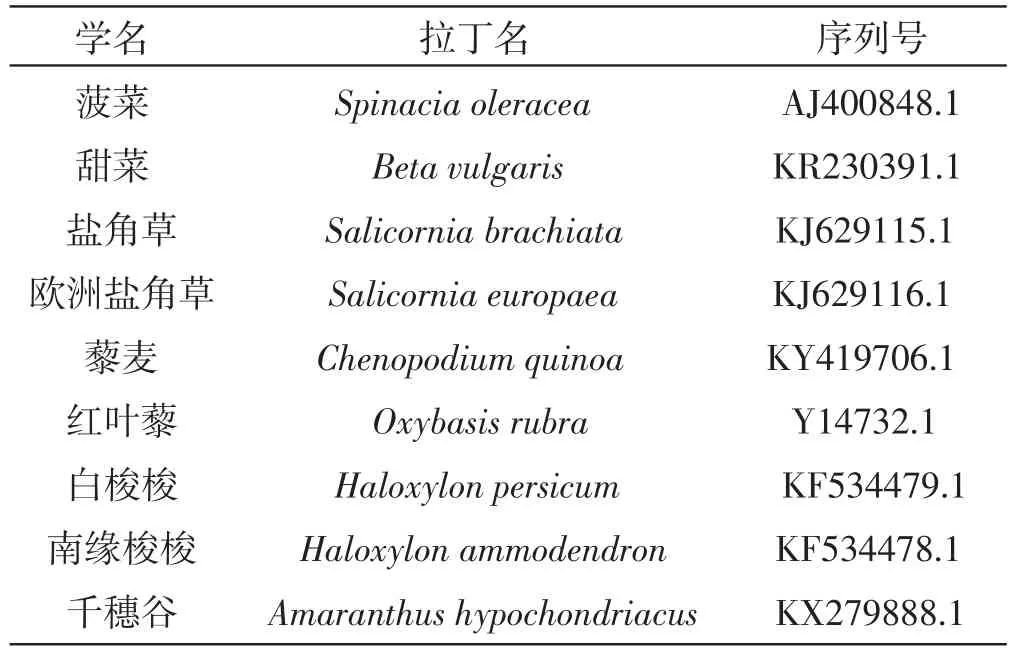
表1 NCBI数据库中下载9个物种psbA基因完整序列
2 结果
2.1 菊叶香藜psbA基因编码序列生物信息学分析
2.1.1 菊叶香藜psbA基因编码蛋白理化性质。基于转录组测序,将注释到psbA基因的unigenes(c1846_g1、c19645_g1、c24547_g1)在NCBI数据库中进行比对,然后拼接在一起,最终的序列长度为1059bp,GC含量为41%,序列已上传到NCBI数据库,序列号为:KY942083。
通过NCBI在线分析软件ORF Finder对核苷酸序列进行分析,预测出氨基酸序列,共353个氨基酸,如下所示:MTAILERRESESLWGRFCNWITSTENRLYIGWFGVL MIPTLLTATSVFIIAFIAAPPVDIDGIREPVSGSLLYGN NIISGAIIPTSAAIGLHFYPIWEAASVDEWLYNGGPYE LIVLHFLLGVACYMGREWELSFRLGMRPWIAVAYSA PVAAATAVFLIYPIGQGSFSDGMPLGISGTFNFMIVFQ AEHNILMHPFHMLGVAGVFGGSLFSAMHGSLVTSSLI RETTENESANEGYRFGQEEETYNIVAAHGYFGRLIFQ YASFNNSRSLHFFLAAWPVVGIWFTALGISTMAFNLN GFNFNQSVVDSQGRVINTWADIINRANLGMEVMHER NAHNFPLDLAAIEAPSING
负电荷残基总数为28,正电荷残基总数为15,其理论分子量为38962.62Da,理论等电点为5.12,不稳定指数是36.33。分子式为C1793H2682N456O493S14,原子总数为5438,脂肪系数为96.77。
2.1.2 菊叶香藜psbA基因编码蛋白氨基酸组分分析。氨基酸组分中最多的为A 35(9.9%),最少的为C 2(0.6%),其中,没有氨基酸K;相对亲水表面为71.67%在0-16%之间,28.33%在16%-100%之间。(如图1所示)

图1 菊叶香藜psbA基因编码蛋白氨基酸组分
2.1.3 菊叶香藜psbA基因编码蛋白二级结构。通过蛋白质二级结构在线预测软件PredictProtein,预测出氨基酸序列的二级结构为α-螺旋57.51%,β-折叠4.82%,β-转角37.68%.
2.1.4 菊叶香藜psbA基因编码蛋白亲疏水性分析。用ProtParam分析软件对菊叶香藜psbA基因编码蛋白的疏水性/亲水性进行预测,其疏水性平均系数为0.350。参照ExpASy的Protscale程序计算菊叶香藜psbA基因编码蛋白的疏水性/亲水性图谱(图2)分析氨基酸残基的得分可知,多肽链的第51号的丙氨酸(Ala)具有最高分值3.189,疏水性最强。第234号天冬酰胺(Asn)具有最低分值-2.133,亲水性最强。
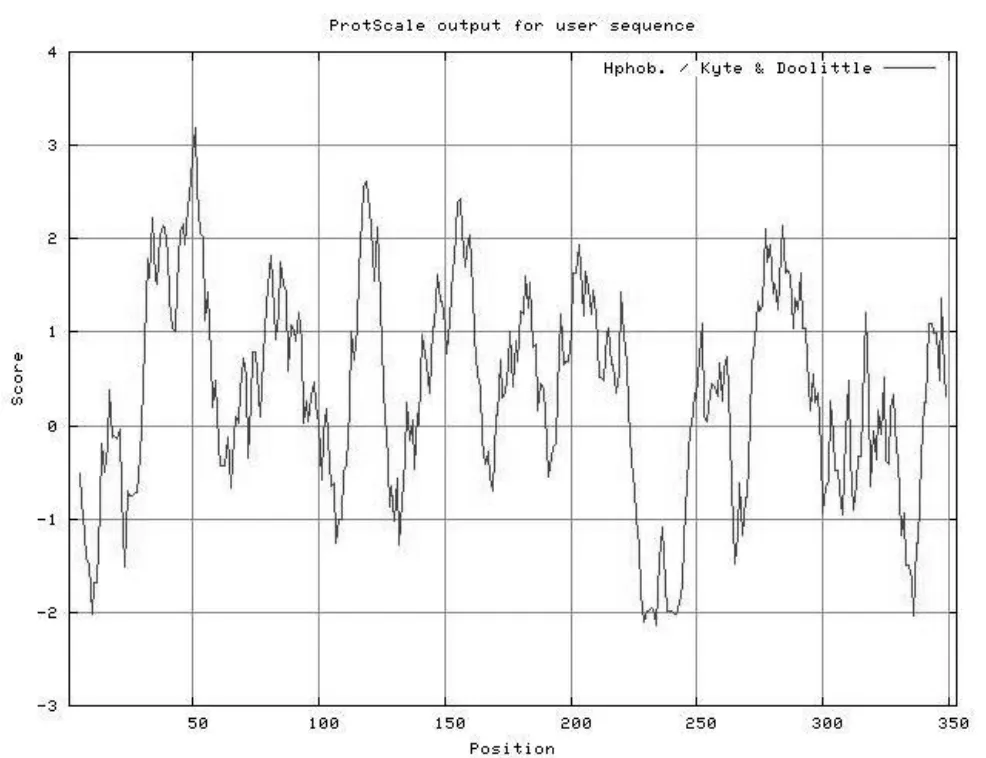
图2 菊叶香藜psbA基因编码蛋白亲疏水性分析
2.1.5 菊叶香藜psbA基因编码蛋白跨膜区域。由图3可知,菊叶香藜psbA基因编码蛋白存在7个跨膜区域,分别位于29~51、71~93、105~127、142~164、171~193、197~219和273~295氨基酸之间,跨膜区分析结果与该蛋白的亲/疏水性分析结果基本一致。
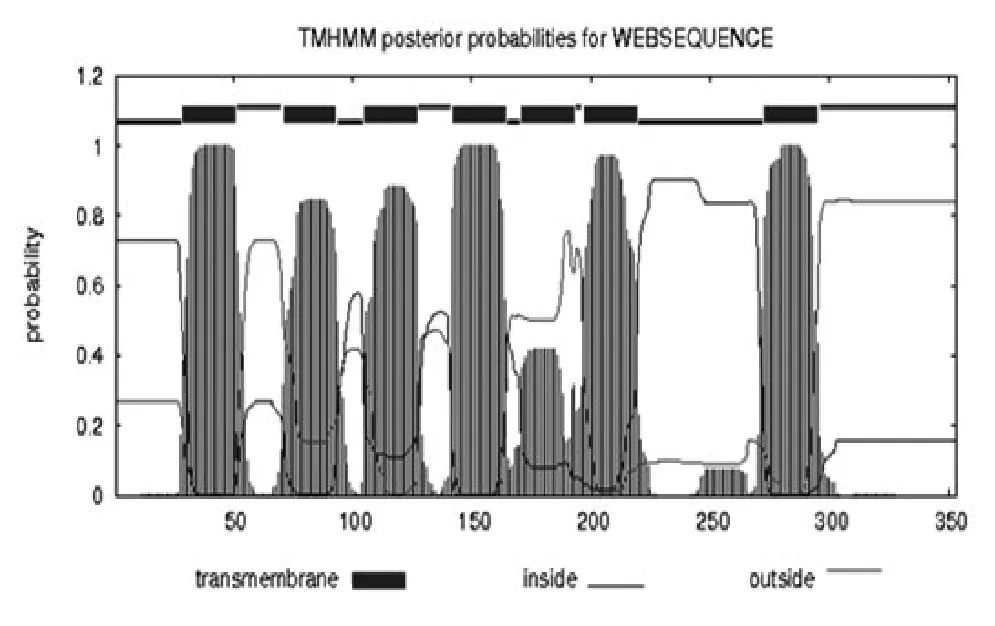
图3 菊叶香藜psbA基因编码蛋白跨膜区域
2.1.6 菊叶香藜psbA基因编码蛋白结构功能域及功能分类。通过Protfun分析软件预测菊叶香藜psbA基因编码蛋白结构功能域及功能分类从分析结果可知(表2),菊叶香藜psbA基因编码蛋白为非酶类(Prob=0.805,odds=1.128)和转运相关蛋白(Prob=0.191,odds=1.749),该蛋白具有翻译、脂肪酸代谢、辅酶因子的生物合成和能量代谢作用的可能性分别为4.760、4.477、3.423和2.195。因此,推测该蛋白的主要作用与转运有关。

表2 psbA基因编码的蛋白质的功能预测
2.1.7 菊叶香藜psbA基因编码蛋白三级结构预测。通过Swiss-Model在线预测软件预测出三级结构,选取Template>30%、GQME值接近1、|QMEAN|<4的模型,预测出的菊叶香藜psbA基因编码蛋白三级结构(图4)。
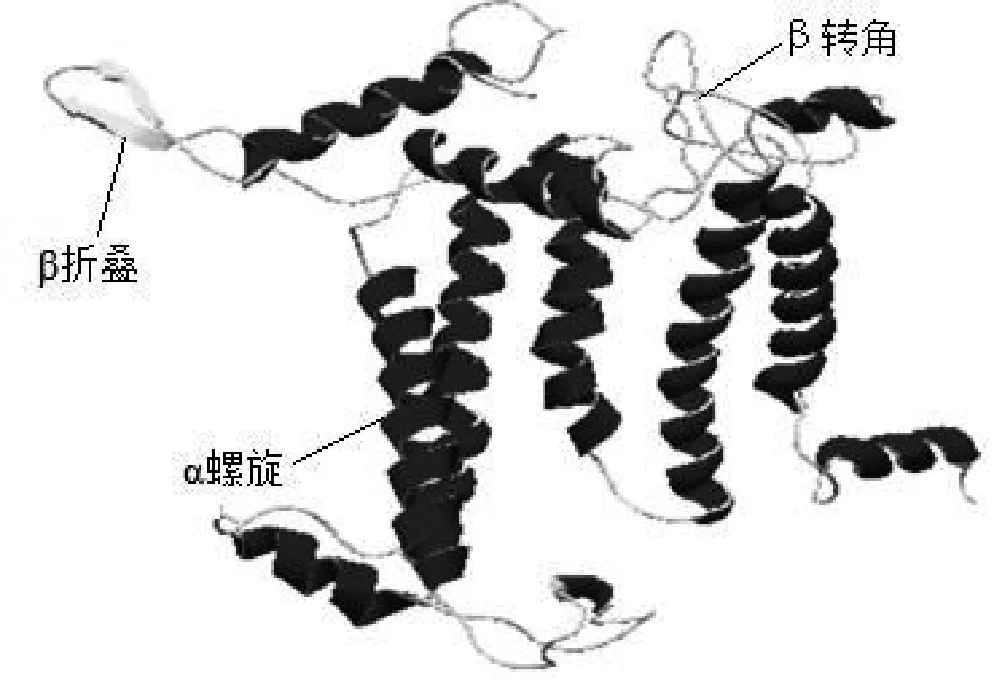
图4 菊叶香藜psbA基因编码蛋白三级结构预测模型
2.1.8 菊叶香藜psbA基因密码子偏好性。通过CUSP和CondoW程序分析菊叶香藜及其近缘种psbA基因的密码子偏好性基本指标。从表3中可以看出,各物种psbA基因在密码子偏好性方面差异较小,其中CBI、GC含量、GC3和GC3s,苋科植物均大于藜科植物,而ENC值,苋科植物均小于藜科植物。GC1、GC2、GC3和GC含量均小于0.5,说明psbA基因密码子各位点均偏好使用含A/T的密码子,而GC3s均小于0.3,说明在同义密码子中,psbA编码序列更偏好使用以A/T结尾的密码子,其中,NNT>NNA>NNC>NNG,NNT远大于NNC。ENC值均位于35-50之间,说明psbA基因密码子偏好性一般,基因表达水平一般。
通过CUSP和CondoW程序分析菊叶香藜psbA基因的密码子偏好性可知,菊叶香藜psbA基因总共使用了49个密码子,23个密码子为偏好性密码子(RSCU>1),其中 GCU、GAU、GAA、GGU、AUU、CUA、UUA、CCU、CAA、CGU、AGU、UCU、ACU和GUA为高频密码子(RSCU>1.5),以GU和CU结尾的密码子全部为高频密码子。在偏好性密码子中,有18个密码子以A/T结尾,这18个密码子Fraction和使用频率(Frequency)都相对较高(表4)。
为探索影响psbA基因密码子偏好性的因素,通过中性绘图和ENC-plot绘图进行分析。中性绘图结果显示(图5所示),GC12与GC3相关性不显著(R2=0.1,P=0.251),说明psbA基因密码子第1、2位和第3位碱基相关性不显著,psbA基因GC含量高度保守,密码子偏好性主要受选择作用影响。
ENC绘图结果显示(图6),所有物种的psbA基因均落在标准曲线下方相对较远的位置,说明密码子的偏好性只受到选择作用的影响。进一步探究影响选择作用的因素,本研究主要分析CAI值和蛋白长度对ENC值的影响,发现CAI值与ENC值表现为极显著的负相关(R2=0.6817,P<0.01,回归方程为:y=-37.053x+51.522),蛋白长度与ENC值表现为完全线性相关(x=353)。因此,选择作用主要受到表达水平和蛋白质长度的影响,psbA基因密码子偏好性受到表达水平和蛋白质长度的的影响。
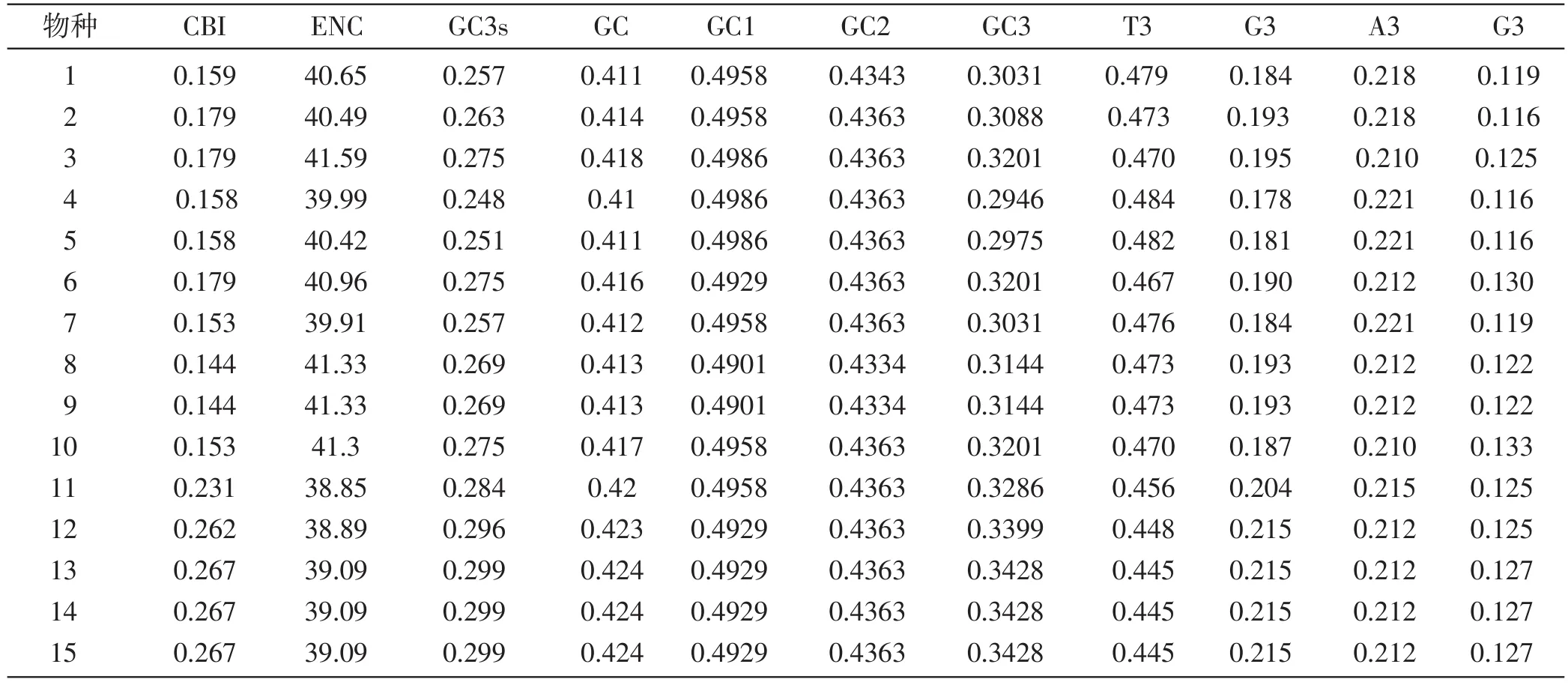
表3 菊叶香藜及其近缘种psbA基因的密码子偏好性基本指标

表4 CUSP和CondoW程序分析菊叶香藜psbA基因的密码子偏好性
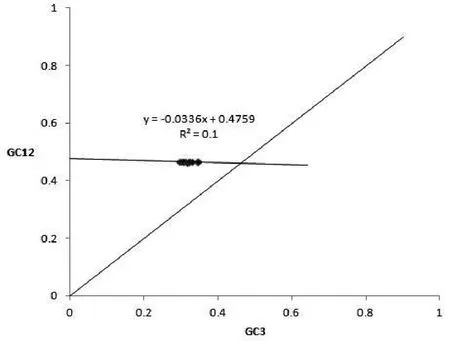
图5 GC3和GC12中性绘图结果

图6 GC3s和ENC绘图结果
2.1.9 菊叶香藜psbA基因系统发生树结果。由菊叶香藜及其近缘物种的psba完整序列生成的系统进化树可知(图7),系统树上聚为两大支,藜科和苋科各聚为一大支,说明在藜科和苋科中,psbA基因存在差异。而对于藜科这一支来说,psbA基因在藜科各属之间差异相对较大;各进化支部分种间自展值为100%,说明psbA序列对于种的鉴定存在一定的局限性。菊叶香藜原为藜科藜属植物,但在近年来的藜属与刺藜属的争议中,被划为藜科刺藜属中,从图7中我们可知,菊叶香藜与藜属植物分别聚为一支,正好与形态学分类上的观点一致。
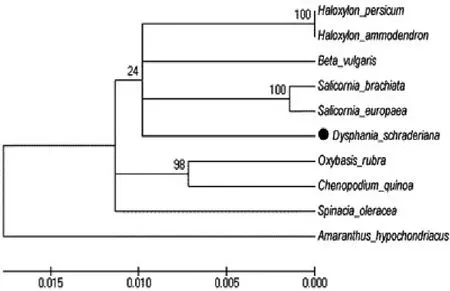
图7 菊叶香藜psbA基因系统发生树结果(“●”为菊叶香藜)
3 讨论
菊叶香藜psbA基因编码序列长度为1059bp,GC含量为41%,共编码353个氨基酸,最多的为丙氨酸(A),为35个(9.9%),不含有赖氨酸,符合psbA基因的特征。二级结构包含α-螺旋(57.51%),β-折叠(4.82%),β-转角(37.68%)。psbA 基因编码蛋白为疏水性蛋白,包含7个跨膜区域,是非酶类转运蛋白。psbA基因密码子偏好性水平相对较弱,主要受到选择作用、表达水平和蛋白质长度的影响,该结果与尚明照等[12]研究结果一致,为进一步研究影响psbA基因密码子偏好性的因素提供依据。在同义密码子中,psbA编码序列更偏好使用以 A/T 结尾的密码子,其中,NNT>NNA>NNC>NNG,NNT远大于NNC,与侯惠静[13]研究结果不一致,与侯士昌等[14]研究结果一致。系统发生树的结果证明菊叶香藜与藜属植物存在差异,支持将其划归到刺藜属中。
