长链非编码RNA基因NRON与肉鸡生长性状关联分析
2018-07-10杜志强董翔宇汪礼建高卓然
杜志强,董翔宇,汪礼建,高卓然,李 辉
(1.农业部鸡遗传育种重点实验室,哈尔滨 150030;2.黑龙江省高校动物遗传育种与繁殖重点实验室,哈尔滨 150030;3.东北农业大学动物科学技术学院,哈尔滨 150030)
长链非编码RNA(lncRNA)是近年新发现的一类长度大于200个核苷酸,缺乏蛋白编码功能,由RNA聚合酶Ⅱ或Ⅲ转录生成的RNA[1],可多层面调控转录后和表观生物学等过程[2-4]。起初,lncRNA被视为转录过程中的“转录噪音”。随后研究发现,lncRNA可作为生物信号分子、miRNA吸附“海绵体”、转录因子引导者、增强子及蛋白支架等,参与转录本可变剪接、基因组印记、染色体沉默、细胞周期调控、蛋白质合成等生物学过程[5-6]。
NRON是抑制NFAT(Nuclear factor of activated T cells,活化T细胞核因子)的lncRNA[7],最初是在一个表达序列标签(EST)测序项目中,通过筛选512个保守非编码RNA序列而被找到[8-9]。NFAT是一类转录因子家族,在免疫反应中对诱导基因转录起重要作用[10]。除T细胞外,免疫细胞多可表达该类蛋白质[11],如B淋巴细胞[12]、肥大细胞、嗜酸性粒细胞等;NFAT也影响心肌、骨骼肌及神经系统发育[9]。此外,NFAT与脂肪细胞分化有关[13-14]。钙离子依赖的钙调蛋白磷酸酶调节NFAT活性[15-16]。随细胞内钙水平升高,钙调磷酸酶(Cn)被激活,直接脱磷酸以过磷酸化形式存在于NFAT蛋白,诱导其转移至细胞核内。转移至核内NFAT蛋白结合于靶基因启动子上,单独或与其他转录因子共同诱导基因表达。与钙调磷酸酶激活通路拮抗的NFAT激酶、糖原合成酶激酶(GSK)、酪蛋白激酶(CK),使NFAT蛋白磷酸化,从核内转出至细胞质[17]。NRON则通过与NFAT形成RNA-蛋白复合物形式调节NFAT脱磷酸作用[18],参与艾滋病病毒HIV-1复制过程[19-20]。目前,鸡NRON功能研究国内外无相关报道。
本研究以东北农业大学肉鸡腹脂双向选择系和爱拔益加(Arbor acres,AA)肉鸡群体为材料,检测NRON单核苷多态性,进一步分析其同生长性状间关联,旨在探讨NRON基因影响肉鸡肌肉和脂肪组织生长发育分子机制。
1 材料与方法
1.1 材料
东北农业大学肉鸡高、低腹脂双向选择品系(NEAUHLF)经21个世代选育,高、低脂系间腹脂重和腹脂率差异显著。本研究以NEAUHLF第19世代鸡群和AA肉鸡随机群体为试验材料,选用高脂系公鸡114只,低脂系公鸡121只,AA群体公鸡母鸡共240只,收集各类生长性状:肉鸡1~7各周龄体重,7周龄屠宰时胴体性状。
组织表达检测样品采集过程如下:从高、低脂系第19世代(G19)肉鸡出生后1周龄开始取组织样品,每周采样一次,直至7周龄。取样分为两组:高脂系公鸡和低脂系公鸡。禁食10 h后称量体重并屠宰,屠宰后称量腹脂重,计算腹脂率(腹脂重/体重)。采集腹脂、大脑、肝脏、肾脏、脾脏、心脏、肌胃、肠系膜周围脂、皮下脂、肌胃周围脂共10种组织。样品于0.75%氯化钠中清洗,液氮中速冻,-80℃保存待用。
1.2 方法
1.2.1引物设计与合成
1.2.1.1Real-Time RT-PCR引物设计
根据Ensembl数据库中鸡NRON基因序列(ENSGALG00000025631)和NFAT基因序列(ENSG ALG00000039300),结合内含子位置信息,选择Primer Premier 5.0设计Real-Time RT-PCR表达检测引物。内参基因TBP根据NCBI数据库信息,使用Primer Premier 5.0设计引物。引物信息见表1。
1.2.1.2酶切引物设计
根据Ensembl数据库鸡NRON基因序列(ENS⁃GALG00000025631),利用在线软件WatCut(http://watcut.uwaterloo.ca/)及Primer Premier 5.0设计酶切引物(见表2)。
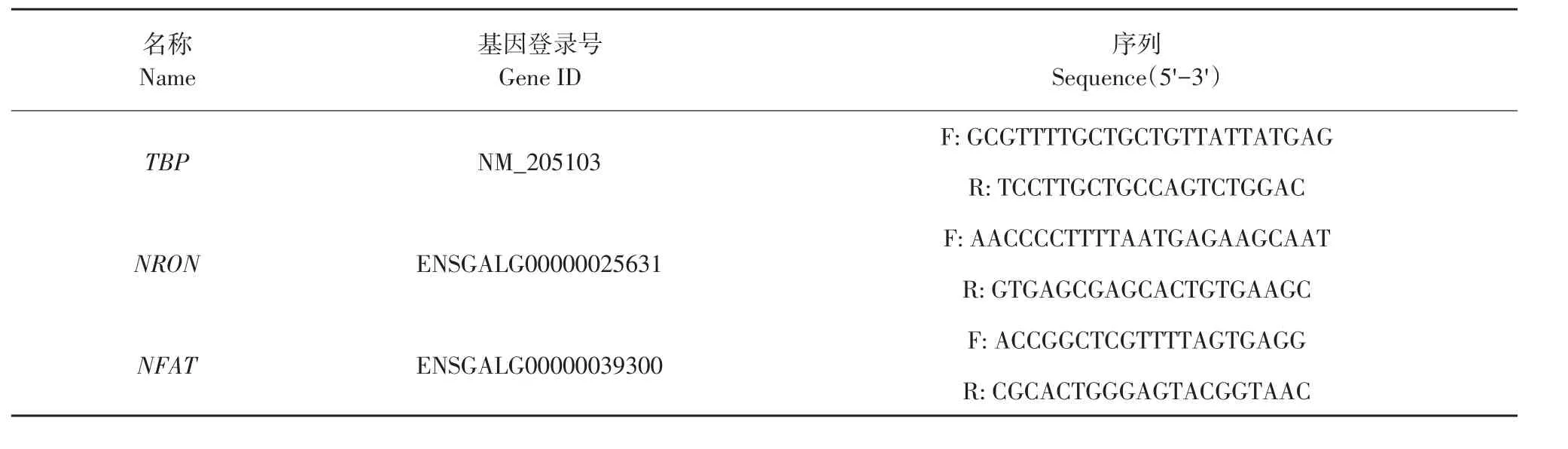
表1 Real-Time RT-PCR表达检测引物Table 1 Primers for Real-Time RT-PCR

表2 酶切引物Table 2 Primers for restriction enzyme cleavage
1.2.2RNA提取及cDNA合成
高、低脂系肉鸡(各5只),分别提取组织总RNA(TRIZOL法),通过琼脂糖凝胶电泳分析,Nano⁃drop检测浓度,确保DNA和RNA质量和完整性。
取1 mL TRIZOL加入研磨(液氮中)组织样品50~100 mg,充分混匀。加入0.2 mL氯仿,离心后,水相转移到新离心管。加入400 mL异丙醇沉淀水相中RNA,离心后移去上清。加入1 mL DEPC处理的75%乙醇洗涤,RNA沉淀后移去上清,DEPC水溶解RNA。-80℃冰箱备用。利用PrimeScript™RT reagent Kit with gDNA Eraser(购自TaKaRa宝生物公司)获得去除基因组cDNA。
1.2.3Real-Time RT-PCR
Real-Time RT-PCR反应体系为:SYBR®Pre⁃mix Ex TaqTM(2×)(购自宝生物工程(大连)有限公司)5μL,ROX Reference DyeⅡ(50×)0.2μL,上、下游引物10μmol·L-1各0.2μL,cDNA模板1μL,ddH2O 3.4μL,总体积10μL。反应条件:95℃预变性10 s,95℃变性5 s,60℃复性延伸34 s,共40个循环。溶解曲线95℃15 s,60℃ 10 min,95℃15 s,每个样品设3孔重复。使用Real-Time RT-PCR仪型号为ABI 7500。
1.2.4PCR扩增
根据设计SNP1和SNP2基因作PCR扩增,PCR扩增体系为:50 ng·μL-1基因组 1 μL,10×PCR Buffer 0.8 μL,10 mol·μL-1上游引物0.2 μL,10 mol·μL-1下游引物 0.2 μL,10 mmol·μL-1dNTP 0.8 μL,Taq DNA 聚合酶0.1 μL,去离子灭菌水6.7 μL。
PCR扩增程序:94℃ 5 min;94℃ 30 s,(55.7~60.7 ℃) 30 s,72 ℃ 30 s,共35个循环;72℃7 min。PCR产物于琼脂糖凝胶电泳检测,以5μL DNA Marker DL 2000为参照。电泳结束后利用凝胶成像系统观察扩增结果。
NRON基因变异位点可被限制性内切酶切割,选用限制性片段长度多态性(Restriction fragment length polymorphisms,RFLP)分型方法。
酶切体系设计为:内切酶10 U·μL-10.1 μL,10·Buffer 2.0 μL,PCR产物0.3~0.5 μg,去离子水加至20 μL。
将上述反应液混匀,于适当温度(依据不同酶而定)水浴中过夜消化,将全部反应液作琼脂糖凝胶电泳,检测PCR产物酶切效果及片段多态性。酶切产物经2%琼脂糖凝胶电泳检测并观察结果。
1.2.6基因效应分析
根据群体特点,构建基因型分析统计模型:
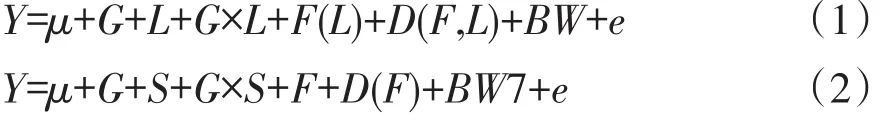
其中,Y为性状观测值,μ为群体均值,G为基因型固定效应,L为品系固定效应,G×L为基因型和品系互作效应,F(L)为品系内家系随机效应,D(F,L)为家系与品系内母鸡随机效应,S为性别固定效应,G×S为基因型和性别互作效应,F为家系随机效应,D(F)为家系内母鸡随机效应,BW(第1或第7周龄体重)为协方差变量,e为随机效应。
模型①适于东北农业大学高、低脂系肉鸡双向选择系群体,模型②适于AA肉鸡随机群体;使用统计软件JMP 7.0检验基因型与性状间相关性,估计性状最小二乘均值。P<0.05为显著,P<0.01为极显著。
2 结果与分析
2.1 鸡NRON基因多态性检测
2.1.1SNP位点发现
在婚姻中,不要试图改造或改变伴侣。人无完人,每个人身上都存在优点,也存在缺点。当初,你之所以选择和对方结婚,肯定是因为对方身上有你欣赏的地方,不妨学会欣赏对方的优点,忽略对方的缺点。一个善解人意的妻子或丈夫,应该尊重对方的个性,不要把自己的意志强加给对方,要给对方保留一定的自由空间,允许对方有自己的社交圈子。
本试验根据19世代高、低脂系肉鸡全基因组重测序数据(未发表结果),发现NRON基因上下游共10个单核苷酸多态性(SNP)位点。为进一步确定SNP真实性,根据Ensemble数据库中鸡NRON序列(ENSGALG00000025631),随机选取19世代高、低脂系肉鸡各3个基因组样本为模板,设计3段引物及PCR方法扩增长度分别为898、891和837 bp序列(覆盖2 757 bp,包括NRON基因和其上下游各1 200 bp序列)。PCR产物测序后与鸡基因组(Gallus 5.0)比对分析,验证10个SNP,后续关联分析使用其中两个SNP位点,1个位于NRON基因,另1个位于NRON基因下游。具体SNP位置信息如图1所示。
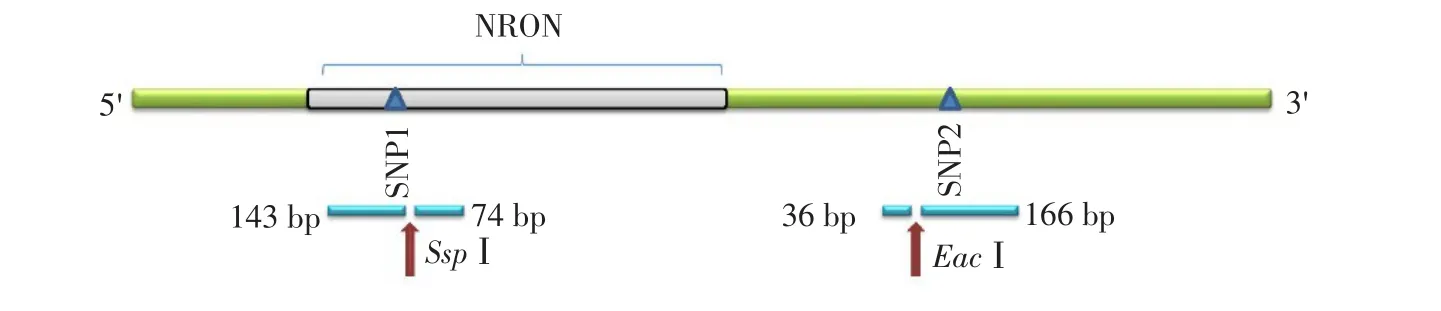
图1 NRON基因SNP相对位置信息Fig.1 Relative positions of SNPs discovered in NRON
2.1.2多态性检测及分析
采用SNP-RFLP技术检测NRON基因两个SNP位点,高、低脂肉鸡和AA肉鸡群体中分别检测3种基因型。检测NRON基因多态性PCR扩增片段产物长度分别为 217 bp(SNP1)和 202 bp(SNP2)。SNP1片段经SspⅠ限制性内切酶酶切后产生长度为143 bp和74 bp片段,根据条带不同分别检测3种基因型,分别命名为GA基因型(217 bp+143 bp+74 bp)、AA基因型(143 bp+74 bp)和GG基因型(217 bp);SNP2片段经EaeⅠ限制性内切酶酶切后产生长度为166 bp和36 bp片段,根据条带不同分别检测到3种基因型,分别命名为GG基因型(202 bp)、CC基因型(166 bp+36 bp)和GC基因型(202 bp+166 bp+36 bp)(见图2)。
关联分析结果见表3~4,AA肉鸡群体中,SNP1位点与生长性状无关联,SNP2位点与心脏重、3周龄和5周龄显著相关。高、低脂系肉鸡群体中,SNP1位点与肌胃重、骨盆宽、胸角性状显著相关,SNP2位点与心脏重、腺胃重、胸宽性状显著相关。同时,AA肉鸡群体中,虽然SNP2位点多态性与腹脂重不相关,但显性效应值大于加性效应值,且显性度为1.3,表明SNP2位点有超显性且影响腹脂重;高、低脂系肉鸡群体中,SNP2位点与胸宽性状显著相关,且显性效应值大于加性效应值,显性度为12.9,表明SNP2位点有超显性且影响胸宽;但在两个群体中,心脏重显性度均不明显。
2.2 连锁不平衡分析
以东北农业大学肉鸡高、低脂双向选择品系第19世代资源群体(G19)235只公鸡和AA群体240只公鸡和母鸡为试验材料,Haploview软件分析SNP1和SNP2在两个群体间连锁不平衡关系。结果表明,AA肉鸡群体中,SNP1和SNP2位点间连锁不平衡程度较强(D'=1.0)(见图3a);而高、低脂系肉鸡群体中,SNP1与SNP2几乎不连锁(D'=0.03,见图3b)。
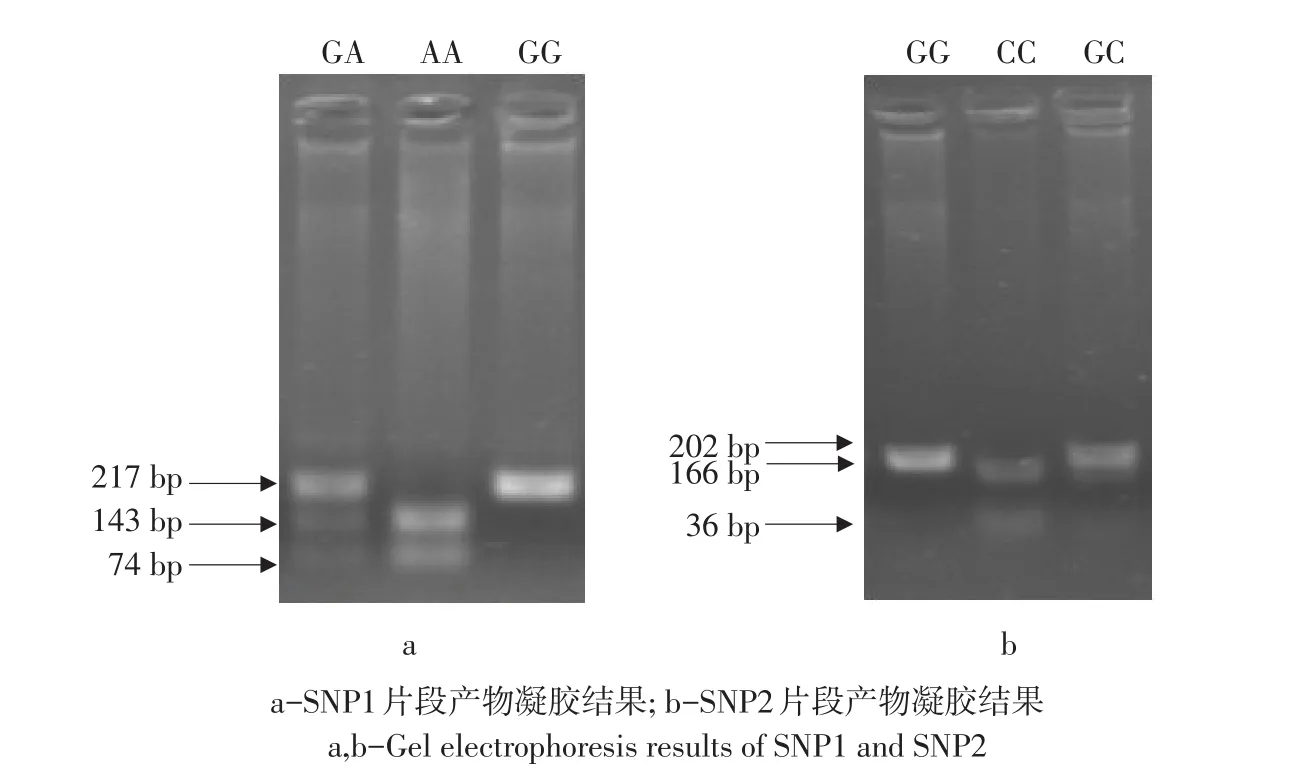
图2 NRON基因SNP多态片段产物凝胶电泳结果Fig.2 Gel electrophoresis of NRON SNP RFLP
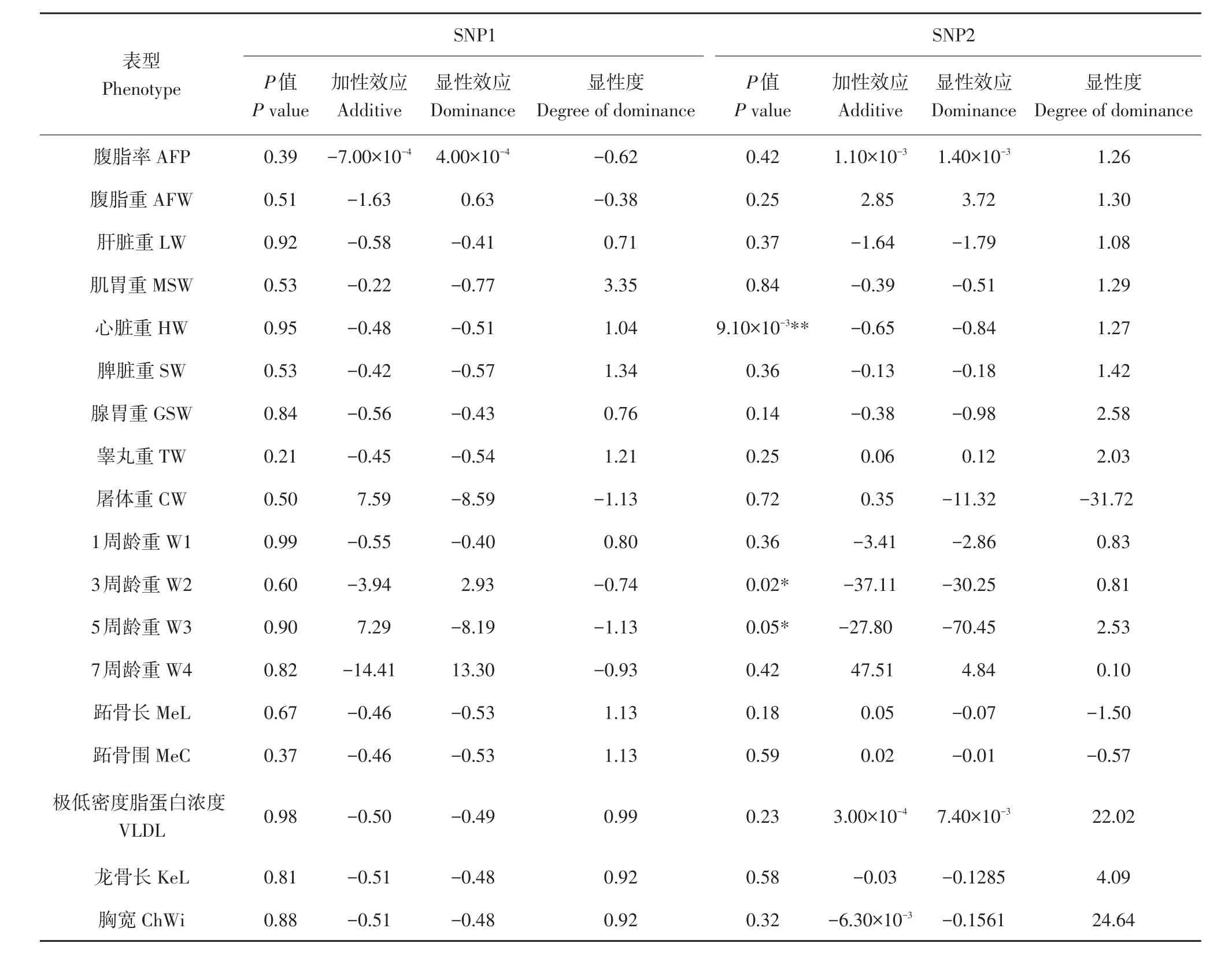
表3 AA肉鸡群体NRON基因SNP多态性与生长性状关联分析Table 3 Association of NRON polymorphisms with growth traits in AA broilers
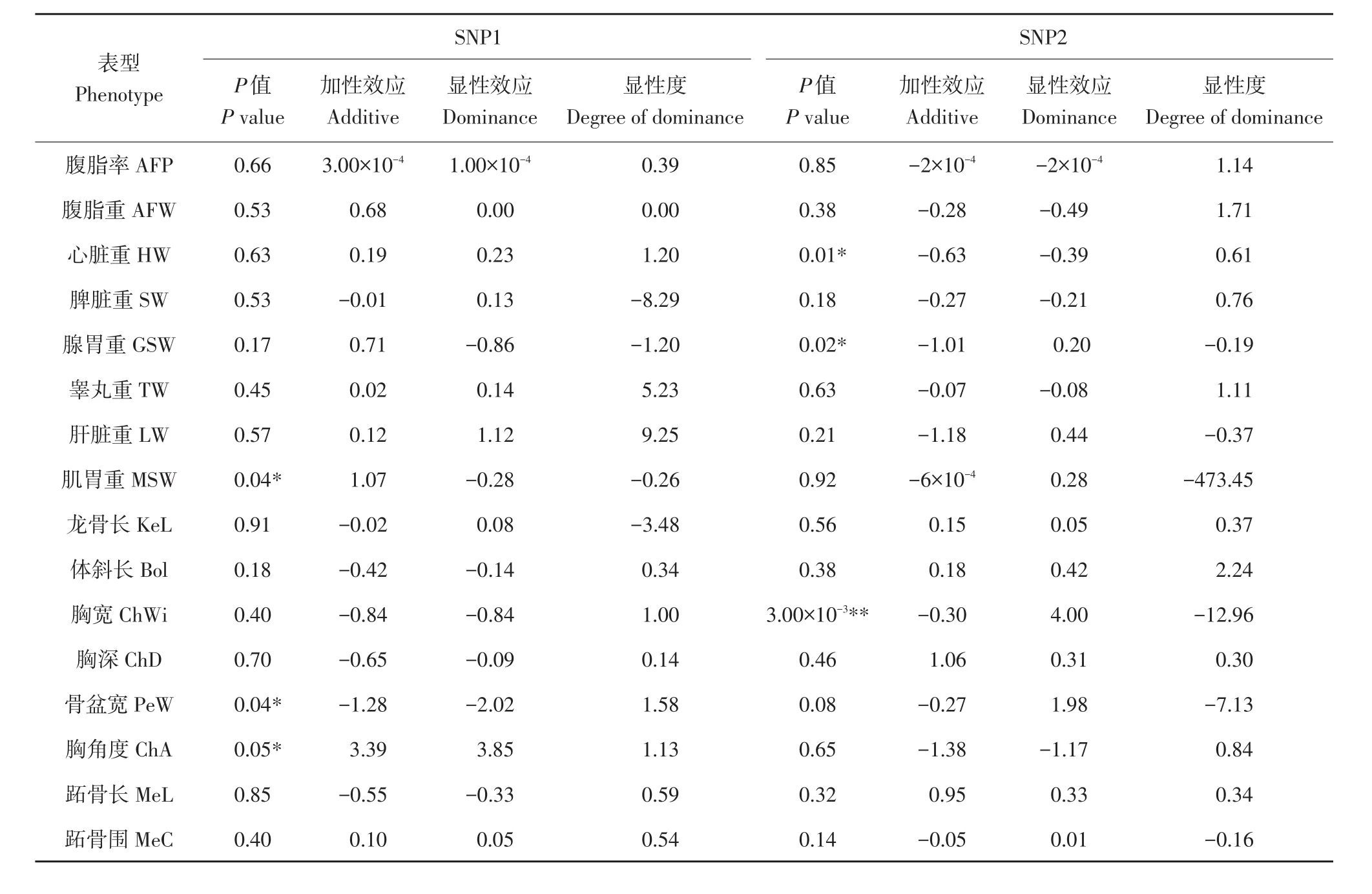
表4 高、低脂系肉鸡群体中NRON基因SNP多态性与生长性状关联分析Table 4 Association of NRON polymorphisms with growth traits in HLF broilers
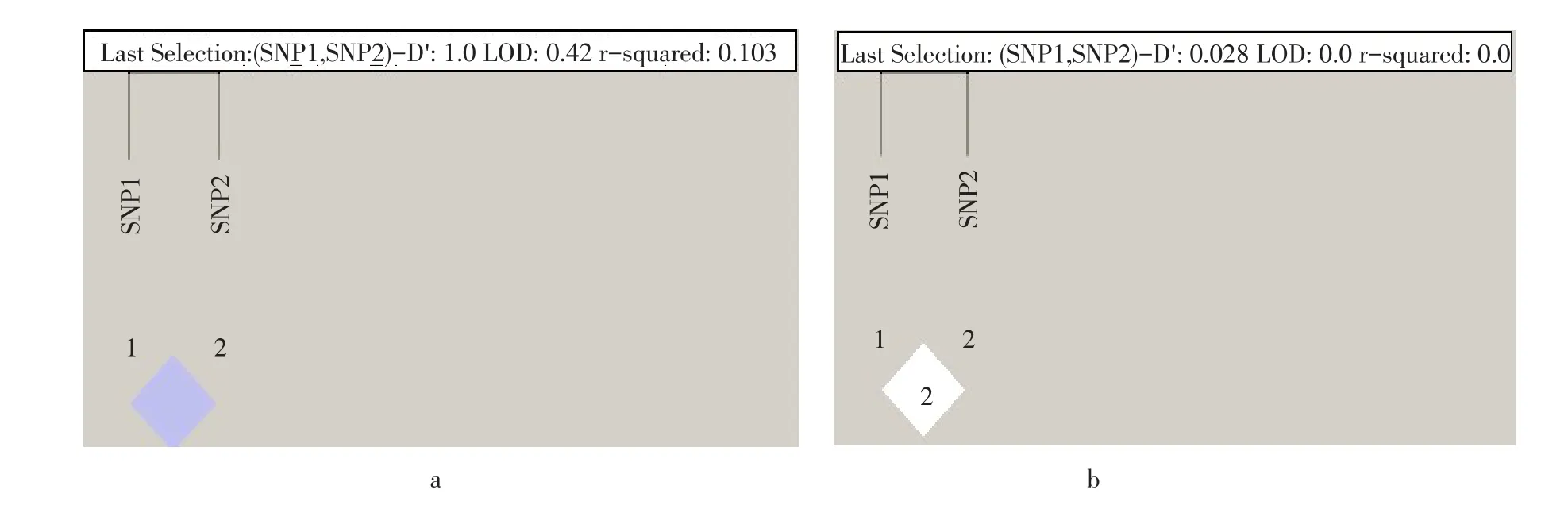
图3 AA和高、低脂系肉鸡群体中NRON SNP连锁不平衡分析Fig.3 Linkage disequilibrium analysis of NRON SNPs in AA and HLF broilers
2.3 在不同组织中鸡NRON基因表达规律
Real-Time RT-PCR检测NRON基因高、低脂系肉鸡腹部脂肪组织表达情况(n=5)。
由图4可知,1、4、7周龄,NRON基因在肉鸡高脂系表达水平显著高于低脂系(P<0.05);且NRON及NFAT基因随周龄增加,表达量呈递增趋势。
同时,检测NRON基因在高、低脂肉鸡心脏、腺胃、肌胃、胸肌、肝脏、睾丸、脾脏组织中表达情况(n=3)。
由图5可知,NRON基因在睾丸和脾脏组织中不表达;肌胃、肝脏中表达差异不显著;心脏、腺胃、胸肌中表达差异极显著(P<0.01),且在心脏和腺胃中,高脂系显著高于低脂系,胸肌中,高脂系显著低于低脂系。
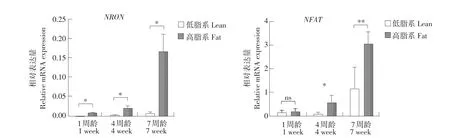
图4 鸡脂肪组织中NRON及NFAT的mRNA表达水平Fig.4 mRNA expression patterns of NRON and NFAT in abdominal adipose tissues
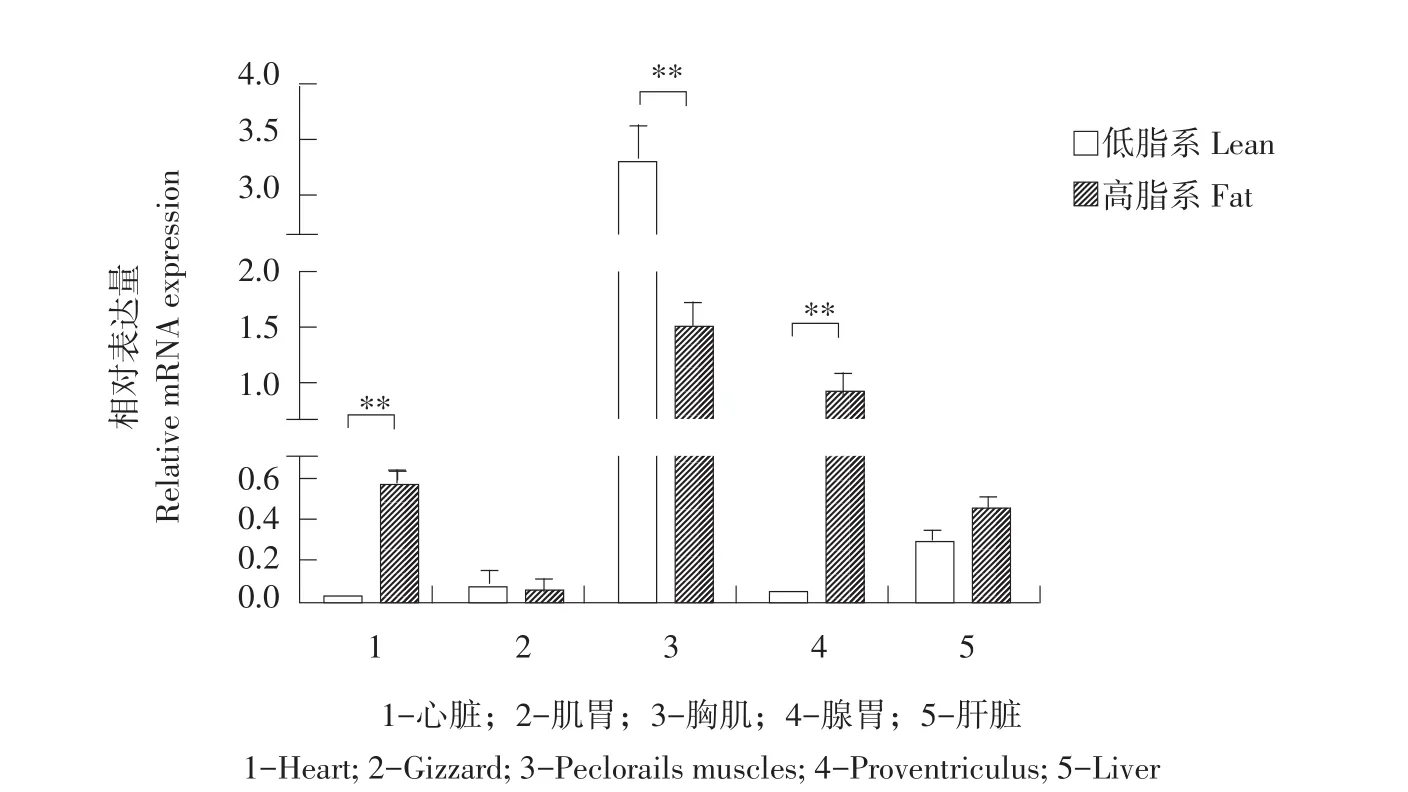
图5 鸡不同组织中NRON的mRNA表达水平Fig.5 mRNA expression patterns of NRON in different tissues
3 讨论与结论
3.1NRON和NFAT与心脏发育
本研究发现NRON与肉鸡心脏重极显著相关,高、低脂系肉鸡中,NRON基因在心脏中表达差异极显著,高脂系表达显著高于低脂系。研究表明NFAT基因为调控心房肌生长发育重要因子,其功能紊乱导致心脏疾病,在人类心脏表达基因中,13%在启动子区有NFAT结合位点,这些基因中20%~40%在心脏病发病前期发生表达修饰[21]。NFAT激活由钙调磷酸酶介导,对于肥厚心肌组织中心肌细胞基因表达有重要作用[22],Putt等研究表明NFAT是心肌肥大反应调节元件[23]。在心肌肥大心脏中,NFAT脱磷酸化增强自身核定位和转录活性[24-25],导致NFAT活性在患心脏病或年老患者中均明显增强[26]。同时,抑制NFAT基因活性可干扰心肌肌钙蛋白基因转录过程,导致心房肌变薄[10]。NRON基因作为NFAT抑制物[9],作为一种新生物标记可预测心脏病[27]。NRON可作研究肉鸡心脏生长发育过程标记基因。
3.2 NRON与肌肉生长发育
除与心脏生长发育密切相关外,NRON与肉鸡其他生长性状(3周龄重、5周龄重、腺胃重、胸宽)也显著相关(P<0.05)。高脂系胸肌中NRON表达水平显著低于低脂系。NRON基因表达研究表明,在人胎盘组织、胸腺、脾脏中NRON高丰度表达,在睾丸、肾脏、大脑和肾上腺组织中也检测到NRON基因较高丰度表达[9]。小鼠骨骼肌、胸腺中NRON基因高丰度表达,脾脏、淋巴组织和肺组织也可检测明显表达[9]。NRON基因Northern杂交结果同其基因表达结果吻合,且NRON转录本在不同组织中有特异性剪接形式,可能有其他未知生物学功能[9]。研究结果暗示NRON与骨骼肌和平滑肌生长发育可能相关。因此,NRON基因与肉鸡体重等生长性状显著相关,胸肌中差异表达,因此可能被作为重要生产性状候选基因,应用于分子育种和肉鸡生产。
3.3 NRON与脂肪组织生长发育
NRON及NFAT基因在东北农业大学高、低脂系肉鸡腹部脂肪组织中表达差异显著(P<0.05),表达趋势一致,均为肉鸡高脂系中mRNA表达水平高于低脂系。NFAT转录因子家族源自REL(c-Rel)-nuclear factor-κB(REL-NF-κB)转录因子家族[28],被认定为活化T细胞中重要组成因子,其受钙调磷酸酶调节特性决定NFAT在多种生物过程均有调节功能[29-31],如非免疫细胞生成[32-33],心肌形成[34-36]和神经回路[36]等。最初并未发现NFAT与脂肪生成相关[14,30],近期Graef等研究发现其在脂肪细胞分化过程中具有重要作用[37-39]。NFAT可与转录因子CCAAT/增强子结合蛋白互作形成复合元件[38-39],调节脂肪生成重要调节因子过氧化物酶增殖体受体γ2(PPARγ2)基因表达[40-41],参与脂肪生成;此外,NFAT与脂肪酸结合蛋白(aP2)同样有关[42-43],在小鼠3T3-L1前脂肪细胞中,NFAT蛋白结合aP2启动子并反式调控aP2转录过程,影响脂肪细胞分化[44]。
作为脂肪生成重要调控因子,去乙酰化酶SIRT1不仅结合NF-κB使其去乙酰化,抑制NF-κB转录激活活性[45],还抑制NFAT活性[46],抑制脂肪生成。NFAT与脂肪分化标志基因脂联素密切相关。NFAT家族成员NFAT3/c4通过结合脂联素启动子区,激活脂联素启动子活性,调控其在小鼠3T3-L1脂肪细胞中表达[47]。综上,NFAT可通过多种分子机制促进脂肪细胞分化,NRON则通过抑制NFAT脱磷酸化而促进脂肪分化过程。
3.4 NRON功能性SNP鉴定
通过连锁不平衡分析发现NRON基因中SNP1和SNP2位点在高、低脂系肉鸡群体中连锁不平衡程度较强,且两个位点与生产性状关联分析结果不同。通过转录因子结合位点在线预测,发现NFAT蛋白结合NRON序列十分接近SNP1(未发表结果)。表明SNP1位点可能影响NRON与NFAT形成RNA-蛋白复合物,参与NRON调控NFAT功能分子生物学过程。本研究仅检测NRON基因单核苷酸多态性位点与生产性状间相关及组织中表达情况,需进一步开展SNP功能性鉴定和相关作用机制分析,揭示NRON与肌肉和脂肪性状间分子调控机制。
