坝上长尾鸡MITF基因核心启动子鉴定与分析
2018-07-04刘小辉彭永东周荣艳张传生李祥龙河北农业大学动物科技学院保定0700河北科技师范学院动物科技学院秦皇岛066004
刘小辉, 彭永东,周荣艳,张传生,李祥龙*(. 河北农业大学 动物科技学院,保定 0700; . 河北科技师范学院 动物科技学院,秦皇岛 066004)
禽羽毛是反映品种和性别的重要表型器官之一,羽毛颜色、色泽、形态等外观性状是定价的重要参考指标,羽色的多样性也是自身的天然保护色。小眼畸形相关转录因子(MITF)是影响羽色形成的重要基因,主要在色素细胞中表达,在黑色素细胞的存活、迁移、增殖和分化中发挥重要作用,在维持黑色素细胞色素生成中至关重要[1],与多个色素相关基因的表达激活相关,直接参与酪氨酸酶基因家族的表达[2-5],MITF对白绒乌鸡体内黑色素沉积起正向调控作用,其高表达可能有助于乌鸡体内黑色素的形成[6],其自身功能性缺失或突变的发生会导致许多物种出现一系列的表型变化,尤其是在色素细胞中,部分突变导致眼的色素缺失、减退及小眼畸形。例如,美国海德伦德貂的MITF突变会引起皮毛色素减退[7],白色獭兔之间或白色和海狸色獭兔之间MITF外显子1、5、7中部分多态与獭兔的毛色有显著关联[8]。鸡MITF基因定位于12号染色体上,约88 kb,含有10个外显子。MITF表达激活依赖于基因上游激活剂和抑制剂对该基因转录和翻译过程的调控,受到部分转录因子和通路的影响[9]。启动子区突变的发生可能会影响转录因子结合位点,进而影响启动子活性。关于猪的MITF启动子已有相关报道[10],而鸡的该基因启动子仍未见报道。
本研究对鸡MITF基因启动子结构活性的分析,为阐明鸡MITF基因的表达调控机制提供理论基础,并为从遗传学角度分析羽色的形成提供思路。
1 材料与方法
1.1 试验样品
试验用坝上长尾鸡的血液样品来源于张家口京星园生态农业有限公司。
1.2 试验试剂
基因组提取试剂盒、Taq酶、PCR纯化试剂盒、DNA Marker、Trans5α 感受态、染色剂、胎牛血清(全式金,中国北京),T载体、DNA 连接液、KpnⅠ、MluⅠ(TaKaRa,中国大连),DNA小量抽提试剂盒、胶回收试剂盒(生工,中国上海),无内毒素质粒大提试剂盒(天根,中国北京),双荧光素酶报告基因检测试剂盒(Promega,美国),DMEM、DPBS(Hyclone,美国),T4连接酶、Opti-MEM、0.25%胰蛋白酶、Lipofectamine®2000(Thermo Fisher Science,美国),pGL3-Basic、pRL-TK(平皓生物技术有限公司,中国北京)、鸡成纤维细胞(DF1)(北京协和细胞资源库,中国北京)。
1.3 试验方法
1.3.1 启动子序列分析 通过UCSC数据库(http://genome.ucsc.edu/)、NCBI数据库(https://www.ncbi.nlm.nih.gov/)、Ensemble数据库(http://asia.ensembl.org/index.html)和BioEdit分析序列,通过ALGGEN PROMO数据库预测转录因子结合位点(http://alggen.lsi.upc.es/cgi-bin/promo_v3/promo/promoinit.cgi?dirDB=TF_8.3)。
1.3.2 PCR扩增和载体构建 从NCBI数据库获取鸡MITF基因上游序列(登录号: NC_006099.4)2 000 bp,利用Primer5设计引物,分析扩增序列KpnⅠ和MluⅠ的酶切位点,并加入保护碱基(表1),扩增出不 同长度的缺失片段。PCR扩增体系:Taq酶0.5 μL,10×TaqBuffer 5 μL,2.5 mmol·L-1dNTPs 4 μL,DNA(50 ng·μL-1)5 μL,上下游引物(10 μmol·L-1)各1 μL,ddH2O补至50 μL。PCR反应条件:95 ℃预变性5 min;95 ℃变性30 s,退火30 s,72 ℃延伸,退火温度及延伸时间见表1,共33个循环;72 ℃延伸10 min,4 ℃保存。PCR产物通过1%琼脂糖凝胶电泳检测。
PCR产物经琼脂糖凝胶回收纯化,连接pMD19-T(Simple)载体并于Trans5α感受态细胞中进行扩繁,KpnⅠ和MluⅠ双酶切阳性T载体,选取目的片段与pGL3-Basic连接,将菌液PCR扩增,有目的条带的菌液送至北京六合华大基因测序,对质粒进行双酶切,对鉴定为阳性的菌液提取无内毒素质粒,并检测其浓度和纯度。
1.3.3 细胞培养和活性检测 控制培养箱的温度为37℃,CO2稳定在5%,待DF1细胞状态好时,铺于24孔板内,24 h观察细胞状态,贴壁80%左右,进行转染,包含构建的重组质粒(0.475 μg)、pRL-TK(0.025 μg)脂质体(1.0 μL)、Opti-MEM共计50 μL的脂质体和质粒孵育5 min,转染到DF1细胞,48 h检测萤火虫与海肾荧光素酶的相对活性。试验进行3次系统重复,每次包含3次机械重复。
1.3.4 统计分析 分别测得萤火虫荧光素酶活性检测值F和海肾荧光素酶活性检测值R,得到相对值F/R,再除以阴性对照pGL3-Basic的相对荧光素酶活性检测的背景值,以消除不同转染批次的系统误差。利用SPSS19.0软件非参数检验法统计分析试验数据。
表1扩增鸡MITF基因启动子的引物信息
Table1TheinformationofprimersamplifyingchickenMITFgenepromoter

名称Name引物序列(5′→3′)Primersequence产物长度/bpLengthTm/℃延伸时间/sExtensiontimep4⁃FGGGGTACCATTGGGAGGGTGGTGTTTTTGC12856080p2⁃FGGGGTACCGGAGCCAGAGGTGGAGTTTC8606055p5⁃FGGGGTACCGTGTAGTTTGATGTTTACCTTCCTTAAGAGTTCC7515847p6⁃FGGGGTACCCTGTAAAACTTGAAATAGTGAGGCATGTTTTCCAG3516025RCGACGCGTCCGACGCTCCTGCTCCTGFGGGGTACCGGAGCCAGAGGTGGAGTTTCp1⁃RCGACGCGTGGGGCAGGCTGGCGGGCA9606860p3⁃RCGACGCGTGGATTCGTGAGGTCATGCTG8105650
表中黑体字部分表示保护碱基,下划线部分表示酶切位点
Bold letters are protective bases, and underlined sequences are restriction enzyme digestion sites
2 结 果
2.1 坝上长尾鸡MITF启动子扩增与序列分析
以坝上长尾鸡基因组为模板,进行PCR扩增获得了鸡MITF基因候选启动子区域1 385 bp,并对扩增产物进行了测序,与UCSC提交的MITF基因序列(Chromosome 12: 15 557 648~15 559 032 bp)相似性为99.9%(图1),说明片段来源正确,可用于试验。将此处外显子1起始位点作为+1。

图1 坝上长尾鸡MITF基因上游序列与UCSC数据库比对结果Fig.1 Alignment of upstream sequence of Bashang Long-tail chicken MITF gene with UCSC database
2.2 坝上长尾鸡MITF启动子区不同缺失片段的扩增
利用不同引物扩增鸡MITF基因上游序列的缺失片段,包括p1(-660~+300 bp)、p2(-660~+200 bp)、p3(-660~+150 bp)、p4(-1 085~+200 bp)、p5(-551~+200 bp)、p6(-151~+200 bp),PCR扩增出的鸡MITF基因的各个片段经1%琼脂糖凝胶电泳检测(图2),与预期长度一致。
2.3 坝上长尾鸡MITF启动子荧光素酶表达载体的构建与活性分析
PCR扩增片段与pMD-19T载体的连接体系: 1 μL pMD-19T vector,1~4 μL PCR回收产物,5 μL Solution I,ddH2O补足至10 μL,16℃连接,重组质粒经过双酶切后通过T4连接酶连接到表达载体pGL3-Basic,将菌液PCR有目的条带的菌液送至北京六合华大基因进行测序,并对重组质粒进行KpnⅠ和MluⅠ双酶切(图3),对鉴定为阳性的菌液提无内毒素质粒,并检测其浓度和纯度。
将重组表达载体转染到DF1细胞中,进行双荧光素酶活性检测,图4表明,构建的P1~P6的活性与阴性对照组差异显著 (P<0.05),报告基因P2(-660~+200 bp)转染活性值最高,该活性值是对照组pGL3-Basic的50倍,从-1 085 bp缺失到-151 bp时,启动子活性在DF1中先升高后降低,表明-660~+200 bp区域为鸡MITF基因的核心启动子区域,含有200 bp的外显子1。
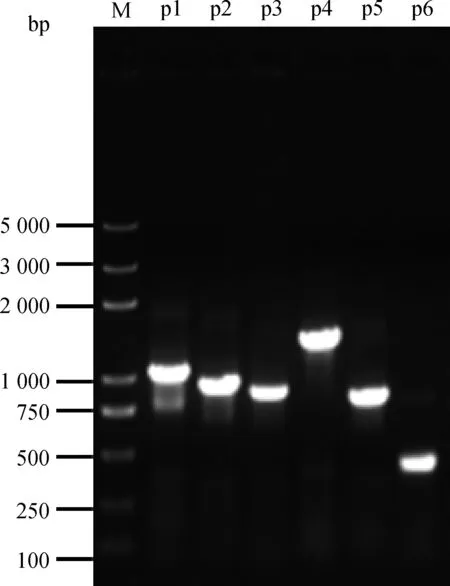
M. DNA相对分子质量标准;p1~p6. 扩增的目的片段M. Trans2K® Plus DNA marker; p1-p6. The amplified target fragments
图2 MITF基因启动子PCR产物琼脂糖凝胶电泳图Fig.2 Agarose gel electrophoresis of PCR products in MITF promoter
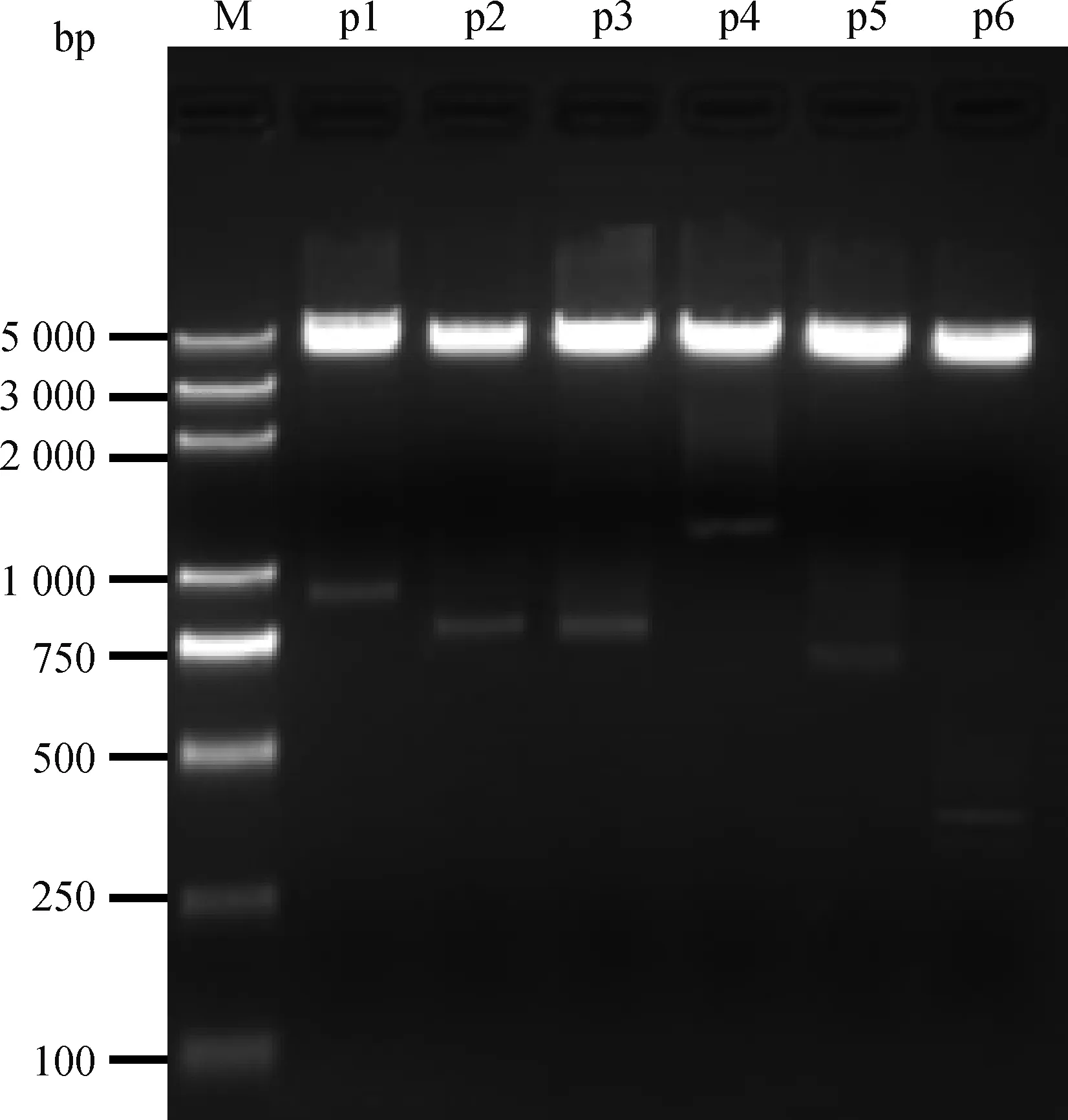
M. DNA相对分子质量标准;p1~p6. 重组质粒酶切鉴定M. Trans2K® Plus DNA marker; p1-p6. Enzyme digestion of each recombinant plasmid
图3 重组质粒双酶切产物琼脂糖凝胶电泳图Fig.3 Agarose gel electrophoresis of recombinant plasmids by dual-enzyme digestion

右图误差线表示标准误,不同质粒间,标有不同小写字母代表差异显著(P<0.05),标有相同小写字母代表差异不显著(P>0.05),图5同The standard error is represented by the error line in the right figure, the different letters on the column indicate the significant difference (P<0.05), and the same letter indicates no significant difference (P>0.05) among the expression vectors, the same as figure5
图4 重组质粒转染DF1细胞后的相对荧光活性值Fig.4 Relative luciferase activity of recombinant plasmids in DF1 cell
2.4 坝上长尾鸡MITF核心启动子区突变
鸡P2上下游引物之间的序列存在突变,UCSC数据库公布了10个单核苷酸多态(single nucleotide polymorphisms,SNPs)位点(图1),以60只坝上长尾鸡DNA为模板,扩增该区域片段,并送至北京六合华大基因测序,发现坝上长尾鸡MITF该区域存在10个突变位点,分别为-579 bp、-505 bp、-274 bp、-220 bp、-203~-202 bp、-98 bp、-46 bp、-14 bp、+1 bp、+28 bp(表2)。
表2核心启动子区突变位点
Table2Mutantsitesinthecorepromoterregion

UCSC参考SNP编号UCSCreferenceSNPID位置/bpPositionUCSC数据库UCSCdatabase坝上长尾鸡BashangLong⁃tailchickenrs739244840-579A/TA/Trs735905475-535A/TArs14983773-505C/TC/Trs317801581-354C/TTrs314129601-294C/TCrs14983774-274A/GA/Grs732406418-220T/AT/A无编号-203~-202ATAT/AT缺失rs317496556-98A/GA/G无编号-46CC/Trs313918239-23A/GGrs316071234-14C/TC/T无编号+1GG/A无编号+28CC/T
2.5 坝上长尾鸡MITF核心启动子区突变载体的构建与活性分析
以坝上长尾鸡的不同个体DNA为模板进行PCR扩增获得突变序列,构建核心启动子区突变载体,分别转染到DF1细胞中,对报告基因进行双荧光素酶活性检测(图5),mut-1~mut-3及P2均显著高于pGL3-Basic活性值(P<0.05),且两两之间差异显著(P<0.05),mut-4与pGL3-Basic活性值差异不显著(P>0.05),均显著低于其他启动子活性值(P<0.05)。

图5 荧光素酶表达载体(P2)及其对应突变载体(mut-1~mut-4)不同位点的碱基组成和活性分析Fig.5 The base composition of different loci and activity analysis of relative luciferase expression vectors (P2) and corresponding mutant vectors (mut-1-mut-4)
2.6 坝上长尾鸡MITF突变位点转录因子结合位点预测
用ALGGEN PROMO数据库对核心启动子区突变区域进行转录因子结合位点预测,碱基突变前后,预测到HOX家族(HOXA3、HOXD8、HOXD9、HOXD10、HOX11)、NF-1、DBP、TFIID和TFIIB转录因子结合位点的相似性发生了变化(图6),例如,-274 A>G导致NF-1与序列结合性增强, -203~-202AT缺失后HOXD9、HOXD10与序列结合性降低。-98C>T导致TFIID、HOXD8与序列结合性增强,-46C>T导致HOXA3与序列结合性略有降低,-14C>T导致NF-1和HOXA3与序列结合性增强,+1G>A导致DBP与序列结合性降低,+28C>T导致TFIIB与序列结合性降低。

图6 转录因子结合位点的相似性Fig.6 The similarity of transcription factor binding sites
3 讨 论
MITF基因是影响羽色形成的重要基因,MITF通过和其他基因互作对皮肤毛色形成以及其他组织色素形成均有作用,其自身功能性缺失或突变的发生会影响基因表达量的改变进而影响表型。启动子区突变的发生可能会影响转录因子结合位点,影响启动子活性。本研究通过鸡MITF基因启动子活性分析,确定-660~+200 bp区域为鸡MITF基因的核心启动子区域。通过启动子区的突变载体mut-1~mut-4活性分析表明,-579 bp、-505 bp、-274 bp、-220 bp、-203~-202 bp、-98 bp、-46 bp、-14 bp、+1 bp、+28 bp位点对MITF基因启动子活性有较大影响,通过ALGGEN PROMO数据库进行转录因子结合位点预测,发现MITF基因核心启动子突变区域HOX家族(HOXA3、HOXD8、HOXD9、HOXD10、HOX11)、NF-1、DBP、TFIID和TFIIB转录因子结合位点的相似性发生了变化。
有研究表明,基因转录时,转录起始复合物(pre-initiation complex, PIC)是启动基因表达的关键,包括RNA聚合酶II以及普遍转录因子TFIIA、TFIIB、TFIID、TFIIE、TFIIF和TFIIH。TFIIB是继TFIID之后加入形成转录起始复合物PIC的重要组成部分,TFIIB有两个结构域,一个可以促进TFIID的TATA结合蛋白(TBP)亚基与RNA聚合酶II结合,决定转录起始,另一个可以介导其他普遍转录因子的介入,TFIIB是一些反式调控元件的作用靶点[11-15]。DPB是重要的转录调控因子,例如感染病毒后,DPB转录本或蛋白的缺失,使得芽病毒和包涵体部分减少[16],DBP可通过DBP-TRIM55-TNF-α-CCL2通路上调单核趋化因子CCL2的表达[17],也可以使IGFBP-1启动子活性增强[18]。由本研究结果分析可知,mut-4活性缺失,存在+1G>A,+28C>T突变,+1位点DBP与序列结合性降低,+28位点TFIIB与序列结合性降低,说明启动子活性缺失可能是由于DBP对MITF有正调控作用,同时,转录因子TFIIB与序列结合性降低,无法形成转录起始复合物而使得转录受到抑制从而影响mut-4启动子的活性。
张涛等[19]的研究结果表明,NF-1对热休克蛋白90的表达起到一定抑制作用,而李丽莎等[20]的研究表明,NF-1为山羊PMEL基因转录的正调控元件。同源框基因(homeobox genes,Hox)表达的蛋白质是重要的转录调节因子,在人、小鼠、绒山羊已有相关研究[21],Hox基因在毛囊生长发育过程中扮演着重要的角色,几乎所有的Hox家族基因均在皮肤和毛囊中表达[22-23]。本试验中,mut-1、P2、mut-2、mut-3活性值依次显著降低(P<0.05),推测启动子活性值改变可能是由于启动子区突变引起了转录因子HOX家族(HOXA3、HOXD8、HOXD9、HOXD10、HOX11)、NF-1、DBP、TFIID和TFIIB与启动子序列的结合性发生了改变,这些转录因子可能对鸡MITF基因表达具有调控作用,进一步结合染色质免疫共沉淀(CHIP)和凝胶阻滞电泳(EMSA)技术,将对研究MITF基因在毛囊组织中的表达调控提供依据。
4 结 论
本研究构建了6个含有不同长度鸡MITF基因启动子片段的pGL3-Basic表达载体及4个核心启动子区突变载体,确定了核心启动子区域为-660~+200 bp,其中-579 bp、-505 bp、-274 bp、-220 bp、-203~-202 bp、-98 bp、-46 bp、-14 bp、+1 bp、+28 bp突变位点对MITF基因启动子活性有较大影响。并且软件预测这些位点突变改变了HOX家族(HOXA3、HOXD8、HOXD9、HOXD10、HOX11)、NF-1、DBP、TFIID和TFIIB与序列的结合性。
参考文献(References):
[1] 赵兵令,王海东,陈天直,等.小鼠毛囊不同生长周期中MITF下游色素相关基因的定位表达及相关性分析[J].中国生物化学与分子生物学报,2017,33(2):198-206.
ZHAO B L,WANG H D,CHEN T Z,et al.Localization and correlation analysis of MITF downstream pigmentation-related genes at different growth cycle of mouse hair follicle[J].ChineseJournalofBiochemistryandMolecularBiology,2017,33(2):198-206.(in Chinese)
[2] VACHTENHEIM J,BOROVANSKY J."Transcription physiology" of pigment formation in melanocytes:central role of MITF[J].ExpDermatol,2010,19(7):617-627.
[3] STEINGRIMSSON E,COPELAND N G,JENKINS N A.Melanocytes and theMicrophthalmiatranscription factor network[J].AnnuRevGenet,2004,38(38):365-411.
[4] 黎 钊,王 平,洪为松,等.正常人黑素细胞MITF对酪氨酸酶相关蛋白的转录调控研究[J].医学研究杂志,2013,42(3):58-62.
LI Z,WANG P,HONG W S,et al.Effect of MITF transcriptional control on family of tyrosinase-related proteins in normal human melanocytes[J].JournalofMedicalResearch,2013,42(3):58-62.(in Chinese)
[5] 杨玉静,张丹瑾,聂瑞强,等.绵羊MITF-M在黑素细胞中过表达后的功能分析[J].中国农业科学,2016,49(21):4214-4221.
YANG Y J,ZHANG D J,NIE R Q,et al.The function analysis of over-expression of oar MITF-M in melanocytes[J].ScientiaAgriculturaSinica,2016,49(21):4214-4221.(in Chinese)
[6] 郑嫩珠,辛清武,朱志明,等.白绒乌鸡MITF基因的cDNA克隆、表达及其对黑色素沉积的影响[J].中国农业科学,2015,48(18):3711-3718.
ZHENG N Z,XIN Q W,ZHU Z M,et al.cDNA cloning and expression of MITF gene and its effect on melanin deposition in silky fowl[J].ScientiaAgriculturaSinica,2015,48(18):3711-3718.(in Chinese)
[7] MARKAKIS M N,SOEDRING V E,DANTZER V,et al.Association ofMITFgene with hearing and pigmentation phenotype in Hedlund white American mink (Neovisonvison)[J].JGenet,2014,93(2):477-481.
[8] 牛晓艳,任克良,曹 亮,等.獭兔MITF基因部分外显子多态性研究[J].山西农业科学,2013,41(11):1231-1234.
NIU X Y,REN K L,CAO L,et al.Research of polymorphisms of parts of exons inMITFgene in rex rabbit[J].JournalofShanxiAgriculturalSciences,2013,41(11):1231-1234.(in Chinese)
[9] HARTMAN M L,CZYZ M.MITF in melanoma:mechanisms behind its expression and activity[J].CellMolLifeSci,2015,72(7):1249-1260.
[10] 张廷焕.猪MITF基因转录调控区域克隆及活性分析[D].雅安:四川农业大学,2014.
ZHANG T H.Cloning and activity analysis of pig microphthalmia-associtated transcription factor (MITF) gene promoter[D].Ya'an:Sichuan Agricultural University,2014.(in Chinese)
[11] KNUTSON B A,HAHN S.TFIIB-related factors in RNA polymerase I transcription[J].BiochimBiophysActa,2013,1829(3-4):265-273.
[12] LIU X,BUSHNELL D A,WANG D,et al.Structure of an RNA polymerase II-TFIIB complex and the transcription initiation mechanism[J].Science,2010,327(5962):206-209.
[13] ORPHANIDES G,LAGRANGE T,REINBERG D.The general transcription factors of RNA polymerase II[J].GenesDev,1996,10(21):2657-2683.
[14] ROEDER R G.The role of general initiation factors in transcription by RNA polymerase II[J].TrendsBiochemSci,1996,21(9):327-335.
[15] 张爱贤,夏云飞.普通转录因子TFⅡB与肝细胞癌相关的研究进展[J].南通大学学报:医学版,2011,31(1):58-61.
ZHANG A X,XIA Y F.Advances in research on the association of general transcription factor II B and hepatocellular carcinoma[J].JournalofNantongUniversity:MedicalSciences,2011,31(1):58-61.(in Chinese)
[16] KATSUMA S.Transcription of dbp from the coding region of theBm17 gene is required for the efficient propagation of Bombyx mori nucleopolyhedrovirus[J].VirusRes,2016,223:57-63.
[17] 吕 杨,陈 蕾,谢院生,等.转录因子DBP对系膜增殖性肾炎免疫炎症反应的作用及其机制研究[C]∥中国中西医结合学会肾脏疾病专业委员会2015年学术年会资料汇编.南宁:中国中西医结合学会肾脏疾病专业委员会,2015:846.
LV Y,CHEN L,XIE Y S,et al.The role of transcriptional factor DBP in immune inflammatory response to mesangial proliferative nephritis and its mechanism[C]∥Information Compilation of the 2015 Academic Annual Meeting of the Professional Committee on Kidney Disease in Chinese and Western Medicine Society.Nanning:Chinese Academy of Integrated Traditional Chinese and Western Medicine Kidney Disease Specialized Committee,2015:846.(in Chinese)
[18] BABAJKO S,GROYER A.Interplay of the liver-enrichedtrans-acting factors,DBP and HNF1,in the transactivation of human IGFBP-1 promoter[J].BiochemBiophysResCommun,1993,196(1):480-486.
[19] 张 涛,张子平,贾锡伟,等.杂色鲍HSP90基因启动子的功能分析[J].水产学报,2017,41(4):490-497.
ZHANG T,ZHANG Z P,JIA X W,et al.Functional analysis of the promoter inHaliotisdiversicolorHSP90 gene[J].JournalofFisheriesofChina,2017,41(4):490-497.(in Chinese)
[20] 李丽莎,彭永东,郑晓宁,等.山羊PMEL基因启动子活性及转录调控元件分析[J].畜牧兽医学报,2017,48(5):826-835.
LI L S,PENG Y D,ZHENG X N,et al.Analysis of the promoter activity and transcriptional regulatory elements of goatPMELgene[J].ActaVeterinariaetZootechnicaSinica,2017,48(5):826-835.(in Chinese)
[21] MORGAN R,SIMPSON G,GRAY S,et al.HOX transcription factors are potential targets and markers in malignant mesothelioma[J].BMCCancer,2016,16(1):85-96.
[22] 金淑清,浦予飞,裘 莹.HOX基因的研究进展[J].癌症进展,2011,9(2):154-158.
JIN S Q,PU Y F,QIU Y.Hox genes mechanisms[J].OncologyProgress,2011,9(2):154-158.(in Chinese)
[23] 李 慧,花保祯.Hox基因及其进化机制的研究进展[J].动物学杂志,2011,46(1):136-142.
LI H,HUA B Z.Hoxgenes and their evolutionary mechanisms[J].ChineseJournalofZoology,2011,46(1):136-142.(in Chinese)
