养殖水体三种大型爆发性海洋藻类孢子/配子实时荧光定量PCR检测方法的建立与应用初探❋
2018-06-20张倩倩包卫洋
缪 栋, 苏 蕾, 张倩倩❋❋, 包卫洋, 龚 骏
(1. 扬州大学环境科学与工程学院,江苏 扬州 225000;2. 中国科学院烟台海岸带研究所,山东 烟台 264003)
“青苔”是对养殖水体中易爆发的藻类生物的俗称,如丝状藻类、水绵、转板藻、石莼等,包含绿藻门,蓝藻门,褐藻门中的多种原核和真核藻类。近年来,海参池塘养殖过程中大型青苔藻类爆发有增加的趋势,已经严重危害到养殖生物的安全,给海参养殖产业带来巨大经济损失[1]。海参养殖环境中大型藻类爆发的危害体现在如下几个方面:与有益微藻(硅藻等)竞争光、营养盐等限制性资源,导致饵料生物生长缓慢乃至缺乏,限制海参的生长;藻类爆发可能伴随毒素的分泌,对养殖生物和其他水生生物造成生理胁迫和化学毒害,严重时导致生物大量死亡[2];青苔的丝状结构会使海参困在其中,影响海参的运动和摄食;另外,藻类死后沉积在池底,在微生物作用下分解并消耗大量溶解氧,引起缺氧和底质腐败,并导致海参缺氧死亡[3-4]。在养殖过程中,当藻类已经长成肉眼可见的青苔时,会进行人工捞除或除草剂清除,但由于藻类丝状体生长迅速,在这些阶段人工干预或清除,效果均不尽理想。若能在青苔大量形成以前检测有关指标并预警,则可能大大降低后期控制的难度,从而减轻青苔爆发的危害。
绿藻门的石莼属(Ulva)和褐藻门的萱藻属(Scytosiphon)是海参养殖过程中常见的青苔藻类。石莼是发生绿潮的典型藻类[5],一般在春季繁殖,生活史中存在单性生殖、无性生殖和有性生殖[6];萱藻(Scytosiphonlomentaria)一般在冬、春季繁殖,存在有性生殖和无性生殖共存的现象[7-8]。2种大型藻类生活史中均产生单细胞的游动配子体和孢子体。青苔藻类生殖细胞的数量可能与后期形成的营养体数量直接相关。研究发现,营养盐丰富时,石莼可释放具有高耐受性、能快速附着的生殖细胞(孢子或配子),对其形成绿潮起到关键作用[9]。对于上述青苔藻类,若能在成体形成前对藻类生殖细胞的数量和动态进行监控,将有助于青苔爆发的早期预警。然而,因孢子体和配子体等生殖细胞个体较小(<10 μm), 在光镜下不易与其他单细胞原生生物(如自养型及混合营养型鞭毛虫)相区别,仅通过形态学观察难以确定其种类或生物量,很难实现对自然水体中这类生殖细胞的数量检测和动态监测。
使用分子生物学方法,例如实时荧光定量PCR(qPCR)技术,通过特异性引物对单细胞微生物的基因拷贝数进行定量,在一定程度上回避了形态学研究中微型生物难以鉴定和定量的难题[10]。利用qPCR技术开展对甲藻(Pyrrophyta)等单细胞赤潮藻的生物量检测,显示出高的特异性和敏感度[11-12]。何闪英等[11]利用18S rDNA特异性引物和Taqman探针,对红色裸甲藻(Gymnodiniumsanguineum)进行了物种鉴定,并构建了qPCR检测方法;其研究发现,qPCR方法的检测下限远低于赤潮爆发时的藻密度,可对赤潮爆发进行预警。然而,利用qPCR技术对大型藻类生殖细胞的检测,已开展的应用性研究还较少,缺乏相关的实验探讨。
核糖体转录间隔区(ITS1-5.8S rDNA-ITS2,简称ITS),是分子生态学常用的分子标记之一,具有丰富的基因变异信息,适用于类群特异性检测和qPCR实验[13-14]。ITS的适用性已在甲藻(Pyrrophyta)和蓝藻(Cyanophyta)等微型藻类的qPCR检测中得以印证[15-16]。在石莼属中,ITS作为分子标记用于区分肠浒苔(Enteromorphaintestinalis)和扁浒苔(Enteromorphacompressa)的多个地理种群[17],体现出种群特异性。对于石莼和萱藻两类大型藻,目前可用的分子标记仅涉及核糖体小亚基基因(18S rDNA)[18],可用的ITS引物较少。
针对以上问题,本工作对山东省烟台市海参养殖池塘中常见的青苔藻(2种石莼和一种萱藻)进行了采集,并在青苔爆发期间对海参养殖水体进行了周年取样,以获取青苔孢子体和配子体。通过扩增藻类的18S rDNA和ITS获得藻体序列,对3种藻类分别设计了以ITS为靶区域的类群/物种特异性引物;建立了qPCR检测方法,以期对养殖水体中非青苔爆发期藻类的孢子和配子体进行定量检测。另外,在本研究中,以萱藻为例定量描述了其孢子体/配子体在6个不同季度水体样品中的相对数量,探索利用此方法对青苔爆发进行早期预警的适用性。
1 材料与方法
1.1 藻类采集和样品来源
本研究所用的藻类(石莼、萱藻及其他对照种类)均采自山东省烟台市云溪村东方海洋海参养殖基地(37°26′44″N, 121°32′4″E)。在2014年12月—2016年3月间每3个月采集养殖水体水样1次(共6次)用于分析孢子体/配子体。每次在养殖池不同位置设置2个采样点(相隔约30 m)作为生物学重复,采集表层水样。每份水样约300 mL,经200 μm孔径的筛绢预过滤后,缓慢抽滤到0.22 μm孔径的醋酸纤维素膜上(直径47 mm;Millipore,美国),进行包含孢子体/配子体在内的微型生物富集,膜样于-80 ℃超低温保存,用于DNA提取。藻类丝状体采集于2015年3月,处于青苔爆发期(优势种类为萱藻)。
1.2 DNA提取和核糖体基因测序
藻类丝状体样品经去离子水冲洗10 min,去掉表面附着物,转移至乘有300 mL 95%乙醇的烧杯中,玻璃棒轻微搅拌30 s;后移至300 mL 0.05%次氯酸钠中,静置漂洗2 min;再转移至 300 mL 70%的乙醇中,静置漂洗2 min;最后用灭菌超纯水冲洗若干次。清洗干净的藻类加入液氮研磨,称取0.5 g研磨产物用于DNA提取。DNA提取采用植物DNA提取试剂盒(Plant Genomic DNA Kit,Tiangen,北京),提取步骤参照试剂盒说明进行。富集有微型生物样品的膜样使用FastDNA®土壤提取试剂盒(Mpbio,美国)进行总DNA提取,两个生物学重复的DNA样品分开处理。
使用真核生物通用引物EukA(5’-TGATCCTTCTGCAGGTTCACCTAC-3’)和EukB (5’-TCCTCCGCTTATTGATATGC-3’)对3种藻类丝状体的核糖体小亚基基因(18S rDNA)进行扩增。PCR扩增使用Fermentas DreamtaqTMDNA 合成酶试剂盒(Thermo Scientific,美国),25 μL体系含有10×缓冲液(Mg2+)2.5 μL,dNTP(10 mmol/L)0.5 μL,引物(10 μmol/L)各1 μL,Dream taq酶(500 U)0.2 μL,灭菌超纯水19.8 μL。PCR扩增程序为:94 ℃预变性5 min;94 ℃变性1 min,56 ℃退火1 min,72 ℃延伸2 min,循环30次;72 ℃延伸10 min。使用ITS通用引物ITS1(5’-TCCGTAGGTGAACCTGCGG-3’)和ITS4(5’-TCCTCCGCTTATTGATATGC-3’)对藻类丝状体转录间隔区(ITS1-5.8S-ITS2)进行扩增,PCR扩增体系同上,扩增程序为94 ℃预变性5 min;94 ℃变性1 min,55 ℃退火40 s,72 ℃延伸90 s,循环35次;72 ℃延伸10 min。PCR扩增产物经琼脂糖凝胶电泳,使用TIANprep Midi 凝胶回收试剂盒(Tiangen,北京)进行回收。回收产物经InsTAclone PCR 克隆试剂盒(Thermo,Winmington,DE,美国)载体连接,转化至DH5а感受态细胞中,进行蓝白斑筛选。挑取白斑菌落进行PCR检测,阳性克隆送至上海生工公司进行双端测序(上海生工科技有限公司,中国)。
1.3 引物设计
使用1.2中测得3种藻类18S rDNA和ITS序列,在NCBI数据库中分别选取序列相似性高于90%的同属或同科物种构建数据集,查找涵盖ITS部分或全长的类群/物种特异性引物。在18S区域,所用数据集包含石莼属新测序列7条,参考序列47条,涵盖石莼属11个物种和石莼科9个属;萱藻数据集包含新测序列1条,萱藻属和相近属参考序列49条。在ITS区域,石莼数据集包含新测序列8条,参考序列49条,涵盖物种Ulvascandinavica,U.armoricana,U.laetevirens,U.fenestrate,U.rigida,U.linza,U.prolifera,Enteromorphaahlneriana等40个石莼属的物种/种群,以及7个Ulvella属种类;萱藻数据集包含新测序列3条,参考序列34条,涵盖Scytosiphonlomentaria的多个种群,S.canaliculatus、S.fascia、S.Petalonia、S.Colpomenia等种属。数据集序列使用在线工具MAFFT(http://mafft.cbrc.jp/alignment/software/)排列后,在18S rDNA右端和ITS区域查找目标物种/类群特异于其他物种的位点,选取的引物序列包含多个特异性位点或以特异性位点为3’端。使用Intergrated DNA Technologies(http://sg.idtdna.com/site)网站对所选特异性引物的GC含量,Tm值,引物二聚体形成,发卡结构产生等参数进行在线评估,筛选高质量引物对。
1.4 引物特异性检验
利用NCBI的BLASTn功能和Primer-BLAST在线软件对引物特异性进行验证。实验室条件下,以5种对照藻类(Ulvacf.linza、Ulvasp.、Scytosiphonlomentaria、U. cf.comperssa、U. cf.flexuosa)基因组DNA为模板进行PCR扩增,检验引物特异性。藻类PCR体系同文中1.2所述,扩增程序为:95 ℃预变性5 min;95 ℃变性30 s,56 ℃退火1 min,72 ℃延伸1 min,循环35次;72 ℃延伸10 min。利用环境水样DNA为模板进行常规PCR实验,进一步验证引物特异性和适用性,所用PCR体系和反应条件同上。藻类和环境水样DNA扩增产物经切胶回收后,克隆测序以确定产物的专一性,克隆测序方法同本文1.2所述。
1.5 实时荧光定量PCR(qPCR)实验
利用上文所得ITS引物,分别进行藻类和环境样品DNA的qPCR实验,所用仪器为ABI 7500fast qPCR仪器(Applied BiosystemsTM,美国)。扩增体系采用SYBR® Premix Ex TaqTMⅡ体系(TaKaRa,日本),20 μL反应体系中含有2×SYBR Green ⅡMix溶液10 μL,DyeⅡ、前后引物(10 μmol/L)各0.4 μL,灭菌超纯水7.8 μL,模板DNA 1 μL。所用程序为:95 ℃预变性7 min;95 ℃变性30 s,56 ℃/58 ℃退火30 s,72 ℃延伸45 s,40个循环;程序在72 ℃时采集信号。
以1.4中藻类常规PCR所得阳性克隆菌液为模板构建质粒标准曲线。使用通用引物M13F和M13R进行质粒PCR扩增,纯化获得含有藻类ITS片段的质粒DNA。将质粒DNA按1∶103~1∶108倍比稀释为7个浓度梯度,每个浓度设置3次重复,分别进行qPCR实验,得出临界循环值(CT)。使用Nanodrop(Quawell,美国)测量不同稀释倍数下质粒的DNA浓度,分别计算得出1 μL质粒模板中含有的 ITS拷贝数,计算公式为:
copies(/μL)=(6.02×1023×(DNA浓度(ng/μL)×10-9))/(DNA长度×660)[10]。
(1)
质粒标准曲线以质粒DNA中ITS拷贝数(log10)为横坐标,CT值(3个重复值的平均数)为纵坐标进行绘制。后续qPCR实验扩增效率根据质粒标准曲线斜率(k)计算,计算公式为:
E=(10-1/k-1)×100%[10]。
(2)
qPCR实验检测藻类和环境样品DNA中的ITS拷贝数。将藻类DNA按1∶10~1∶108倍比稀释,环境水样DNA采用原始浓度,每个稀释度或样品设置三次重复,取1 μL DNA溶液为模板进行qPCR实验,获取模板对应的CT值。参照质粒标准曲线,通过CT值估算模板中所含ITS拷贝数。藻类DNA以梯度稀释倍数(log10)为横坐标,ITS拷贝数(log10)为纵坐标绘制散点图。养殖水样DNA以时间跨度为横坐标,ITS拷贝数为纵坐标绘制柱状图。
2 实验结果
2.1 藻类形态特征和核糖体基因序列
3种藻类的丝状体形态,表层细胞结构如图1所示。藻1黄绿色至深绿色,带状,主支明显,无分支(见图1A),表层细胞四角形至六角形,直径约8~20 μm(见图1C),类似《中国海藻志》中缘管石莼特征(Ulvacf.linza,又名缘管浒苔Enteromorphalinza)[19];藻2鲜绿色,薄叶状,叶片形状不规则,有波皱(见图1D),细胞呈多边形,直径约12~25 μm(见图1F),仅能确认为石莼属[19],未能鉴定到种(Ulvasp.);藻3褐色至深褐色,直径1~2 mm,藻体呈圆柱形,多室配子囊位于叶状体表面或分散各处(见图1H),表面细胞多边形,含有色素体,直径约12~20 μm(见图1I),依《中国黄渤海海藻》,吻合萱藻(Scytosiphonlomentaria)特征[20]。
提取的丝状体藻类基因组DNA浓度,藻1为16.6 ng/μL,藻2为6.3 ng/μL,藻3为16 ng/μL。共获得藻1、藻2、藻3的18S rDNA克隆序列分别有2、4、1条;获得藻1、藻2、藻3的ITS序列分别有1、7、3条。NCBI数据库比对结果显示,藻1中18S rDNA和ITS序列与缘管石莼(U.linza)和浒苔(U.prolifera)的相似度均为99%;藻2 序列与多个石莼物种相似度达99%;藻3序列与萱藻相似度为99%~100%。所测藻类18S rDNA和ITS基因序列已提交GenBank,序列号为KY446810-KY446828。

图1 3种藻类的丝状体形态、表层细胞结构(A~C,缘管石莼类似种;D~F,石莼未定种;G~I,萱藻 )Fig.1 The blades and cells in surface view of three algal species(A~C, Ulva cf. linza; D~F, Ulva sp.; G~I, Scytosiphon lomentaria)
2.2 引物设计及其特异性检测
在ITS1和ITS2区域,3种藻类各获得1对类群/种类特异性引物,分别为Ul_linF171/Ul_linR496(缘管石莼类似种);Ul_rigF79/Ul_rigR543(石莼未定种);Scy_lomF269/Scy_lomR818(萱藻)。引物所在位置和目标片段长度如图2所示。引物长度、GC含量以及适用退火温度(Tm)见表1。
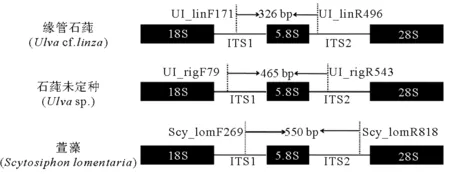
图2 3种藻类ITS区域特异性引物示意图Fig.2 Schematic diagram of the specific primers designed for amplifying the ITS rDNA regions of three algal species
BLASTn和Primer-BlAST在线检验引物特异性,结果显示:在允许0~3个位点错配时,引物Ul_linF171/Ul_linR496仅能匹配到Ulvalinza和U.prolifera两物种,具有种类特异性;引物对Ul_rigF79/Ul_rigR543仅能匹配到U.armoricana、U.sandinavica、U.rigida、U.laetevirens、U.fenestrate、U.lactuca6个物种,具有类群特异性;引物对Scy_lomF269/Scy_lomR818仅能匹配本研究的萱藻(S.lomentaria)种群序列和NCBI库中萱藻物种的部分种群,具有种群特异性。
利用藻类DNA样品进行常规PCR检测,所得琼脂糖凝胶电泳结果显示,3对引物扩增藻类本身均可获得单一高亮条带,对照藻类均无条带(见表2)。所得扩增产物进行克隆测序,分别获得克隆序列缘管浒苔类似种8条,石莼未定种4条,萱藻4条。该部分序列结合2.1中所得藻类ITS序列,进行种群内ITS序列差异分析,发现种群内ITS序列相似度在缘管浒苔类似种为98.8%(4个位点突变),石莼未定种为98.67%(6个位点突变),萱藻为100%,ITS序列在3种藻类中种群内较保守,可用作类群/物种分子标记。

表1 扩增三种藻类ITS rDNA区域的特异性PCR引物参数
对原位养殖水体6个季度的样品DNA检测结果显示,两对石莼引物,Ul_linF171/Ul_linR496和Ul_rigF79/Ul_rigR543,对水样DNA无扩增结果;萱藻引物Scy_lomF269/Scy_lomR818可获得单一条带,且目标片段长度吻合。该条带进行切胶测序,所得2条序列与2.1中萱藻ITS序列相似度为99.8%(1个位点突变),支持扩增片段来自所研究萱藻种群的孢子或配子体。

表2 特异性引物扩增藻类和原位水样DNA结果Table 2 Results of primers sets evaluated by PCR on the genomic DNA of algae and environmental water samples
注:“+”和“—”表示引物对DNA扩增结果:“+”表示有单一条带,“—”表示无条带;*原位水样来自2014年12月—2016年3月间6次采样,“++”“--”是指每个检查两个生物学重复。“+” and “—” indicate the result of primers targeted DNA,“+”indicates positive and presenting single band, “—”indicates negative;water samples were collected from December 2014 to March 2016*,“++” and“--” indicate each sample has two biological replications.
2.3 实时荧光定量PCR结果
使用质粒DNA所含ITS拷贝数的log10对数值为横坐标,临界循环值CT为纵坐标,建立的标准曲线如图3 A,C,E所示。其中,缘管石莼类似种质粒标准曲线回归方程为y=-3.3975x+ 37.127,回归系数R2=0.997 8(见图3A),扩增效率为96.9%;石莼未定种质粒标准曲线回归方程为y=-3.4787x+ 37.503,回归系数R2= 0.996 1(见图3C),扩增效率为93.8%;萱藻质粒样品标准曲线回归方程为y=-3.430 1x+ 36.74,回归系数R2= 0.998 8(见图3E),扩增效率为95.7%。
ITS特异性引物对藻类DNA的qPCR检测结果显示,3种藻类熔解曲线均显示单峰。qPCR检出ITS拷贝数(log10值,y)与藻类DNA稀释倍数(x)的相关性见图3B、D、E所示。在1∶10~1∶105(石莼未定种1∶10~1∶106)的稀释倍数下,3种藻类检出ITS拷贝数(log10值)与稀释倍数呈线性关系,且相关性较高(R2> 0.99),表明qPCR检出的ITS拷贝数可准确反映样品中的藻类DNA含量。在稀释倍数高于105(石莼未定种106)时,藻类ITS拷贝数检出值不随稀释倍数发生降低,该节点DNA浓度视为qPCR方法可检出的藻类DNA的最低浓度,缘管石莼类似种和萱藻分别为1.66×10-4ng/μL,对应200和400个ITS基因拷贝数;石莼未定种为6.6×10-6ng/μL,对应200个ITS基因拷贝数。

(A,B.缘管石莼类似种;C,D.石莼未定种;E,F 萱藻。A, B. Ulva cf. linza; C, D. Ulva sp.; E, F. Scytosiphon lomentaria.)
以海参养殖池不同时期的原位水样DNA为模板,使用萱藻特异性引物进行qPCR检测,获得的熔解曲线显示单峰,表明PCR过程中无非特异性扩增。根据萱藻质粒标准曲线计算样品DNA所含孢子/配子体ITS拷贝数,以ITS拷贝数为指标对不同时期水样中萱藻DNA含量进行比较(见图4)。结果显示,2014年12月份水样中萱藻孢子/配子体ITS拷贝数约为1.0×107拷贝/mL,2015年3月份达到最高,约2.5×107拷贝/mL;自2015年6月起萱藻孢子/配子体ITS拷贝数降低至5.0×106拷贝/mL,此后均低于4.0×106拷贝/mL(见图4)。比较可得,萱藻爆发月份(2015年3月)前一时期(2014年12月)水体中藻类孢子/配子量高于其他非爆发月份;2016年3月份未发生萱藻爆发,当月及其前期(2015年12月份)孢子量均处于较低水平。
3 讨论
针对海参养殖池塘的大型藻类(青苔)灾害爆发,目前常用的防治办法有人工捞除,加入除草剂清除等,耗费大量人工和物力财力。然而藻类一旦爆发,人工捞除后会伴随残余藻类组织或丝状体的再次大量增殖,效果并不明显。除草剂的加入对于水体中的有益藻类、养殖生物(如海参)都会带来严重的刺激作用,破坏水体的生态平衡。治理青苔时机选择宜早不宜晚,使用分子生物学方法在爆发前期和非爆发期对大型藻类孢子数量进行监控和预测,具有重要的现实意义。基于基因层面的检测方法在大型危害藻类孢子和配子检测中的应用还有限,不过它在部分微型浮游藻类的研究中已经展示出巨大的应用潜力[11-12, 21-22],是未来环境检测使用的重要技术手段之一。本工作初步尝试利用qPCR技术,结合特异性引物对海参养殖水体的藻类孢子/配子进行监测,显示了一定的可行性和灵敏度。
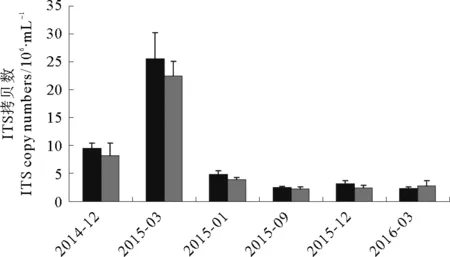
(黑色和灰色表示每个月份两组生物学重复。The black and grey columns indicate the two duplicates in a sampling period.)
图4 表层水样中不同时期萱藻孢子/配子体ITS拷贝数
Fig.4 The temporal variations in ITS abundance ofScytosiphonlomentariaspores in surface water samples
核糖体DNA(rDNA)是真核生物研究中最常用的分子标记之一[13]。相对于18S rDNA的高度保守性,转录间隔区(ITS)具有较高的进化速率,适用于区分低阶元如属级以下的物种或类群[15-17]。Blomster等[17]曾使用ITS特异性引物和探针对肠浒苔和扁浒苔两个形态相似种进行区分,并获得了较好的效果。本工作针对ITS区域设计的萱藻特异性引物显示出较高的物种(种群)特异性,在后续研究中也可应用于萱藻的物种(种群)鉴定,与同属的形态相似物种进行区分。缘管石莼类似种的ITS引物除Ulvalinza外,还匹配NCBI库中多条注释为Ulvaprolifera的序列,原因为Ulvaprolifera序列与缘管石莼在ITS区具有一致的特异性位点,共同区别于其他藻类,但二者无法通过特定的特异性位点区分。因缺乏准确形态鉴定的Ulvaprolifera材料,该引物对Ulvaprolifera的实际扩增效果还有待检测。在后续研究中采用ITS分子标记对Ulvalinza和Ulvaprolifera进行区分,需结合藻类特异性蛋白基因等标记,使用双重或多重PCR方法进行扩增检测[23]。
qPCR方法对基因的最低检出量,反映其检出藻类细胞的灵敏程度,该检出量与基因在细胞基因组/转录组中的含量,引物结合模板的难易程度等有关。何闪英等[11]使用18S rDNA 引物检测红色裸甲藻,最低检出量为130个藻细胞;Touzet等[24]使用ITS物种特异性引物可最低检测出1个亚历山大藻细胞(Alexandriumminutum);而邱圆圆等[25]使用微囊藻素合成酶基因(mcyE)和16S rDNA对微囊藻的定量研究,最低检出限为50个基因拷贝。本工作选择的ITS靶区域位于核糖体RNA基因,为真核细胞中含量最丰富的基因,易于扩增和检出。qPCR方法对3种藻类的最低检出量,缘管石莼类似种和萱藻为1.66×10-4ng基因组DNA,分别对应200和400个ITS拷贝数;在石莼未定种中为6.6×10-6ng DNA,对应200个ITS拷贝数,该结果与已报道的微型藻类的最低检出限具有可比性,呈现出一定的灵敏度。有关石莼和萱藻单个孢子体或配子体中所含rDNA拷贝数的信息还有待研究,用以推断qPCR方法可最低检出的孢子或配子体的细胞数量。
在对水体样品DNA的检测中,两组石莼ITS引物均未获得PCR扩增产物。所用水体样品富集了大量尺寸>0.22 μm的微型生物,包括细菌、原生生物、单胞藻类(硅藻,甲藻等)和其他大型藻类的生殖细胞,两组引物未对其他藻类产生非特异性扩增,在一定程度上说明两组引物具有较好的特异性。另一方面,在本研究所值青苔爆发期间(2015年3月),优势藻类为萱藻,石莼种类数量相对较少,成体多沉积于水体底层,推断其游动孢子体和配子体多出现在藻类丝状体或叶状体附近,因数量较少,未能到达采样水层。
本工作的主要目的是对ITS特异性引物进行设计和验证,对使用qPCR方法检测大型藻类孢子和配子体的可行性进行探讨,故在实际样品检测方面仅做了初步尝试。然而,所得养殖水样的检测结果给出了一些启示:首先,非青苔爆发期萱藻孢子/配子量被检出,且低于爆发期(见图4),表明qPCR方法的灵敏程度满足对非爆发期萱藻孢子量的检测;其次,不同月份萱藻游动孢子/配子量的变化规律与养殖水体的萱藻爆发规律吻合(见结果2.3),萱藻爆发前期伴随水体中孢子量的增高,初步表现出对藻类爆发的预测作用。本工作得出的萱藻爆发规律仍需更详尽的周年采样和实验设计进行验证,需要综合考虑多种因素对大型藻类爆发规律的影响,如养殖活动,季节变化引起的水体温度、营养盐、微生物群落的波动等。
4 结语
本工作针对海参养殖水体中常见的三种大型青苔藻,以高分辨率的ITS区为目标设计类群/物种特异性引物,并测试了其在qPCR实验中的适用性,以期对水体中藻类的孢子量/配子量进行检测。实验中分别测试了qPCR方法对3种藻类的最低检出量,并初步尝试监测海参养殖水体中萱藻孢子量的变化规律,表现出一定的可行性和灵敏度。未来对养殖水体青苔藻类孢子的数量检测和动态监测研究,需测试最适采样水层和样品采集量,并综合多种因素设计采样计划。青苔藻类爆发前期的孢子数量需要统计,以作为监控和预警的标准。在检测到孢子体数量超出标准时,需及时采取措施对来年的青苔爆发进行预防,如适时安排水体循环、减少营养盐的摄入,增加有益藻类细胞竞争等生物、物理、化学手段。
参考文献:
[1] 王印庚, 荣小军. 我国刺参养殖存在的主要问题与疾病综合防治技术要点 [J]. 齐鲁渔业, 2004, 21(10): 29-31.
Wang Y G, Rong X J. Primary problems and techniques of disease prevention in the culture of sea cucumber in China [J]. Shangdong Fisheies, 2004, 21(10): 29-31.
[2] Landsberg J H. The effects of harmful algal blooms on aquatic organisms [J]. Reviews in Fisheries Science, 2002, 10(2): 241-243.
[3] Hiraoka M, Ohno M, Kawaguchi S, et al. Crossing test among floatingUlvathalliforming ‘green tide’ in Japan [J]. Hydrobiologia, 2004, 512(1-3): 239-245.
[4] Lapionte B E, Barile P J, Littler M M, et al. Macroalgal blooms on southeast Florida coral reefs: II. Cross-shelf discrimination of nitrogen sources indicates widespread assimilation of sewage nitrogen [J]. Harmful Algae, 2005, 4(6): 1106-1122.
[5] 邓邦平, 徐韧, 刘材材, 等. 江苏陆地海岸线绿潮藻种类组成及分布特征 [J]. 环境科学学报, 2015, 35(1): 137.
Deng Bangping, Xu Ren, Liu Caicai, et al. Composition and distribution chacteristics of ‘green tide’ algae growing on settled sedimnet in land coastline area of Jiangsu Province [J]. Acta Scientiae Circumstantiae, 2015,35(1): 137.
[6] 马家海, 嵇嘉民, 徐韧, 等. 长石莼(缘管浒苔) 生活史的初步研究 [J]. 水产学报, 2009, 34(1): 45-52.
Ma J H, Ji J M, Xu R, et al. Preliminary study on life history ofUlvalinzaLinnaeus [J]. Journal of Fisheries of China, 2009, 34(1): 45-52.
[7] 邢永泽, 宫相忠, 尹宝树. 萱藻不同发育阶段形态学及生活史的研究 [J]. 中国海洋大学学报(自然科学版), 2010, 40(8): 98-102.
Xing Y Z, Gong X Z, Yin B S. The morphology and life history ofScytosiphonlomentaria[J]. Periodical of Ocean University of China, 2010, 40(8): 98-102.
[8] Li X L, Zhang Z Y, Dong S L, et al. Sexual fusion and life history ofScytosiphonlomentaria(Scytosiphonaceae, Phaeophyceae) in Dalian, China [J]. Journal of Ocean University of China, 2011, 10(2): 170-176.
[9] Tan I H, Blomster J, Hansen G, et al. Molecular phylogenetic evidence for a reversible morphogenetic switch controlling the gross morphology of two common genera of green seaweeds,UlvaandEnteromorpha[J]. Molecular Biology and Evolution, 1999, 16(8): 1011-1018.
[10] Liu X H, Gong J. Revealing the diversity and quantity ofPeritrichciliatesin environmental samples using specific primer-based PCR and quantitative PCR [J]. Microbes and Environments, 2012, 27(4): 487-503.
[11] He S Y, Yu Z G. A real-time PCR method for rapid detection ofGymnodiniumsanguineum[J]. 浙江大学学报(农业与生命科学版), 2009, 35(2): 119-126.
[12] 于志刚, 米铁柱, 姚鹏, 等. 赤潮藻鉴定与定量检测方法进展 [J]. 中国海洋大学学报(自然科学版), 2009, 39(5): 1067-1076.
Yu Z G, Mi T Z, Yao P, et al. Advances in identification and quantification of harmful algae [J]. Periodical of Ocean University of China, 2009, 39(5): 1067-1076.
[13] Hills D M, Dixon M T. Ribosomal DNA: Molecular evolution and phylogenetic inference [J]. Quarterly Review of Biology, 1991, 66(4): 411-453.
[14] Markmann M, Tautz D. Reverse taxonomy: An approach towards determining the diversity of meiobenthic organisms based on ribosomal RNA signature sequences [J]. Philosophical Transactions of the Royal Society B: Biological Sciences, 2005, 360(1462): 1917-1924.
[15] 张宝玉, 王广策, 张炎, 等. 东海原甲藻(Prorocentrumdonghaiense) 和海洋原甲藻 APBM(P.micansAPBM) 的5. 8S rDNA及其转录间隔区(ITS) 的克隆和序列分析[J]. 海洋与湖沼, 2004, 35(3): 264-272.
Zhang B Y, Wang G C, Zhang Y, et al. Cloning and sequence analysis of 5. 8S rDNA and its region fromProrocentrumdonghaienseandP.micansAPBM [J]. Oceanologia Et Limnologia Sinica, 2004, 35(3): 264-272.
[16] Janse I, Kardinaal W E A, Meima M, et al. Toxic and nontoxicMicrocystiscolonies in natural populations can be differentiated on the basis of rRNA gene internal transcribed spacer diversity [J]. Applied and Environmental Microbiology, 2004, 70(7): 3979-3987.
[17] Blomster J, Hoey E, Maggs C, et al. Species-specific oligonucleotide probes for macroalgae: Molecular discrimination of two marine fouling species ofEnteromorpha(Ulvophyceae) [J]. Molecular Ecology, 2000, 9(2): 177-186.
[18] Dong M, Zhang X, Chi X, et al. The validity of a reference gene is highly dependent on the experimental conditions in green algaUlvalinza[J]. Current genetics, 2012, 58(1): 13-20.
[19] 丁兰平. 中国海藻志 [M]. 北京: 科学出版社, 2013: 51-52.
Ding L P. Flora Algaram Marinarum Sinicatrum[M]. Beijing: Science Press, 2013: 51-52.
[20] 曾呈奎. 中国黄渤海海藻 [M]. 北京: 科学出版社, 2009: 347-351.
Zeng C K. Seaweads in Yellow Sea and Bohai Sea of China[M]. Beijing: Science Press, 2009: 347-351.
[21] Park T G, Park Y T. Detection ofCochlodiniumpolykrikoidesandGymnodiniumimpudicum(Dinophyceae) in sediment samples from Korea using real-time PCR [J]. Harmful Algae, 2010, 9(1): 59-65.
[22] Penna A, Battocchi C, Garc S E, et al. Detection of microalgal resting cysts in European coastal sediments using a PCR-based assay [J]. Deep Sea Research Part II: Topical Studies in Oceanography, 2010, 57(3): 288-300.
[23] Ouahid Y, Del Campo F F. Typing of toxinogenicMicrocystisfrom environmental samples by multiplex PCR [J]. Applied Microbiology and Biotechnology, 2009, 85(2): 405-412.
[24] Touzet N, Keady E, Raine R, et al. Evaluation of taxa-specific real-time PCR, whole-cell FISH and morphotaxonomy analyses for the detection and quantification of the toxic microalgaeAlexandriumminutum(Dinophyceae), Global Clade ribotype [J]. FEMS Microbiology Ecology, 2009, 67(2): 329-341.
[25] Qiu Y, Yuan T, Kon T, et al. Rapid detection and quantitation of microcystin-producingMicrocystisusing real-time PCR [J]. Journal of Molecular Biomarkers and Diagnosis, 2013, 5(006): 2155-9929.
