基于线粒体DNA控制区高变区的黄河流域光泽黄颡鱼群体遗传学分析❋
2018-06-20金斌松高天翔
宋 娜 , 周 伟 , 金斌松 , 高天翔
(1. 中国海洋大学水产学院,山东 青岛 266003; 2. 南昌大学生命科学学院,江西 南昌 330031 3. 浙江省海洋渔业资源可持续利用技术研究重点实验室,浙江 舟山 316021)
光泽黄颡鱼(Pelteobagrusnitidus)属于鲶形目(Siluriformes)鲿科(Bagridae)黄颡鱼属(Pelteobagrus),是一种中小型经济鱼类,分布在我国的黑龙江至闽江水系及朝鲜半岛[1]。目前分布在我国的黄颡鱼属鱼类有5种,主要栖息于江河、湖泊的中下层,体型均为中小型,因其肉质细嫩、味道鲜美而成为重要的经济捕捞对象[2]。近年来,由于天然水域环境污染、水利设施的建设及酷渔滥捕现象严重,大型经济鱼类资源衰退严重,包括黄颡鱼属在内的一些小型鱼类在渔获物中所占比例曾逐渐上升,但随着捕捞强度的加大,黄颡鱼属鱼类资源开始衰退,部分水域已发现黄颡鱼(P.fulvidraco)、光泽黄颡鱼等种类出现种群结构小型化现象[3-4]。
目前光泽黄颡鱼的研究资料较为缺乏,主要集中在食性[5-6]、生物学[7]、系统发育[8-9]等方面,而光泽黄颡鱼群体遗传学分析仅见丁言伟[8]采用ND4基因对27个样本进行的遗传多样性分析,结果检测到较高水平的遗传多样性。在已开发的多种分子标记中,DNA序列的直接测序能够准确反应个体间碱基的变异,是目前使用较为广泛的遗传学分析手段之一[10]。线粒体DNA控制区具有碱基替代速率较快、结构简单、严格遵循母系遗传、几乎不发生重组等优点[11],在种群遗传学和种内微观水平的系统发育关系等研究领域有着广泛的应用[10, 12]。近年来随着光泽黄颡鱼资源的逐渐衰退和其养殖业的逐渐发展,开展其遗传背景的相关研究势在必行。本研究采集了黄河水系3个地点的光泽黄颡鱼样品,基于线粒体DNA控制区高变区序列对其遗传多样性、遗传结构和群体历史动态进行研究,以期为光泽黄颡鱼的资源保护和开发提供资料。
1 材料与方法
1.1 样品采集
本研究于2013—2015年采集到3个地点的光泽黄颡鱼样品:黄河陕西渭南段(WN)、黄河河南洛阳小浪底水库(LY)、黄河河南开封段(KF),共79个个体;采集到江西鄱阳湖(PYL)的长须黄颡鱼(P.eupogon)用于遗传学对比分析,共8个个体(见图1和表1)。
1.2 DNA提取
采用传统酚-氯仿方法提取光泽黄颡鱼和长须黄颡鱼基因组DNA,用超纯水将乙醇沉淀后的DNA溶解,-20℃保存备用。
1.3 PCR扩增与产物测序
采用引物L15923 (5’-TTAAAGCATCGGTCTTGTAA-3’)[13]和H16500(5’-GCCCTGAAATAGGAACCAGA-3’)[14]对2种黄颡鱼线粒体DNA(mtDNA)控制区的高变区(Hypervariable region I; HVRI)进行扩增。纯化、回收后的PCR产物送到上海桑尼生物科技有限公司采用ABIPrism 3700自动测序仪进行双向测定。
1.4 数据分析
所有序列均由Dnastar软件包(DNASTAR, Inc., Madison, USA)进行编辑、校对。序列比对采用Dnastar软件包、ClustalX 2.1[15]和Mega 7.0[16]进行。基于BIC衍生的决策论(Decision-theoretic performance-based; DT)搜索策略,采用jModeltest 2.1.6[17]计算HVRI碱基替代模型,本研究中基于决策论法计算得到序列替换模型为HKY。
使用ARLEQUIN 3.5软件[18]分析计算分子多态性指数(如单倍型数目、多态位点数、基因多样度、核苷酸多样度、平均两两碱基差异数等)及群体间遗传分化指数FST、确切P检验等。单倍型邻接关系树(neighbor-joining tree; NJ)使用MEGA 7.0软件构建[16]。通过ARLEQUIN 3.5软件[18]计算核苷酸不配对分布(mismatch distribution),分别模拟群体数量(Demographic / sudden expansion)和空间扩张(range / spatial expansion)。运用Beast 1.8软件[19]中贝叶斯天际线(Bayesian skyline plot; BSP)检测群体历史群体大小变动。
2 结果
本研究中79个个体的光泽黄颡鱼共扩增得到长度为517 bp的mtDNA序列片段,其中包括103 bp的tRNA和414 bp的控制区第一高变区(HVRI)片段。在103 bp的tRNA片段上,并未检测到变异位点。将所有序列经过比对后选择414 bp的HVRI同源片段进行后续分析。
光泽黄颡鱼在414 bp的HVRI片段上,共检测到6处核苷酸替换,分别为5个转换,1个颠换。6个多态位点共定义了5个单倍型,Hap1是共享个体数最多的单倍型,在开封与渭南群体中均检测到5个单倍型的存在,不存在群体特有单倍型,Hap2未在洛阳群体中检测到(见表2)。79个个体的碱基含量分别为:C: 22.47%、T: 29.53%、A: 33.56%、G: 14.44%,A+T碱基含量明显大于C+G的含量。
Note:①Kaifeng;②Luoyang;③Weinan;④Total;⑤Poyang Lake;⑥ID;⑦Sampling date;⑧Number;⑨Number of polymorphic loci;⑩Number of haplotypes;Haplotype diversity;Nucleotide diversity;The mean number of pairwise nucleotide differences
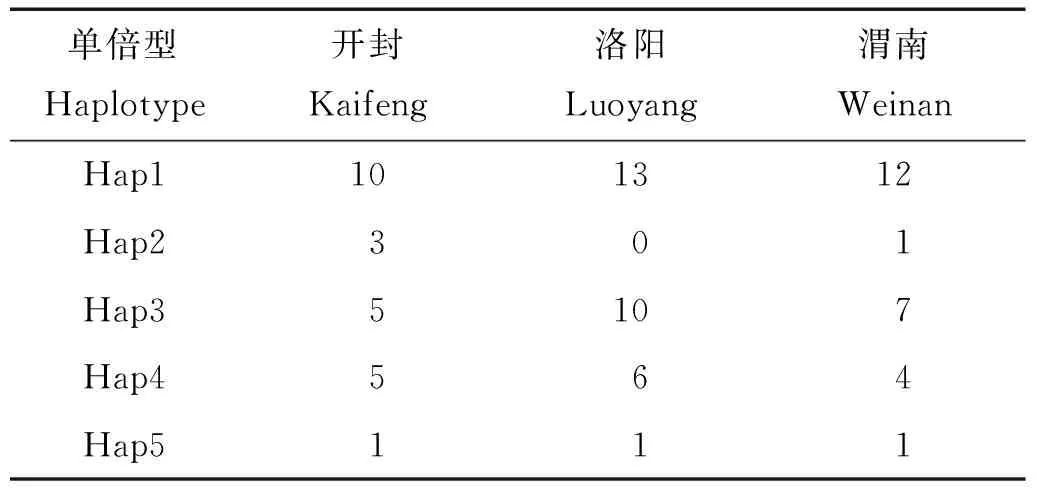
表 2 本研究中光泽黄颡鱼3个群体的单倍型频率分布表Table 2 Haplotype distribution of three P. nitidus populations
光泽黄颡鱼3个群体的单倍型多样度为0.682 8±0.043 7~0.753 6±0.056 2,核苷酸多样度为0.002 5±0.001 9~0.003 4±0.002 4,以开封、渭南、洛阳的顺序逐渐降低,总的单倍型多样度为0.694 9±0.030 4,核苷酸多样度为0.002 8±0.002 0,遗传多样度水平处于中等水平(见表1)。长须黄颡鱼鄱阳湖群体表现出较低的遗传多样度水平(单倍型多样度为0.464 3±0.200 0;核苷酸多样度为0.001 2±0.130 1),这可能与较少的样品数量有一定关系(见表1)。
以长须黄颡鱼的3个单倍型为外群,构建光泽黄颡鱼5个单倍型的邻接关系树(见图2),结果显示两种黄颡鱼分别聚为2个分支,2个分支间的净遗传距离为0.109。与邻接关系树的谱系结构一致,光泽黄颡鱼的单倍型最小跨度树也比较简单,没有分支与地理位置相对应(见图3)。

(分支上的数字表示支持率,支持率<60%的未列出。Bootstrap with <60% were not shown.)
图2 基于光泽黄颡鱼和长须黄颡鱼控制区单倍型构建的邻接关系树
Fig. 2 NJ tree forP.nitidusandP.eupogoncontrol region haplotypes
光泽黄颡鱼3群体的遗传分化指数FST值为-0.0302~0.0038,且统计检验的结果均不显著,表现出较小的遗传分化;确切P检验结果也均不显著,表明两两群体间存在随机交配现象。
对光泽黄颡鱼黄河群体进行群体历史动态检验,结果显示Tajima’sD值为不显著较小的负值,Fu’sFs值为不显著较小的正值,黄河群体的光泽黄颡鱼并不显著偏离中性(见表3)。数量和空间扩张模型核苷酸不配对分布表现出单峰分布,并呈现出“L”型分布(见图4)。Beast下进行3次不同替换速率分布模型模拟,BSP曲线显示黄河流域光泽黄颡鱼有效群体大小发生了近期的下降,表现出近期的瓶颈效应(见图5)。

(*圆圈面积与单倍型频率成正比,短划线代表单倍型间的核苷酸替换数目。*The sizes of circles are proportional to haplotype frequency. Perpendicular tick marks on the lines joining haplotypes represent the number of nucleotide substitutions.)
图3 光泽黄颡鱼控制区单倍型的最小跨度树
Fig. 3 Unrooted minimum spanning trees showing genetic relationship among control region haplotypes ofP.nitidus.

图4 光泽黄颡鱼黄河群体数量扩张核苷酸不配对分布图Fig.4 Mismatch distribution for demographic expansion based on mtDNA control region sequences for 3 populations of P. nutidus
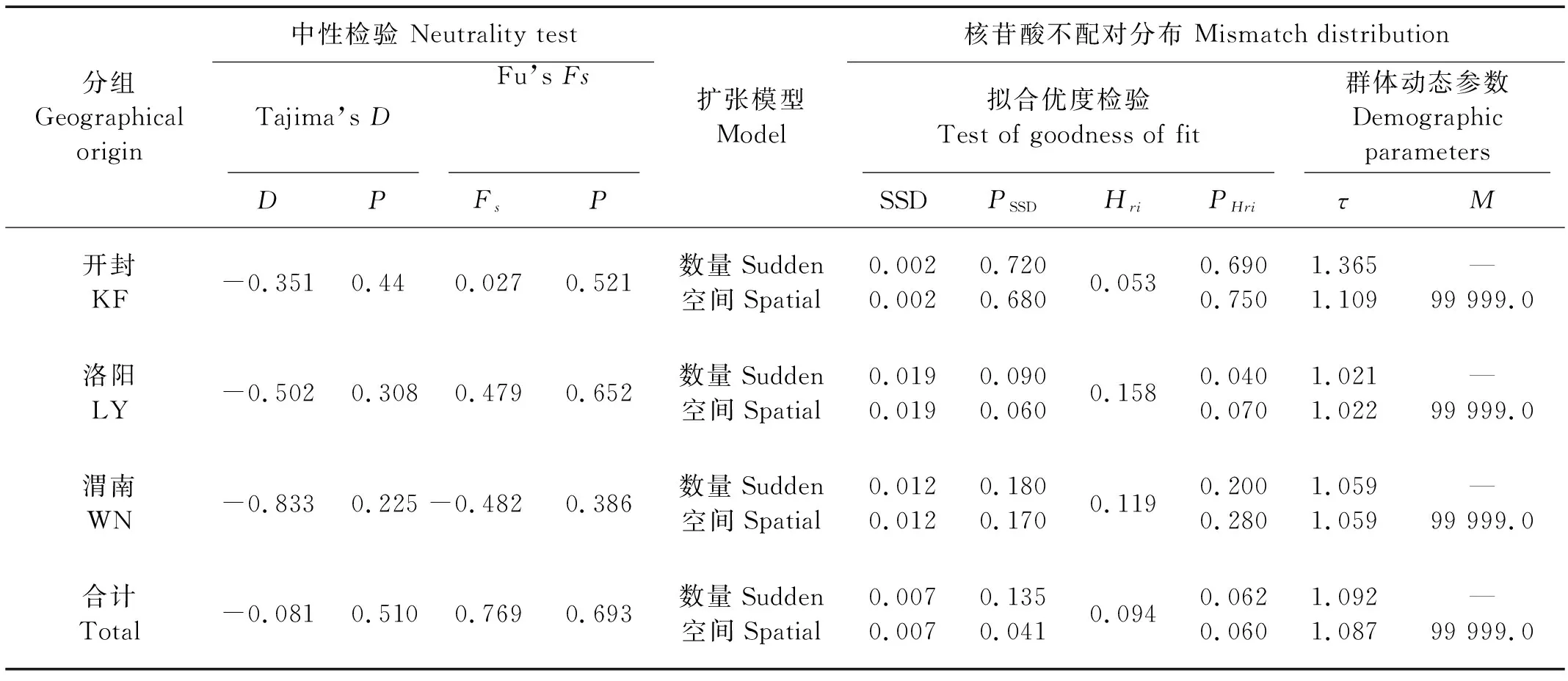
分组Geographicalorigin中性检验 Neutrality testTajima’s DFu’s FsDPFsP扩张模型Model核苷酸不配对分布 Mismatch distribution拟合优度检验Test of goodness of fit群体动态参数Demographic parametersSSDPSSDHriPHriτM开封KF-0.3510.440.0270.521数量Sudden空间Spatial0.0020.0020.7200.6800.0530.6900.7501.3651.109—99 999.0洛阳LY-0.5020.3080.4790.652数量Sudden空间Spatial0.0190.0190.0900.0600.1580.0400.0701.0211.022—99 999.0渭南WN-0.8330.225-0.4820.386数量Sudden空间Spatial0.0120.0120.1800.1700.1190.2000.2801.0591.059—99 999.0合计Total-0.0810.5100.7690.693数量Sudden空间Spatial0.0070.0070.1350.0410.0940.0620.0601.0921.087—99 999.0
3 讨论
遗传多样性通常是物种长期进化的结果,是物种或其群体持续生存并适应不断变化的环境而不断进化的前提。通常物种的遗传多样性或变异性越丰富,则表明该物种的进化潜力越大,对环境改变响应的进化能力就越强[20]。因此物种的开发与保护策略的制定必须建立在对其遗传多样性及种群遗传结构充分了解与认识的基础上[21]。物种遗传多样性和遗传结构的研究主要依赖于分子标记的发展,而分子标记能够直接反应DNA水平上的遗传变异,是一种检测物种群体遗传结构和遗传多样性的有效手段[22]。目前,利用线粒体DNA序列对黄颡鱼群体遗传多样性研究较多[23-26],而该属其他种类的遗传多样性研究却鲜有报道。本研究中光泽黄颡鱼呈现中等程度的遗传多样性水平(h=0.694 9±0.030 4,π=0.002 8±0.002 0),与已报道的黄颡鱼的遗传多样性水平相当[26],可能是由于2种鱼类的分布区域、生活史特征相似导致其具有相似的遗传多样性和遗传结构。丁言伟[8]基于线粒体DNA的ND4序列检测到光泽黄颡鱼较高的遗传多样性,可能是其所采集的27个样本覆盖了不同的水系导致。本研究中光泽黄颡鱼的采样地点仅分布在黄河水系,因此存在较低的遗传多样性水平。两两群体间的遗传分化指数计算结果也显示黄河水系3个群体间存在广泛的基因交流,同一水系的淡水鱼类通常具有相似的遗传特征[27],因此在遗传管理上应作为一个管理单元进行管理。为进一步了解光泽黄颡鱼的种群遗传结构,在后续的研究中需采集其他水系的样品进行比较分析。

图5 光泽黄颡鱼3个群体控制区序列的BSP分析Fig. 5 Bayesian skyline plots showing changes through time of effective population size based on mtDNA control region sequences for 3 populations of P. nutidus
Rogers和Harpending[28]对核苷酸不配对分布数量扩张模型模拟发现,发生瓶颈效应之后群体有效种群数量维持在较小数量时也会在核苷酸不配对分布上对于数量扩张模型表现出相同的分布信号,但是其研究只是定性的描述了两者的区分。Peter等[29]研究表明瓶颈效应会导致种群近期基因交流的现象,存在遗传结构和种群下降会导致相同的遗传谱系。核苷酸不配对分布和中性检验由于基于分离位点和单倍型频率推导群体历史动态,有时不能充分利用DNA序列的动态历史信息[30-31]。本研究群体历史动态显示核苷酸不配对分布数量和空间模型分布为单峰,BSP曲线显示光泽黄颡鱼群体存在近期有效种群数量的下降。因此本研究表明光泽黄颡鱼黄河群体并未表现出群体的近期扩张事件,反而表现出近期的瓶颈效应,有效种群数量降低。
光泽黄颡鱼群体79尾个体只表现出6个简约信息位点,且遗传多样性指数计算结果表明光泽黄颡鱼中等程度的遗传变异。这一方面可能与光泽黄颡鱼自身的喜静、沉性卵等生活习性有关,另一方面更大可能是光泽黄颡鱼近期的瓶颈效应导致遗传信息的丢失,产生大量的低突变碱基和较少的高突变位点。但是本研究基于的HVRI序列的灵敏度较低,也可能导致序列多态较低,从而表现出瓶颈效应的假象。光泽黄颡鱼黄河群体是否确实存在近期的瓶颈效应需要进一步采用较高灵敏度的分子标记手段进一步确认。
4 结语
本研究基于线粒体DNA控制区第一高变区序列对黄河流域的3个光泽黄颡鱼群体进行了遗传学分析,研究结果提示黄河流域的光泽黄颡鱼在遗传管理上应作为一个管理单元进行管理。中等的遗传多样度及近期有效种群数量的瓶颈效应则提示应加大对黄河流域光泽黄颡鱼的保护,以防光泽黄颡鱼有效种群的衰减。
致谢:感谢介子林研究员、王益昌高级工程师等为本实验提供光泽黄颡鱼样品。
参考文献:
[1] 褚新洛, 莫天培, 戴定远. 中国动物志[M]. 北京: 科学出版社, 1999: 152-156.
Chu X L, Zheng B S, Dai D Y. Fauna Sinica (Teleostei): Siluriformes [M]. Beijing: Science Press, 1999: 152-156.
[2] 童芳芳, 汤明亮, 杨星, 等. 用RAPD和SCAR复合分子标记对黄颡鱼属进行种质鉴定[J]. 水生生物学报, 2005 , 29(4): 465-468.
Tong F F, Tang M L, Yang X, et al. Species authentication ofPelteobagrusby multiplex molecular marker RAPD and SCAR[J]. Acta Hydrobiologica Sinca, 2005, 29(4): 465-468.
[3] 邱春刚, 刘景祯, 刘丙阳, 等. 汤河水库黄颡鱼的生物学及其资源利用[J]. 水产科学, 2002, 19(2): 28-30.
Qiu C G, Liu J Z, Liu B Y, et al. Biology and resource utilization ofPelteobagrusfulvidracoin Tanghe Reservoir[J]. Fisheries Science, 2000, 19(2): 28-30.
[4] 谌海虎. 赣江南昌段光泽黄颡鱼的年龄、生长和繁殖特性的初步研究[D]. 武汉:华中农业大学, 2010.
Chen H H. Preliminary Studies on Age, Growth and Reproduction of Yellow CatfishPelteobagrusnitidusin Nanchang Section of Ganjiang River [D]. Wuhan:Huazhong Agricultural University, 2010.
[5] 袁刚, 茹辉军, 刘学勤. 洞庭湖光泽黄颡鱼食性研究[J]. 水生生物学报, 2011, 35(3): 270-275.
Yuan G, Ru H J, Liu X Q. Feeding habits ofPelteobagrusnitidusin Lake Dongting[J]. Acta Hydrobiologica Sinca, 2011, 35(3): 270-275.
[6] 刘其根, 吴杰洋, 颜克涛, 等. 淀山湖光泽黄颡鱼食性研究[J]. 水产学报, 2015, 39(6): 859-866.
Liu Q G, Wu J Y, Yan K T, et al. Feeding habits ofPelteobagrusnitidusin Dianshan Lake[J]. Journal of Fisheries of China, 2015, 39(6): 859-866.
[7] 耿龙, 高春霞, 韩婵, 等. 淀山湖光泽黄颡鱼的生物学初步研究[J]. 上海海洋大学学报, 2014, 23(3): 435-440.
Geng L, Gao C X, Han C, et al. Primary biological study of yellow catfish (Pelteobagrusnitidus) in Dianshan Lake[J]. Journal of Shanghai Ocean University, 2014, 23(3): 435-440.
[8] 丁言伟. 黄颡鱼属(硬骨鱼纲, 鲿科)鱼类分子系统发育及种群遗传结构的研究[D]. 武汉:华中农业大学, 2005.
Ding Y W. Molecular Phylogenetics and Population Genetic Structure of GenusPelteobagrus(Teleostei, Bagridae) [D]. Wuhan: Huazhong Agricultural University, 2005.
[9] Ku X, Peng Z, Diogo R, et al. MtDNA phylogeny provides evidence of generic polyphyleticism for East Asian bagrid catfishes[J]. Hydrobiologia, 2007, 579(1): 147-159.
[10] Avise J C. Phylogeography: Retrospect and prospect[J]. Journal of Biogeography, 2009, 36(1): 3-15.
[11] Meyer A. Evolution of mitochondrial DNA in fishes[J]. Biochemistry and Molecular Biology of Fishes, 1993, 2: 1-38.
[12] Liu J X, Gao T X, Wu S F, et al. Pleistocene isolation in the Northwestern Pacific marginal seas and limited dispersal in a marine fish,Chelonhaematocheilus(Temminck & Schlegel, 1845) [J]. Molecular Ecology, 2007, 16(2): 275-288.
[13] Iguchi K, Tanimura Y, Nishida M. Sequence divergence in the mtDNA control region of amphidromous and landlocked forms of ayu[J]. Fisheries Science, 1997, 63(6): 901-905.
[14] Inoue J G, Kumazawa Y, Miya M, et al. The historical biogeography of the freshwater knifefishes using mitogenomic approaches: a mesozoic origin of the Asian notopterids (Actinopterygii: Osteoglossomorpha) [J]. Molecular Phylogenetics and Evolution, 2009, 51(3): 486-499.
[15] Larkin M A, Blackshields G, Brown N P, et al. Clustal W and Clustal X version 2. 0[J]. Bioinformatics, 2007, 23(21): 2947-2948.
[16] Kumar S, Stecher G, Tamura K. MEGA7: molecular evolutionary genetics analysis version 7. 0 for bigger datasets[J]. Molecular Biology and Evolution, 2016, 33(7): 1870.
[17] Posada D. JModel test: phylogenetic model averaging[J]. Molecular Biology and Evolution, 2008, 25(7): 1253-1256.
[18] Excoffier L, Lischer H E L. Arlequin suite ver 3. 5: a new series of programs to perform population genetics analyses under Linux and Windows[J]. Molecular Ecology Resources, 2010, 10(3): 564-567.
[19] Drummond A J, Rambaut A. BEAST: Bayesian evolutionary analysis by sampling trees[J]. BMC Evolutionary Biology, 2007, 7(1): 214.
[20] Laikre L, Palm S, Ryman N. Genetic population structure of fishes: Implications for coastal zone management[J]. Ambio, 2005, 34(2): 111-119.
[21] Falk D A, Holsinger K E. Genetics and Conservation of Pare Plants[M]. USA: Oxford University Press, 1991.
[22] Whitehead A, Anderson S L, Kuivila K M. Genetic variation among interconnected populations ofCatostomusoccidentalis: Implications for distinguishing impacts of contaminants from biogeographical structuring[J]. Molecular Ecology, 2003, 12(10): 2817-2833.
[23] Watanabe K, Nishida M. Genetic population structure of Japanese bagrid catfishes[J]. Ichthyological Research, 2003, 50(2): 140-148.
[24] 钟立强, 刘朋朋, 潘建林,等.长江中下游5个湖泊黄颡鱼(Pelteobagrusfulvidraco)种群线粒体细胞色素b基因的遗传变异分析[J].湖泊科学, 2013, 25(2): 302-308.
Zhong L Q, Liu P P, Pan J L, et al. Genetic variation analysis of yellow catfish (Pelteobagrusfulvidraco) from five lakes in the middle and lower reaches of the Yangtze River based on Mitochondrial DNA Cytb[J]. Journal of Lake Sciences, 2013, 25(2): 302-308.
[25] 刘朋朋, 钟立强, 潘建林,等.基于线粒体D-loop区分析黄颡鱼(Pelteobagrusfulvidraco)五个淡水湖泊群体的遗传多样性[J].海洋与湖沼, 2013, 44(3): 728-733.
Liu P P, Zhong L Q, Pan J L, et al. Population genetics in mitochondrial DNA control region for five fresh-water yellow catfishPelteobagrusfulvidraco[J]. Oceanologia et Limnologia Sinica, 2013, 44(3): 728-733.
[26] 周伟, 王俊, 金斌松, 等. 黄颡鱼群体遗传变异分析[J]. 水产学报, 2016, 40(10): 1531-1541.
Zhou W, Wang J, Jin B S. Genetic variation based on the mitochondrial DNA control region of yellow catfish (Pelteobagrusfulvidraco) [J]. Journal of Fisheries of China, 2016, 40(10): 1531-1541.
[27] 苏军虎, 张艳萍, 娄忠玉, 等. 基于线粒体控制区序列的黄河上游厚唇裸重唇鱼种群遗传结构[J]. 生态学报, 2012, 32(13): 4191-4198.
Su J H, Zhang Y P, Lou Z Y, et al. Genetic structure of Gymnodiptychus pachycheilus from the upper reaches of the Yellow River as inferred from mtDNA control region[J]. Acta Ecologica Sinica, 2012, 32(13): 4191-4198.
[28] Rogers A R, Harpending H C. Population growth makes waves in the distribution of pairwise genetic differences[J]. Molecular Biology and Evolution, 1992, 9(3): 552-569.
[29] Peter B M, Wegmann D, Excoffier L. Distinguishing between population bottleneck and population subdivision by a Bayesian model choice procedure[J]. Molecular Ecology, 2010, 19(21): 4648-4660.
[30] Fitzpatrick S W, Brasileiro C A, Haddad C F B, et al. Geographical variation in genetic structure of an Atlantic coastal forest frog reveals regional differences in habitat stability[J]. Molecular Ecology, 2009, 18(13): 2877-2896.
[31] Hu Z M, Li W, Li J J, et al. Post-Pleistocene demographic history of the North Atlantic endemic Irish mossChondruscrispus: Glacial survival, spatial expansion and gene flow[J]. Journal of Evolutionary Biology, 2010, 24(3): 505-517.
