盐碱胁迫及植物耐盐碱分子机制研究
2018-06-19,,
, ,
(1.中国科学院 东北地理与农业生态研究所,黑龙江 哈尔滨 150081;2.中国科学院大学,北京 100049)
0 引 言
土地盐碱化是世界范围内普遍存在的问题,严重影响植物的生长和发育,限制农作物的质量和产量。全世界有8.31亿hm2的土壤因盐碱度过高而不能被有效利用。其中,4.34亿hm2为碱化土壤,3.97亿hm2为盐化土壤[1]。位于我国东北的松嫩平原是世界上盐碱地分布最广的地区之一,有盐碱地373万hm2,且正以每年1.4%的速率逐年递增。

水稻是世界范围内重要的粮食作物,在我国种植面积达4.5亿hm2,居世界第二位。盐胁迫对水稻的伤害包括营养生长和生殖生长的各个时期:萌发期可降低种子发芽率,延迟种子发芽时间;孕穗期阻碍幼穗的分化,减少分蘖数,延长分蘖的时间,影响种子灌浆,从而降低水稻的产量和品质等[3]。碱胁迫对水稻的伤害主要表现为水稻幼苗植株矮小,生长迟缓,细胞质膜损伤,甚至导致幼苗的死亡。我国盐碱地面积分布广泛,而水稻属于盐碱敏感型作物,土地盐碱化严重制约着水稻的种植面积及产量,限制了我国的农业生产发展。随着世界人口不断增加,土地盐碱化日益加剧,粮食安全问题已成为亟待解决的问题。因此,了解植物耐盐碱机制,挖掘耐盐碱基因,培育耐盐碱植物新品种,对于开发利用这些盐碱地资源,保证我国农业持续高效发展和粮食安全,具有重大的战略意义和现实意义。
1 盐碱胁迫对植物伤害的生理分析
1.1 离子毒害
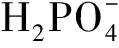
1.2 渗透胁迫
正常情况下,植物细胞渗透压高于土壤溶液。植物细胞利用这种高渗透压来吸收土壤溶液中的水分和必需的矿物质。在盐胁迫下,由于高浓度盐分的存在,降低了土壤的水势,植物细胞的渗透压低于土壤溶液的渗透压,使植物体内水分外渗导致植物缺水,从而降低了植物对水和钾、钙等矿物质的吸收能力[6],引发渗透胁迫。渗透胁迫的特点是对作物伤害迅速,但是持续时间短,植株的幼嫩组织或器官易受到危害。
1.3 氧化胁迫
细胞内严重的离子失衡将引起渗透胁迫,导致细胞严重失水,细胞内环境紊乱,而这种离子失衡又进而引发活性氧的产生,造成氧化胁迫。在盐碱条件下,离子毒害和活性氧(Reactive oxygen species,ROS)常常伴随发生,细胞内活性氧大量积累,植物体内会产生大量的氧自由基,从而导致膜脂被氧化伤害,DNA链断裂以及蛋白质受到损伤等,使得植物细胞丧失正常的生理功能。
1.4 高pH胁迫

研究表明,植物根系对周围高 pH 值环境的应答主要是有机酸的分泌。在碱胁迫条件下,棉花、沙棘及苜蓿植株茎和叶中的Na+累积明显高于盐胁迫。但若当胁迫强度超过根系的调节能力时,大量的Na+涌入到植物的茎叶之中,则会造成细胞内叶绿体片层结构被破坏,叶绿素含量降低,多种酶失活和气孔导度降低等。尽管对耐碱机制仍未有定论,但是毋庸置疑,碱胁迫对植物造成的伤害比盐胁迫更为严重和复杂,所以具有更重要的研究价值。
2 耐盐碱植物资源及其相关研究
长期处于不断变换的自然环境使植物进化出了复杂的适应系统,只有增强对环境尤其是逆境的适应能力才能维持其正常生存。因此,为了揭示植物的碱胁迫应答过程和耐碱机理,科学家们大多从天然的耐碱植物着手,以其为研究对象深入探究其生理适应机制。
尽管盐碱胁迫对植物的伤害巨大,但仍有部分植物能够在盐碱地中生存,是研究植物耐碱机制的理想材料,如星星草(Puccinelliatenuiflora)[7]、羊草(Leymuschinensis)[8-9]、刚毛怪柳(Tamarixhispida)[10-11],碱茅(Puccinelliadistans)[12]及野生大豆[13]等。星星草是生长在草原、盐渍化土壤碱斑周围的多年生禾本科草本植物,能够在pH值10以上、表土含盐量5%以上的土壤中生长良好,具有较强的耐盐、碱能力。这些植物资源的发现使科研工作者在挖掘耐盐碱关键基因,揭示植物耐盐碱作用机制,培育耐盐碱作物新品种及改良盐碱地等方面取得了较大进展。研究发现星星草不仅拥有较强的耐碱能力,对土壤碱性环境还存在着有效的调节机制,目前人工播种星星草成为当前治理盐碱化草地行之有效的手段[14]。
结合较为先进的技术手段,如芯片数据、转录组测序等,通过对这些物种的深入研究,已获得大量盐碱胁迫应答基因,并对耐盐碱植物的耐盐碱机理有了初步的认识。刘桂丰等通过芯片杂交的方法获得了碱胁迫(Na2CO3)下星星草的基因表达谱[15]。王玉成等通过差异显示技术对星星草在碱胁迫条件下的基因表达进行了分析,并获得7个在碱胁迫下差异表达的基因片段[16]。Kobayashi等通过对比耐碱品种星星草和一种对碱敏感的一年生早熟禾(Poaannua)在碱胁迫条件下的转录组,发现表达量变化的基因数目在耐碱品种星星草中远低于碱敏感品种早熟禾,推测在星星草中,调节碳酸氢钠耐性的基因组成型表达[17]。并且,与铁离子吸收相关的同源基因(Metal-nicotianamine transporter和NAAT-A)在星星草中上调表达而在盐碱敏感品种早熟禾中则下调表达,这种不同应答机制说明星星草在碱胁迫条件下能够更好地适应Fe缺乏症状。Ma等通过对不同程度碱胁迫条件下处理6个月的羊草的根,地下茎和地上部分的离子含量(K+、Na+和Ca2+)进行分析,揭示了盐碱胁迫条件下羊草在维持离子平衡上的作用机制,结果表明根中Na+含量较低,而对K+和Ca2+的选择吸收性较强,保证了较高的K+/Na+和Ca2+/Na+比,是羊草适应碱胁迫的重要机制[18]。
3 植物应答盐碱胁迫的分子机制
植物本身进化出多种盐碱胁迫应答的分子机制,主要包括以下几个方面:
3.1 离子平衡对植物应答盐碱胁迫的影响
盐碱胁迫会对植物产生离子毒害,植物根系在受到离子胁迫时会通过调节细胞内离子的进出来维持细胞内外或组织内外的离子稳态。
3.1.1 Na+-K+对植物应答盐碱胁迫的影响。受到盐碱胁迫的水稻,细胞内Na+浓度降低、K+浓度迅速升高,且随着盐碱溶液浓度的升高,水稻幼苗体内的Na+/K+比下降。植物体内存在过多的 Na+又导致了K+的缺乏,并且会抑制植物对其他离子(Ca2+、Mg2+)的吸收[19]。碱胁迫干扰 Na+-K+吸收和代谢是碱害的生理基础,植物如果能够有效地控制 Na+-K+的吸收和转运就能够在碱胁迫条件下生存,换言之维持Na+-K+平衡可能就是植物抗碱的最终体现。
在盐胁迫或碱胁迫下,根周围环境Na+浓度远远超过了其生长需求的范围,大部分植物在盐胁迫下都积累了大量Na+。研究表明,在碱胁迫下,高pH导致根外质子亏缺,抑制依赖质子梯度的Na+外排作用,使植物根系出现Na+积累的倾向[20]。这些积累的Na+都会被区隔到液泡中或排出体外,因此,植物Na+的区隔化和外排作用成为了Na+代谢的核心过程,也在植物抗盐碱过程中起决定性的作用。主要由两类蛋白完成这两个过程,既细胞膜上的盐超敏感蛋白1(SOS1)介导 Na+外排,液泡膜上的Na+/H+逆向转运蛋白(NHX)介导Na+区隔到液泡,这两个蛋白均受钙依赖的SOS2/SOS3激酶复合体调控。研究表明,盐胁迫下钙离子的增加被钙离子传感器SOS3感知,SOS3蛋白与SOS2蛋白激酶相互作用,SOS2-SOS3复合物激活一个质膜上Na+/H+的反向转运体SOS1蛋白,从而重新建立细胞内钠稳态。
3.1.2 铁对植物应答盐碱胁迫的影响。铁缺乏症是碱胁迫对植物的主要影响之一,因此在碱胁迫下,植物能获取更多的铁离子至关重要。植物通常采用两种策略进行铁离子的吸收和转运,即还原策略(策略I)和螯合策略(策略II)。大多数双子叶植物和单子叶非禾本科植物通常采用还原策略吸收铁离子,即通过酸化将质子释放到根际,增加Fe3+的溶解度,然后通过膜结合铁还原酶氧化酶将Fe3+还原成Fe2+,Fe2+通过转运蛋白从土壤中转运到根细胞中[21]。而单子叶禾本科植物通常采用螯合策略对铁离子进行吸收和转运,即根际分泌植物铁载体,例如能与Fe3+离子高亲和力结合的麦根酸,形成Fe3+-植物铁载体复合物,在缺铁情况下从根际周围获得铁[22-23]。

大麦是最耐缺铁症的禾本科植物,Murata等人在2006已确定一个植物铁离子转运子HvYS1[25],该基因与ZmYS1有72.7%相似度。HvYS1蛋白的细胞定位和底物特异性与ZmYS1[26]不同,说明HvYS1是特殊的转运子,在大麦的根中参与主要的离子吸收过程。水稻是对盐碱中度敏感的作物,在盐碱胁迫条件下,水稻品种东稻4相较于吉粳88表现出了较强的盐碱胁迫耐性,除了叶绿素含量较高,根系较发达等,东稻4还表现出了较强的根际酸化能力和较高的铁离子含量[27]。进一步研究发现OsIRO2、OsIRT1及OsNAS1等铁缺乏诱导基因在碱胁迫条件下的表达量显著高于吉粳88。这些结果表明,具有发达的根系和高效吸收铁离子能力显著增强东稻4的盐碱胁迫耐性。
3.1.3 钙对植物应答盐碱胁迫的影响。钙是植物生长发育所必需的营养素之一,它是细胞壁和细胞膜中各种结构的重要组成部分。钙除了在正常条件下起着重要的作用外,在不同的发育条件和盐胁迫条件下,钙作为植物的主要次级信使分子发挥着重要的作用。细胞内pH值的变化,与细胞内Ca2+浓度等变化相一致,引起植物广泛的细胞功能,包括植物防卫反应的信号转导。一般认为,盐胁迫和其他应激一样,在细胞膜上,即细胞外或细胞内被感知,然后触发细胞内信号级联,包括产生次级信使分子,如Ca2+和质子。Ca2+和质子信号的多样性和复杂性是由植物的胁迫性质和植物对指定胁迫的耐受能力决定的。在盐胁迫条件下,不同的耐盐或盐敏感植物中,细胞内Ca2+和质子在细胞水平和全株水平上都存在[28]。
3.2 渗透调节对植物应答盐碱胁迫的影响


3.3 抗氧化调节对植物应答盐碱胁迫的影响

研究发现,SOD活性与抗坏血酸-谷胱甘肽循环相结合,对缓解盐胁迫和碱胁迫诱导的番茄叶片氧化胁迫起着重要作用[21]。Guan等发现在酵母、拟南芥和水稻中超量表达水稻LDS1型锌指蛋白OsLOL5 显著提高了其耐盐碱胁迫的能力。在水稻中超量表达OsLOL5的水稻在碱胁迫下表现出较强的耐性,包括较高的地上部分和鲜重。氧化胁迫相关基因OsAPX2、OsCAT、OsCu/Zn-SOD以及OsRGRC2基因的表达量在转基因水稻中显著上调,表明OsLOL5可能通过调节活性氧清除途径提高了水稻对盐碱胁迫的耐性[29]。并且最近有研究发现,外源激素2,4-D能够通过调节水稻根的抗氧化能力和离子转运能力缓解盐胁迫的伤害[30]。
3.4 氮代谢对植物应答盐碱胁迫的影响

3.5 有机酸对植物应答盐碱胁迫的影响

研究表明,在碱胁迫下,虎尾草分泌有机酸不仅可以维持细胞内pH稳定和离子平衡,还在根外pH调节中起重要作用。虎尾草根系在碱化土壤环境中,通过分泌有机酸,还可能通过吸收Na+交换出H+及呼吸释放CO2等其他作用在根表面或皮层质外体空间进行pH调节,将碱胁迫所致的高pH屏蔽在根外,使根系结构与功能基本不受影响[37]。碱胁迫下水稻仅分泌少量的有机酸,无法有效地调节根周围微环境pH,碱胁迫的高pH就会迅速破坏根结构和功能,明显地抑制水稻根生长,甚至导致根细胞大批死亡解体。研究表明,碱胁迫下水稻中的有机酸以苹果酸、柠檬酸和草酸为主。碱胁迫下根系积累草酸用于离子平衡可能是水稻根系对缺氧环境的一种适应策略,水稻根系长期生长在水生环境,可能已经进化出以低耗氧为主的代谢途径[20]。
3.6 激素调节对植物应答盐碱胁迫的影响
脱落酸(ABA)、乙烯、生长素、茉莉酸及水杨酸等在协调植物适应逆境胁迫过程中具有重要的作用[38-41]。ABA诱导基因RD29A、RD29B及COR47等在植物响应干旱、冷和盐碱胁迫过程中发挥重要作用,其蛋白产物在植物细胞中的累积使植物更好地适应逆境环境,常用来作为盐碱或其他逆境胁迫下的Marker基因[38,42]。大豆乙烯应答转录因子GmERF4不仅受乙烯诱导,还受茉莉酸、水杨酸、盐及干旱等非生物胁迫诱导,在烟草中超量表达GmERF4提高了对盐和干旱胁迫的耐性[43]。研究表明,外源ABA提高水稻幼苗的盐胁迫耐性[44]。进一步研究表明,ABA能一定程度的降低碱胁迫所引起的幼苗质膜的损伤,并能提高水稻地上部分的相对含水量,减轻渗透胁迫对水稻幼苗的伤害,促进其生长;其次,ABA降低水稻体内Na+/K+比值,缓解由于碱胁迫下 Na+含量过高而引起的离子毒害,避免对植株新陈代谢造成干扰。高pH胁迫导致水稻根部损伤,是碱胁迫抑制水稻生长的主要原因,ABA对碱胁迫下水稻根长和根表面积的影响有缓解作用。同时,在非胁迫条件下,ABA处理对水稻株高存在抑制作用,其浓度越大抑制越明显,而在碱胁迫条件下,ABA对水稻株高存在促进作用,说明植物激素只有在一定浓度范围内才能发挥有效的作用[45]。
同样,研究发现生长素的过量累积是碱胁迫抑制根伸长的原因之一。通过对模式植物拟南芥在高pH条件下根生长受到抑制的作用机制分析发现,碱胁迫条件下植物根中的乙烯含量增加,进一步诱导生长素的产生,IAA合成的增多导致其在根尖中过度累积从而抑制根的伸长[46]。该研究指出,由AUX1介导的生长素的合成和累积参与了该碱胁迫应答过程,然而,生长素不能影响碱胁迫条件下乙烯激素的累积[46]。近期,有研究进一步证明了这个观点,通过对野生大豆基因GsERF71的耐碱功能分析发现GsERF71在碱胁迫条件下能够调节拟南芥根中生长素的累积,并且通过调节细胞质膜上的H+-ATPase提高了超表达拟南芥对碱胁迫的耐性[47]。
4 水稻铁转移与耐盐碱的研究
铁是植物体内新陈代谢过程所必需的离子元素,包括光合作用和呼吸作用。碱胁迫条件下植株通常表现出明显的由缺铁导致的失绿症状,主要因为虽然土壤中铁含量丰富,但由于碱性土壤的pH增高,铁大多以Fe3+形式存在,溶解度降低,不溶于水而无法为植物所用[23],从而限制了植物的光合作用,严重抑制了植物的生长和产量。
植物通常采用两种策略进行铁离子的吸收和转运。研究表明,根据铁离子的吸收形式不同,禾本科植物水稻主要采用策略II从土壤中获取铁离子,即在铁缺乏条件下,植物体内会大量的合成植物铁载体(Phytosideropres,PS),例如天然Fe3+螯合剂-麦根酸植物铁载体(MAs),从植物根系分泌到根际土壤中,与土壤中难溶的Fe3+直接结合形成螯合物后,被转运至细胞内,再释放出Fe3+,以供植物进行正常的生命代谢活动[48]。近年来,有研究表明水稻既可以运用策略II,通过分泌PS,螯合、吸收和转运Fe3+,也可以利用策略I中的Fe2+转运系统,直接从土壤中吸收Fe2+,由OsIRT1/2等铁转运蛋白将Fe2+转运到细胞内,再通过其它的转运蛋白将Fe2+运输到各个细胞器和组织中加以利用[22]。因此,研究人员通过研究相关基因和分子机制,旨在通过增加水稻对铁离子的吸收,改良水稻在盐碱土壤中生长的耐受性。
MAs是植物铁载体,能够螫合植物根际周围土壤中的Fe3+,在禾本科植物吸收转运铁的过程中发挥着重要的作用。NAS(尼克酰胺合成酶)、NAAT(尼克酰胺转氨酶)和DMAS(脱氧麦根酸合成酶)是在MAs合成过程中发挥重要作用的关键酶,决定着水稻根系MAs的合成量[49-51]。首先,SAM(S-腺苷-L-甲硫氨酸)在NAS作用下合成NA(尼克酰胺),在NAAT和DMAS催化下形成DMA(脱氧麦根酸),最后双加氧酶IDS2和IDS3将其转化为MAs(麦根酸)。通过输出转运蛋白OsTOM1、OsTOM2,将合成的MAs运输到根际周围螯合土壤中游离的Fe3+[52],形成Fe3+-MAs复合物。再由铁运输载体OsYSL15、OsYSL16把复合物从根际土壤转运到根表皮细胞中,之后,铁运输载体OsYSL15、OsYSL16和OsYSL18负责长距离运输,把根细胞内的Fe3+-MAs复合物从木质部和韧皮部运输到地上部[51,53-55]。
水稻吸收转运铁主要是通过MAs螯合Fe3+,在缺铁条件下,水稻根系也能产生和分泌数量较少的MAs,但与之相比,大麦根系所能分泌的MAs相对较多。因此,研究人员通过农杆菌介导的方法将大麦基因组中两个编码MAs合成关键酶的基因HvNAAT-A和HvNAAT-B转入了水稻中,以此来改良水稻的耐碱性。研究表明,在碱性土壤中,HvNAAT的异源表达使转基因水稻植株表现出较强的耐碱性,相对野生型植株而言,转基因水稻植株表现出较高的NAAT活性,根系分泌更多的DMA,且生长状态良好,没有出现老叶与旗叶失绿的症状,同时在pH8.5的碱性土壤中增产4.1倍[56]。与此相同,研究人员在水稻中异源表达大麦调控MAs合成的基因HvNAS1、HvIDS3,水稻转基因株系在苗期也表现出明显的耐碱性[57-58]。进一步研究发现,水稻调控MAs合成的基因OsNAS2的水稻过表达植株对铁缺乏的耐受性增强,并使其在碱性条件下生长良好,在一定程度下增强了水稻的耐碱性[59]。其他与MAs合成相关的基因过表达也被证明能提高植株对石灰性土壤的耐受性。因此,通过转基因方法提高水稻对低铁胁迫的耐受性是提高石灰性土壤条件下水稻农业生产力的有效途径。
IDEF1是禾本科植物缺铁响应基因中的中心转录调节子,位于其他缺铁因子反应级联的上游,铁缺乏的初期参与铁的吸收和利用。研究显示,OsIDEF1既可以通过直接与Fe2+等金属离子结合,在缺铁时发挥传感器的作用;又可以通过识别水稻中铁缺乏响应顺式元件的CATGC序列来调节水稻对铁缺乏的响应。进一步研究表明,OsIDEF1的表达水平与铁吸收转运的相关基因OsIRO2、OsYSL15、OsIRT1、OsYSL2、OsNAS1、OsNAS2、OsNAS3和OsDMAS1的诱导水平呈正相关关系。因此,研究人员得到的转基因IDEF1过表达水稻植株,不但对缺铁表现出较强的耐受性,而且同时增强了其耐碱性。转录因子IDEF2与铁缺乏响应顺式元件结合,参与铁缺乏基因的调控,在植物铁稳态中发挥着重要的作用。转录因子OsIDEF1和OsIDEF2均受缺铁诱导,OsIDEF1、OsIDEF2的表达增强导致石灰性土壤中植物铁载体分泌增加,对低铁有效性的耐受性增强[57]。
HRZs/BTS是禾本科植物和非禾本科植物中的E3泛素连接酶和铁缺乏的负调控因子。与OsIDEF1类似,水稻OsHRZ1和OsHRZ2可以直接结合铁离子和锌离子,是缺铁条件下,负调控铁的获得的铁结合传感器,也是铁积累有效的调节者,同时具有泛素化活性,参与蛋白的降解或修饰[60-65]。进一步研究发现,铁稳态正调节因子OsPRI1与OsHRZ1互作,且能被OsHRZ1泛素化,作为OsHRZ1-OsPRI1-OsIRO2/3信号转导级联的一部分,介导水稻对铁缺乏的响应,正调控OsIRO2和OsIRO3的表达[66]。OsIRO2、OsIRO3和 OsbHLH133[67]是碱性螺旋-环-螺旋( basic helix 1oop helix,bHLH)的转录因子,在水稻铁离子的吸收转运中发挥着重要的作用。其中,OsIRO2能正向调控铁离子的吸收和转运,而OsIRO3和OsbHLH133负向调控铁离子的吸收。研究表明,转录因子OsIRO2正向调控水稻根系MAs的分泌量,在石灰性土壤长期栽培条件下,水稻过表达OsIRO2植株不仅能增强OsNAS1、OsNAS2、OsNAS3、OsNAAT1、OsDMAS1等MAs合成相关基因的表达,使得水稻根系MAs的分泌增多-在缺铁条件下,过表达植株分泌的DMA是正常植株的1.7倍,水稻过表达OsIRO2植株还能增强Fe3+-DMA转运载体OsYSL1的表达,使得转基因株系对低铁利用的耐受性增强,且在石灰性土壤中积累的铁多于正常植株,并表现出一定程度的耐碱能力[68-71]。
水稻除了通过策略II以Fe3+-MAs螯合物形式吸收转运铁,也可以通过策略I直接转运游离态的Fe2+。水稻的生长发育时期大部分处于水中,根际周围的Fe2+含量比其他禾本科植物相对较多,为了更好地适应缺铁环境,水稻也进化出了相应的Fe2+吸收机制。研究表明,铁运输载体OsIRT1、OsIRT2、OsNRAMP1和OsNRAMP5均可以直接转运游离态的Fe2+。其中,OsIRT1在此过程中发挥了主要作用,是铁的一种功能性金属转运体,并积极参与水稻对铁的吸收,特别是在限制条件下[23,72-75]。将酵母Fe3+螯合还原基因refre1/372融合至铁调控转运体OsIRT1的启动子下游,导入水稻植株中,提高了水稻根系细胞铁还原酶的活性,从而增强了水稻在碱性土壤中对缺铁的耐受性[57]。
此外,以策略I植物矮牵牛为研究对象,通过引入大麦参与策略II途径的铁转运载体HvYSL1和PS,DMA,增强了矮牵牛在碱性条件下对缺铁的耐受性。研究表明,在高pH条件下,植物根际周围存在着难溶的Fe3+,MAs有效性提高,可以促进水稻对铁的吸收。外源性DMA的使用不仅在促进环境铁的吸收中起着重要的作用,还可以促进水稻的生长,并能协调铁和硝酸盐的同化。外源DMA的使用提高了水稻对铁的选择性吸收,促进植株体内铁的迅速转运,并上调硝酸还原酶的活性,还促进了水稻幼苗在高pH条件下的生长恢复,且SAM依赖的代谢途径部分参与了碱胁迫[76]。综上,通过增强水稻对铁离子的吸收和转运功能可以显著提高水稻对缺铁的耐受性,尤其是碱胁迫导致的缺铁耐受性,提高其在盐碱土壤中的生存能力。
5 总结与展望
盐碱逆境严重限制着世界范围内的农业生产与发展,因此,挖掘耐盐碱基因、了解植物耐盐碱的分子机制已经成为重要的挑战。目前,随着多种天然耐盐碱植物如星星草[7]、碱茅[12]及野生大豆[13]等的挖掘与利用,以及基因芯片、转录组测序及QTL定位等新技术方法有效结合,参与植物多方面如离子平衡调节、抗氧化、有机酸分泌及铁离子的吸收转运等生长代谢过程的耐盐碱新基因被发现并鉴定出来[57],为耐盐碱作物新品种的培育提供了多种重要的基因资源。然而在植物耐盐碱分子机制研究方面,相对于中性盐胁迫,关于碱性盐胁迫的信号传导机制研究的报道还比较少,尤其是碱胁迫的感知受体以及信号传递过程仍然未知,需要科研人员进一步深入的探索。
此外,我国是人口大国,盐碱地面积的不断扩增严重制约了我国农业的生产与发展,威胁到了我国粮食安全,但是目前对于我国重要粮食作物水稻的盐碱耐性改良方面的研究仍较少,多数研究集中于通过增加铁离子的吸收提高水稻的耐碱性。水稻育种专家主要通过传统的育种方式选育出耐盐碱的优良品种,然而这种培育方式周期较长且没有可预见性,其原因在于水稻耐盐碱品种的培育所能利用的种质资源和基因资源匮乏。因此,水稻耐盐碱基因的挖掘和利用尤为重要,仍需要结合现代测序,QTL定位等进行更深入的探索。此外,水稻耐盐碱的分子机制及其信号调控网络仍未被清楚的认识,且目前对耐盐碱分子机理的研究看法多样,植物在不同生育过程响应盐碱逆境的程度不同,需要重视并开展水稻盐碱胁迫不同阶段各类应答基因的研究,如上游传感器、转录调控因子、蛋白激酶及离子转运蛋白等。因此,进一步挖掘耐盐碱水稻品种,鉴定新的耐盐碱基因,通过探索多种途径提高水稻的耐盐碱能力将成为我们未来研究的重点方向。
参考文献(References):
[1] JIN H,KIM H R,PLAHA P,et al.Expression profiling of the genes induced by Na2CO3and NaCl stresses in leaves and roots ofLeymuschinensis[J].Plant Science,2008,175(6): 784-792.
[2] ZHANG L P,WANG X F,SHI Q H,et al.Differences of physiological responses of cucumber seedlings to NaCl and NaHCO3stress[J].Chinese Journal of Applied Ecology,2008.8,19(8): 1854-1859.
[3] 祁栋灵,郭桂珍,李明哲,等.水稻耐盐碱性生理和遗传研究进展[J].植物遗传资源学报,2007,8(4): 486-493.
QI D L,GUO G Z,LI M Z,et al.Progress of physiology and genetic research on saline-alkaline tolerance in rice[J].Journal of Plant Genetic Resources,2007,8(4): 486-493.
[4] HASEGAWA P M,BRESSAN R A,ZHU J K,et al.Plant cellular and molecular responses to high salinity[J].Annual Review of Plant Physiology and Plant Molecular Biology,2000,51: 463-499.
[5] MUNNS R,TESTER M.Mechanisms of salinity tolerance[J].Annual Review of Plant Biology,2008,59: 651-681.
[6] MUNNS R,JAMES R A,LAUCHLI A.Approaches to increasing the salt tolerance of wheat and other cereals[J].Journal of Experimental Botany,2006,57(5): 1025-1043.
[7] 张 恒.星星草(Puccinelliatenuiflora)叶绿体Na2CO3胁迫应答的生理学与定量蛋白质组学研究[D].哈尔滨: 东北林业大学,2012.
ZHANG H.Physiological and quantitative proteomics analysis ofPuccinelliatenuiflorachloroplast in response to Na2CO3[D].Harbin:Northeast Forestry University,2012.
[8] 孙 芳.羊草多糖的分析及其对羊草盐碱耐受性的影响[D].长春:东北师范大学,2010.
SUN F.Analysis of the polysaccharides fromLeymuschinensisand its effect on salinity and alkalinity tolerance[D].Changchun:Northeast Normal University,2010.
[9] JIN H,PLAHA P,PARK J Y,et al.Comparative EST profiles of leaf and root ofLeymuschinensis,a xerophilous grass adapted to high pH sodic soil[J].Plant Science,2006,170(6): 1081-1086.
[10] WANG C,GAO C Q,WANG L Q,et al.Comprehensive transcriptional profiling of NaHCO3-stressedTamarixhispidaroots reveals networks of responsive genes[J].Plant Molecular Biology,2014,84(1-2): 145-157.
[11] GAO C Q,WANG Y C,LIU G F,et al.Expression profiling of salinity-alkali stress responses by large-scale expressed sequence tag analysis inTamarixhispid[J].Plant Molecular Biology,2008,66(3): 245-258.
[12] 陈冠宜.盐地碱蓬和小花碱茅对NaCl和Na2CO3抗性的比较研究[D].济南: 山东师范大学,2011.
CHEN G Y.Comparative study on the resistance ofSuaedesalsaL.andPuccinelliatenuifloraunder different concentrations of NaCl and Na2CO3stress[D].Jinan: Shandong Normal University,2011.
[13] 葛 瑛,朱延明,吕德康,等.野生大豆碱胁迫反应的研究[J].草业科学,2009,26(2): 47-52.
GE Y,ZHU Y M,LÜ D K,et al.Research on responses of wild soybean to alkaline stress[J].Pratacultural Science,2009,26(2): 47- 52.
[14] 殷立娟,祝 玲.东北盐生草甸五种习见牧草苗期抗盐碱性的比较[J].四川草原,1988,9(2): 43-49.
YIN L J,ZHU L.Comparison of salt and alkalinity resistance of five species of forage grass in northeast salt meadow[J].Journal of Sichuan Grassland,1988,9(2): 43-49.
[15] LIU G F,CHU Y G,WANG Y C,et al.Study of expression profiles ofPuccinelliatenuifloragenes under NaHCO3stress by means of the cDNA microarray[J].Acta Botanica Boreali-Occidentalia Sinica,2005,25(5): 887-892.
[16] 王玉成,杨传平,刘桂丰,等.差异显示技术研究NaHCO3胁迫下星星草基因表达[J].植物学通报,2005,22(3): 307-312.
WANG Y C,YANG C P,LIU G F,et al.Studies of gene expression inPuccinelliatenuifloraunder NaHCO3stress using differential display technology[J].Chinese Bulletin of Botany,2005,22(3): 307-312.
[17] KOBAYASHI S,SATONE H,TAN E,et al.Transcriptional responses of a bicarbonate-tolerant monocot,Puccinelliatenuiflora,and a related bicarbonate-sensitive species,Poaannua,to NaHCO3stress[J].International Journal of Molecular Sciences,2014,16(1): 496-509.
[18] MA H Y,LIANG Z W,YANG H Y,et al.Ion adaptive mechanisms ofLeymuschinensisto saline-alkali stress[J].Journal of Food Agriculture and Environment,2011,9(3-4): 688-692.
[19] 张 唤,黄立华,李洋洋,等.东北苏打盐碱地种稻研究与实践[J].土壤与作物,2016,5(3): 191-197.
ZHANG H,HUANG L H,LI Y Y,et al.Research and experience of rice planting in saline-sodic soil in Northeast China[J].Soils and Crops,2016,5(3): 191-197.
[20] 杨春武.虎尾草和水稻抗碱机制研究[D].长春: 东北师范大学,2010.
YANG C W.Mechanisms of alkali tolerance inChlorisvirgateand rice(Oryzasativa)[D].Changchun:Northeast Normal University,2010.
[21] GONG B,WEN D,VANDENLANGENBERG K,et al.Comparative effects of NaCl and NaHCO3stress on photosynthetic parameters,nutrient metabolism,and the antioxidant system in tomato leaves[J].Scientia Horticulturae,2013,157: 1-12.
[22] ISHIMARU Y,SUZUKI M,TSUKAMOTO T,et al.Rice plants take up iron as an Fe3+-phytosiderophore and as Fe2+[J].The Plant Journal,2006,45(3): 335-346.
[23] GOMEZ-GALERA S,SUDHAKAR D,PELACHO A M,et al.Constitutive expression of a barley Fe phytosiderophore transporter increases alkaline soil tolerance and results in iron partitioning between vegetative and storage tissues under stress[J].Plant Physiology and Biochemistry,2012,53: 46-53.
[24] MARTINEZ-CUENCA M R,LEGAZ F,FORNER-GINER M,et al.Bicarbonate blocks iron translocation from cotyledons inducing iron stress responses inCitrusroots[J].Journal of Plant Physiology,2013,170(10): 899-905.
[25] ZHENG L Q,FUJII M,YAMAJI N,et al.Isolation and characterization of a barley yellow stripe-like gene,HvYSL5[J].Plant and Cell Physiology,2011,52(5): 765-774.
[26] CURIE C,PANAVIENE Z,LOULERGUE C,et al.Maize yellow stripe1 encodes a membrane protein directly involved in Fe(III) uptake[J].Nature,2001,409(6818): 346349.
[27] LI Q,YANG A,ZHANG W H.Efficient acquisition of iron confers greater tolerance to saline-alkaline stress in rice(OryzasativaL.)[J].Journal of Experimental Botany,2016,67(22): 6431-6444.
[28] KADER M A,LINDBERG S.Cytosolic calcium and pH signaling in plants under salinity stress[J].Plant Signalingand Behavior,2010,5(3): 233-238.
[29] GUAN Q J,MA H Y,WANG Z J,et al.A rice LSD1-like-type ZFP geneOsLOL5 enhances saline-alkaline tolerance in transgenicArabidopsisthaliana,yeast and rice[J].BMC Genomics,2016,17: 142.
[30] ISLAM F,FAROOQ M A,GILL R A,et al.2,4-D attenuates salinity-induced toxicity by mediating anatomical changes,antioxidant capacity and cation transporters in the roots of rice cultivars[J].Scientific Reports,2017,7(1): 10443.
[31] YANG C W,XU H H,WANG L L,et al.Comparative effects of salt-stress and alkali-stress on the growth,photosynthesis,solute accumulation,and ion balance of barley plants[J].Photosynthetica,2009,47(1): 79-86.
[32] WANG H,WU Z,CHEN Y,et al.Effects of salt and alkali stresses on growth and ion balance in rice(OryzasativaL.)[J].Plant Soil and Environment,2011,57(6): 286-294.
[33] WANG H,LIN X,CAO S,et al.Alkali tolerance in rice(OryzasativaL.):Growth,photosynthesis,nitrogen metabolism,and ion homeostasis[J].Photosynthetica,2015,53(1): 55-65.
[34] 王 欢.碱胁迫下水稻氮代谢调节机制[D].长春: 东北师范大学,2013.
WANG H.Regulatory mechanisms of nitrogen metabolism under alkali stress in Rice(OryzasativaL.)[D].Changchun: Northeast Normal University,2013.
[36] CHEN W,FENG C,GUO W,et al.Comparative effects of osmotic-,salt- and alkali stress on growth,photosynthesis,and osmotic adjustment of cotton plants[J].Photosynthetica,2011,49(3): 417-425.
[37] YANG C W,GUO W Q,SHI D C.Physiological roles of organic acids in alkali-tolerance of the alkali-tolerant halophyteChlorisvirgata[J].Agronomy Journal,2010,102(4): 1081-1089.
[38] MSANNE J,LIN J S,STONE J M,et al.Characterization of abiotic stress-responsiveArabidopsisthalianaRD29AandRD29Bgenes and evaluation of transgenes[J].Planta,2011,234(1): 97-107.
[39] MIZOI J,SHINOZAKI K,YAMAGUCHI-SHINOZAKI K.AP2/ERF family transcription factors in plant abiotic stress responses[J].Biochimica et Biophysica Acta(BBA)-Gene Regulatory Mechanisms,2012,1819(2): 86-96.
[40] MÜLLER M,MUNNÉ-BOSCH S.Ethylene response factors: A key regulatory hub in hormone and stress signaling[J].Plant Physiology,2015,169(1): 32-41.
[41] ZHU Q,ZHANG J T,GAO X S,et al.TheArabidopsisAP2/ERF transcription factorRAP2.6 participates in ABA,salt and osmotic stress responses[J].Gene,2010,457(1-2): 1-12.
[42] LIU A L,YU Y,DUAN X B,et al.GsSKP21,aGlycinesojaS-phase kinase-associated protein,mediates the regulation of plant alkaline tolerance and ABA sensitivity[J].Plant Molecular Biology,2015,87(1-2): 111-124.
[43] ZHANG G Y,CHEN M,CHEN X P,et al.Isolation and characterization of a novel EAR-motif-containing geneGmERF4 from soybean(GlycinemaxL.)[J].Molecular Biology Reports,2009,37(2):809.
[44] GURMANI A R,BANO A,ULLAH N,et al.Exogenous abscisic acid(ABA) and silicon(Si) promote salinity tolerance by reducing sodium(Na+) transport and bypass flow in rice(Oryzasativaindica)[J].Australian Journal of Crop Science,2013,7(9): 1219.
[45] 魏立兴.外源脱落酸对水稻(OryzasativaL.)盐碱胁迫的诱抗效应及其生理机制[D].北京: 中国科学院大学,2015.
WEI L X.Priming effect and the mechanism of abscisic acid on saline-alkaline stress tolerance in rice(OryzasativaL.)[D].Beijing: University of Chinese Academy of Sciences,2015.
[46] LI J,XU H H,LIU W C,et al.Ethyleneinhibits root elongation during alkaline stress through AUXIN1 and associated changes in auxin accumulation[J].Plant Physiology,2015,168(4): 1777-1791.
[47] YU Y,DUAN X B,DING X D,et al.A novel AP2/ERF family transcription factor fromGlycinesoja,GsERF71,is a DNA binding protein that positively regulates alkaline stress tolerance inArabidopsis[J].Plant Molecular Biology,2017,94(4-5): 509-530.
[48] ROBERTS L A,PIERSON A J,PANAVIENE Z,et al.Yellow stripe1.expanded roles for the maize iron-phytosiderophore transporter[J].Plant Physiology,2004,135(1): 112-120.
[49] CHENG L J,WANG F,SHOU H X,et al.Mutation in nicotianamine aminotransferase stimulated the Fe(II) acquisition system and led to iron accumulation in rice[J].Plant Physiology,2007,145(4): 1647-1657.
[50] INOUE H,TAKAHASHI M,KOBAYASHI T,et al.Identification and localisation of the rice nicotianamine aminotransferase geneOsNAAT1 expression suggests the site of phytosiderophore synthesis in rice[J].Plant Molecular Biology,2008,66(1-2): 193-203.
[51] KAKEI Y,ISHIMARU Y,KOBAYASHI T,et al.OsYSL16 plays a role in the allocation of iron[J].Plant Molecular Biology,2012,79(6): 583-594.
[52] NOZOYE T,NAGASAKA S,KOBAYASHI T,et al.Thephytosiderophore efflux transporter TOM2 is involved in metal transport in rice[J].Journal of Biological Chemistry,2015,290(46): 27688-27699.
[53] KOIKE S,INOUE H,MIZUNO D,et al.OsYSL2 is a rice metal-nicotianamine transporter that is regulated by iron and expressed in the phloem[J].The Plant Journal,2004,39(3): 415-424.
[54] LEE S,CHIECKO J C,KIM S A,et al.Disruption ofOsYSL15 leads to iron inefficiency in rice plants[J].Plant Physiology,2009,150(2): 786-800.
[55] AOYAMA T,KOBAYASHI T,TAKAHASHI M,et al.OsYSL18 is a rice iron(III)-deoxymugineic acid transporter specifically expressed in reproductive organs and phloem of lamina joints[J].Plant Molecular Biology,2009,70(6): 681-692.
[56] TAKAHASHI M,NAKANISHI H,KAWASAKI S,et al.Enhanced tolerance of rice to low iron availability in alkaline soils using barley nicotianamine aminotransferase genes[J].Nature Biotechnology,2001,19(5): 466-469.
[57] NISHIZAWA N.Enhancing tolerance of rice to alkaline soils using genes involved in Fe acquisition in plants[J].Journal of Japanese Society for Extremophiles,2008,7(2): 15-19.
[58] SUZUKI M,MORIKAWA C K,NAKANISHI H,et al.Transgenic rice lines that include barley genes have increased tolerance to low iron availability in a calcareous paddy soil[J].Soil Scienceand Plant Nutrition,2008,54(1): 77-85.
[59] LEE S,KIM Y S,JEON U S,et al.Activation of ricenicotianaminesynthase2(OsNAS2) enhances iron availability for biofortification[J].Molecules and Cells,2012,33(3): 269-275.
[60] KOBAYASHI T,OGO Y,ITAI R N,et al.The transcription factor IDEF1 regulates the response to and tolerance of iron deficiency in plants[J].Proceedings of the National Academy of Sciences,2007,104(48): 19150-19155.
[61] KOBAYASHI T,NISHIZAWA N K.Iron sensors and signals in response to iron deficiency[J].Plant Science,2014,224: 36-43.
[62] OGO Y,KOBAYASHI T,ITAI R N,et al.A novel NAC transcription factor,IDEF2,that recognizes the iron deficiency-responsive element 2 regulates the genes involved in iron homeostasis in plants[J].The Journal of Biological Chemistry,2008,283(19): 13407-13417.
[63] KOBAYASHI T,OGO Y,AUNG M S,et al.The spatial expression and regulation of transcription factors IDEF1 and IDEF2[J].Annals of Botany,2010,105(7): 1109-1117.
[64] KOBAYASHI T,ITAI R N,OGO Y,et al.The rice transcription factor IDEF1 is essential for the early response to iron deficiency,and induces vegetative expression of late embryogenesis abundant genes[J].The Plant Journal,2009,60(6): 948-961.
[65] KOBAYASHI T,NAGASAKA S,SENOURA T,et al.Iron-binding haemerythrin RING ubiquitin ligases regulate plant iron responses and accumulation[J].Nature Communications,2013,4: 2792.
[66] ZHANG H M,LI Y,YAO X N,et al.POSITIVE REGULATOR OF IRON HOMEOSTASIS1,OsPRI1,facilitates iron homeostasis[J].Plant Physiology,2017,175(1):543-554.
[67] WANG L,YING Y H,NARSAI R,et al.Identification of OsbHLH133 as a regulator of iron distribution between roots and shoots inOryzasativa[J].Plant Cell and Environment,2013,36(1): 224-236.
[68] OGO Y,ITAI R N,NAKANISHI H,et al.Isolation and characterization of IRO2,a novel iron-regulated bHLH transcription factor in graminaceous plants[J].Journal of Experimental Botany,2006,57(11): 2867-2878.
[69] OGO Y,ITAI R N,NAKANISHI H,et al.The rice bHLH protein OsIRO2 is an essential regulator of the genes involved in Fe uptake under Fe-deficient conditions[J].The Plant Journal,2007,51(3): 366-377.
[70] ZHENG L Q,YING Y H,WANG L,et al.Identification of a novel iron regulated basic helix-loop-helix protein involved in Fe homeostasis inOryzasativa[J].BMC plant biology,2010,10: 166.
[71] OGO Y,ITAI R N,KOBAYASHI T,et al.OsIRO2 is responsible for iron utilization in rice and improves growth and yield in calcareous soil[J].Plant Molecular Biology,2011,75(6): 593-605.
[72] TAKAHASHI R,ISHIMARU Y,SENOURA T,et al.The OsNRAMP1 iron transporter is involved in Cd accumulation in rice[J].Journal of Experimental Botany,2011,62(14): 4843-4850.
[73] BUGHIO N,YAMAGUCHI H,NISHIZAWA N K,et al.Cloning an iron-regulated metal transporter from rice[J].Journal of Experimental Botany,2002,53(374): 1677-1682.
[74] ISHIMARU Y,TAKAHASHI R,BASHIR K,et al.Characterizing the role of rice NRAMP5 in manganese,iron and cadmium transport[J].Scientific Reports,2012,2: 286.
[75] SASAKI A,YAMAJI N,YOKOSHO K,et al.Nramp5 is a major transporter responsible for manganese and cadmium uptake in rice[J].The Plant Cell,2012,24(5): 2155-2167.
[76] ARAKI R,KOUSAKA K,NAMBA K,et al.2′-Deoxymugineic acid promotes growth of rice(OryzasativaL.) by orchestrating iron and nitrate uptake processes under high pH conditions[J].The Plant Journal,2015,81(2): 233-246.
