跨损伤合成途径中真核生物DNA聚合酶δ转换机制的研究进展
2018-06-13周亚竟
陈 炜,周亚竟
(江苏大学 生命科学研究院,镇江 212013)
每天细胞内仅因内源因素(如活性氧自由基、代谢产物等)就会引起高达数以万计的DNA损伤,每个损伤均可导致复制叉进程受阻,这些阻碍可以通过各种损伤修复途径得以克服,或通过损伤耐受机制激活跨损伤合成(Translesion synthesis, TLS)聚合酶,在损伤DNA加成物(adduct)对称新链的相应位置加上核苷酸,或通过模板交换而旁路绕过这些损伤。相较于RNA、蛋白质、代谢物等,DNA位于信息传递的顶层,是细胞内唯一无法被取代的大分子,因此,损伤的DNA完全依赖于修复。如果这些损伤未能被及时修复或被不正确的修复,将引起基因突变、错误插入、删除、或者染色体紊乱,最终导致人类多种疾病的发生。因此,准确的DNA复制和及时的损伤修复在真核生物维持其染色体稳定、防止遗传变异和人类多种疾病,特别是癌症的发生等方面起着极其重要的作用。
1 DNA聚合酶Pol δ与染色体DNA复制和损伤修复
生命的本质在于遗传信息从上代到下代的正常传递,生物体在进化过程中发展出一套精确机制以保证“忠实”的DNA复制。真核生物染色体DNA复制主要由3种B-家族复制性聚合酶(Replicative polymerases)来完成,即:聚合酶α、δ和ε(Pol α、Pol δ和 Pol ε),当双螺旋亲代DNA分子解链后,Pol α/primase分别在复制叉的前导链和滞后链上合成一小段低保真度的RNA/DNA引物而启动DNA的合成,复制叉上的分工实验表明,在前导链(Leading strand)上主要是由Pol ε来主导子代DNA的合成,而在滞后链(Lagging strand)上则是由Pol δ来完成[1]。但也有观点认为,Pol δ可能均参与了两条链上子代DNA的合成过程[2],但是,目前还不清楚体内(in vivo)这两种复制聚合酶在复制叉上的具体分工和明确的复制机制。
Pol δ作为一个同时拥有5′-3′聚合酶催化活性和3′-5′核酸外切酶活性(框架阅读校对功能,prof-reading)的高保真聚合酶,除了通过它的高度复制保真特性在染色体结构稳定和避免遗传变异方面扮演关键角色外,几乎所有的DNA修复过程均涉及Pol δ的参与,如:Pol δ通过链上置换(Strand displacement)机制协同Fen-1在错配修复途径(Mismatch repair)中起重要作用,此过程需要p68亚基的参与[3]。在跨损伤修复途径中,Pol δ和特定的TLS聚合酶通过一种聚合酶转换机制(Polymerases switching mechanism)来完成跨损伤合成。一般观点认为,X-家族聚合酶Pol β是负责碱基切除修复(Base excision repair, BER)最主要的修复聚合酶,但本实验室和其他同行的研究表明,Pol δ在BER途径中扮演着关键角色[4-5]。大量研究也表明,Pol δ通过DNA的重新合成和缺口填补在核苷酸切除修复(Nucleotide excision repair, NER)以及双链断裂修复的同源重组(Homologous recombination, HR)途径中也起着重要作用[6]。
2 真核生物DNA聚合酶Pol δ 的结构与功能
通过对人源、酵母(S.pombe和S.cerevisiae)Pol δ在遗传学和生物化学等领域的大量研究,目前对真核生物Pol δ的结构和功能已有了较充分的了解。类似于S.pombe的Pol δ(各亚基分别为:Pol3、Cdc1、Cdc27、Cdm1),人源Pol δ也是由4个亚基组成的异源四聚体(各亚基分别为:p125、p50、p68、p12),而在酿酒酵母S.cerevisiae中尚未发现对应的第4亚基,由前3种亚基(Pol3p、Pol31p、Pol32p)构成异源三聚体。根据2001年发表的真核生物Pol δ亚基统一命名法,真核生物Pol δ的催化亚基被命名为A-亚基,其它3个辅助亚基分别为B-、C-、D-亚基[7]。
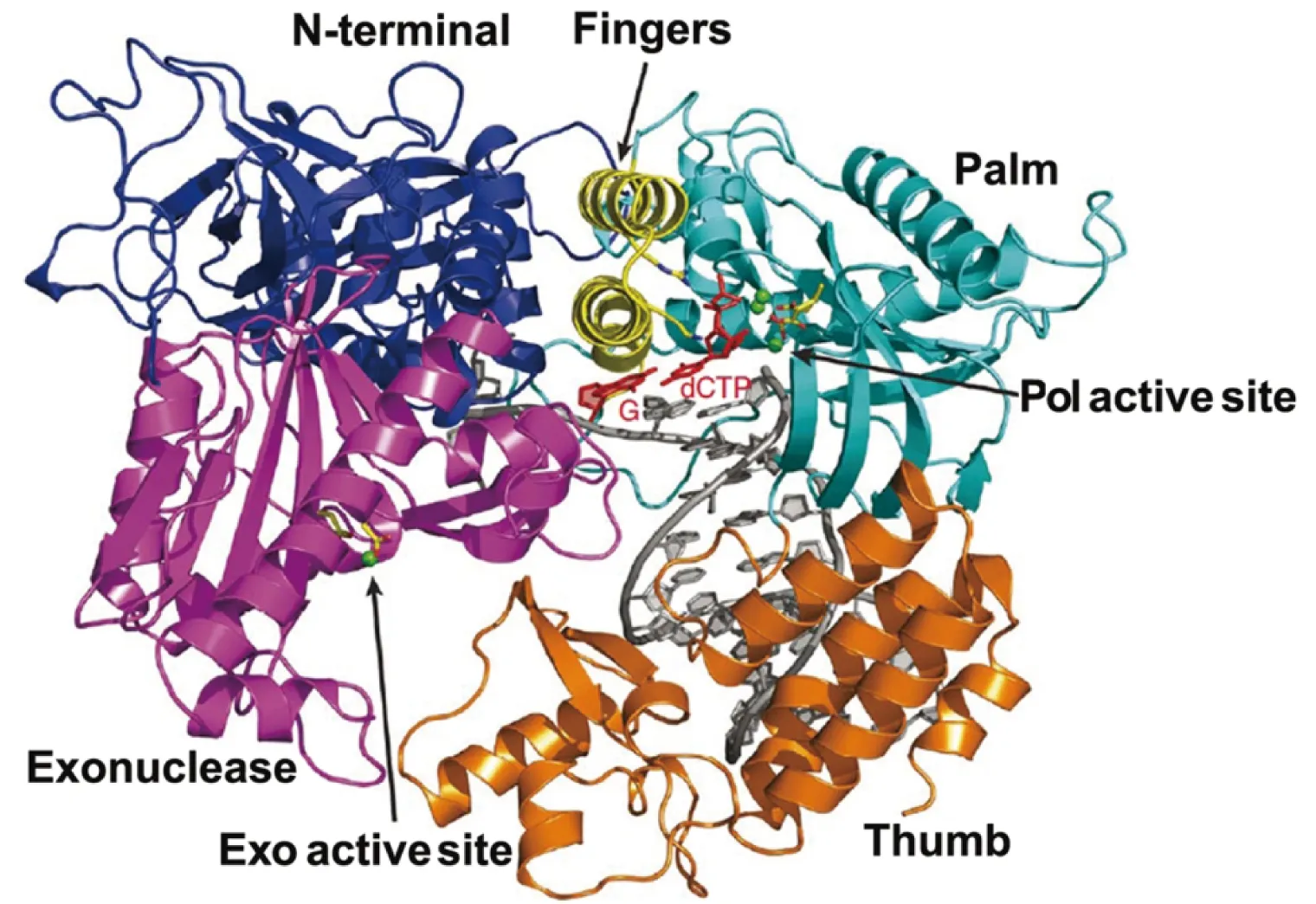
图1 酵母S.cerevisiae Pol3p-DNA-dCTP三元复合物晶体结构卡通示意图[8]Fig 1 Crystal structure of the Pol3p-DNA-dCTP ternary complex of S.cerevisiae
酵母S.cerevisiaeA-亚基催化核心域的“手掌”“手指”“拇指”、核酸外切酶、N-端域分别以青、黄、橙、洋红、深蓝颜色表示,DNA以灰色表示;模板上G和即将加入的dCTP以红色表示,钙离子以绿色表示,催化(Pol)和核酸外切(Exo)活性中心如箭头所指
目前还没有人源A-亚基p125晶体三维结构分析信息的报道,但通过对S.cerevisiaePol δ的A-亚基(Pol3p)催化核心域(catalytic core)与模板/引物及嵌入dCTP核苷酸所形成的三元复制复合物晶体进行的三维结构分析如图1所示[8],其催化核心域具有如所有已知的DNA聚合酶所特有的右手型构象,在C-端域(C-terminal domain, CTD)的非结构化“拴绳”(an unstructured tether)末端含有一个富含半胱氨酸域和一个亚域,这个亚域对与B-亚基的互作反应至关重要[9],也就是说,B-亚基与A-亚基CTD的亚域发生捆绑反应;而富含半胱氨酸域构成两个锌指基序ZnF1和ZnF2,其ZnF1对A-亚基与增殖细胞核抗原PCNA的捆绑及介导Pol δ的持续合成至关重要。
由人源B-亚基p50与C-亚基p68 N-末端残基形成的复合物晶体三维结构分析显示[10],p50亚基显示有较大的、约12 868 Å2磷酸二酯酶状(Phosphodiesterase-like, PDE)的表面结构域,且含有许多突出部和柔性回路,这使得p50亚基特别适合作为对接平台连接其它亚基和众多调控蛋白;又由于构成p50亚基PDE结构域的双β-折叠(β-sheet)之一通过平行的β-股(β-strands)与p68的β-折叠相连,形成扩展了的β-折叠,因此,p68 N-端可以看作是p50亚基的延伸。B-亚基p50作为一个理想的“脚手架”(Scaffold),除了链接其它3个亚基外,还可以通过延伸的C-亚基介导Pol δ在复制调控、跨损伤修复、中断复制(Break-induced replication, BIR)等过程中的功能[11]。
对人源p12亚基而言,它同时与p125的CTD域和p50亚基捆绑而形成稳定的三角结构,p12起到稳定Pol δ空间结构的功能[12]。因此,真核生物Pol δ复合体的结构被确定为:A-、B-、D-亚基相互“捆绑”形成稳定的三角结构,而C-亚基只能和B-亚基发生“捆绑”反应,形成B-亚基的延伸。
Pol δ作为染色体DNA复制功能的研究总是和PCNA相关联,此时,3个PCNA分子形成三聚体的环状滑行夹协同Pol δ的复制功能,同时,PCNA作为对接平台功能,通过募集-离散相关蛋白(复合体),在各种DNA的损伤修复过程也发挥着至关重要的作用。所有的Pol δ 4个亚基均被报道含PCNA捆绑反应域与PCNA发生捆绑反应,其中以p68与PCNA反应最强,而p50与PCNA的反应最弱[13]。在我们曾经提出过的(如图2)Pol δ-PCNA复制复合体分子反应模型中,p125、p68和p12 3个亚基分别与三聚体PCNA中一个分子起反应,当缺失了p12亚基时,p50亚基有可能成为与PCNA反应的备选分子。但由于p50与PCNA的亲和能力太过微弱以及Pol δ复合体空间结构的改变,p50亚基也可能无法与PCNA反应,使得三聚体中空出一个PCNA分子。在某些场合,如DNA的损伤应答,空出的这个PCNA分子是否可能作为对接平台的功能,通过募集其它修复蛋白,如Y-家族聚合酶等,参与到DNA的损伤修复过程等,均有待进一步深入研究。

图2 人源Pol δ-PCNA复制复合物的蛋白-蛋白互作反应模型Fig 2 A model of the network of protein-protein interactions for human Pol δ-PCNA complexes
A:由四亚基与PCNA构成的复制复合物Pol δ4-PCNA。Pol δ内部各亚基之间以及PCNA三聚体之间的反应以实线表示,而各亚基与PCNA之间的反应以三实线表示。B:由缺失了p12亚基的三亚基与PCNA构成的复制复合物Pol δ3-PCNA。p50可能提供一个与PCNA反应的备选分子,但也可能由于亲和能力太过微弱而无法与PCNA发生互作反应,使得三聚体中空出一个PCNA分子以备他用。本图源自文献[13]并作相应修改
3 跨损伤修复途径中Pol δ 与TLS聚合酶转换机制的激活
尽管复制性DNA聚合酶Pol δ具有紧凑缜密的催化活性中心和3′-5′核酸外切酶活性的框架阅读功能以保证DNA复制的高度持续合成能力和高保真性,DNA还是易受到各种内源或外源因素导致的损伤,据估计每天仅因内源因素就会产生数以万计的损伤[14]。为维护染色体DNA结构的完整性和遗传稳定性,细胞被赋予一整套如前面所提及的各种复杂的修复途径,以便在复制启动前消除这些损伤,但是,还是有相当数量的损伤会逃逸这些精确设计的修复途径而存在于基因组中,使得细胞S-期复制叉进程“熄火”、染色体异常甚至细胞死亡,因此,细胞进化出一种“旁路通过”(by-pass)受损DNA的损伤耐受机制(或称为复制后修复机制,DNA damage tolerance, DDT)来恢复DNA的正常复制,避免这些逃逸了的损伤所致的影响。一种损伤耐受机制涉及模板交换的无错(error-free)途径,即:利用无损的姐妹链,而不是损伤链作模板继续DNA的复制[15];另一种被广泛深入研究的是有错的(error-prone)TLS修复途径,TLS聚合酶在模板的损伤点对面加入核苷酸以延伸引物,但不修复这些损伤,即所谓的“旁路通过”,这虽然增加了复制错误的频率,却能够使S-和G2-期的复制叉进程得以恢复[16]。
哺乳动物介导跨损伤合成的主要聚合酶包括Y-家族的4个成员(REV1、Pol η、Pol ι 和Pol κ)以及B-家族成员之一的Pol ζ,有别于复制聚合酶,这些TLS聚合酶的催化活性中心比较松弛,能够容纳畸变了的损伤碱基,且持续合成能力低,不含3′-5′核酸外切酶活性的框架阅读能力[17]。当复制进程遇到损伤点(域)时,复制暂停,TLS聚合酶被募集并取代复制体(replisome)中Pol δ,在完成跨损伤合成后离开,Pol δ重新被募集回到引物末端继续新的合成,这就是有错TLS修复途径的一个关键步骤——聚合酶转换机制,但对在损伤点(域)聚合酶如何进行转换的机制存在争议。有观点认为,在Pol δ和Pol ζ介导的跨损伤修复中是通过催化亚基共享辅助亚基在损伤点(域)进行了转换[18],但这种模型无法解释其它TLS聚合酶在损伤点(域)与Pol δ是如何进行交换的。
通过对PCNA泛素化修饰及修饰后调控真核细胞应对DNA损伤的深入研究,另外的观点认为,是PCNA的泛素化-去泛素化修饰介导了TLS修复途径的聚合酶转换。酵母中,PCNA在K164残基位点被Rad6/Rad18缀合酶-连接酶(E2/E3)泛素反应调控系统单泛素化修饰(这里,以PCNA-Ub1表示被单泛素化修饰了的PCNA),在Pol η 和Pol ζ介导的跨损伤修复途径中扮演重要角色[19],PCNA-Ub1特异性地与Pol η发生亲和结合,但未经修饰的PCNA则不能[20],PCNA-Ub1能够特异性激活Pol η和REV1,促进跨损伤合成[21]。所有的TLS聚合酶C-末端存在高度进化保守的泛素结合域UBM(Ubiquitin-binding motif)或泛素结合锌指UBZ(Ubiquitin-binding zinc finger),TLS聚合酶通过UBM和UBZ与单泛素化了的PCNA-Ub1亲和结合被募集到损伤区的复制点,在没有DNA损伤情况下,PCNA的单泛素化修饰被去泛素化酶USP1所抑制,复制性聚合酶Pol δ和TLS聚合酶之间的转换被阻止,一旦发生DNA损伤,TLS途径被激活,PCNA的单泛素化修饰调控TLS聚合酶在损伤点(域)的募集[22]。
4 跨损伤修复途径中可能存在未知的聚合酶转换激活机制
尽管“Pol δ-TLS聚合酶之间进行转换是跨损伤修复途径的核心”已成目前为被广泛接受的观点,但对“PCNA单泛素化-去泛素化修饰介导了聚合酶的交换”存在争议。相反的研究表明,组成分子环状滑行夹的3个PCNA分子能够同时被单泛素化修饰,值得注意的是,单泛素化了的PCNA-Ub1并不影响复制聚合酶Pol δ的活性,也未破坏Pol δ-PCNA的捆绑结构,对TLS聚合酶Pol η的活性无增强作用;无论是未经修饰的PCNA还是泛素化了的PCNA-Ub1,它们对TLS聚合酶Pol ζ 和REV1的活性均并无任何刺激作用,因此,这些研究指出,在损伤点(域),Pol δ并不是通过PCNA被TLS聚合酶取代,PCNA的单泛素化修饰并不是构成聚合酶转换的核心,而可能是通过其它的某些未知功能而影响着TLS进程,推测可能是某个Pol δ相关的未知因子介导了TLS聚合酶在损伤点的募集[22-24]。
人源和酵母PCNA-Ub1晶体三维结构的X-衍射分析均显示PCNA和泛素的结构与它们结合前的游离形式几乎没有太大区别。人源PCNA结构卡通模型如图3所示,被Rad6/Rad18介导单泛素修饰的赖氨酸残基K164位于PCNA环状滑行夹的背面,而DNA合成则是在滑行夹的正面发生,即沿着引物的5′→3′前进方向进行。
所有的真核Y-家族聚合酶均含有非规范的(non-canonical)PIP盒(REV1除外,它通过自身BRCT域与PCNA发生反应),TLS聚合酶通过它们的PIP盒与PCNA发生亲和反应。TLS聚合酶PIP盒肽段与PCNA捆绑的晶体结构分析表明,Pol η的PIP盒与PCNA的亲和能力要比其UBZ域与PCNA-Ub1上泛素分子的亲和能力要强得多[22,26]。因此,在遇到损伤时,TLS聚合酶不太可能通过亲和能力较弱的UBM或UBZ域与环状滑行夹背面的泛素发生亲和而被募集到PCNA环状滑行夹正面朝向的损伤点(域)与Pol δ发生交换。这也从另一个侧面证明了由PCNA的泛素化修饰激活聚合酶转换机制的可能性较低。
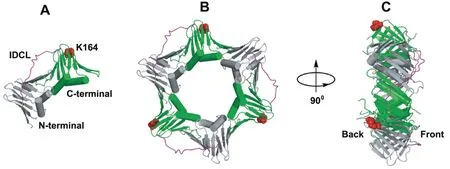
图3 人源PCNA三维结构的卡通模型Fig 3 Crystal structure of human PCNA
A:单分子PCNA由N-端(灰色)和C-端(绿色)的两个独立域组成,两个独立域之间通过域间连接环(interdomain connecting loop,IDCL)相连(洋红),164位的赖氨酸残基(K164)以红色球状表示。B和C:分别表示三聚体PCNA的正视图和侧视图,3个PCNA单分子头尾相连,构成显著不同的两个面,由C-端凸出部构成的一面被称为正面(Front side),或称C-端面(C-terminal face)。侧视图显示这两个面的显著不同,右向的正面(C-端面)含IDCL,在正常DNA合成过程负责与复制性聚合酶的反应,而发生单泛素化修饰的K164位于左向的背面。本图源自文献[25]并作相应修改
5 展望
前期的研究表明,在人源细胞几乎所有的、由各种因子诱导产生的DNA损伤中,Pol δ均以缺失了p12亚基的三聚体形式Pol δ3以应对,且鉴定出两种能够介导p12经多聚泛素化-蛋白酶体途径降解的E3连接酶RNF8和CRL4Cdt2[27-29]。我们的最新研究还发现,RNF8还能够介导Pol δ的p50亚基被单泛素化修饰(未发表的数据)。然而,细胞内的p50亚基通过什么E2/E3泛素化修饰调控系统被单泛素化修饰?修饰后的p50在缺失了p12亚基的Pol δ三聚体中扮演什么角色?与TLS聚合酶的互作关系?在跨损伤合成途径中如何调控Pol δ与TLS聚合酶之间的转换?这些问题将成为我们今后需要进行研究的重点。
癌症是一种由遗传和表观遗传改变、以细胞异常增殖及转移为特点的一大类疾病,DNA修复功能缺陷是许多自发性肿瘤和遗传性癌症的主要特征之一;与早(衰)老症相对应的转基因鼠模型研究还表明,未修复的DNA损伤积累也与早(衰)老症及老年性疾病密切相关,最终阐明跨损伤合成途径聚合酶Pol δ与TLS聚合酶交换机制,将为今后以Pol δ作为一种小分子抑制剂潜在的新靶标、设计新的肿瘤诊断方法和治疗手段来抗击癌症、防治衰老等相关疾病提供理论依据。
[1]BURGERS P M.Polymerase dynamics at the eukaryotic DNA replication fork [J].Journal of Biological Chemistry, 2009, 284(7): 4041-4045.
[2]PAVLOV Y I,SHCHERBAKOVA P V.DNA polymerases at the eukaryotic fork-20 years later [J].Mutation Research/Fundamental & Molecular Mechanisms of Mutagenesis, 2009, 685(1-2): 45-53.
[3]KADYROV F A, GENSCHEL J, FANG Y, et al.A possible mechanism for exonuclease 1-independent eukaryotic mismatch repair [J].Proc Natl AcadSci U S A, 2009, 106(21): 8495-8500.
[4]PARSONS J L, PRESTON B D, O’CONNOR T R, et al.DNA polymerase delta-dependent repair of DNA single strand breaks containing 3′-end proximal lesions [J].Nucleic Acids Research, 2007, 35(4): 1054-1063.
[5]ZHOU Y, CHEN H, LI X, et al.Production of recombinant human DNA polymerase delta in aBombyxmoribioreactor [J].PLoS One, 2011, 6(7): e22224.
[6]PRINDLE M J, LOEB L A.DNA polymerase delta in dna replication and genome maintenance [J].Environmental and Molecular Mutagenesis, 2012, 53(9): 666-682.
[7]MACNEILL S A, BALDACCI G, BURGERS P M, et al.A unified nomenclature for the subunits of eukaryotic DNA polymerase δ [J].Trends in Biochemical Sciences, 2001, 26(1): 16-17.
[8]SWAN M K, JOHNSON R E, PRAKASH L, et al.Structural basis of high-fidelity DNA synthesis by yeast DNA polymerase delta [J].Nature Structural & Molecular Biology, 2009, 16(9): 979-986.
[9]BROCAS C, CHARBONNIER J B, DHÉRIN C, et al.Stable interactions between DNA polymerase δ catalytic and structural subunits are essential for efficient DNA repair [J].DNA Repair, 2010, 9(10): 1098-1111.
[10]BARANOVSKIY A G, BABAYEVA N D, LISTON V G, et al.X-ray structure of the complex of regulatory subunits of human DNA polymerase delta [J].Cell Cycle, 2008, 7(19): 3026-3036.
[11]LYDEARD J R, JAIN S, YAMAGUCHI M, et al.Break-induced replication and telomerase-independent telomere maintenance require Pol32 [J].Nature, 2007, 448(7155): 820-823.
[12]LI H, XIE B, ZHOU Y,et al.Functional roles of p12, the fourth subunit of human DNA polymerase δ [J].Journal of Biological Chemistry, 2006, 281(21): 14748-14755.
[13]WANG Y,ZHANG Q,CHEN H, et al.P50, the small subunit of DNA polymerase delta, is required for mediation of the interaction of polymerase delta subassemblies with PCNA [J].PloS One, 2011, 6(11): e27092.
[14]DERKS K W J,HOEIJMAKERS J H J, POTHOF J.The DNA damage response: the omics era and its impact [J].DNA Repair, 2014, 19: 214-220.
[15]CHANG D J, CIMPRICH K A.DNA damage tolerance: when it′s ok to make mistakes [J].Nature Chemical Biology, 2009, 5(2): 82-90.
[16]WATERS L S, MINESINGER B K, WILTROUT M E, et al.Eukaryotic translesion polymerases and their roles and regulation in DNA damage tolerance [J].Microbiology & Molecular Biology Reviews, 2009, 73(1): 134-154.
[17]GUO C, KOSAREKSTANCEL J N, TANG T S, et al.Y-family DNA polymerases in mammalian cells [J].Cellular & Molecular Life Sciences, 2009, 66(14): 2363-2381.
[18]BARANOVSKIY A G, LADA A G, SIEBLER H, et al.DNA polymerases delta and zeta switching by sharing the accessory subunits of DNA polymerase delta [J].Journal of Biological Chemistry, 2012, 287(21):17281-17287.
[19]HOEGE C, PFANDER B, MOLDOVAN G L, et al.RAD6-dependent DNA repair is linked to modification of PCNA by ubiquitin and SUMO [J].Nature, 2002, 419(6903): 135-141.
[20]KANNOUCHE P L, WING J, LEHMANN A R.Interaction of human DNA polymerase eta with monoubiquitinated PCNA: a possible mechanism for the polymerase switch in response to DNA damage [J].Molecular Cell, 2004, 14(4): 491-500.
[21]LEHMANN A R, NIIMI A, OGI T, et al.Translesion synthesis: Y-family polymerases and the polymerase switch [J].DNA Repair, 2007, 6(7): 891-899.
[22]CHUN A C, JIN D Y.Ubiquitin-dependent regulation of translesion polymerases [J].Biochemical Society Transactions, 2010, 38(Pt 1): 110-115.
[23]ACHARYA N, YOON J H, GALI H, et al.Roles of PCNA-binding and ubiquitin-binding domains in human DNA polymerase η in translesion DNA synthesis [J].Proceedings of the National Academy of Sciences of the United States of America,2008, 105(46): 17724-17729.
[24]HARACSKA L, UNK I, PRAKASH L, et al.Ubiquitylation of yeast proliferating cell nuclear antigen and its implications for translesion DNA synthesis [J].Proceedings of the National Academy of Sciences,2006, 103(17): 6477-6482.
[25]HEDGLIN M, BENKOVIC S J.Regulation of Rad6/Rad18 activity during DNA damage tolerance [J].Biophysics, 2015, 44: 207-228.
[26]HISHIKI A, HASHIMOTO H, HANAFUSA T, et al.Structural basis for novel interactions between human translesion synthesis polymerases and proliferating cell nuclear antigen [J].Journal of Biological Chemistry, 2009, 284(16): 10552-10560.
[27]ZHANG S, ZHOU Y, SARKESHIK A, et al.Identification of RNF8 as a ubiquitin ligase involved in targeting the p12 subunit of DNA polymerase δ for degradation in response to DNA damage [J].Journal of Biological Chemistry, 2013, 288(5): 2941-2950.
[28]ZHANG S, ZHAO H, DARZYNKIEWICZ Z, et al.A novel function of CRL4(Cdt2): regulation of the subunit structure of DNA polymerase δ in response to DNA damage and during the S phase [J].Journal of Biological Chemistry, 2013, 288(41): 29550-29561.
[29]ZHANG Q, ZHANG Q, CHEN H, et al.Multiple forms of human DNA polymerase delta sub-assembling in cellular DNA transactions [J].Current Protein & Peptide Science, 2016, 17(8):746-755.
