改良碱裂解法和煮沸法提取金黄色葡萄球菌DNA效果的比较
2018-06-08邹治情王俊玲陈思谢雨DinshKumar吴亮姜旭淦阴晴陈盛霞许化溪
邹治情, 王俊玲, 陈思, 谢雨, Dinsh Kumar K,吴亮,*, 姜旭淦, 阴晴, 陈盛霞, 许化溪
(1. 江苏大学医学院, 江苏 镇江 212013; 2. 江苏大学附属医院检验科, 江苏 镇江 212001)
金黄色葡萄球菌是临床常见的革兰阳性菌,可产生多种毒力因子,引起感染者食物中毒、假膜性肠炎、烫伤样皮肤综合征、毒素休克综合征、化脓性炎症和败血症等,给患者造成极大损害[1]。近年来随着抗生素的广泛应用,金黄色葡萄球菌耐药现象日益严重,临床耐甲氧西林的金黄色葡萄球菌(methicillin-resistantStaphylococcusaureus, MRSA)分离率逐年提升。目前金黄色葡萄球菌的分离鉴定和耐药性分析需要5~7 d,方法烦琐且耗时耗力,不能满足临床诊断需求。PCR法等分子生物学技术是目前公认最具发展前景的细菌鉴定和耐药性分析技术,具有高灵敏度、高特异性及操作简便迅速等优点[2]。但PCR法检测结果的灵敏度和可信度依赖模板DNA质量,因此获得高质量的金黄色葡萄球菌基因组DNA对于开展PCR检测工作十分重要[3]。
金黄色葡萄球菌具有厚重的细胞壁,煮沸、冻融和溶菌酶裂解等常规方法难以有效破坏其细胞壁[4]。此外大部分临床菌株还能分泌耐热核酸酶,经煮沸后耐热核酸酶仍具有降解细菌核酸的能力[5]。因此金黄色葡萄球菌基因组DNA提取难度较大,使用常规方法提取细菌基因组DNA产率极低,无法用于PCR扩增。本研究中我们参考植物细胞基因组DNA碱裂解提取法并加以改进,建立了一种简单、经济、有效的提取方法,现报告如下。
1 材料和方法
1.1 菌种
10株金黄色葡萄球菌株由江苏大学附属医院检验科微生物室鉴定分离,均经VITEKⅡCompact全自动细菌鉴定仪(法国生物梅里埃公司)鉴定为MRSA菌。所有细菌均先涂布于血琼脂培养基活化,再挑取单个菌落接种于水解酪蛋白培养基(Mueller-Hinton, MH),经37℃连续振摇培养16 h后获得新鲜菌液。细菌菌落和新鲜菌液用于后续细菌基因组DNA提取。
1.2 主要试剂
血琼脂培养平板和MH培养基(法国生物梅里埃公司);酵母浸出液和胰蛋白胨(英国OXIOD公司);溶菌酶(美国Amersco公司);20 mg/mL蛋白酶K溶液(美国Merck公司)。2×PCR预混液(Dye Plus)购自南京Vazyme公司;琼脂糖为西班牙进口分装;TanonTMDNA 标准参照物购自上海天能公司。改良碱裂解法提取试剂包括3种:溶液1中含有25 mol/L Tris·HCl,10 mmol/L EDTA-Na2和50 mmol/L葡萄糖;溶液2中含有0.2 mol/L NaOH和10%(w/v)SDS,该液现配现用;溶液3中含有醋酸钾150 mg,冰醋酸11.5 mL,加灭菌双蒸水至100 mL,室温下保存备用。其他试剂均为国产分析纯。
1.3 引物
根据李克诚等[6]报道合成spa基因扩增引物,spa-F: 5′-TAAAGACGATCCTTCGGTGAGC-3′,spa-R:5′-CAGCAGTAGTGCCGTTTGCTT-3′,预测产物长度为n×170 bp。根据黄辉等[7]报道合成mecA基因和femB基因扩增引物,mecA-F:5′-GGCATTCGTGTCACAATCG-3′,mecA-R:5′-CTGGAACTTGTTGAGGAGAG-3′, 预测产物长度为310 bp;femB-F:5′-TTACAGAGTTAACTGTTACC-3′,femB-R: 5′-ATACAAATCCAGCACGCTCT-3′,预测产物长度为651 bp。上述引物由苏州涨迅生物公司合成。
1.4 细菌基因组DNA提取方法
1.4.1 直接煮沸法 在1.5 mL灭菌Ep管中加入200 μL灭菌双蒸水,从血琼脂平板上挑取1个菌落与灭菌双蒸水充分混合。将上述Ep管煮沸10 min后,经14 000 r/min离心10 min,吸取上清液作为PCR反应模板。
1.4.2 改良碱裂解法 按“1.1”方法扩增细菌,吸取200 μL新鲜菌液经离心收集菌体沉淀,在上述菌体沉淀中加入180 μL溶液1并充分重悬,再加入20 μL溶菌酶溶液(20 mg/mL),37℃孵育6 h消化细菌的细胞壁。在上述溶液中再加入20 μL蛋白酶K溶液(20 mg/mL),60 ℃水浴1 h。加入新鲜配制的300 μL溶液2,室温中反复上下颠倒10次并静置10 min以充分裂解细菌。最后加入300 μL溶液3,上下颠倒5次充分混匀后于冰上放置5 min,以12 000 r/min离心5 min,取上清液600 μL,加入异丙醇500 μL,室温中上下颠倒5次后静置沉淀基因组DNA,10 min后以12 000 r/min离心5 min,弃去上清液。在沉淀中加入1 mL 70%乙醇,上下颠倒2次后于12 000 r/min离心2 min并弃去上清液。打开离心管盖在室温下放置10 min除去残留乙醇,加入200 μL灭菌蒸馏水重新溶解金黄色葡萄球菌基因组DNA,用于后续PCR检测。
1.5 PCR检测
在PCR管中依次加入12.5 μL 2×PCR预混液(Dye Plus),各引物0.3 μL,煮沸法制备模板和改良碱裂解法制备模板均为1 μL,以灭菌蒸馏水补足体系至25 μL。PCR反应条件:94℃预变性3 min,94℃变性 45 s,spa基因扩增时以55℃退火60 s,mecA基因扩增时以50.6 ℃退火45 s,femB基因扩增时以57 ℃退火45 s,所有PCR扩增均以72 ℃延伸1 min,共30个循环。PCR反应结束后,产物经10 g/L琼脂糖凝胶电泳,在凝胶成像系统下观察并照相记录。
2 结果
2.1 spa基因扩增结果
以改良碱裂解法制备的金黄色葡萄球菌DNA为模板,所有样本可扩增出spa基因;以直接煮沸法制备的DNA为模板,仅3号样本扩增出spa基因。见图1。
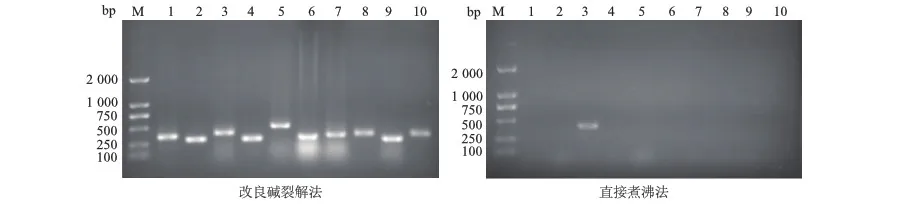
M:DNA标准参照物;1-10:10株金黄色葡萄球菌菌株
2.2 mecA基因扩增结果
以改良碱裂解法制备的细菌DNA为模板,所有样本可扩增出mecA基因;以直接煮沸法制备的DNA为模板,可见8号和9号样本扩增出明亮的mecA基因,3号和7号样本可见隐约的mecA基因。见图2。
2.3 femB基因扩增结果
以改良碱裂解法制备的金黄色葡萄球菌DNA为模板,可扩增出3号、4号、6号和9号样本的femB基因;以直接煮沸法制备的DNA为模板,无样本扩增出femB基因。见图3。
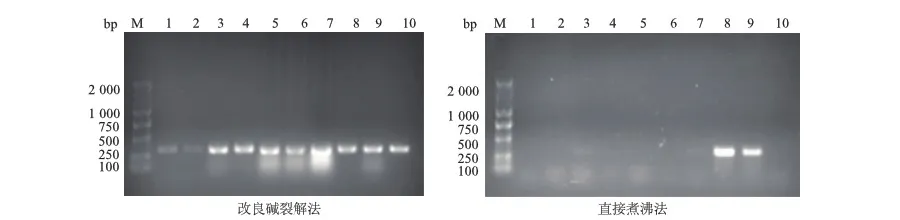
M:DNA标准参照物;1-10:10株金黄色葡萄球菌菌株

M:DNA标准参照物;1-10:10株金黄色葡萄球菌菌株
3 讨论
常规方法难以有效破坏金黄色葡萄球菌坚固的细胞壁,使得基因组DNA提取效率极低,无法用于PCR法检测[8]。虽然目前已有多种金黄色葡萄球菌基因组DNA提取方法,包括SDS裂解法、CTAB法、反复冻融裂解法等,但都存在操作复杂、耗时长、提取效率低等缺点[9]。现有的DNA抽提试剂盒虽然可以有效提取金黄色葡萄球菌基因组DNA,但使用成本高,增加了患者的经济负担,也阻碍了分子生物学技术在金黄色葡萄球菌研究中的推广使用[3]。
与金黄色葡萄球菌相似,植物细胞也具有坚固的细胞壁。常规PCR检测多采用碱裂解法破坏细胞壁提取植物细胞基因组DNA,而无需使用液氮研磨等方式[10]。本研究中我们借鉴植物细胞基因DNA的碱裂解提取法并降低了提取试剂中NaOH浓度,建立了改良碱裂解法提取金黄色葡萄球菌基因组DNA技术。该方法不仅可以有效裂解金黄色葡萄球菌细胞壁,同时可以抑制细菌释放的核酸酶活性,具有较高的DNA提取效率,并能够缩短操作时间,完全可以满足临床检验科开展后续分子生物学检测的要求,值得推广使用。
煮沸法是目前最常用的细菌基因组DNA抽提方法,该方法无需其他试剂,方便快捷,广泛用于各种细菌PCR检测前的模板制备[11]。本研究中我们采用PCR法检测了MRSA菌中spa基因、mecA基因和femB基因。我们的研究结果表明,与改良碱裂解法相比,直接煮沸法制备模板扩增出来的spa基因、mecA基因和femB基因的标本数量明显低于改良碱裂解法。我们推测,由于金黄色葡萄球菌细胞壁较厚,单纯地煮沸并不能有效裂解细菌。同时临床分离获得的金黄色葡萄球菌多为致病菌,自身还分泌耐热核酸酶,该酶在煮沸后并不会失活,仍可以降解细菌裂解所释放出的核酸,因此煮沸法所获得的模板DNA量较少,导致PCR法无法检测出细菌相关基因[12]。改良碱裂解法中,我们借鉴大肠埃希菌质粒抽提方法,在DNA抽提体系中加入SDS和NaOH,这两种试剂不仅可以裂解细菌,还将整个体系pH值调整至8.0以上,可有效地抑制核酸酶活性,防止细菌DNA被降解。
由于单纯煮沸无法裂解金黄色葡萄球菌,普通溶菌酶裂解效果也较差,许多学者使用特殊的葡萄球菌酶裂解金黄色葡萄球菌,但该酶成本高昂[13]。本研究中,我们通过摸索发现,普通溶菌酶只需将37℃孵育时间延长至6 h,再配合使用碱裂解法即可获得理想的裂解效果。该方法虽然延长了抽提基因组DNA的时间,但避免使用较为昂贵的葡萄球菌酶,节省了成本,建议推广使用。
[ 1 ] 王凤玲, 侯英荣,冯秀河. 耐甲氧西林金黄色葡萄球菌耐药基因及致病毒素基因的研究进展[J]. 中国感染与化疗杂志, 2010, 10(1):72-75.
[ 2 ] Mehta MS, McClure JT, Mangold K, et al. Performance of 3 real-time PCR assays for direct detection ofStaphylococcusaureusand MRSA from clinical samples[J]. Diagn Microbiol Infect Dis, 2015, 83(3):211-215.
[ 3 ] Sabat A, Malachowa N, Miedzobrodzki J, et al. Comparison of PCR-based methods for typingStaphylococcusaureusisolates[J]. J Clin Microbiol, 2006, 44(10):3804-3807.
[ 4 ] 张涛涛, 王兰,龚频,等. 金黄色葡萄球菌基因组DNA提取方法的优化[J]. 食品研究与开发, 2014, 35(3):8-10.
[ 5 ] 冯飞, 梁景龙,苏丽婷,等. 不同方法对金黄色葡萄球菌基因组DNA提取效果的比较[J]. 广东农业科学, 2010, 37(10):153-155.
[ 6 ] 李克诚, 李琼, 夏菲,等. 耐甲氧西林金黄色葡萄球菌spa基因分型[J].疾病监测,2012,27(11):877-880.
[ 7 ] 黄辉, 陈颖, 安如俊,等. MRSA中mecA及femB基因的检测与耐药相关性[J]. 重庆医学, 2009,3(1):54-56.
[ 8 ] Huletsky A,Giroux R, Rossbach V,et al.New real-time PCR assay for rapid detection of methicillin-resistantStaphylococcusaureusdirectly from specimens containing a mixture of staphylococci[J]. J Clin Microbiol, 2004, 42(5):1875-1884.
[ 9 ] Maes N, Magdalena J, Rottiers S, et al. Evaluation of a triplex PCR assay to discriminateStaphylococcusaureusfrom coagulase-negative Staphylococci and determine methicillin resistance from blood cultures[J]. J Clin Microbiol, 2002, 40(4):1514-1517.
[10] Lartigue C, Glass JI, Alperovich N, et al. Genome transplantation in bacteria: changing one species to another[J]. Science, 2007, 317(5838):632-638.
[11] 李筱婷, 陈卓君, 许文涛, 等. 一种适于PCR扩增的植物基因组快速提取新方法[J]. 农业生物技术学报, 2010, 18(2): 394-399.
[12] Pilsczek FH, Salina D, Poon KK, et al. A novel mechanism of rapid nuclear neutrophil extracellular trap formation in response toStaphylococcusaureus[J].J Immunol,2010,185(12):7413-7425.
[13] Francius G, Domenech O, Mingeot-Leclercq MP, et al. Direct observation ofStaphylococcusaureuscell wall digestion by lysostaphin[J]. J Bacteriol, 2008, 190(24):7904-7909.
