原位微宇宙法研究温带森林土壤真菌群落构建的驱动机制
2018-06-07李毳马转转乔沙沙刘晋仙柴宝峰
李毳,马转转,乔沙沙,刘晋仙,柴宝峰,
1. 山西财经大学环境经济学院,山西 太原 030006;2. 山西大学黄土高原研究所,山西 太原 030006;3. 山西大学绿色发展中心,山西 太原 030006
群落的生物多样性及时空格局是生态学研究的中心议题之一(Tedersoo et al.,2012)。传统生态位理论(Gause,1934)认为,物种对资源的竞争所导致的生态位分化是维持群落多样性的主要机制,最经典的案例是Lack在Galapagos群岛上对达尔文莺(Coerebini)的研究,鸟嘴的形态反映了鸟类食性的分化,是生态位分化的直接证据。另外,热带森林土壤营养的可利用性对群落结构的决定作用,以及温带森林中的物种聚集(aggregation)与环境因子的相关性,这些都说明群落结构受到环境因子的影响(Kaiser et al.,2010;Kraft et al.,2008)。由此认为,群落的结构特征可以通过环境因子得以预测,是一个确定性过程(deterministic processes)。近年来备受关注的中性理论(Whitfield,2002),承认群落内物种对资源的竞争,但认为群落内所有处于同一营养级的物种的属性是等价的,迁入、迁出和成种过程对群落结构具有决定作用,是一个随机性过程(stochastic processes)(Hubbell,2006),并得到一些野外实验数据的支持(Volkov et al.,2004;Zhou et al.,2008)。Gravel et al.(2006)将以上两种观点整合为连续体(continuum)假说,认为生态位理论和中性理论是一个连续体的两个极端,现实的自然群落位于连续体中的一个特定位点,所以目前的焦点集中于确定性过程和随机性过程在群落构建中的相对重要性(Dumbrell et al.,2010)。已有的数据大多基于植物和动物,而针对微生物群落的研究起步较晚(Guo et al.,2015)。微生物群落结构和功能的研究近年来备受关注(Fuhrman,2009;Zhou et al.,2012)。有观点认为,微生物群落构建不存在扩散限制(Finlay,2002),环境选择决定了微生物群落的构建过程(Gulay et al.,2016),也有学者质疑此观点,认为微生物群落的相似度随地理距离增加而衰减(Nemergut et al.,2013;Zinger et al.,2014),如丛枝菌根真菌、子囊真菌(Martiny et al.,2011)。在全球尺度上对 4个热带森林和区域尺度上对3个温带森林的外生菌根真菌的研究,也证实扩散限制影响了真菌群落的构建(Hazard et al.,2013)。这些相互矛盾的观点说明,微生物群落生态学的理论亟待进一步完善。
土壤微生物种类丰富,数量多,且有极强的扩散能力,是验证中性理论的理想材料。山西庞泉沟不同海拔高度的森林土壤真菌群落中,子囊菌门和担子菌门居多,子囊菌多为腐生菌,可分解难降解的有机质;红盖伞属和红菇属是重要的外生菌根,与多种树种共生,这些真菌在不同植被类型的土壤微生物群落中占据的比例不同(乔沙沙等,2017)。本文利用 PCR-DGGE技术和微宇宙实验,对庞泉沟5种典型森林的土壤真菌群落结构开展研究,分析局域环境因子和区域源群落的多样性对温带森林土壤真菌群落构建的影响,为揭示微生物群落多样性维持机制提供支持。
1 材料与方法
1.1 研究区概况
庞泉沟国家自然保护区位于吕梁山脉中段( 37°48'~37°56'N , 111°24'~111°32'E ), 面 积 为10443.5 hm2,属暖温带大陆性季风气候,年均降水量822.6 mm,年均温4.3 ℃。试验样地包括高海拔的华北落叶松(Larix gmelinii var.principis-rupprechtii),低海拔的华北落叶松、油松(Pinus tabulaeformis)、白杄(Picea meyeri)、青杄(P. wilsonii)、白桦(Betula platyphylla)和红桦(B.albosinensis)林等。样地分布及具体信息见表1。
1.2 实验设计与样品采集
1.2.1 实验设计
微宇宙实验参照Bell(2010)的方法。土壤样品取自保护区内不同海拔、不同植被类型的10个样地(表 1),作为区域土壤微生物物种库;在 10个样地固定样方(1 m×1 m)中按对角线取0~15 cm厚度土壤混合,再从每个样地的土壤样品中各取50 g,充分混匀,加入1 L灭菌水,振荡10 min,灭菌滤纸过滤后得到接种液。根据海拔高度和群落类型,选择1、2、3、4、8、9共6个样地,每个样地取15 cm深处土壤,混匀后称取15 g,灭菌处理,然后喷洒5 mL接种液,混合均匀,置于50 mL无菌离心管中,0.22 μm滤膜封口,作为微生物的源群落,分别置于相应的6个样地,呈45°斜角埋于土壤15 cm深处,原地培养12个月,带回实验室,同时取样地土壤样品为对照。
1.3 样品分析
1.3.1 土壤理化性质测定
用 HANNA HI3221(msoil∶Vwater=1∶2.5)测定土壤pH值;用vario MACRO cube元素分析仪测定土壤总碳(TC)和总氮(TN)含量;用CleverChem 380全自动间断化学分析仪测定速效磷(AP)、NH4+的含量;用重铬酸钾容量法(鲁如坤,2000)测定土壤有机质(SOM);用激光衍射粒度分析仪(Matersizer 3000 Laser Diffraction Particle Size Analyzer,UK)测定土壤粒度;参照关松荫的方法测定蔗糖酶的酶活性(关松萌,1986)。
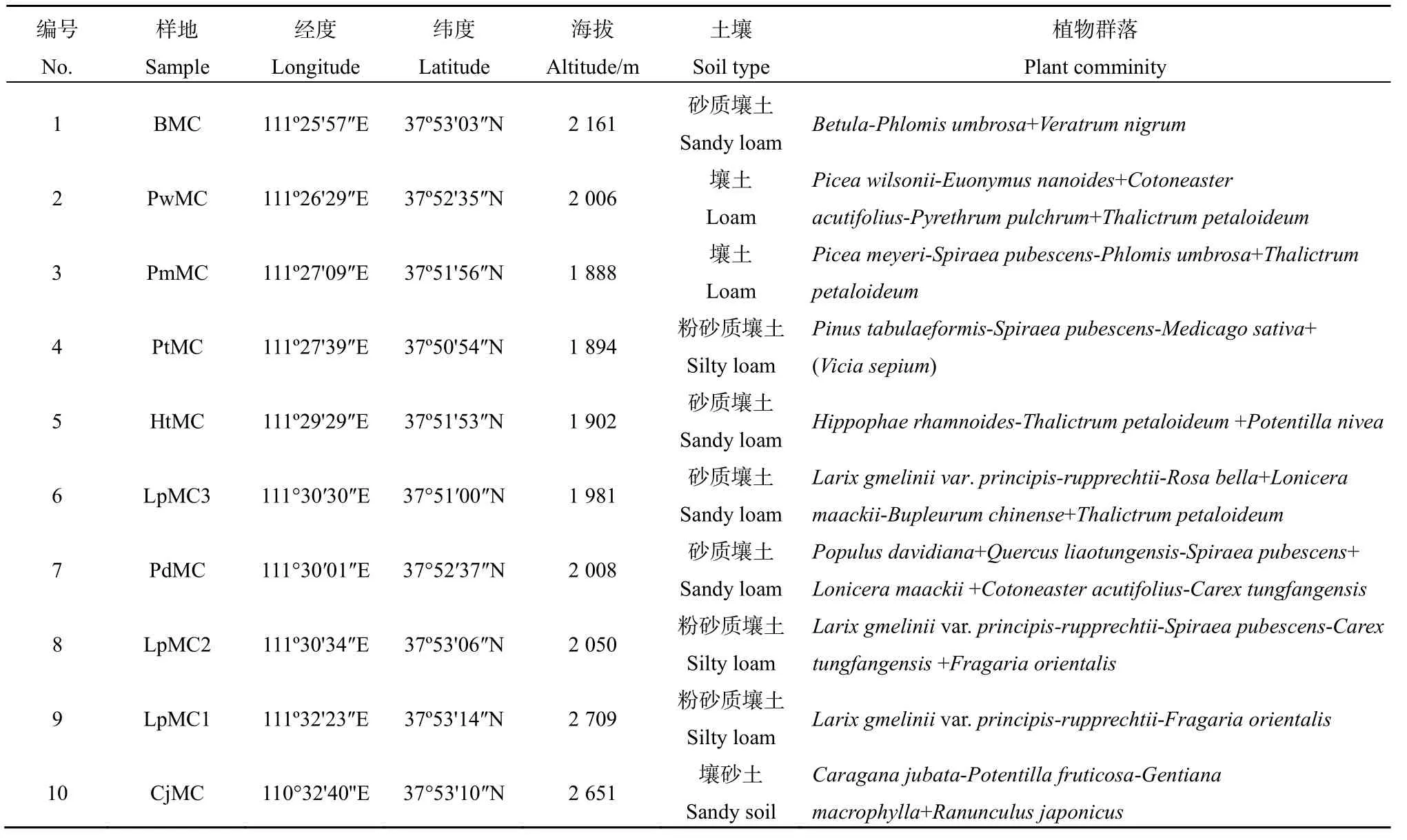
表1 样地土壤和植物群落概况Table 1 The information of soil and plant communities in samples of this study
1.3.2 土壤宏基因组提取和PCR扩增
用 E.Z.N.A.Soil DNA Kit试剂盒(Omega Bio-tek,Inc.)提取土壤宏基因组,用特异性引物NS1:GTAGTCATATGCTTGTCTC 和 GCFung:5'-CGCCCGCCGCGCCCCGCGCCCGGCCCGCCG CCCCC GCCCCATTCCCCGTTACCCGTTG-3'扩增真菌 18S rDNA 片段。PCR反应体系:2.5 μL 10×EasyTaq Buffer;0.25 U Taq 酶(500 U);2 μL 2.5 mmol dNTPs;2 μL DNA(10 ng 左右);引物(10 μmol·L-1)各 0.3 μL;加灭菌双蒸水至 25 μL。RCR反应条件:94 ℃ 4 min,94 ℃ 30 s,58 ℃ 20 s,72 ℃ 20 s,30次循环,72 ℃ 7 min,10 ℃保存。
1.3.3 变性梯度凝胶电泳分析
变性梯度凝胶电泳(DGGE),采用8%的聚丙烯酰胺凝胶,变性剂的质量分数范围为26%~31%。电泳条件:1×TAE电泳缓冲液,60 V,60 ℃,电泳12 h。银染液染色,获得DGGE条带图谱。利用Quantity One(version 4.52)软件分析图谱,用条带数和条带灰度值计算 Shannon-Wiener多样性指数(H′)、Pielou均匀度指数(J)和丰富度。

式中,ni是第 i条条带的多度;n为样品中所有条带的总多度;S为样品中的总条带数(Magurran et al.,2011)。
1.4 数据分析
运用SPSS 19.0中的单因素方差分析法比较真菌群落多样性指数、均匀度指数及丰富度,采用LSD检验进行多重比较;运用Canoco 5软件进行主坐标分析;运用Gephi 0.9.1分析群落种间网络关系;运用Ri386 3.3.1进行Mantel分析和偏Mantel分析。
2 结果与分析
2.1 环境因子对真菌群落结构形成的影响
6个微宇宙样地的土壤真菌群落 α-多样性见表2。LpMC1中真菌群落Shannon指数和丰富度均为最大,而BMC中真菌群落多样性指数均为最小,LpMC1、LpMC2及PwMC的Shannon指数、均匀度指数均没有显著差异(P<0.05)。各样地微宇宙土壤真菌群落的 3个多样性指数总体上均大于样地土壤,微宇宙土壤真菌Shannon多样性、丰富度和均匀度的平均值分别为 2.59、18.14和0.89,而对照样地分别为2.35、14.65、0.88。微宇宙真菌群落多样性指数均有所提高,说明源群落对新形成的真菌群落结构有影响,区域物种迁移过程可能影响局域群落结构,即真菌群落结构受到扩散限制的影响。
微宇宙土壤的真菌群落的 β-多样性平均值为0.51(0.62~0.44),而6个对照样地的平均值为0.54(0.75~0.36)。样地 LpMC1 和 PmMC 最大,为 0.75,LpMC1和PwMC最小,为0.36,LpMC1和LpMC2之间、LpMC2和PwMC之间的β-多样性值分别为0.38和0.40。由此可见,微宇宙土壤的α多样性显著提高,而β多样性减小,在一定程度上表现出趋同效应,可见“种源”对微宇宙土壤真菌群落结构的影响。由此说明,环境因子驱动群落发生趋异效应的能力是有限的,群落的形成历史(种源)对群落结构的形成具有重要作用。
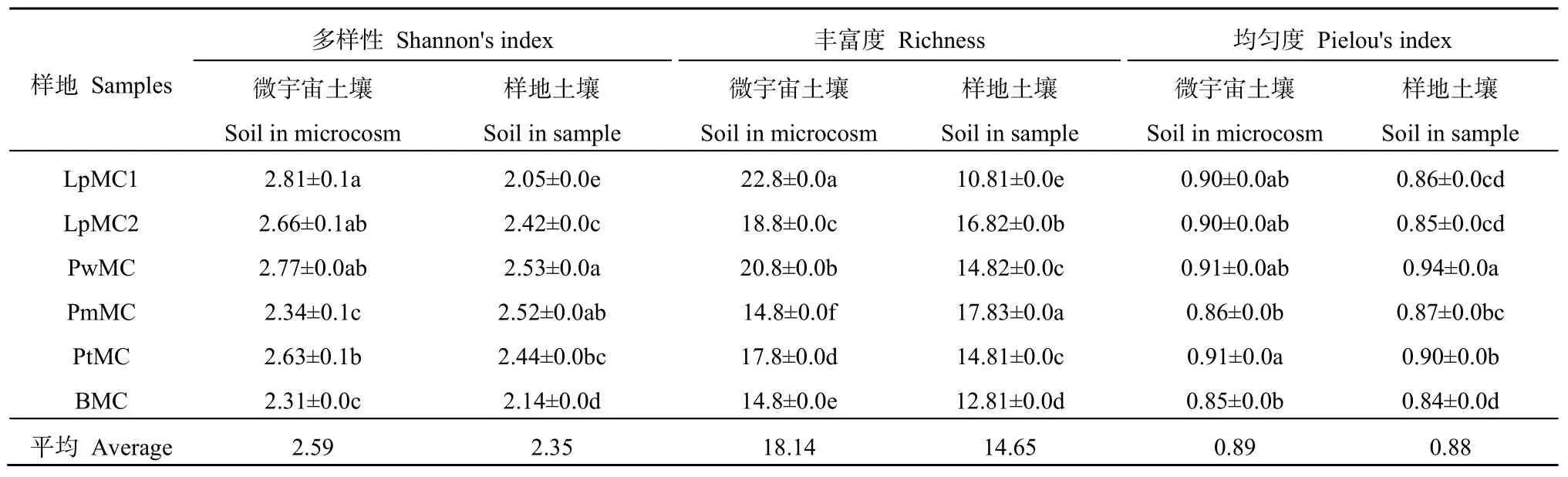
表2 置于不同样地土壤中的“微宇宙”土壤真菌群落多样性Table 2 Diversity of fungal communities in microcosm soils in different plant communities
微宇宙试验忽略了空间变量对微生物群落的影响,土壤真菌群落的相对丰度与环境因子及植物优势度呈显著正相关(表3)。整体而言,微宇宙土壤真菌群落结构受到土壤营养状况、温度、水分、pH以及地表植被优势度的影响,相关性最大的为土壤有机质(SOM,Rm=0.5908,P=0.001),其次为有效磷(Rm=0.476,P=0.001)、土壤含水量(Rm=0.388,P=0.001),它们均对其具有极显著的影响,另外,土壤 pH、蔗糖酶和温度亦对其有显著影响。对于样地土壤真菌群落,土壤有机质的影响也是较大的(R=0.6595,P=0.001),但土壤有效磷(R=0.613,P=0.001)和土壤含水量(R=0.5664,P=0.001)的影响更为显著。植被优势度对样地真菌的影响显著(R=0.3256,P=0.003),而对微宇宙土壤真菌的影响不显著。总体而言,土壤因子和植被对样地土壤和微宇宙土壤真菌群落均有一定的影响。Mantel分析(表4)表明,土壤环境因子和植被因子均对微宇宙土壤真菌群落有极显著影响(P<0.01),R值分别为0.4936、0.2652。偏Mantel分析表明,控制植被变量后,土壤变量对真菌群落的影响仍是极显著的(Rm=0.4862,R=0.464)。
2.2 生物因素对真菌群落结构的影响
用中介中心性(betweenness centrality,BC)分析土壤真菌群落不同类群间的相互关系(图1),有显著相关性的真菌类群共有 160对(B1~B160)。对照样地土壤真菌类群间相互关系总体表现为排斥大于共存(图1a),共有6个关键类群,分别为B1、B11、B12、B17、B22、B23;而微宇宙土壤真菌类群间相互关系则明显表现出共存大于排斥(图1b),共有5个关键类群,分别为B14、B16、B32、B30、B8。这 5个关键类群不是存在于所有微宇宙土壤中,如类群B16、B30均存在于高海拔落叶松林,低海拔落叶松林、青杄林、白杄林的微宇宙土壤中,类群B8出现在高海拔、低海拔落叶松林及白杄林的微宇宙土壤中。总体而言,微宇宙土壤真菌群落间相互作用比对照样地土壤更为简单,且关键类群数减少,这可能是因为要形成一个稳定的微生物群落需要经历更长的时间,而微宇宙土壤微生物群落代表的是群落形成初期(1 a)的状态。
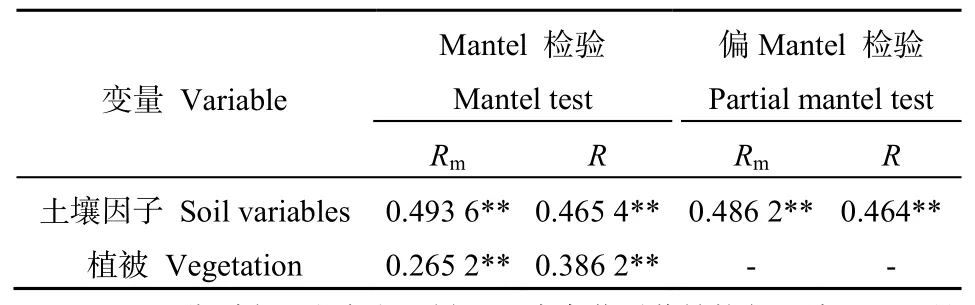
表4 真菌群落结构与不同变量因子的Mantel检验Table 4 Mantel test analysis of the relationship between the structure of fungal communities and soil and plant variables
微宇宙试验表明,环境因子(包括土壤理化因素和植被因素)及生物因素(种间作用)在驱动土壤真菌群落动态过程中具有重要作用,但群落历史或区域过程对群落结构的形成也有一定的影响。
3 讨论
局域环境分拣(species sorting effect)与区域扩散限制(dispersal limitation)的相对重要性是微生物群落生态学研究的一个焦点(Chase,2010;Ricklefs,1987),我们认为这两种过程对土壤真菌群落结构都具有重要的影响。(1)低海拔华北落叶松土壤真菌群落的Shannon指数和丰富度指数都低于高海拔华北落叶松(表 2),与 Siles et al.(20016)的研究结果一致,高海拔土壤真菌群落的丰度和Shannon指数较高,且多样性指数与土壤pH呈显著正相关。细菌群落多样性随海拔升高而降低是因为高海拔针叶林林土壤中,尽管总碳和腐殖质含量较高,但多数为难分解物质,而真菌偏好低营养、难分解以及高碳氮比的有机物,对不良环境的耐受能力远远大于细菌,而且真菌更适应于寒冷环境(Robinson,2001)。不过也有研究显示,随着海拔升高,真菌群落物种多样性呈下降趋势(Bahramm et al.,2012)。(2)土壤温度、水分、pH值等是影响微生物群落结构的关键因子(BBahram et all.,2015),本研究结果表明,环境因素在驱动真菌群落构建过程中具有非常重要的作用(表3),土壤温度、含水量、有机质、pH值、总碳、总氮、有效磷、以及植被优势度对样地及微宇宙土壤真菌群落相对丰度均具有显著影响(P<0.05)。(3)关于环境因子对微生物群落结构的影响,一般认为,土壤微生物群落结构在海拔梯度上主要受地表植被类型的影响(Chenn et al.,20177)。一些真菌类群如丛枝菌根菌可与植物共生,进而影响其他微生物类群。也有研究提出,植被本身不直接影响土壤微生物群落的结构,但可通过影响环境因子间接影响群落结构(Tedersooet al.,20144)。本研究结果与Ping et all.(2017)的研究结果一致,森林土壤真菌群落结构同时受到土壤环境因子和空间格局的影响,环境因素对其影响更为明显。
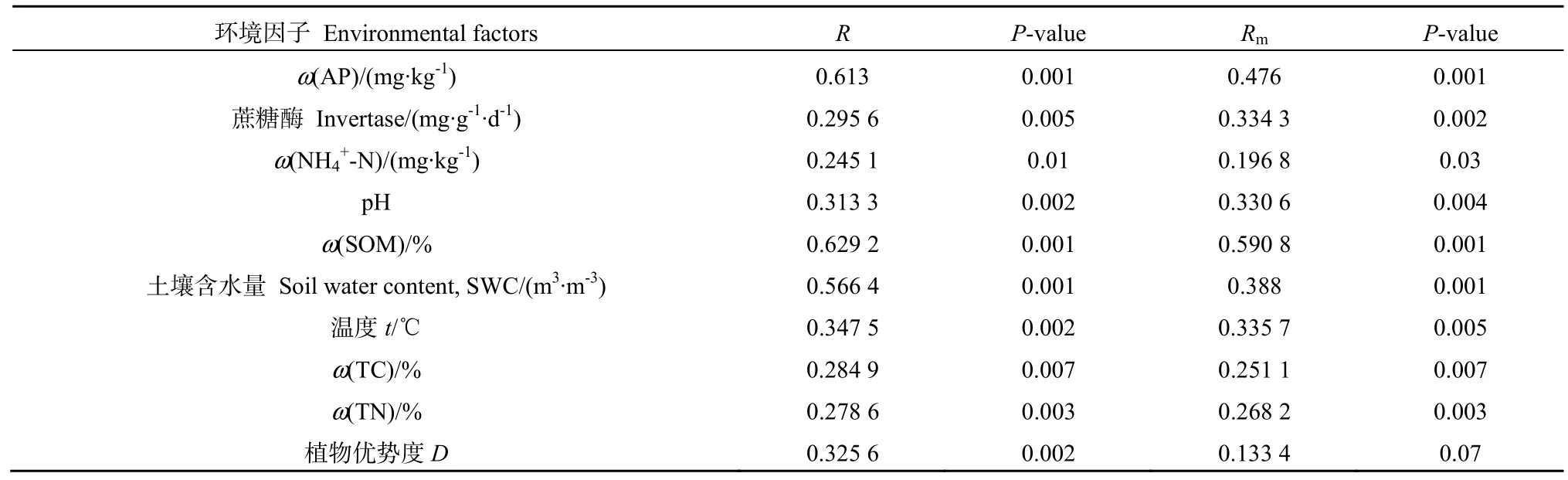
表3 真菌群落结构与土壤环境因子关系的Mantel检验Table 3 Mantel test analysis of the relationship between structure of fungal communities and soil edaphic factors
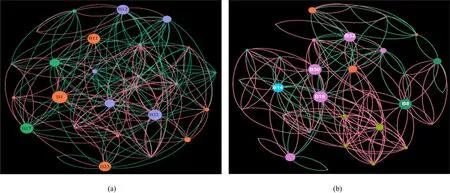
图1 样地土壤(a)和微宇宙土壤(b)真菌群落类群间关系网络图Fig. 1 Thee network map of iinterspecific relationships of fungalcommunities
微生物因具有极强的扩散能力而呈全球性分布,即“everything is everywwhhere,but theenvironmentt seleects”,这一假设引起了广泛争论(Wit et al.,20006)。Tedersooo et al.(20014)就土壤真菌的全球多样性和生物地理格局进行了研究,认为在全球尺度上真菌大多具有较强的生物地理格局,且很可能是由扩散限制和环境因子所驱动的。Raapoport法则指出大型动植物物种多样性呈现纬度梯度,之后发现该法则同样适用于海洋细菌(Sul etal.,2013)。利用转接实验研究微生物扩散到新环境中的群落构建方式,探讨在相同环境条件下,不同来源的微生物群落是否呈现趋同现象,或是经过人为扩散的微生物群落由于起始群落组成的差异,发生趋异现象。有研究认为其在科水平呈现趋同效应,而在种水平则呈现趋异效应,起始群落组成的差异是导致类群分化的主要原因(Karrayanni et al.,2017)。本研究针对不同海拔高度的几种植被类型土壤真菌群落,利用微宇宙转接实验,将整个区域的土壤微生物作为源群落,将其分别置于不同的土壤生境中达一年以上,发现微宇宙土壤真菌群落α多样性显著提高,而β多样性减小,趋同效应明显。认为区域真菌源群落对微宇宙土壤真菌群落的结构具有显著的影响,群落历史对群落结构的形成具有重要的作用。
除环境因子,生物间共存模式对群落结构也有影响,扩散的真菌类群受到新的环境条件和种间竞争的双重影响。微生物群落中的种间作用方式以及关键类群的改变会对群落结构产生影响(Barberan et al.,2012)。BC分析显示,与源样地土壤相比,人为扩散的微宇宙土壤真菌类群间的关系发生了改变,关键类群减少(图1b),类群间共存作用大于排斥作用,在一定程度上影响局域真菌群落的构建过程。
4 结论
在局域尺度上,确定性过程在温带森林土壤真菌群落构建过程中占主导作用,而随机性过程具有重要影响。土壤因子和生物因素作为确定性因素决定了真菌群落的结构,扩散限制一定程度上对真菌群落的结构产生影响。人为扩散的真菌群落的结构和多样性受到源群落的多样性特征和环境因子的双重影响。
BAHRAM M, PEAY K G, TEDERSOO L. 2015. Local-scale biogeography and spatiotemporal variability in communities of mycorrhizal fungi [J]. The New Phytologist, 205(4): 1454-1463.
BAHRAM M, POLME S, KOLJALG U, et al. 2012. Regional and local patterns of ectomycorrhizal fungal diversity and community structure along an altitudinal gradient in the Hyrcanian forests of northern Iran[J]. The New Phytologist, 193(2): 465-473.
BARBERAN A, BATES S T, CASAMAYOR E O, et al. 2012. Using network analysis to explore co-occurrence patterns in soil microbial communities [J]. The ISME Journal, 6(2): 343-351.
BELL T. 2010. Experimental tests of the bacterial distance-decay relationship [J]. The ISME Journal, 4(11): 1357-1365.
CHASE J M. 2010. Stochastic community assembly causes higher biodiversity in more productive environments [J]. Science, 328(5984):1388-1391
CHEN Y L, XU T L, VERESOGLOU S D, et al. 2017. Plant diversity represents the prevalent determinant of soil fungal community structure across temperate grasslands in northern China [J]. Soil Biology & Biochemistry, 110: 12-21.
DUMBRELL A J, NELSON M, HELGASON T, et al. 2010. Relative roles of niche and neutral processes in structuring a soil microbial community [J]. The ISME Journal, 4(3): 337-345.
FINLAY B J. 2002. Global dispersal of free-living microbial eukaryote species [J]. Science, 296(5570): 1061-1063.
FUHRMAN J A. 2009. Microbial community structure and its functional implications [J]. Nature, 459(7244): 193-199.
GAUSE G F. 1934. Experimental analysis of vito volterra's mathematical theory of the struggle for existence [J]. Science, 79(2036): 16-17.
GRAVEL D, CANHAM C D, BEAUDET M, MESSIER C. 2006.Reconciling niche and neutrality: the continuum hypothesis [J].Ecology Letters, 9(4): 399-409.
GULAY A, MUSOVIC S, ALBRECHTSEN H J, et al. 2016. Ecological patterns, diversity and core taxa of microbial communities in groundwater-fed rapid gravity filters [J]. The ISME Journal, 10(9):2209-2222.
GUO G, KONG W, LIU J, et al. 2015. Diversity and distribution of autotrophic microbial community along environmental gradients in grassland soils on the Tibetan Plateau [J]. Applied Microbiology and Biotechnology, 99(20): 8765-8776.
HAZARD C, GOSLING P, VAN DER GAST C J, et al. 2013. The role of local environment and geographical distance in determining community composition of arbuscular mycorrhizal fungi at the landscape scale [J]. The ISME Journal, 7(3): 498-508.
HUBBELL S P. 2006. Neutral theory and the evolution of ecological equivalence [J]. Ecology, 87(6): 1387-1398.
KAISER C, KORANDA M, KITZLER B, et al. 2010. Belowground carbon allocation by trees drives seasonal patterns of extracellular enzyme activities by altering microbial community composition in a beech forest soil [J]. The New Phytologist, 187(3): 843-858.
KARAYANNI H, MEZITI A, SPATHARIS S, et al. 2017. Changes in microbial (bacteria and archaea) plankton community structure after artificial dispersal in grazer-free Microcosms [J]. Microorganisms,5(31): 1-14.
KRAFT N J, VALENCIA R, ACKERLY D D. 2008. Functional traits and niche-based tree community assembly in an Amazonian forest [J].Science, 322(5901): 580-582.
MAGURRAN A E, MCGILL B J. 2011. Biological Diversity: frontiers in measurement and assessment [M]. New York: Oxford University Press.
MARTINY J B, EISEN J A, PENN K, et al. 2011. Drivers of bacterial beta-diversity depend on spatial scale [J]. Proceedings of the National Academy of Sciences of the United States of America, 108(19):7850-7854.
NEMERGUT D R, SCHMIDT S K, FUKAMI T, et al. 2013. Patterns and processes of microbial community assembly [J]. Microbiology and Molecular Biology Reviews: 77(3): 342-356.
PING Y, HAN D, WANG N, et al. 2017. Vertical zonation of soil fungal community structure in a Korean pine forest on Changbai Mountain,China [J]. World Journal of Microbiology & Biotechnology, 33(1): 12.
RICKLEFS R E. 1987. Community diversity: relative roles of local and regional processes [J]. Science, 235(4785): 167-171.
ROBINSON C H. 2001. Cold adaptation in Arctic and Antarctic fungi [J].New Phytologist, 151(2): 341-353.
SILES J A, MARGESIN R. 2016. Abundance and diversity of bacterial,archaeal, and fungal communities along an altitudinal gradient in alpine forest soils: what are the driving factors? [J]. Microbial Ecology, 72(1): 207-220.
SUL W J, OLIVER T A, DUCKLOW H W, et al. 2013. Marine bacteria exhibit a bipolar distribution [J]. Proceedings of the National Academy of Sciences of the United States of America, 110(6): 2342-2347.
TEDERSOO L, BAHRAM M, PÕLME S, et al. 2014. Global diversity and geography of soil fungi [J]. Science, 346(6213): 1-11.
TEDERSOO L, BAHRAM M, TOOTS M, et al. 2012. Towards global patterns in the diversity and community structure of ectomycorrhizal fungi [J]. Molecular Ecology, 21(17): 4160-4170.
VOLKOV I, BANAVAR J R, MARITAN A, HUBBELL S P. 2004.Neutral theory: the stability of forest biodiversity [J]. Nature,427(6976): 696; discussion 696-697.
WHITFIELD J. 2002. Neutrality versus the niche [J]. Nature, 417(6888):480-481.
WIT D R, BOUVIER T. 2006. ‘Everything is everywhere, but, the environment selects’; what did Baas Becking and Beijerinck really say? [J]. Environmental Microbiology, 8(4): 755-758.
ZHOU J Z, XUE K, XIE J P, et al. 2012. Microbial mediation of carbon-cycle feedbacks to climate warming [J]. Nature Climate Change, 2(2): 106-110.
ZHOU S R, ZHANG D Y. 2008. A nearly neutral model of biodiversity [J].Ecology, 89(1): 248-258.
ZINGER L, BOETIUS A, RAMETTE A. 2014. Bacterial taxa-area and distance-decay relationships in marine environments [J]. Molecular Ecology, 23(4): 954-964.
关松萌. 1986. 土壤酶及其研究方法[M]. 北京: 农业出版社.
鲁如坤. 2000. 土壤农业化学分析方法[M]. 北京: 中国农业科技出版社.
乔沙沙, 周永娜, 柴宝峰, 等. 2017. 关帝山森林土壤真菌群落结构与遗传多样性特征[J]. 环境科学, 38(6): 314-324.
