籼稻C84和粳稻春江16B重组自交系遗传图谱构建及籽粒性状QTL定位与验证
2018-05-25周梦玉宋昕蔚徐静付雪李婷朱雨晨肖幸运毛一剑曾大力胡江朱丽任德勇高振宇郭龙彪钱前吴明国林建荣张光恒
周梦玉 宋昕蔚 徐静 付雪 李婷 朱雨晨 肖幸运 毛一剑 曾大力 胡江 朱丽 任德勇 高振宇 郭龙彪 钱前 吴明国 林建荣 张光恒
水稻是世界主要粮食作物之一,超过一半的人口以稻米为主食[1]。随着世界人口增加,耕地面积不断减少,环境压力逐渐加大,提高水稻单产成为解决世界粮食安全问题的有效途径之一。而随着人们生活水平提高,对稻米品质的要求也越来越高。水稻粒型不仅与水稻产量相关,还与稻米的外观品质、加工品质、蒸煮和食味品质存在密切的关系[2]。因此,开展粒型性状QTL研究,对克隆调控水稻粒型性状关键基因、揭示产量构成、探索粒型与稻米品质相关性具有重要的理论意义和应用价值。
迄今为止,国内外的许多研究者们利用不同类型的遗传群体构建了多种遗传图谱。McCouch等[3]利用水稻基因组测序成果,采用电子PCR的方法绘制了一张包含2740个SSR分子标记连锁图谱。Xu等[4]利用窄叶青8号与京系17籼粳杂交获得的永久性DH群体,构建了一个包含108个位点的水稻遗传图谱。张启军等[5]选用4个测序水稻品种作为作图亲本,两两组合,绘制了6张微卫星连锁图谱。庄杰云等[6]利用24个SSR标记构建了103个水稻主要栽培品种的分子指纹图谱数据库。徐建军[7]以一套用籼稻品种9311为受体、粳稻品种日本晴为供体构建的128个染色体片段代换系为材料,利用多元回归分析方法,绘制Bin图谱。作图群体为F2群体或回交群体时,属于暂时性群体,只能使用一个有性世代,难以长期维持。相比之下,重组自交系(RIL)群体和加倍单倍体(DH)群体由于遗传上的纯合性,遗传干扰少,可永久继代保存,适于进行各种持续性研究[8]。
遗传图谱的构建,为水稻数量性状调控基因的定位与克隆提供了便利。研究者们利用各种不同类型的遗传群体,报道了粒长、粒宽、粒厚和粒重等粒型相关性状的QTL或基因。其中,粒长QTL达103个,粒宽QTL达95个,粒厚QTL数量相对较少[9],另有控制粒重QTL 233个。方先文等[10]以云南省地方籼稻品种苜蓿和江苏省优质粳稻品种南京46为材料,构建了包含202个SSR标记的分子遗传连锁图谱,并研究了RIL群体中粒长、粒宽、籽粒长宽比和粒厚等粒型相关性状。Xia等[11]为了阐明籽粒形态的遗传基础,利用籼稻品种Jin 23B和圆粳稻品种QingGuAi构建的一个回交群体,共检测到10个粒型性状的QTL,其中qGW1、qGS3和qGS7三个对籽粒形态有较大影响的QTL在两年内均被检测到。Zhao等[12]为了解“南阳占”大粒形态的遗传基础,利用南阳占和珍汕97B的重组自交系群体2年内共检测到53个QTL,分布在11条染色体上。其中,粒宽和粒长的QTL分别在第2和第3染色体上检测到的频率最高。Zhou等[13]用蜀恢527作为轮回亲本与小粒品种杂交并回交,利用BC2F2群体将控制水稻粒长的主效QTLLk-4(t)作为单个孟德尔因子,定位在第3染色体着丝粒附近遗传距离为1.4 cM的范围内。这些粒型主效QTL定位加速推进了水稻籽粒性状调控基因的挖掘和高产分子育种的进程。目前GL7、srs-3、GS3、GL3.1、GL4、GS5、GW2、qSW5、GW5和Ghd7[14-23]等多个水稻粒型相关主效QTL已被成功克隆,gw3.1、qGL7、qGL7-2、TGW3b和gw9.1[24-28]等也已完成精细定位。但是,由于构建图谱所使用的群体不同,亲本材料遗传背景存在差异,在基因组覆盖面、标记疏密程度和具体标记类型等方面均有所不同,其QTL定位结果往往难以直接比较,不利于进一步开展QTL精细定位和基因克隆,这也限制了水稻粒型主效QTL快速、全面的克隆与挖掘。
本研究利用超级稻春优84的两个在遗传背景上存在较大差异的亲本晚粳稻品种春江16B(CJ16B)和广亲和中籼恢复系C84作为群体杂交亲本,杂交并连续自交7代构建衍生的重组自交系作为遗传作图群体,筛选含有158个SSR和STS分子标记构建遗传图谱。通过对水稻粒型相关性状进行QTL定位、分析和验证,进一步挖掘水稻重要农艺性状的调控基因,完善水稻产量建成调控网络的分子机制,为水稻新种质资源的筛选鉴定和高产分子设计育种利用奠定材料基础、提供理论依据。
1 材料与方法
1.1 群体材料及遗传图谱构建
以籼稻恢复系品种C84为父本、粳稻品种春江16B为母本杂交获得F1,2013年夏从F1代单株上收取自交种,采用单粒传法连续自交7代,获得C84/春江16B重组自交系群体(RIL),从中选取表型稳定的188个株系用于遗传图谱的构建。选取在C84和春江16B间呈多态性,并均匀分布到12条染色体上的69对SSR(simple sequence repeats)和89对STS(sequence tagged site)标记,对188个重组自交系的基因型进行分析鉴定,构建遗传连锁图谱。
1.2 材料种植及农艺性状调查
亲本C84和春江16B及RIL群体,分别于2016年夏和2016年冬,种植在浙江省杭州市中国水稻研究所富阳试验基地和海南省陵水市中国水稻研究所南繁基地,行株距为20 cm×20 cm,每株系种植4行,每行6株。按照常规大田管理方法进行管理,并对病虫草害进行防治。
成熟期对亲本C84和春江16B及其188个重组自交系的粒型性状进行鉴定。挑选灌浆饱满的籽粒,利用考种仪(万深SC-G型,杭州万深检测科技有限公司)测定粒长、粒宽、千粒重等,每株系3次重复,取平均值[29]。粒厚用游标卡尺测量,每株系8次重复,取平均值。
1.3 遗传图谱构建与QTL分析
用MapDraw 2.1进行标记间的连锁分析,绘制遗传连锁图谱[30]。通过SAS 9.2对双亲和重组自交系群体的粒型性状进行统计,利用QTLNetwork 2.1进行QTL计算与分析。以P=0.005为阈值统计该标记处是否存在与性状有关的QTL,采用Kosambi函数将重组值转化成遗传距离。QTL的命名参照McCouch等[31]的命名方式。
1.4 双亲基因克隆序列差异分析及dCAPs分子标记差异位点验证
对双亲QTL区间内他人已克隆的6个粒型主效QTL等位基因进行测序,分析其序列差异,根据编码区cDNA差异位点设计dCAP标记验证。
2 结果与分析
2.1 重组自交系亲本C84与春江16B粒型比较
对重组自交系亲本C84和春江16B的粒型性状在两个环境条件下进行比较分析,发现籽粒长度、宽度、厚度、长宽比和千粒重等在杭州和陵水两个环境下都存在一定程度的差异,但各性状变化趋势和差异程度不尽相同(表1)。C84和春江16B的粒长在杭州分别为8.2 mm和7.4 mm,在陵水为7.8 mm和7.0 mm,两者均相差0.8 mm,群体亲本C84和春江16B粒长在陵水相对杭州略有下降,均减少0.40 mm,降幅分别为4.87%和5.41%,群体株系平均粒长下降了0.18 mm。粒宽的变化趋势则不一样,粳稻亲本春江16B在两地没有差异,均为3.2 mm,而籼稻亲本C84的粒宽则陵水比杭州增加0.2 mm,增幅达6.89%,群体株系平均粒宽增加0.11 mm,差异显著。双亲籽粒厚度差异极显著,不同环境变化不大,粒长、粒宽的改变直接影响籽粒长宽比,籼稻亲本C84、粳稻亲本春江16B和群体株系的长宽比平均值陵水比杭州分别下降了10.71%、4.35%和5.95%。粒型改变往往导致千粒重发生变化,虽然两亲本在杭州和陵水的千粒重并没有显著的差异,但是群体株系的千粒重显著增加,由杭州的22.17 g增加到陵水的22.98 g,增幅达3.65%。结果显示,双亲在籽粒形态上均存在显著差异,且受环境影响不大,因此,构建的重组自交系群体可以用于粒型相关性状的QTL定位。
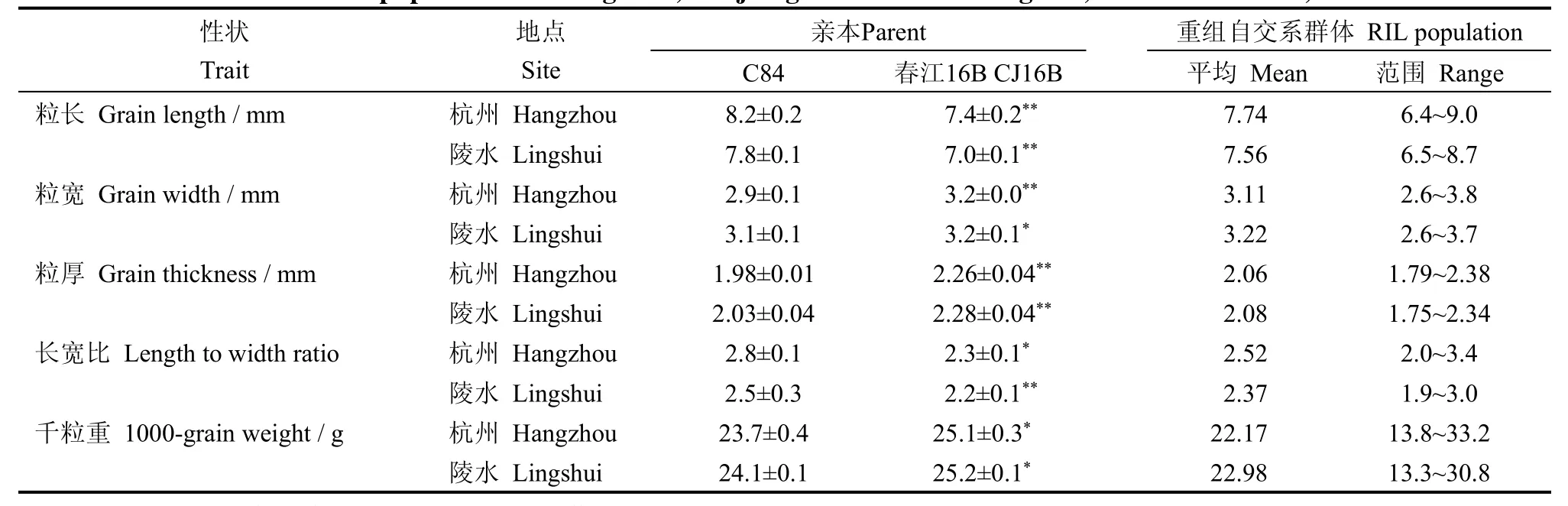
表1 双亲(C84和春江16B)及其RIL群体2016年在杭州和陵水的粒长、粒宽、粒厚、长宽比及千粒重Table 1.Grain length,width,thickness,length-to-width ratio and 1000-grain weight in C84 and CJ16B and their recombinant inbred line population in Hangzhou,Zhejiang Province and Lingshui,Hainan Province,China in 2016.
2.2 重组自交系群体粒型性状分布及相关性分析
分别对在杭州、陵水两个环境下的双亲及群体粒型性状进行统计。结果表明,无论是在杭州还是陵水,群体中不同株系粒长、粒宽、粒厚、长宽比及千粒重测定值均表现为连续分布,且有一定数量超亲株系存在,基本符合正态分布(图1)。说明粒型为多基因控制的数量性状,符合QTL定位的要求。

图1 C84/CJ16B重组自交系群体粒型相关性状分布Fig.1.Frequency distributions of grain shape traits in C84/CJ16B recombinant inbred lines.

表2 C84/CJ16B重组自交系群体粒型性状间相关性分析Table 2.Correlationship analysis on grain shape traits in C84/CJ16B recombinant inbred lines.
对C84/春江16B重组自交系群体粒型性状间的相关性进行分析,结果表明同一性状在不同的环境中呈极显著正相关(表2)。杭州与陵水的群体株系的粒长、粒宽和籽粒长宽比等性状均呈极显著的正相关,其相关系数分别为0.809、0.817和0.854;但粒厚及千粒重相对其他粒型性状在两地间的相关性较低,相关系数仅为0.270和0.529。推测这可能与双亲的遗传背景、群体大小以及杭州、陵水特定环境中的光照条件存在一定的关系,同时,群体中不同株系对环境敏感性差异可能会影响籽粒的充实度,造成粒厚及千粒重较大程度分离所致。
2.3 遗传图谱的构建
利用均匀分布在水稻12条染色体上的158个SSR和STS多态标记对RIL群体的188个株系的基因型进行鉴定,构建了总长度为1428.4 cM的遗传连锁图谱,平均每条染色体覆盖长度为119.03 cM,相邻分子标记间的平均距离为9.04 cM,平均每条染色体上的标记数13.2个(表3)。第1染色体及第4染色体上分子标记相对较多,分别达17和18个,第9和第10染色体上分子标记相对较少,分别只有10个和9个。第1染色体平均遗传距离最大,达11.34 cM,第5染色体平均遗传距离最小,为7.13 cM。所有标记在染色体上分布较为均匀,但也存在一些间隔较大的区域(如第3染色体的H3-3-RM1479之间和第12染色体的RM1337-RM3739之间)和标记较为密集的区域(如第2染色体的H2-3-RM262之间,第4染色体的H4-2-H4-8之间和第5染色体的H5-1-H5-6之间)。整体来看,距离适中,构建的遗传连锁图谱适合进行水稻相关农艺性状的QTL定位。

表3 本研究所用的标记在水稻12条染色体上分布Table 3.Distribution of the markers used in the study on 12 chromosomes of rice.
2.4 粒型性状的QTL定位
利用构建的遗传连锁图谱,结合RIL群体188个株系及其亲本的粒长、粒宽、粒厚、籽粒长宽比、千粒重等5个粒型性状在杭州、陵水的表型值,采用复合区间作图法,对籽粒形态进行了QTL分析,共检测到30个粒型相关QTL(表4、图2)。其中,粒长QTL 9个,粒宽QTL 5个,粒厚QTL 5个,长宽比QTL 6个和千粒重QTL 5个,单位点贡献率3.51%~17.25%。
共检测到9个粒长QTL,贡献率介于5.75%~14.40%,亲本C84及春江16B均有增加粒长的增效等位基因,但来自C84的增效等位基因数目比来自春江16B的要多,这与亲本C84的籽粒长度大于春江16B相符。其中qGL10在两地均能被检测到,位于第10染色体上RM6370-RM5689区间内,杭州和陵水的贡献率分别为11.97%和10.33%,其增效等位基因均来源于亲本C84,该区间已有粒长调控基因OsPCR1[32]被克隆。qGL1、qGL2.1和qGL2.2,qGL5、qGL11贡献率均小于10%,其中,qGL2.1区间内已有粒长调控基因FUWA[33]被克隆,qGL5相应区间内已有粒长调控基因JMJ703[34],SRS3/OsKinesin-13A/sar1[35]被克隆。qGL7.1、qGL7.2及qGL12分别解释粒长10.09%、14.40%、11.04%的遗传变异。
共检测到5个粒宽QTL,除qGW1增效等位基因来源于亲本C84之外,其余增效等位基因均来源于亲本春江16B,且加性效应值较大。qGW2.1和qGW2.2分别在两地被检测到,区间位置相邻,贡献率分别为9.58%和9.34%。粒宽QTL中贡献率最高的是qGW5,其在两年的试验中被重复检测到,位于RM17954-H5-7标记区间内,贡献率分别为12.28%和11.44%,该区间内已有粒宽调控基因qGW5[36]被克隆。qGW1和qGW6贡献率分别为3.51%和7.06%,尚未见报道,可能是新的QTL位点。
共检测到5个粒厚QTL。与粒宽相同,除qGT1增效等位基因来源于籼稻亲本C84之外,其余增效等位基因均来源于母本春江16B。其中,位于第1染色体上RM8097-RM6703区间的qGT1在两地均能被检测到,杭州和陵水的贡献率分别为12.75%和12.59%,该区间与千粒重QTLqTGW1.2重叠,可能受同一基因控制,说明千粒重与粒厚关系密切。qGT2.2定位于第2染色体H2-5-H2-6区间内,解释9.51%遗传变异,该区间同时定位到qGL2.2及qGLW2.2。qGT2.1、qGT6和qGT9贡献率均小于10%,至今均尚未见报道,可能是影响籽粒形态发育相关的新QTL调控位点。

表4 RILs的粒型相关性状的QTL定位结果Table 4.QTLs for grain shape-related traits of recombinant inbred lines.
在杭州和陵水分别检测到了2个和5个控制籽粒长宽比的QTL。其中,qGLW5在杭州和陵水两地均能被检测到,贡献率分别为17.25%及12.75%。qGLW2.1、qGLW2.2及qGLW2增效等位基因均来源于父本C84,位置相邻,分别解释籽粒长宽比13.88%、15.61%、12.95%的遗传变异。qGLW11及qGLW12贡献率相对较低,分别为7.79%和6.25%,增效等位基因来源于母本春江16B。
共检测到5个控制千粒重的QTL。位于第5染色体上qTGW5,加性效应值及贡献率最高,贡献率达10.55%,其增效等位基因来源于亲本C84,能增加千粒重1.2472 g。qTGW1.1和qTGW1.2分别在杭州及陵水两地被检测到,位置相近,加性效应及贡献率也基本一致,其增效等位基因均来源于C84,qTGW1.1区间内已有千粒重相关调控基因OsTRBF3[36]被克隆。加性效应及贡献率最低的qTGW9,贡献率仅为4.55%,增效等位基因来源于C84,能增加千粒重0.8719 g。qTGW4加性效应值为1.1454 g,贡献率达7.41%。

图2 水稻粒型相关性状QTL在染色体上的分布Fig.2.Distribution of QTLs related to grain shape traits in rice chromosome.
利用杭州和陵水两个群体检测到的QTL,部分与已报道的QTL相同或相近,部分为首次报道。由于数量性状的表现受基因、环境及其互作的影响,同一性状在不同的定位群体中表现不一致,且同一群体在不同年份、不同环境中的表现结果也会有所不同。
2.5 双亲已克隆粒型主效QTL等位基因序列差异分析及分子标记验证
为了进一步验证利用春江16B和C84构建的RIL连锁遗传图谱进行QTL定位的可靠性及挖掘重要粒型调控新基因的可能性,我们对定位获得的粒形主效QTL区间,比较已克隆基因在双亲中的等位基因序列差异。本研究检测到的粒型主效QTL区间内共有6个已知克隆基因,第1染色体上的千粒重调控基因OsTRBF3已被克隆基因,第2染色体上的粒长调控基因FUWA已被克隆,第5染色体上共有3个已知被克隆基因,分别是粒长调控基因SRS3/OsKinesin-13A/sar1,粒宽调控基因qGW5和JMJ703(既调控粒长也调控粒宽性状),第10染色体上粒长调控基因OsPCR1为已被克隆基因。分别对双亲已知基因进行克隆测序。表5列出了相关基因外显子编码区序列差异,可见除JMJ703基因在外显子编码区无差异之外,双亲在另外5个基因多个位点都表现出碱基差异,相应位点氨基酸也不同。有两个位点还由于碱基缺失导致编码区翻译移码,基因功能发生变化。我们又根据双亲目标基因测序的差异位点设计dCAPs标记进行验证,每个目标基因选取一个差异位点,设计分子标记(表6),酶切验证,发现上述基因对应碱基位点的确存在碱基差异(图 3)。
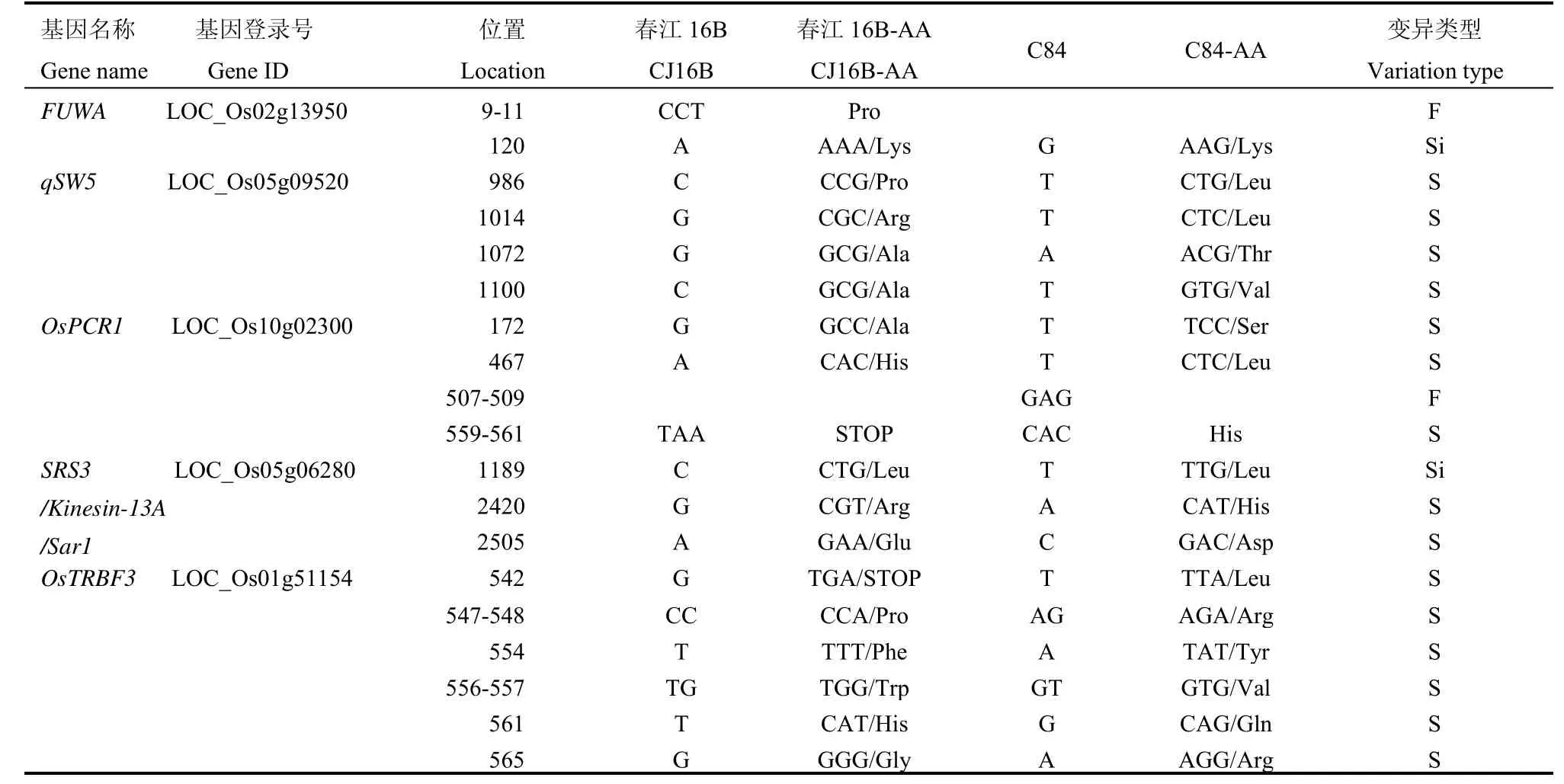
表5 双亲(C84和春江16B)已克隆粒型主效QTL等位基因序列差异分析Table 5.Sequence variance analysis of the cloned grain shape main effect QTL allele in the two parents(C84 and CJ16B).
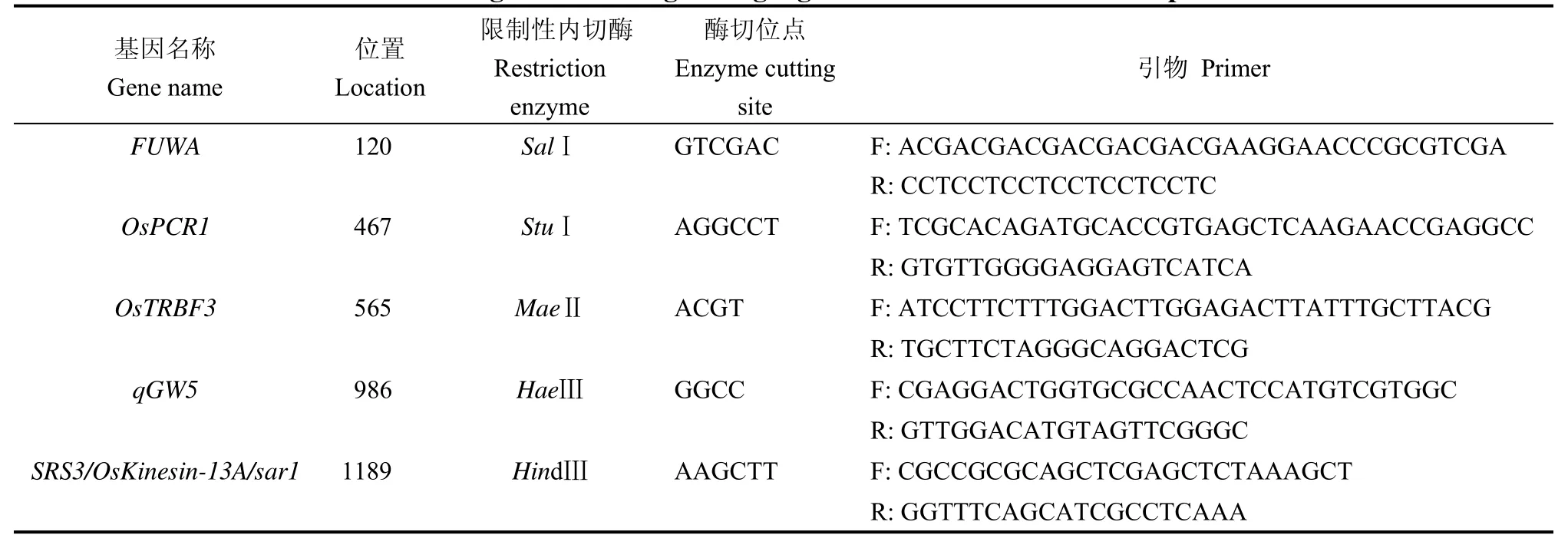
表6 双亲目标基因差异位点dCAPs分子标记引物设计Table 6.dCAPs molecular marker designed according to target gene difference site of the two parents C84 and CJ16B.

图3 差异位点设计dCAPs分子标记验证结果Fig.3.Results of dCAPs markers in difference site verifying.
3 讨论
水稻籽粒形态是多途径、多基因调控的复杂遗传性状的综合体现。本研究通过对杭州和陵水两个环境的籽粒性状比对,同一性状在不同环境之间具有较高的相关性,说明粒型性状总体受环境影响较小,具较高的遗传力,但不同性状受环境影响存在差异。从表型上看,粒长相对粒宽、粒厚、千粒重更容易受环境的影响。究其原因可能主要是由群体亲本籼粳亚种遗传背景差异、主效QTL/基因的等位变异以及微效QTL与环境互作协同调控的结果。
我们利用C84/春江16B重组自交系群体共定位到的30个与粒型性状相关QTL,有5个QTL区间覆盖了6个已克隆的主效粒型QTL/基因,其中qGL10、qGW5、qGT1和qGL/GW5在两地中能同时被检测到,贡献率均稳定在10%以上,分别覆盖已克隆粒型基因OsPCR1、qGW5和JMJ703(JMJ703同时调控籽粒的长度与宽度的发育)。其中qGL2.1、qGL7.1、qGW2.1、qGT2.1和qGL/GW2.2是在杭州群体中被检测到,qGL2.2、qGL7.2、qGW2.2、qGT2.2和qGLW2是在陵水群体中被检测到的,同一染色体上相应QTL两个标记位置相邻、加性效应方向一致、贡献率也基本相同,我们推测可能是受同一个QTL调控,由于环境效应导致定位发生偏离。另外,我们发现RM17954-H5-7区间内同时存在粒长、粒宽、长宽比和千粒重的QTL,说明同一区间中可能存在多个影响籽粒形态发育的调控基因,或者存在“一因多效”的可能性。
利用不同材料构建遗传群体定位的QTL结果往往存在一定的差异。我们定位到的9个调控粒长QTL的区间中,在3个区间内已有粒型调控基因克隆,分别为第2染色体上的FUWA,第5染色体上的JMJ703、SRS3/OsKinesin-13A/sar1和第10染色体上的OsPCR1。FUWA在禾本科作物中进化保守,通过限制细胞周期过程,能使籽粒变短、变宽、变厚[33]。JMJ703是水稻中一种活性H3K4特异性去甲基酶,参与控制转座子的活性,突变体种子长、宽以及厚度都下降[37]。SRS3在发育的器官中有高表达,影响细胞纵向长度,调控水稻种子长度[38];OsKinesin-13A通过影响颖壳大小,进而调控水稻颖果大小;sar1突变体的籽粒长度以及其他器官如节间、叶片以及根部等均变短[35]。OsPCR1主要在水稻幼苗期的根中以及生殖期的第I和II节间表达,与野生型相比,OsPCR1敲除株系籽粒变轻,粒长变短,粒重下降12%[32]。上述已知克隆基因对水稻粒型均有显著的调控作用。另有4个粒长QTLqGL-1[39]、qGL2-2[40]、qGL-11[39]和qGL-12[40]等与已报道的水稻粒型基因/QTL区间有重叠;同样,5个粒宽QTL定位区间中,除了qGW1区段内有1个基因(qGW5/JMJ703)被克隆外,qGW1、qGW6也有重叠区间的粒宽QTL报道[41,42];而目前已挖掘的水稻粒厚QTL数量相对较少,在定位区间内未发现与已知粒厚QTL位置重叠。在所有检测到的千粒重QTL中,除qTGW1.1为检测到的新位点外,其余4个区间存在重叠区段的位点qTGW1-2/qTGW1-3[43]/Kw1-2[44]/tgwt1[45]、qGW-1[46]、gw4[47]、qTGWT-5[48]/gw5b/Gwt5a[49,50]等也已先后被报道。在该区间内已有编码单MYB组蛋白家族的端粒重复序列结合因子基因OsTRBF3被克隆,它影响籽粒千粒重[36]。说明千粒重是一个籽粒形态与稻米胚乳发育相关的籽粒综合性状指标,除了受环境和遗传因素影响外,粒长、粒宽、粒厚等形态性状发育与水稻千粒重也密切相关。另外,在我们的定位结果中,除了以上多个已知的粒型相关主效QTL相同或相邻外,还发现了多个新的粒型相关QTL调控位点,其中,粒长4个、粒宽3个、粒厚5个及千粒重1个。由此,我们推测不同群体间同一性状主效QTL定位结果的异同与构建群体的双亲间亲缘关系远近、形态性状多态性、差异等位基因的多寡以及连锁图谱的标记数目存在密切关系。水稻粒型QTL的定位与克隆为发展分子选择标记、聚合有利基因及水稻超高产分子设计育种顺利实施奠定理论基础和提供基因新资源。
同时,我们结合C84、春江16B对定位区间内的5个已知粒型等位基因序列进行分析并根据差异位点开展分子标记验证,证实双亲目标等位基因间确实存在编码氨基酸的差异,我们推测该差异等位基因即为该QTL定位区间内具粒型调控作用的目标基因。说明利用C84/春江16B构建的重组高代自交系群体可以成功的用于水稻粒型性状或者其他重要农艺性状的QTL的定位与挖掘。后期通过双亲全基因组SNPs扫描,加密区间内的标记数目,从而饱和该区段的内的标记数,以提高不同性状QTL检测准确性,加速目标基因的精细定位和图位克隆。
[1]Tian Z X,Qian Q,Liu Q Q,Yan M X,Liu X F,Yan C J,Liu G F,Gao Z Y,Tang S Z,Zeng D L,Wang Y H,Yu J M,Li J Y.Allelic diversities in rice starch biosynthesis lead to a diverse array of rice eating and cooking qualities.Proc Natl Acad Sci USA,2009,106(51):21760-21765.
[2]李孝琼,韦宇,高国庆,邓国富,郭嗣斌.水稻遗传图谱构建及粒形相关性状的QTL定位.南方农业学报,2014,45(7):1154-1159.Li X Q,Wei Y,Gao G Q,Deng G F,Guo C B.Construction of genetic map and mapping quantitative trait loci for grain shape-related traits in rice.J South Agric,2014,45(7):1154-1159.(in Chinese with English abstract)
[3]McCouch S R,Teytelman L,Xu Y,Lobos K B,Clare K,Walton M,Fu B,Maghirang R,Li Z,Xing Y,Zhang Q,Kono L,Yano M,Fjellstrom R,DeClerck G,Schneider D,Cartinhour S,Ware D,Stein L.Development and mapping of 2240 new SSR markers for rice(Oryza sativaL.).DNA Res,2002,9(6):257-279.
[4]Xu J C,Zhu L,Chen Y,Lu C F,Cai H W.Construction of a rice molecular linkage map using a double hploid(DH)population.J Gen Genom,1994,21(3):205-214.
[5]张启军,叶少平,虞德容,李平,吕川根,邹江石.六张水稻遗传连锁图谱的比较分析.西南农业学报,2005,18(5):584-592.Zhang Q J,Ye S P,Yu D R,Li P,Lu C G,Zou J S.Comparison of six rice genetic linkage maps.Southwest China J Agric Sci,2005,18(5):584-592.(in Chinese with English abstract)
[6]庄杰云,施勇烽,应杰政,鄂志国,曾瑞珍,陈洁,朱智伟.中国主栽水稻品种微卫星标记数据库的初步构建.中国水稻科学,2006,20(5):460-468.Zhuang J Y,Shi Y F,Ying J Z,Dong Z G,Zeng R Z,Chen J,Zhu Z W.Construction and testing of primary microsatellite database of major rice varieties in China.Chin J Rice Sci,2006,20(5):460-468.(in Chinese with English abstract)
[7]Xu J J,Zhao Q,Du P N,Xu C W,Liu Q Q,Feng Q,Wang B H,Tang S Z,Gu M H,Han B,Liang G H.Developing high throughput genotyped chromosome segment substitution lines based on population whole-genome re-sequencing in rice(Oryza sativaL.).BMC Genomics,2010,11(1):656.
[8]沈利爽,何平,徐云碧,谭震波,陆朝福,朱立煌.水稻DH群体的分子连锁图谱及基因组分析.植物生态学报,1998,40(12):1115-1122.Shen L S,He P,Xu Y B,Tan Z B,Lu C F,Zhu L H.Genetic molecular linkage map construction and genome analysis of rice doubled.Plant Acta Ecol Sin,1998,40(12):1115-1122.(in Chinese with English abstract)
[9]Huang R Y,Jiang L G,Zheng J S,Wang T S,Huang Y M,Wang H C,Hong Z L.Genetic bases of rice grain shape:So many genes,so little known.Trends Plant Sci,2013,18(4):218-226.
[10]方先文,张云辉,肖西林,张所兵,林静,汪迎节.基于重组自交系群体的水稻粒形QTL定位.江苏农业学报,2017,33(2):241-247.Fang X W,Zhang Y H,Xiao X L,Zhang S B,Lin J,Wang Y J.Mapping of QTLs for grain shape using recombinant inbred lines in rice(Oryza sativaL.).Acta Agric Jiangsu,2017,33(2):241-247.(in Chinese with English abstract)
[11]Xia D,Zhou H,Qiu L,Jiang H,Zhang Q L,Gao G J,He Y Q.Mapping and verification of grain shape QTLs based on an advanced backcross population in rice.PloS One,2017,12(11):e0187553.
[12]Zhao D,Li P B,Wang L Q,Sun L,Xia D,Luo L J,Gao G J,He Y Q,Zhang Q L.Genetic dissection of large grain shape in rice cultivar‘Nanyangzhan’and validation of a grain thickness QTL(qGT3.1)and a grain length QTL(qGL3.4).Mol Breed,2017,37(3):42.
[13]Zhou L Q,Wang Y P,Li S G.Genetic analysis and physical mapping ofLk-4(t),a major gene controlling grain length in rice,with a BC2F2 population.Acta Gen Sin,2006,33(1):72-79.
[14]Wang Y X,Xiong G S,Hu J,Jiang L,Yu H,Fang Y X,Xu E B,Ye W J,Liu R F,Jing Y H,Zhu X D,Qian Q,Jing Y H,Xu J,Zeng L J,Xu J,Meng X B,Chen H Q,Wang Y H,Li J Y.Copy number variation at theGL7locus contributes to grain size diversity in rice.Nat Genet,2015,47(8):944-948.
[15]Wu T,Shen Y Y,Zheng M,Chen Y L,Feng Z M,Liu X,Chen Z J,Wang J L,Wan J M,Yang C Y,Liu S J,Lei C L,Jiang L.GeneSGL,encoding a kinesin-like protein with transactivation activity,is involved in grain length and plant height in rice.Plant Cell Rep,2014,33(2):235-244.
[16]Takano-Kai N,Doi K,Yoshimura A.GS3participates in stigma exsertion as well as seed length in rice.Breed Sci,2011,61(3):244-250.
[17]Qi P,Lin Y S,Song X J,Shen J B,Huang W,Shan J X,Zhu M Z,Jiang L W,Gao J P,Lin H X.The novel quantitative trait locusGL3.1controls rice grain size and yield by regulating Cyclin-T1;3.Cell Res,2012,22(12):1666-1680.
[18]Wu W,Liu X,Wang M,Meyer R S,Luo X,Ndjiondjop M N,Tan L,Zhang J,Wu J,Cai H,Sun C,Wang X,Wing R A,Zhu Z.A single-nucleotide polymorphism causes smaller grain size and loss of seed shattering during African rice domestication.Nat Plants,2017,3:17064.
[19]Li Y B,Fan C C,Xing Y Z,Jiang Y H,Luo L J,Sun L,Shao D,Xu C J,Li X H,Xiao J X,He Y Q,Zhang Q F.Natural variation inGS5plays an important role in regulating grain size and yield in rice.Nat Genet,2011,43(12):1266-1292.
[20]Song X J,Huang W,Shi M,Zhu M Z,Lin H X.A QTL for rice grain width and weight encodes a previously unknown RING-type E3 ubiquitin ligase.Nat Genet,2007,39(5):623-630.
[21]Yan S,Zou G H,Li S J,Wang H,Liu H Q,Zhai G W,Guo P,Song H M,Yan C J,Tao Y Z.Seed size is determined by the combinations of the genes controlling different seed characteristics in rice.Theor Appl Genet,2011,123(7):1173-1181.
[22]Liu J F,Chen J,Zheng X M,Wu F Q,Lin Q B,Heng Y Q,Tian Peng,Cheng Z J,Yu X W,Zhou K N,Zhang X,Guo X P,Wang J L,Wang H Y,Wan J M.GW5acts in the brassinosteroid signalling pathway to regulate grain width and weight in rice.Nat Plants,2017,3:17043.
[23]Xue W Y,Xing Y Z,Weng X Y,Zhao Y,Tang W J,Zhou H J,Yu S B,Xu C B,Li X H,Zhang Q F.Natural variation inGhd7is an important regulator of heading date and yield potential in rice.Nat Genet,2009,40(6):761-767.
[24]Li J,Thomson M,McCouch S R.Fine mapping of a grain-weight quantitative trait locus in the pericentromeric region of rice chromosome 3.Genetics,2004,168(4):2187.
[25]Bai X F,Luo L J,Yan W H,Mallikarjuna R K,Zhan W,Xing Y Z.Genetic dissection of rice grain shape using a recombinant inbred line population derived from two contrasting parents and fine mapping a pleiotropic quantitative trait locusqGL7.BMC Genet,2010,11(1):16.
[26]Shao G N,Tang S Q,Luo J,Jiao G A,Wei X J,Tang A,Wu J L,Zhuang J Y,Hu P S.Mapping ofqGL7-2,a grain length QTL on chromosome 7 of rice.J Gen Genom,2010,37(8):523-531.
[27]Liu T M,Shao D,Kovi M R,Zhong X Y.Mapping and validation of quantitative trait loci for spikelets per panicle and 1,000-grain weight in rice(Oryza sativaL.).Theor Appl Genet,2010,120(5):933-942.
[28]Xie X,Jin F,Song M H,Suh J P,Hwang H G.Fine mapping of a yield-enhancing QTL cluster associated with transgressive variation in anOryza sativa×O.rufipogoncross.TheorApplGenet,2008,116(5):613-622.
[29]陈建华,姚青,谢绍军,孙成效,朱智伟.机器视觉在稻米粒型检测中的应用.中国水稻科学,2007,21(6):669-672.Chen J H,Yao Q,Xie S J,Sun C X,Zhu Z W.Detection of rice shape based on machine vision.Chin J Rice Sci,2007,21(6):669-672.(in Chinese with English abstract)
[30]刘仁虎,孟金陵.MapDraw在Excel中绘制遗传连锁图的宏.遗传,2003,25(3):317-321.Liu R H,Meng J L.MapDraw:A micro for drawing genetic linkage maps based on given genetic linkage data.Hereditas,2003,25(3):317-321.(in Chinese with English abstract)
[31]McCouch S R.Gene nomenclature system for rice.Rice,2008,1(1):72-84.
[32]Song W Y,Lee H S,Jin S R,Ko D,Martinoia E,Lee Y.RicePCR1influences grain weight and Zn accumulation in grains.PlantCell&Environ,2015,38(11):2327-2339.
[33]Chen J,Gao H,Zheng X M,Jin M,Weng J F,Ma J.An evolutionarily conserved gene,FUWA,plays a role in determining panicle architecture,grain shape and grain weight in rice.Plant J,2015,83(3):427-438.
[34]Liu X Y,Zhou S L,Wang W T,Ye Y R,Zhao Y,Xu Q T,Zhou C,Tan F,Cheng S F,Zhou D X.Regulation of histone methylation and reprogramming of gene expression in the rice inflorescence meristem.Plant Cell,2015,27(5):1428-1444.
[35]Zhu Y D,Ling T L,Tang L,Song Y,Bai J K,Shan J H,ChangJY,TaiW.OsKinesin-13Aisanactive microtubule depolymerase involved in glume length regulation via affecting cell elongation.Sci Rep,2015,5(10):9457.
[36]Byun M Y,Hong J P,Kim W T.Identification and characterization of three telomere repeat-binding factors in rice.Biochem Biophy Res Comm,2008,372(1):85-90.[37]Cui X K,Jin P,Cui X,Gu L F,Lu Z K,Xue Y M,Wei L Y,Qi J F,Song X W,Luo M,An G,Cao X F.Control of transposon activity by a histone H3K4 demethylase in rice.Proc Natl Acad Sci USA,2013,110(5):1953-1958.
[38]Kitagawa K,Kurinami S,Oki K,Abe Y,Ando T,Kono I,Yano M,Kitano H,Iwasaki Y.A novel Kinesin 13 protein regulating rice seed length.Plant&Cell Physiol,2010,51(8):1315-1329.
[39]Yoshida S,Ikegami M,Kuze J,Sawada K,Nashimoto Z,Nakamura C,Ishii T,Kamijima O.QTL analysis for plant and grain characters of sake-brewing rice using a doubled haploid population.Breed Sci,2002,52(4):309-317.
[40]Li Z F,Wan J M,Xia J F,Zhai H Q.Mapping quantitative trait loci underlying appearance quality of rice grains(Oryza sativaL.).J Gen Genom,2003,30(3):251-259.
[41]Huang N,Parco A,Mew T,Magpantay G,McCouch S,Guiderdoni E,Xu J,Subudhi P,Angeles E R,Khush G S.RFLP mapping of isozymes,RAPD and QTLs for grain shape,brown planthopper resistance in a doubled haploid rice population.Mol Breed,1997,3(2):105-113.
[42]Wan X Y,Wan J M,Weng J F,Jiang L,Bi J C,Wang C M,Zhai H Q.Stability of QTLs for rice grain dimension and endosperm chalkiness characteristics across eight environments.Theor Appl Genet,2005,110(7):1334.
[43]Hittalmani S,Huang N,Courtois B,Venuprassad R,Shashidhar H E,Zhuang J Y,Zheng K L,Liu G F,Wang G C,Sidhu J S,Srivantaneeyakul S,Sinqh V P,Baqali P G,Prasanna H C,McLaren G,Khush G S.Identification of QTL for growth-and grain yield-related traits in rice across nine locations of Asia.Theor Appl Genet,2003,107(4):679-690.
[44]Gao Y M,Zhu J,Song Y S,He C X,Shi C H,Xing Y Z.Analysis of digenic epistatic effects and QE interaction effects QTL controlling grain weight in rice.J Zhejiang Univ Sci,2004,5(4):371-377.
[45]Lin H X,Qian H R,Zhuang J Y,Lu J,Min S K,Xiong Z M,Huang N,Zheng K L.RFLP mapping of QTLs for yield and related characters in rice(Oryza sativaL.).Theor Appl Genet,1996,92(8):920-927.
[46]Cho Y C,Suh J P,Choi I S,Hong H C,Baek M K,Hong H C,Kim Y G,Choi H,Moon H P,Hwang G H.QTLs analysis of yield and its related traits in wild rice relativeOryza rufipogon.Treat Crop Res,2003(4):19-29.
[47]Hua J,Xing Y,Wu W,Xu C,Sun X,Yu S,Zhang Q.Single-locus heterotic effects and dominance by dominance interactions can adequately explain the genetic basis of heterosis in an elite rice hybrid.Proc Natl Acad Sci USA,2003,100(5):2574.
[48]Zhuang J Y,Fan Y Y,Rao Z M,Wu J L,Xia Y W,Zheng K L.Analysis on additive effects and additiveby-additive epistatic effects of QTLs for yield traits in a recombinant inbred line population of rice.Theor Appl Genet,2002,105(8):1137-1145.
[49]Hua J P,Xing Y Z,Xu C G,Sun X L,Yu S B,Zhang Q.Genetic dissection of an elite rice hybrid revealed that heterozygotes are not always advantageous for performance.Genetics,2003,162(4):1885-1895.
[50]谭震波,沈利爽,陆朝福,陈英,朱立煌,周开达,袁祚廉.水稻再生能力和头季稻产量性状的QTL定位及其遗传效应分析.作物学报,1997,23(3):289-295.Tan Z B,Shen L S,Yuan Z L,Lu C F,Chen Y,Zhou K D,Zhu L H,Zuo W X B.Identification of QTLs for ratooning ability and grain yield traits of rice and analysis of their genetic effects.Acta Agron Sin,1997,23(3):289-295.(in Chinese with English abstract)
