小麦抗病分子标记数据库构建及生物信息学分析
2018-05-18王长彪赵兴华任永康牛瑜琦唐朝晖
赵 霞 ,王长彪 ,赵兴华 ,刘 江 ,韩 斌 ,任永康 ,牛瑜琦 ,唐朝晖
(1.山西大学生物工程学院,山西太原030006;2.山西省农业科学院生物技术研究中心,山西太原030031;3.山西省农业科学院作物科学研究所,山西太原030031)
小麦(Triticum aestivumL.)是世界上重要的粮食作物之一[1]。近几年,小麦病害发生严重,如白粉病、各种锈病等,抗病育种一直是育种家们的研究内容,因此,培育高产优质的小麦新品种势在必行[2-3]。传统的育种方式费工费时,从分子水平改良种质资源的方式正一步步受到人们的重视[4]。近年来,分子标记技术[5]在小麦研究中取得重大进展[6]。
生物信息学综合应用生物学知识、数学模型及计算机运算,对核酸序列和蛋白质进行研究,旨在获得具有生物学意义的信息[7]。随着生物信息学技术的快速发展,基因的电子定位发展了起来[8],其是利用日益丰富的生物信息学资源,运用生物信息学方法,将目的基因定位到染色体实际位置上的一种技术,较传统的定位技术更简便、更快捷、成本更低[9]。分子生物学与生物信息学技术的结合为小麦的研究提供了有效的方法[10]。
本研究旨在寻找与小麦抗白粉病、抗锈病相关的SSR和STS标记,构建小麦抗病分子标记数据库,并将其定位在中国春小麦的全基因组数据库中,把抗病标记进行物理定位,为基因精细定位、克隆基因等方面奠定基础。然后对能够成功定位的基因进行有效的序列分析、功能预测、功能注释及KEGG分析等生物信息学研究,以期获得与小麦白粉菌、锈菌互作进程中的基因表达信息,为研究小麦病害的发病机理、发掘抗病相关新基因及抗病基因的精细定位和分子标记辅助育种奠定基础,对进一步培育抗病小麦品种具有重要的理论实践意义。
1 材料和方法
1.1 材料
供试的51份小麦材料中,41份由山西省农业科学院作物科学研究所提供,分别是运旱618、运旱 20410、石家庄 8 号、SY-9571、良星 99、良星 66、济麦 22、山农 20、百农 64、兰考 906、保丰 104、扬麦 21、中麦 247、皖麦 38、中麦 175、舜麦 1718、京冬 8 号、MY559、周麦 28、周麦 30、周麦 32、京冬17、京冬 18、京冬 24、长 4640、长 6135、长 6452、长6878、太 5902、太 10604、晋麦 66、晋麦 70、晋麦84、晋太 170、晋太 182、宁麦 5 号、石麦 15、济南17、洛旱2号、河农6425、中优9507;3份由山东农业大学提供,分别是 98M71,UC1041,Moro;7 份由西北农林科技大学提供,分别是先麦12、丰尤8号、百农 121、郑 132、郑 369、兰考 165、阜 0608。
从相关文献中查找小麦抗白粉病、抗锈病相关标记,共368个。
1.2 方法
小麦抗病基因定位:在Linux平台上,将368个抗病标记采用re-PCR方法将其定位到中国春小麦全基因组序列中,统计结果并利用MapChart2.1软件进行作图。
使用Fgenesh软件对能进行电子定位的序列进行功能基因预测。使用Blast2go软件对获得的功能基因进行基因的GO注释及KEGG的代谢通路分析。
从预测出的功能基因中随机选取159对引物进行真实PCR检测。采用CTAB法提取小麦幼苗叶片DNA,随后用紫外分光光度计检测其质量和浓度,再进行 PCR反应(10 μL 体系:1.5 μL 50 ng模板 DNA,1 μL 10×buffer,0.2 mol/L dNTP,0.12 U Taq聚合酶,0.75 μmol/L引物),反应程序为:94℃预变性5 min;94℃变性30 s,退火45 s(退火温度据引物温度而定,一般55~60℃),72℃延伸45 s,共35个循环。最后用8%非变性聚丙烯酰胺(PAGE)凝胶电泳对PCR扩增产物进行分离检测,上样2 μL并在220 V电压下运行1 h后,银染后统计结果,读带后利用NTSYS软件的UPGMA聚类分析法构建系统发育树,探究遗传规律。
2 结果与分析
2.1 电子定位结果
统计结果发现,共有247个与抗病相关的基因被定位到了相应染色体上,其中定位到2B染色体上的最多。同时发现,有的同一个标记定位在不同染色体上或同一个标记在同一条染色体的不同部位。具体对分子标记的电子定位效果进行统计,其中,在1A染色体上的分子标记定位率(定位率即成功进行电子定位后每条染色体上的总标记与未定位前每条染色体上的总标记的百分比)为60%,1B为 65.5%,1D为 72.7%,2A 为 57.9%,2B 为 64.3%,2D 为 82.1% ,3A 为 55.6% ,3B 为 66.7% ,3D 为12.5%,4A 为 84.6%,4B 为 80.0%,4D 为 38.5%,5A为 37.5%,5B 为 69.2%,5D为 89.5%,6A 为 91.7%,6B 为 40.0% ,6D 为 6.7% ,7A 为 50.0% ,7B 为51.7%,7D为90.9%。电子定位的结果表明,电子定位能够为抗病基因的克隆、表达和功能研究等提供重要的信息,也可为进一步研究小麦抗病机制等提供基础数据。
2.2 小麦基因功能预测
将可以成功进行定位的247个小麦抗病基因利用基因功能预测软件Fgenesh进行基因预测,共预测出108个功能基因。
2.3 小麦功能基因的序列分析
将所得的108个基因的核酸序列用Blast2go软件进行功能注释分类,其中,成功注释的有73个功能基因,未成功注释的有35个功能基因。共得到89条GO术语信息,其中,属于生物学途径P的有51条,细胞组件C的23条,分子功能F的有15条。GO注释分类的结果表明,在生物学途径中最多的是生物过程与细胞过程,其次就是各种代谢过程,如有机物代谢过程、细胞代谢过程、初级代谢过程、大分子代谢过程等;从细胞组成的角度来看,大多数功能基因都是在细胞、胞内、细胞器及细胞质中起作用的;在分子功能中,作用最多的是分子功能,其次是催化活性和结合功能,充分说明小麦细胞内正常活动离不开包括辅基结合、蛋白结合、辅酶结合、ATP结合、DNA结合和RNA结合等各种结合功能及酶的催化活性。
2.4 基于KEGG的代谢通路分析
运用KEGG数据库对获得的功能基因进行代谢通路分析,共有16个功能基因编码的13种酶涉及到11个生物化学途径。相关的酶有差向异构酶、硝基苯磷酸酯酶、转化酶、α-葡萄糖苷酶、二磷酸合酶、水解酶、磷酸酶、磷酸合酶。参与的生物化学途径有戊糖和葡萄糖醛酸相互转化、氨基苯甲酸盐降解、淀粉和蔗糖代谢、萜类骨架生物合成、脂肪酸伸长率、光合生物固碳、Th1和Th2细胞分化、T细胞受体信号通路、半乳糖代谢、磷酸戊糖途径、抗生素生物合成。本研究认为,在小麦的各种代谢途径中,主要以淀粉、蔗糖代谢为主。具体代谢分析结果列于表1。

表1 小麦功能基因基于KEGG的代谢通路分析
2.5 聚类分析
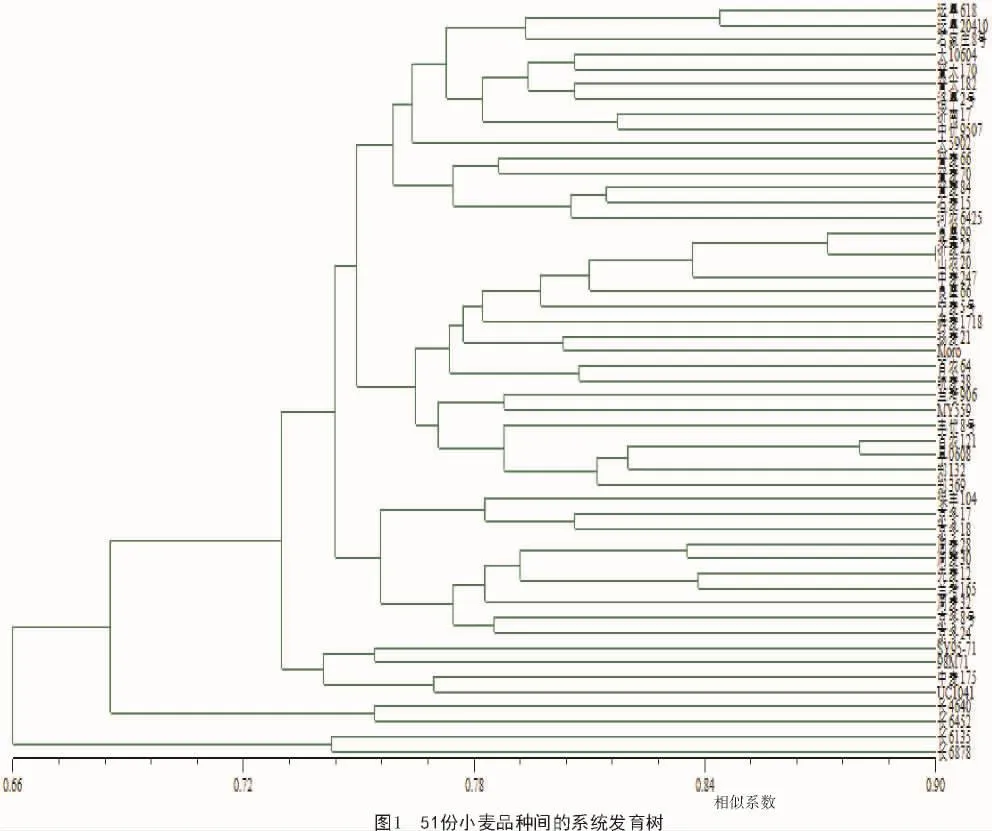
构建的系统发育树如图1所示,从结果可以看出,其遗传相似系数为 0.66~0.90,平均为 0.78。从图1可看出,济麦22和山农20亲缘关系最近。在相似系数为0.75时,可以把51份材料分为Ⅸ大类,其中,第Ⅰ类中运旱618与运旱20410是高感病品种,可以推测与其聚在一起的有可能是高感病品种;第Ⅲ类中大部分品种都是高抗品种[11-15];第Ⅳ类中除了MY559是高抗叶锈病品种外[16],其余都是来自西北农林科技大学的高抗条锈病与高抗白粉病的品种[17];第Ⅵ类中SY-9571是感病品种[11];第Ⅷ类与第Ⅸ类都是长麦系列。以上聚类信息为小麦抗病基因标记在小麦品种鉴定中的应用提供了理论依据,是小麦品种在分子水平上表现出的遗传多样性,为小麦多种品种共同抗病奠定了基础。
3 结论
种业是农业生产的源头,发展小麦种业是保障我国粮食安全的根本[18]。由于长期的人工选择和栽培,导致小麦大量优异基因丢失,导致当今小麦遗传基础狭窄、脆弱,易受病虫害的侵染,限制了小麦产量的提高[19]。本研究通过电子定位技术将368个小麦抗病标记定位到中国春小麦全基因组序列中[20],并利用Fgenesh,Blast2go软件以及KEGG等生物信息学手段对小麦抗病基因进行生物信息学分析,这些为小麦的抗病育种提供了一定的理论依据。随着分子生物学和生物信息学的发展,基因工程抗病育种和分子辅助抗病育种正在逐步变为现实,这些对加快和促进小麦抗病育种、培育小麦优良品种具有重要意义[21-22]。多基因聚合育种可以避免单一抗性品种抗性丧失带来的危险,且可以获得持久抗性,因此,在今后也可以通过多基因聚合来克服栽培品种抗性过快丧失这一难题[23]。
参考文献:
[1]李树超.中国小麦生产的发展趋势、制约因素及战略对策[J].中国农村经济,1998(8):41-44.
[2]韩斌,王长彪,任永康,等.小麦分子育种研究进展Ⅱ.与小麦品质相关的功能基因[J].山西农业科学,2015,43(2):233-236.
[3]PAUXE,SOURDILLE P,MACKAYI,et al.Sequence-based marker development in wheat:Advances and applications to breeding[J].Biotechnology Advances,2011,30(5):1071-1088.
[4]LANDJEVA S,KORZUNV,BÖRNERA.Molecular markers:actual and potential contributions to wheat genome characterization and breeding[J].Euphytica,2007,156(3):271-296.
[5]韩斌,王长彪,任永康,等.小麦分子育种研究进展IV.小麦相关分子标记[J].山西农业科学,2015,43(4):489-492.
[6]MARONE D,LAIDOG,GADALETA A,et al.A high-density consensus map of A and B wheat genomes[J].Theoretical and Applied Genetics,2012,125(8):1619-1638.
[7] ROOS D S.Bioinformatics-trying to swim in a sea of data[J].Sciences,2001,291:1260-1261.
[8]SCHULERG D.Electronic PCR:bridging the gap between genome mapping and genome sequencing[J].Trends in Biotechnology,1998,16:456-459.
[9]王冬冬,朱延明,李勇,等.电子克隆技术及其在植物基因工程中的应用[J].东北农业大学学报,2006,37(3):403-408.
[10]邱平.小麦抗条锈病SSHcDNA文库ESTs的生物信息学分析[D].雅安:四川农业大学,2010.
[11]张小辉,畅志坚,乔麟轶,等.99份小麦地方品种抗白粉病种质发掘及其分子鉴定[J].山西农业科学,2017,45(5):692-698.
[12]李继发,邓志英,孙福来,等.小麦新品种“山农20”抗病基因的分子检测[J].作物学报,2014,40(4):611-621.
[13]苑翠玲.栽培及野生小麦中条锈病抗性基因的定位和利用[D].泰安:山东农业大学,2014.
[14]刘金栋,陈新民,何中虎,等.小麦慢白粉病QTL对条锈病和叶锈病的兼抗性[J].作物学报,2014,40(9):1557-1564.
[15]张林,张梦雅,高颖,等.山东省12个主栽小麦品种(系)抗叶锈性分析[J].植物遗传资源学报,2017,18(4):676-684.
[16]原宗英,武英鹏.山西省小麦区试品种抗病性鉴定[J].山西农业科学,2016,44(9):1338-1341.
[17]郭晓雪.小麦新品种(系)的抗病性鉴定和丰优8号白粉病抗性的遗传分析[D].杨凌:西北农林科技大学,2016.
[18]王晓蓉,贾宝红,信丽媛,等.天津现代种业体系建设的构想与建议[J].天津农业科学,2015,21(6):28-31.
[19]刘妍,田蓉蓉,王天佑,等.2个小麦品种的遗传性状分析及分子标记筛选[J].河南农业科学,2017,46(10):13-20.
[20]梁昭全.电子定位的原理与方法 [J].分子植物育种,2004,2(4):581-592.
[21]赵洁茹,纪兆华.浅谈农业生物信息学的应用促进农业发展[J].科技传播,2010(9):95-103.
[22]陈蕾.生物信息学在农学研究领域中的应用 [J].考试周刊,2011(52):192-193.
[23]兰素缺,李光威,孟雅宁,等.小麦抗白粉病基因pm30种质改良及鉴定[J].华北农学报,2017,32(Z):7-11.
