盐、干旱胁迫下水稻相关miRNA的鉴定及表达分析
2018-05-09文慧丽赵亚帆王博博金玉蔓孙红正赵全志
彭 廷,文慧丽,赵亚帆,王博博,金玉蔓,孙红正,赵全志
(河南粮食作物协同创新中心,河南省水稻生物学重点实验室,河南农业大学 农学院,河南 郑州 450002)
植物在其生命周期中常受到生物胁迫和非生物胁迫的影响。其中,干旱和盐胁迫经常发生,对作物的生长、发育及产量造成不利影响[1-2]。植物处于高浓度盐离子或者干旱胁迫环境下,会同时受到渗透胁迫、离子胁迫和氧化胁迫,最终引起细胞凋亡,甚至造成植株死亡。miRNA是一类长度为18~24 nt的非编码小分子RNA,通过切割或者翻译抑制负调控下游靶基因的表达[3-5]。作为植物级联调控的上游,miRNA在植物生长的各个阶段发挥重要调节功能[6]。近年来,植物miRNA在感受逆境胁迫并产生适应性的过程中发挥重要调控作用,有关植物miRNA响应干旱胁迫和盐胁迫的报道越来越多[7-13]。其中,miR169负调控植物对干旱胁迫的耐受程度[14],miR159是植物应答干旱胁迫的上游调控因子[13],烟草miR160在受到干旱胁迫后表达量下调,而其靶基因的表达量上调,从而提高了转基因烟草对干旱的耐受性,miR167在拟南芥干旱处理下表达量上调,miR164、miR528、miR444、miR319、miR397等一些保守miRNA的表达量在植物受到干旱或者高盐胁迫下发生一定程度的诱导或者下调[15-18]。
然而,前人对于植物miRNA响应非生物胁迫的研究多集中在采用大规模测序的手段对胁迫处理下特定时间点的研究;另外,对单个响应非生物胁迫的miRNA研究大部分是针对单一胁迫处理下miRNA表达量及靶基因变化情况,而对不同胁迫处理下不同miRNA随时间变化情况的研究比较少,且miRNA及靶基因在植物不同组织表达情况的报道较少。目前,研究miRNA表达常用的方法有Northern杂交,微阵列(Microarray),半定量RT-PCR和实时定量PCR(qRT-PCR)等。其中,qRT-PCR检测基因表达的灵敏度高、快捷、高效,已经成为检测基因表达量的重要方法之一。为筛选出更多与盐胁迫、干旱胁迫相关的miRNA,本研究采用定量PCR的方式对水稻10个保守miRNA及其靶基因在盐胁迫和干旱胁迫处理下不同时间点的表达模式及miRNA与靶基因的相关性进行分析,以期为研究植物对逆境胁迫的响应和适应性及分子设计育种提供理论依据。
1 材料和方法
1.1 试验材料
供试水稻品种为粳稻品种日本晴(OryzasativaL. ssp.japonica),选取饱满一致的种子进行试验。首先种子用15%双氧水进行表面消毒10 min,蒸馏水冲洗5~6次;然后在28 ℃恒温培养箱黑暗培养48 h,选取露白种子单粒放置于96孔PCR板中,之后幼苗培养于28 ℃恒温培养箱,14 h光照,10 h黑暗。3 d更换一次营养液,14 d幼苗用于盐(200 mmol/L NaCl)和干旱(20%PEG)处理。
1.2 试验方法
1.2.1 植物总RNA提取 分别于胁迫后0,3,6,12,24,48 h选取对照(未作处理的水稻)、200 mmol/L NaCl 和20%PEG处理幼苗的根和地上部样品,并立即置于液氮中,-80 ℃保存。总RNA的提取用RNA提取试剂盒(TransGen Biotech,Tranzol Plant,ET1201-01)。
1.2.2 反转录 使用HiFi-MMLV cDNA Kit(康为世纪,CW0744A)反转录成cDNA。用于检测miRNA表达量的cDNA,采用Stem-loop的方法进行反转录,所用引物为miRNA特异反向引物与内参反向引物,引物序列详见表1。用于miRNA靶基因反转录的引物为试剂盒自带PrimerMix,反转录体系为20 μL。

表1 miRNA和靶基因表达量鉴定所用引物及序列 Tab.1 Primers used in miRNA and its target(s)

表1(续)
1.2.3 miRNA及靶基因表达量分析 将反转录后的cDNA稀释20倍,10个水稻保守miRNA分别为miR156、miR159、miR160、miR164、miR167、miR169、miR171、miR319、miR398、miR1848,用于实时荧光定量PCR测定。20 μL反应体系:5 μL模板、10 μL UltraSYBR mixture、1 μL正向引物、1 μL反向引物和3 μL水,以β-actin为内参基因。miRNA定量采用Stem-loop方法进行,所用引物为miRNA正向引物与通用引物stem-loop_U。靶基因定量引物为该基因的正向引物与反向引物。实时荧光定量PCR在CFX 96 Real Time System上进行,反应程序为:95 ℃预变性10 min;95 ℃变性30 s,60 ℃退火30 s,72 ℃延伸30 s,40个循环。所有样品均设置3次重复,相对表达量用2-ΔΔCt方法计算,引物详见表1。
1.2.4 数据分析 试验所得数据采用Microsoft Excel 2007进行整理和分析,相关性和显著性借助SPSS 20.0进行分析,利用MultiExperimentViewer(MeV)-version 4.8.1 进行Heatmap作图。
2 结果与分析
2.1 NaCl和PEG处理对水稻幼苗的影响
从图1可以看出,NaCl和PEG处理后与对照相比,水稻幼苗均出现叶片萎蔫,且NaCl和PEG处理相比,胁迫处理24 h后NaCl处理的表型较PEG处理更为严重。

图1 NaCl(A)和PEG(B)处理24 h后水稻幼苗表型 Fig.1 Phenotypes of rice seedlings at 24 h under NaCl (A) and PEG (B) treatments
2.2 水稻幼苗miRNA的表达量随NaCl处理时间的变化趋势
miRNA定量结果表明,与对照相比,10个miRNA(miR156、miR159、miR160、miR164、miR167、miR169、miR171、miR319、miR398、miR1848)NaCl处理后,根部除miR159、miR319、miR398和miR1848在3 h以及miR319在24 h的表达量下调外,所有miRNA在其他时间点的表达量都呈上调趋势(图2-A)。进一步分析表明,根部miRNA的表达量随处理时间的延长分为4类:(Ⅰ)miR167表达量在处理24 h后出现峰值,且表达量比对照提高了7.24倍;(Ⅱ)miR164表达量上调,在处理6 h后出现峰值;(Ⅲ)miR156、miR159、miR160、miR171、miR169这一类miRNA的表达量先下调后上调,在处理12 h后出现峰值,且miR160表达量增加幅度最大,比0 h提高8.3倍;(Ⅳ)miR319、miR398和miR1848,分别在NaCl处理后6 h和24 h表达量达到最高。这一类miRNA的表达量先下调后上调再下调,且下调的谷底出现在处理后3 h。
与根部miRNA表达量情况不同的是,NaCl处理后地上部大多数miRNA的表达量呈下调趋势(图2-B)。在处理后6 h,所有miRNA的表达量与对照相比均下调。进一步分析表明,地上部miRNA的表达量随处理时间的延长分为4类:(Ⅰ)miR156和miR160的表达量除在处理后12 h明显上调之外,与对照相比,其余时间点均呈下调趋势。(Ⅱ)miR159、miR164、miR169、miR171、miR398的表达量在处理后的大部分时间呈下调趋势,其中miR159和miR398在处理6 h后表达量下调最为显著,分别为对照的3.00%和5.00%;(Ⅲ)miR167的表达量在处理后在3 h表达量下调最为显著,12 h后表达量稳定上调,在24 h出现峰值;(Ⅳ)miR319和miR1848的表达量基本呈先下降后上升的趋势,且在处理后48 h出现峰值。

图2 根(A)和地上部(B)miRNA的表达量随NaCl处理时间的变化趋势 Fig.2 Expression patterns analysis of different miRNAs in root (A) and shoot (B) under NaCl treatment
2.3 水稻幼苗miRNA的表达量随PEG处理时间的变化趋势
从图3可以看出,PEG处理后,无论是根部还是地上部,miRNA的表达量大部分呈下降趋势,但是具体表达情况,根部与地上部的表达量又有所不同(图3-A)。进一步分析表明,根部miRNA的表达量随处理时间的延长可以分为5类:(Ⅰ)miR164的表达量除在处理后6 h明显上调之外,其他时间点均下调;(Ⅱ)miR159和miR160的表达量可分为一类,其表达量在处理后6 h和48 h有所上调,其他时间点均表现为下调;(Ⅲ)miR169的表达量除在处理后6 h略有上调之外,其余时间点均下调;(Ⅳ)miR398在处理后12 h略有上调,其余时间点均下调。(Ⅴ)miR156、miR167、miR171、miR319、miR1848的表达量在处理后的所有时间点均下调。
PEG处理后地上部miRNA的表达量与根部的略有不同(图3-B),具体表现为地上部的miRNA表达量随处理时间的延长可以分为4类:(Ⅰ)miR156、miR159、miR160、miR164和miR171可分为一大类,其表达量在处理后的所有时间点均表现为下调;(Ⅱ)miR398的表达量在处理后的24 h和48 h略有上调;(Ⅲ)miR169和miR319的表达量在处理48 h后显著上调,分别上调至对照的4.45,5.40倍;(Ⅳ)miR167和miR1848的表达量呈现波动性变化,在处理后6,24,48 h上调,且在处理后的24,48 h时达到峰值。
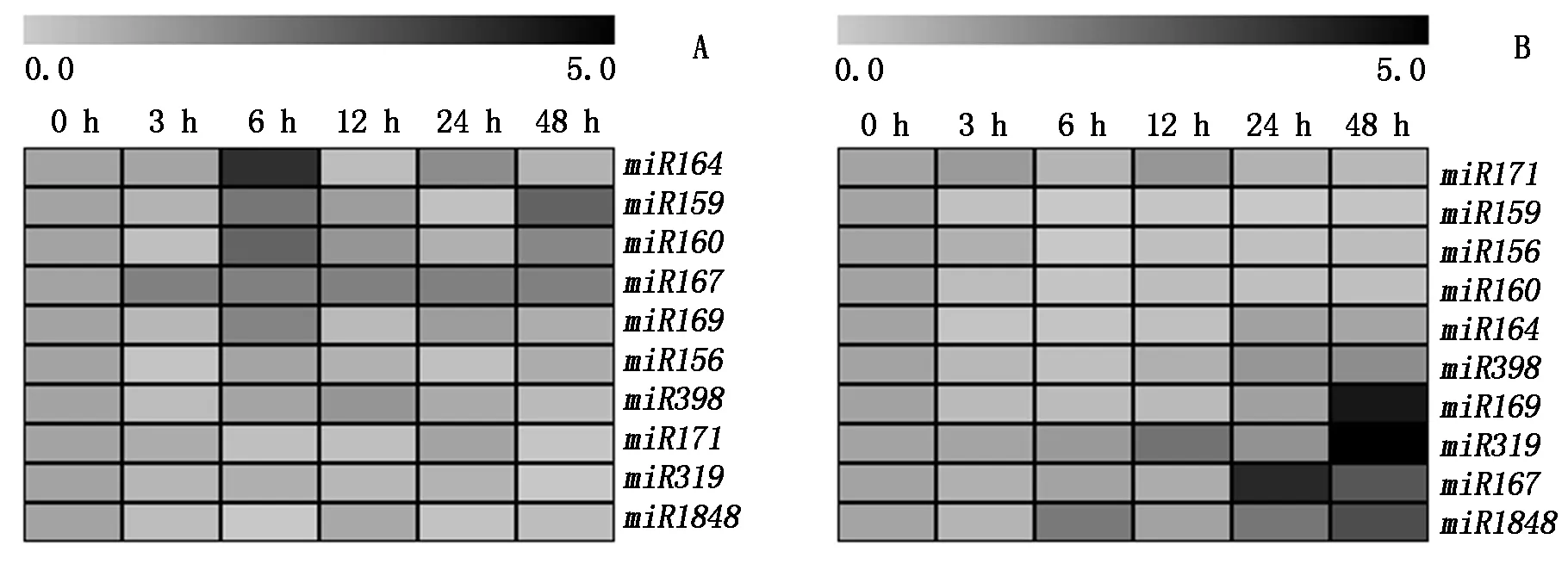
图3 根(A)和地上部(B)miRNA的表达量随PEG处理时间的变化趋势 Fig.3 Expression patterns analysis of different miRNAs in root (A) and shoot (B) under PEG treatment
2.4 水稻幼苗miRNA靶基因的表达量随NaCl处理时间的变化趋势
从图4可以看出,大多数miRNA靶基因的表达量随处理时间的延长呈上调趋势。对根部miRNA靶基因的表达量进一步分析表明,不同靶基因的表达量变化趋势不尽一致(图4-A)。具体可以分为4类:(Ⅰ)OsOMTN6的表达量下调趋势明显,且波谷出现在处理后24 h;(Ⅱ)OsARF8的表达量与对照相比也呈现下调趋势,但波谷出现的时间为处理后3 h,其余时间点无明显变化;(Ⅲ)OsARF16、OsARF18、OsSPL13、OsSPL14、OsSOD2、OsGAMYB、OsNF-YA可以聚为一类,且其表达量呈现波动性变化,除OsGAMYB的表达量一直下调外,其他基因的表达量在处理后均有上调的时间点,其中OsSPL13、OsSPL14、OsSOD2、OsNF-YA的峰值出现在处理后12 h;(Ⅳ)OsCYP51G3、OsGRAS、OsPCF5和OsPCF6因其在根部的表达量太低,未能检测到。
图4-B结果表明,地上部miRNA靶基因的表达量随处理时间的延长可以分为4类:(Ⅰ)OsARF16的表达量除在处理后3 h下调外,其余时间点均上调,上调的峰值出现在胁迫后12 h,上调倍数达到36倍;(Ⅱ)OsSOD2、OsARF18、OsGAMYB3个靶基因的表达量随处理时间的延长基本上呈现递增趋势,峰值出现在胁迫后48 h,其中OsSOD2上调趋势明显,上调倍数达6.06倍;(Ⅲ)OsSPL13、OsSPL14、OsOMTN6、OsARF8、OsNF-YA的表达量在胁迫之后呈现波动变化;(Ⅳ)OsCYP51G3、OsGRAS、OsPCF5、OsPCF6的表达量在地上部表达量过低,均未检测到。

图4 根(A)和地上部(B)miRNA靶基因的表达量随NaCl处理时间的变化趋势 Fig.4 Expression patterns analysis of miRNA targets in root(A) and shoot (B) under NaCl treatment
2.5 水稻幼苗miRNA靶基因的表达量随PEG处理时间的变化趋势
从图5-A可以看出,PEG处理后miRNA靶基因的表达量随处理时间的延长大致可以分为3大类:(Ⅰ)OsSPL13、OsSPL14、OsSOD2的表达量在处理后3 h出现波谷,处理后6 h开始出现上调;(Ⅱ)OsARF16、OsARF18、OsOMTN6、OsARF8、OsGAMYB、OsNF-YA的表达量在处理后,与对照相比呈现下调趋势或基本保持不变。(Ⅲ)与NaCl胁迫下根部类似,OsCYP51G3、OsGRAS、OsPCF5、OsPCF6因其表达量太低未检测到。
通过对PEG处理下地上部miRNA靶基因表达量分析,其表达量随处理时间的延长大致可以分为5类(图5-B):(Ⅰ)OsARF16的表达量在处理后48 h内一直呈上调趋势,且峰值出现在胁迫后12 h,是对照的6.77倍;(Ⅱ)OsSPL13和OsSOD2的表达量峰值都出现在48 h。其中OsSPL13表达量在48 h之前,除6 h和24 h略微升高外,其他时间略低于对照。OsSOD2的表达量在处理后48 h内呈现波动性变化,其中大部分时间点表达量高于对照,且峰值都出现在处理后48 h;(Ⅲ)OsARF8和OsOMTN6的表达量在处理后表达量一直处于下调状态,且OsOMTN6在胁迫后的6,24 h下调幅度较大;(Ⅳ)OsARF18、OsSPL14、OsGAMYB、OsNF-YA的表达量在处理后3 h出现上调,且出现峰值,之后随着处理时间的增加出现不同程度的下调,其中OsARF18和OsGAMYB的表达量在胁迫后48 h再次上调;(Ⅴ)OsCYP51G3、OsGRAS、OsPCF5和OsPCF6因其在地上部的表达量太低,未能检测到。
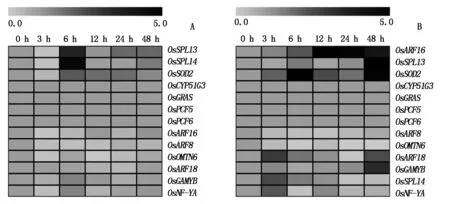
图5 根(A)和地上部(B)miRNA靶基因的表达量随PEG处理时间的变化趋势分析 Fig.5 Expression patterns analysis of miRNA targets in root(A) and shoot (B) under PEG treatment
2.6 NaCl和PEG处理后miRNA表达量与其靶基因表达量的相关分析
本研究利用SPSS 20.0对NaCl和PEG处理后miRNA与靶基因的表达量进行了相关性分析。结果表明,NaCl胁迫处理下,根部miR156的表达量与其靶基因OsSPL13、OsSPL14的表达量存在显著正相关关系,miR159和miR160的表达量与其靶基因OsGAMYB和OsARF16表达量之间也存在显著正相关关系,而其他miRNA的表达量与靶基因的表达量相关性不显著;地上部miR160的表达量与其靶基因OsARF16的表达量存在显著正相关关系,而其他miRNA的表达量与其靶基因的表达量相关性不显著。PEG处理下,根部miR169表达量与其靶基因OsNF-YA表达量之间存在显著负相关关系,而其他miRNA的表达量与其靶基因的表达量相关性不显著;而地上部所有检测的miRNA与其靶基因的表达量之间的相关性均不显著(表2)。
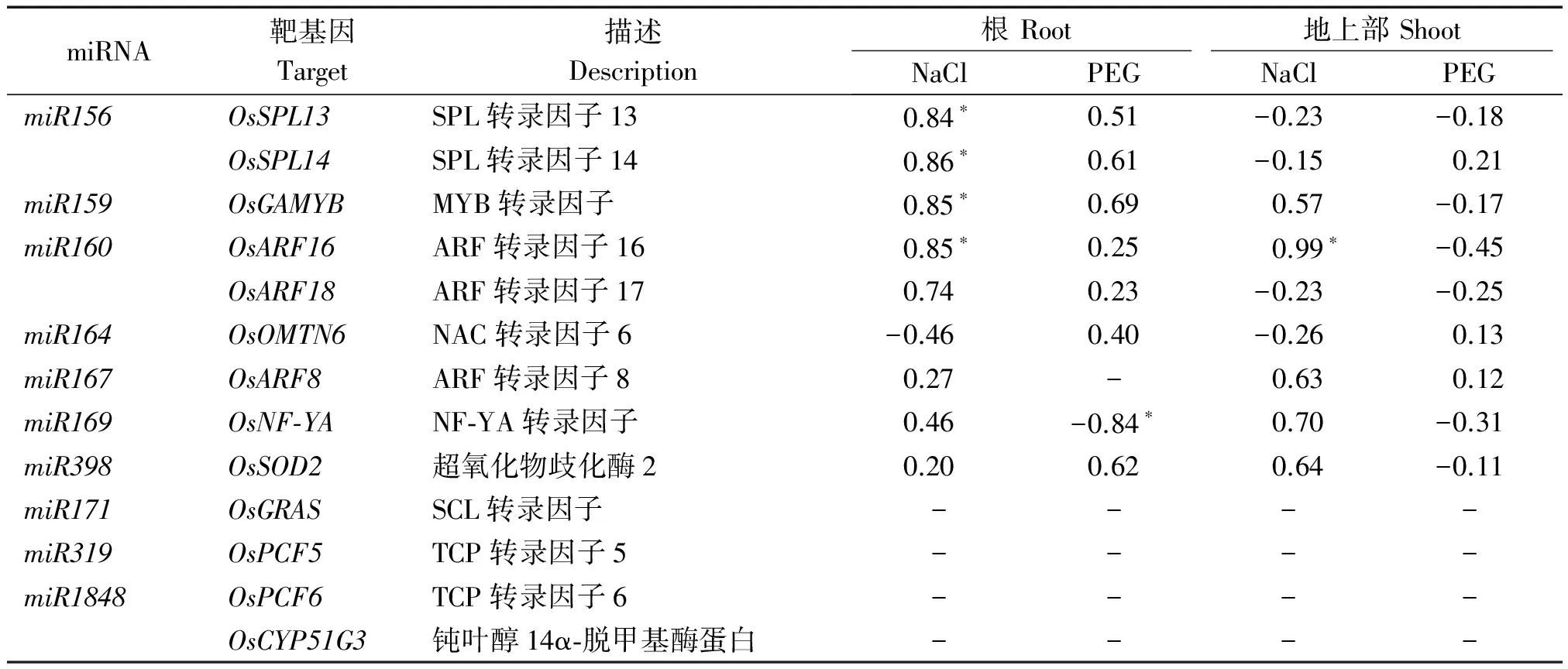
表2 miRNA表达量与靶基因表达量的相关性分析 Tab.2 Correlation coefficient analysis of expressions between miRNA and its target(s)
注:*、**.差异达到显著和极显著。
Note:*and**.Significant at 5% and 1% probability levels,respectively.
3 结论与讨论
植物miRNA在多种逆境胁迫下均有响应,且在感受植物逆境胁迫之后做出的适应性调整过程中发挥重要作用。本研究利用实时定量PCR的方法,对10个miRNA在水稻幼苗盐和干旱胁迫不同时间段的表达模式进行分析,发现这10个miRNA均参与了水稻幼苗对盐和干旱胁迫的响应。NaCl胁迫处理下,大部分miRNA在根部的表达量在多数时间点呈上调趋势,说明NaCl胁迫诱导这10个miRNA在根部的表达。PEG胁迫处理后,除地上部在胁迫后期miR167、miR169、miR319和miR1848表达量上调之外,其余大部分miRNA的表达量都呈下调趋势。说明这10个miRNA不仅能参与NaCl胁迫调控,还参与到干旱胁迫的调控网络中,但其调控机制可能存在差异。
miRNA在盐和干旱胁迫下的表达模式存在差异,同时在相同胁迫下根部与地上部的表达量又有所不同,并且随着胁迫时间的延长,单一miRNA的表达量也是呈现时序性表达。miR156在受到干旱胁迫中表达量下调[19],这与本研究中miR156的表达量在PEG胁迫下根部与地上部表达量均下调是一致的。而在NaCl胁迫处理下,无论是根部或者是地上部,miR156的表达量随处理时间的延长,既有下调,也有上调。miR167在受到PEG胁迫处理后,在根部表现为下调表达,在地上部以及NaCl胁迫处理后随胁迫时间的增加表现为时序性变化,而在烟草干旱胁迫下的48,72 h,miR167的表达量上调[20],在玉米中的表达量在干旱处理后24 h是下调表达,而在处理后48,72 h上调表达[21]。说明miRNA的表达存在明显的组织特异性和时序性。因此,在研究miRNA对环境响应的试验中,应考虑miRNA在环境响应中的时序性特征,单一测定某一时间点miRNA的表达量进而得出miRNA对环境的响应规律是不科学的。
本研究所筛选的10个与盐和干旱胁迫相关的miRNA,除miR398的靶基因编码超氧化物歧化酶蛋白和miR1848靶基因编码钝叶醇14α-脱甲基酶蛋白基因外,其余8个miRNA的靶基因均为转录因子。已有研究认为,miR156通过负调控靶基因SPLs调节叶和芽的发育及生长阶段的转换[22],miR160、miR164、miR167则是通过ARFs家族和NAC等转录因子调控一些生理过程[23-25]。本研究发现,miR156、miR160、miR164、miR167及其靶基因在受到盐胁迫和干旱处理后表达量无论在根部还是在地上部均存在显著变化,且出现差异的时间点不同说明这些miRNAs与靶基因也参与到植物胁迫响应中。miR169通过负调控其靶基因NF-YA家族转录因子参与到调控植物对干旱的耐受程度通路[26-27],miR171很可能参与干旱信号通路[28],miR398在植物抵御氧化胁迫中起着举足轻重的作用[29],miR1848在油菜素内酯介导的种子发育及盐胁迫方面发挥重要作用[30],在盐和干旱胁迫处理后这3个miRNA的表达量也存在时序性变化,且这些miRNA在响应盐和干旱胁迫中发挥重要的功能。
植物miRNA通过近似完美匹配其靶基因的方式,在转录后水平上切割或者在翻译水平上抑制其靶基因的表达,进而对下游的代谢通路起到重要的调控作用[3-4]。本研究中miRNA的表达量与其靶基因表达量之间的关系有正相关、负相关、不相关,说明miRNA与靶基因共同调控植物逆境胁迫的复杂性[31]。在miRNA与下游靶基因的对应关系中,单一miRNA可能调控多个下游靶基因,这些靶基因可能含有相似的结构域[32],也可能是隶属不同家族的蛋白[33];同时一个靶基因也会受到不同miRNA的调控[34],以及miRNA与靶基因之间存在的反馈调节关系[35],增加了研究miRNA与靶基因共同调控植物逆境胁迫的难度。未来研究应集中在深入地研究这些响应植物逆境胁迫的miRNA如何发挥调控作用及其之间的相互关系和调控网络,以便更好地利用这些miRNA,通过相应的生物技术手段应用于作物育种和生产实践。
参考文献:
[1] Sahi C,Singh A,Kumar K,et al. Salt stress response in rice:genetics,molecular biology,and comparative genomics[J]. Functional & Integrative Genomics,2006,6(4):263-284.
[2] Tambo J A,Abdoulaye T. Climate change and agricultural technology adoption:the case of drought tolerant maize in rural Nigeria[J]. Mitigation and Adaptation Strategies for Global Change,2012,17(3):277-292.
[3] Bartel D P. MicroRNAs:genomics,biogenesis,mechanism,and function[J]. Cell,2004,116(2):281-297.
[4] Bartel D P. MicroRNAs:target recognition and regulatory functions[J]. Cell,2009,136(2):215-233.
[5] Achkar N P,Cambiagno D A,Manavella P A. miRNA biogenesis:a dynamic pathway[J]. Trends in Plant Science,2016,21(12):1034-1044.
[6] Jones-Rhoades M W,Bartel D P,Bartel B. MicroRNAs and their regulatory roles in plants[J]. Annual Review of Plant Biology,2006,57(1):19-53.
[7] Liu H H,Tian X,Li Y J,et al. Microarray-based analysis of stress-regulated microRNAs inArabidopsisthaliana[J]. RNA,2008,14(5):836-843.
[8] Ding D,Zhang L,Wang H,et al. Differential expression of miRNAs in response to salt stress in maize roots[J]. Annals of Botany,2009,103(1):29-38.
[9] Fan G,Wang L,Dong Y,et al. Genome of paulownia (Paulowniafortunei) illuminates the related transcripts,miRNA and proteins for salt resistance[J]. Scientific Reports,2017,7(1):1285.
[10] Mondal T K,Ganie S A. Identification and characterization of salt responsive miRNA-SSR markers in rice (Oryzasativa)[J]. Gene,2014,535(2):204-209.
[11] Ren Y,Chen L,Zhang Y,et al. Identification and characterization of salt-responsive microRNAs inPopulustomentosaby high-throughput sequencing[J]. Biochimie,2013,95(4):743-750.
[12] Wang Y G,An M,Zhou S F,et al. Expression profile of maize microRNAs corresponding to their target genes under drought stress[J]. Biochemical Genetics,2014,52(11/12):474-493.
[13] Yang J,Zhang N,Mi X,et al. Identification of miR159s and their target genes and expression analysis under drought stress in potato[J]. Computational Biology and Chemistry,2014,53:204-213.
[14] Ni Z,Hu Z,Jiang Q,et al.GmNFYA3,a target gene of miR169,is a positive regulator of plant tolerance to drought stress[J]. Plant Molecular Biology,2013,82(1/2):113-129.
[15] 艾 佳,李永光,王 涛,等.植物逆境microRNA的研究进展 [J]. 基因组学与应用生物学,2014(5):1154-1160.
[16] 马风勇,朱永兴,石晓霞,等. 植物miRNA抗逆性研究进展 [J]. 西北农林科技大学学报:自然科学版,2012(5):217-223.
[17] 王 维,张玉娟,陈 洁,等. 植物逆境胁迫相关miRNA研究进展[J]. 生物技术通报,2015(1):1-10.
[18] 许振华,谢传晓. 植物microRNA与逆境响应研究进展[J]. 遗传,2010,32(10):1018-1030.
[19] Jian X,Zhang L,Li G,et al. Identification of novel stress-regulated microRNAs fromOryzasativaL.[J]. Genomics,2010,95(1):47-55.
[20] Wei L Y,Zhang D F,Xiang F,et al. Differentially expressed miRNAs potentially involved in the regulation of defense mechanism to drought stress in maize seedings[J]. International Journal of Plant Sciences,2009,170(8):979-989.
[21] Huang S Q,Xiang A L,Che L L,et al. A set of miRNAs fromBrassicanapusin response to sulphate deficiency and cadmium stress[J]. Plant Biotechnology Journal,2010,8(8):887-899.
[22] Jiao Y,Wang Y,Xue D,et al. Regulation of OsSPL14 by OsmiR156 defines ideal plant architecture in rice[J]. Nature Genetics,2010,42(6):541-544.
[23] Raman S,Greb T,Peaucelle A,et al. Interplay of miR164,CUP-SHAPED COTYLEDON genes and LATERAL SUPPRESSOR controls axillary meristem formation inArabidopsisthaliana[J]. Plant Journal,2008,55(1):65-76.
[24] Xiong G L,Xie K,Hou X.MIR164 gene that controls plant root system development and fertility and use thereof [M]. USA:Google Patents,2010.
[25] Liu H,Jia S,Shen D,et al. Four AUXIN RESPONSE FACTORs down-regulated by microRNA167 are associated with growth and development inOryzasativa[J]. Functional Plant Biology,2012,39(9):736-744.
[26] Zhao B,Ge L,Liang R,et al. Members of miR-169 family are induced by high salinity and transiently inhibit the NF-YA transcription factor[J]. BMC Molecular Biology,2009,10:29.
[27] Zhang X,Zou Z,Gong P,et al. Over-expression of microRNA169 confers enhanced drought tolerance to tomato[J]. Biotechnology Letters,2011,33(2):403-409.
[28] Hwang E W,Shin S J,Yu B K,et al. miR171 family members are involved in drought response inSolanumtuberosum[J]. Journal of Plant Biology,2011,54(1):43-48.
[29] Lu Y Z,Feng Z,Bian L Y,et al. miR398 regulation in rice of the responses to abiotic and biotic stresses depends on CSD1 and CSD2 expression[J]. Functional Plant Biology,2011,38(1):44-53.
[30] Xia K,Ou X,Tang H,et al. Rice microRNA osa-miR1848 targets the obtusifoliol 14α-demethylase geneOsCYP51G3 and mediates the biosynthesis of phytosterols and brassinosteroids during development and in response to stress[J]. The New Phytologist,2015,208(3):790-802.
[31] Yang C,Li D,Mao D,et al. Overexpression of microRNA319 impacts leaf morphogenesis and leads to enhanced cold tolerance in rice (OryzasativaL.) [J]. Plant,Cell & Environment,2013,36(12):2207-2218.
[32] Schommer C,Bresso E G,Spinelli S V,et al. Role of microRNA miR319 in plant development [J]. Signaling & Communication in Plants,2012,15:29-47.
[33] Wu J,Yang R,Yang Z,et al. ROS accumulation and antiviral defence control by microRNA528 in rice[J]. Nature Plants,2017,3(1):16203.
[34] Wang L,Sun S,Jin J,et al. Coordinated regulation of vegetative and reproductive branching in rice[J]. Proceedings of the National Academy of Sciences of the United States of America,2015,112(50):15504-15509.
[35] Meng Y,Chen D,Ma X,et al. Mechanisms of microRNA-mediated auxin signaling inferred from the rice mutant osaxr[J]. Plant Signaling & Behavior,2010,5(3):252-254.
