多光谱数据融合技术对绒柄牛肝菌产地的鉴别
2018-04-20刘鸿高李杰庆王元忠
姚 森,李 涛,刘鸿高,李杰庆,*,王元忠*
野生食用菌富含多糖、蛋白质、维生素、膳食纤维和多种矿质元素,经常食用可以增强免疫力、促进人体新陈代谢,能够有效预防和治疗肿瘤、痢疾、水肿等疾病,兼具食药用价值[1-3]。其味道鲜美,口感细腻,已远销欧美等国家,极具开发价值[4-5]。云南被称为“食用菌王国”,种类繁多,分布广泛,因生长环境不同,其代谢产物种类及含量发生变化,导致同种类不同产地食用菌品质存在差异[6]。对其进行产地鉴别,为食用菌资源开发与利用提供参考依据,同时为野生食用菌的质量评价提供基础。
目前,对食用菌鉴别分析方法除了简单的形态学方法外,光谱法和分子生物法应用较广泛[7-8]。Li Yan等[9]采用紫外光谱法结合化学计量学对3 个不同产地野生茯苓进行快速鉴别,结果表明其紫外光谱具有指纹特性,结合聚类分析可准确区分不同产地茯苓;Lian Bin等[10]采用分子生物法,根据内转录间隔区(internal transcribed spacer,ITS)设计引物,成功鉴别区分了美味牛肝菌(Boletus edulis Bull.)和其他蘑菇。单一的光谱技术对样品信息提取率低,容易受到其他因素(CO2、H2O等)干扰;分子生物法需要昂贵的仪器设备,不适宜推广应用。
数据融合技术将多种仪器获得的数据进行优化、整合,丰富样品信息,实现仪器间的互补,获得更全面、可靠的数据,结合化学计量学构建判别模型,对样品进行系统的分析[11-13]。数据融合分为初级融合、中级融合和高级融合,初级数据融合是将不同仪器获得的数据进行简单的串联,形成更全面的数据集[14];中级融合也称为特征级融合是将不同来源的数据经过特征提取,并对选取的特征变量(如主成分、变量在投影方向的重要程度等)进行整合,去除干扰信息,从而获得更加丰富、系统的数据集[15-16];高级融合亦被称作决策级融合,通过两个或两个以上分类模型得出最优鉴别结果[17-18]。在已有的研究中,仅有10%的融合技术采用了高级数据融合[19]。在本研究中,采用初级融合和中级融合技术已经达到了预期效果,无须对高级融合技术进行进一步研究。
本实验采用紫外光谱法和红外光谱法,紫外指纹图谱技术根据峰形、吸收波长、吸光度等差异,结合化学计量学方法可用于分析物质间差异具有操作简便、快速、整体性强、成本低等特点[20-21];红外光谱能有效反映出样品的组分特征,是一种综合性检测方法,广泛应用于食品质量控制、物种鉴别等方面,具有快速、简便、样品用量少等优点[22-23]。通过二阶导数(second derivative,2D)、标准正态变量(standard normal variate,SNV)、多元散射校正(multiplicative scatter correction,MSC)、Savitzky-Golay平滑(15点平滑)等方法对原始光谱数据进行优化处理,采用偏最小二乘判别分析(partial least square discriminant analysis,PLS-DA)和支持向量机(support vector machine,SVM)分析比较紫外光谱、红外光谱和数据融合技术对不同产地绒柄牛肝菌的区分效果,构建快速、有效鉴别牛肝菌产地的方法,同时为食品质量监控提供理论基础和科学依据。
1 材料与方法
1.1 材料
实验所用5 个产地绒柄牛肝菌(B. tomentipes Earle.)采自云南省和四川省,所有样品均由云南农业大学农学与生物技术学院刘鸿高博士鉴定为绒柄牛肝菌,共96份,样品详细信息见表1。

表1 牛肝菌样品信息Table 1 Information about B. tomentipes samples
1.2 仪器与设备
UV-2550型紫外-可见分光光度计 日本岛津公司;Frontier型傅里叶变换红外光谱仪(配备氘化硫酸三甘肽晶体检测器) 美国Perkin Elmer公司;SY3200-T型超声波清洗仪 上海声源超声波仪器设备有限公司;YP-2型压片机 上海市山岳科学仪器有限公司;FW-100型高速粉碎机 天津市华鑫仪器厂;80目标准筛盘浙江上虞市道墟五四仪器厂。
1.3 样品处理和光谱采集
样品采集后清洗干净,50 ℃恒温烘干,粉碎后过80 目标准筛,贮存备用。准确称取0.1 g牛肝菌样品粉末置于试管中,加入10 mL氯仿溶剂,30 ℃条件下超声提取30 min,3 层滤纸过滤,取清液置于石英比色皿中备用。紫外-可见分光光度计预热30 min后对样品提取液进行紫外光谱扫描,重复扫描2 次,取平均光谱,扫描间隔1 nm。扫描前使用氯仿溶液进行基线扫描。
按1∶100的比例,准确称量(1.5±0.2)mg牛肝菌样品和(150±20)mg溴化钾粉末,放入玛瑙研钵充分混合研磨成细粉,将细粉倒入压制磨具中压制成厚度均匀的薄片。将红外光谱仪预热30 min后进行样品测定,样品重复测定2 次,取平均光谱;扫描前使用空白样本扣除CO2和H2O的干扰。
1.4 数据分析
傅里叶变换红外光谱仪和紫外-可见分光光度计在采集光谱信息时,会受背景噪音、散光等干扰信息[24-25]。为消除无关信息干扰,红外原始光谱通过OMNIC 8.0软件进行平均光谱、自动基线校正、纵坐标归一化等预处理,紫外原始光谱采用UV probe 2.34软件进行平滑等预处理;同时,采用2D、SNV、MSC和Savitzky-Golay平滑(15 点平滑)分别对两种光谱进行优化处理。
原始光谱经预处理后,选取具有指纹特性的光谱信息进行串联,形成一个包含大量变量的独立数据矩阵,完成低级融合。采用SIMCA-P+13.0软件对红外光谱和紫外光谱数据分别进行PLS-DA,提取特征变量,变量在投影方向的重要程度(variables important in projection,VIP)表示变量对分类效果的贡献大小。当VIP值大于1时,表明该值对应的变量对区分效果的影响较大,能够代表样品的整体信息[26],整合两种光谱数据的VIP值大于1的波段,进行中级融合。为了消除融合过程中受量纲干扰,在数据融合前进行归一化处理。
SIMCA-P+13.0软件对光谱数据进行2D、SNV、MSC、Savitzky-Golay平滑等优化处理;Origin 8.0软件对图形进行优化;通过SIMCA-P+13.0软件和MATLAB R2014a软件分别进行PLS-DA和SVM判别分析,建立鉴别模型,比较分类结果。
2 结果与分析
2.1 紫外光谱分析
在样品紫外光谱采集过程中,容易受到溶剂、仪器、环境等外部因素的干扰,采用UV probe 2.34软件对紫外图谱进行平滑等预处理。由于在190~250 nm波长内干扰严重,400 nm以后无明显特征吸收峰,且牛肝菌紫外光谱的特征吸收峰集中在250~400 nm波长范围内,因此选取250~400 nm波段的151 个变量作为样品信息用于区分牛肝菌产地。如图1所示,在250~400 nm范围内5 个不同产地绒柄牛肝菌样品的光谱图峰形相似度高,在250~350 nm区间有明显的特征吸收峰,265、273、283、295 nm附近为样品共有峰,表明不同地区牛肝菌的化学组分相似。5 个不同产地绒柄牛肝菌样品的峰强、峰位和峰面积存在差异,尤其在250~300 nm区间差异明显,具有指纹特性,能够作为区分不同地区绒柄牛肝菌的依据。
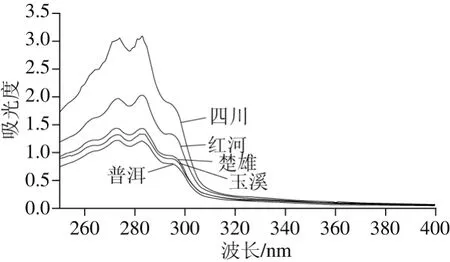
图1 绒柄牛肝菌平均红外光谱Fig. 1 Average UV absorption spectra of B. tomentipes
2.2 红外光谱分析
釆用OMNIC 9.0软件对96 份牛肝菌红外光谱进行平滑、基线校正和纵坐标归一化等预处理。牛肝菌红外光谱在4 000~400 cm-1波段内有明显的特征吸收峰,具有指纹特性,可以用于牛肝菌的鉴别分析。样品预处理后的红外指纹图谱如图2所示,不同产地绒柄牛肝菌的红外光谱较为相似,共有峰波数大致相同,红外光谱在3 325 cm-1附近的强吸收峰归属为蛋白质、多糖、纤维素等O—H伸缩振动或者是蛋白质中N—H伸缩振动;2 927 cm-1附近吸收峰主要为多糖、蛋白质等甲基对称伸缩振动;在1 547、1 453、1 402、1 375、1 259、1 081 cm-1及1 029 cm-1等波数附近有明显吸收峰,但不同产地样品的吸收峰强度有差异。1 547 cm-1附近吸收峰为C=O伸缩振动,为蛋白质酰胺I带;1 453 cm-1附近归属为亚甲基的弯曲振动;1 402、1 375 cm-1及1 259 cm-1等附近为多糖、蛋白质等的C—O—H弯曲振动和亚甲基的变形振动;1 081、1 029 cm-1附近分别为糖类的C—O和C—C伸缩振动;1 000~600 cm-1波段有多个弱吸收峰,主要为糖类异构体的特征峰[4,27]。

图2 绒柄牛肝菌平均红外光谱Fig. 2 Average FTIR spectra of B. tomentipes
2.3 特征变量提取
通过筛选VIP值得到对预测能力贡献大的变量,是一种常用的提取潜在变量方法,光谱技术结合VIP筛选已经在食品鉴别分析方面得到广泛应用[28-29]。采用SIMCA 13.0软件紫外光谱和红外光谱数据进行PLS-DA,对紫外光谱和红外光谱分别进行2D+Savitzky-Golay平滑(15 点平滑)和2D+SNV+MSC优化处理,并提取VIP值大于1的波段。图3和图4中a、b分别代表预处理后光谱指纹图谱和VIP值大于1的波段,由图3a和图4a可知,经预处理后,图谱的特征吸收峰更加明显,图谱平滑,噪音减小;图3b和图4b表示该波段对鉴别绒柄牛肝菌产地具有较大贡献,可以代表牛肝菌整体化学信息,选取对应的变量作为特征变量进行中级融合。

图3 预处理后紫外光谱VIP得分图Fig. 3 Scores plot for VIP of UV spectra after preprocessing
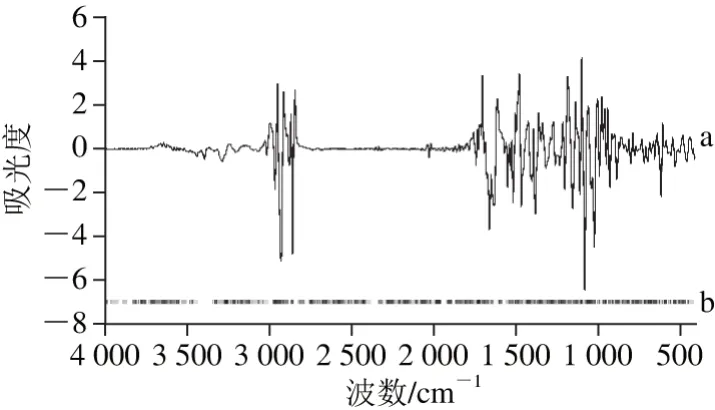
图4 预处理后红外光谱VIP得分图Fig. 4 Scores plot for VIP of IR spectra after preprocessing
2.4 PLS-DA
光谱技术结合PLS-DA法已被广泛应用于饮料、食用油等食品检测和质量评价[30-32]。采用PLS-DA对绒柄牛肝菌产地进行鉴别分析,随机选取32 个绒柄牛肝菌样品(约样品量的1/3)作为预测集,其余64 个样品作为训练集。对紫外光谱、红外光谱、初级融合和中级融合数据分别建立PLS-DA判别模型。如表2所示,红外光谱、紫外光谱、初级融合和中级融合数据级分别建立PLSDA模型,对绒柄牛肝菌产地的预测正确率为56.25%、56.25%、62.50%和81.25%。紫外光谱和红外光谱技术对未知产地牛肝菌的分类效果相同,且紫外光谱技术建立模型更加稳定。初级数据融合技术对不同产地牛肝菌的分类正确率高于单独的紫外光谱技术和红外光谱技术,表明初级融合技术丰富了光谱数据,获得了更加完整的光谱信息,增加了分类正确率。中级数据融合训练集和预测集的正确率分别达到98.44%和81.25%,正确率高于单一光谱技术,分类效果优于初级融合,表明中级融合技术建立的判别模型比单独光谱技术和初级融合技术更稳定、可靠。
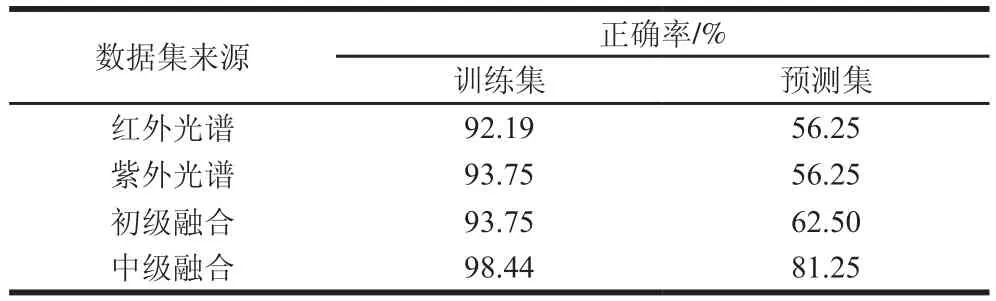
数据集来源 正确率/%训练集 预测集红外光谱 92.19 56.25紫外光谱 93.75 56.25初级融合 93.75 62.50中级融合 98.44 81.25
2.5 SVM分析
SVM判别模型在材料鉴别[33]、食品分析[34]等领域应用广泛,在本研究中,SVM建模选取训练集和预测集的方法同2.4节,基于红外光谱、紫外光谱、低级融合和中级融合数据分别建立SVM判别模型。如图5所示,4 种方法分别有3、11、4、1 个样品区分错误,表明紫外光谱技术建立SVM模型对绒柄牛肝菌产地鉴别效果最差,中级融合技术建立SVM模型对绒柄牛肝菌产地分类效果最佳。
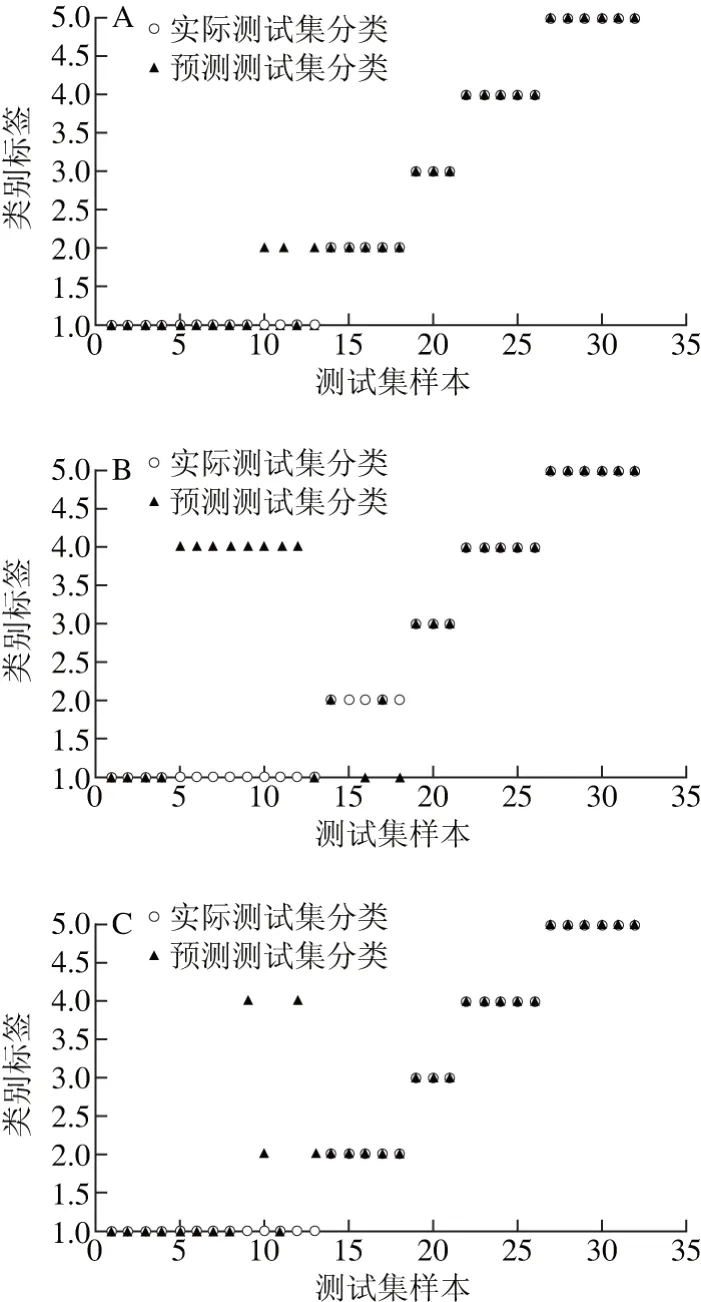

图5 SVM对测试集的实际分类和预测分类Fig. 5 Actual and SVM predicted categories of the test set samples
训练集正确率代表模型的稳定性和可靠性,预测集正确率代表模型对不同产地牛肝菌的区分效果。如表3所示,产地预测正确率分别为90.63%、65.63%、87.50%和96.88%。此外,4 个数据集建模的训练集正确率皆为96.88%,表明4 个模型的稳定性高;初级融合对不同产地的预测正确率高于单个紫外光谱技术,低于单独的红外光谱技术,表明初级融合丰富了光谱数据,同时简单的数据串联将无效信息相互叠加,降低分类正确率;中级融合的分类正确率高于初级融合,证明中级融合在整合数据过程中去除了无效信息,避免两种光谱信息的互相干扰,提高分类正确率。比较表2和表3可知,采用中级融合技术建立SVM判别模型,模型稳定、可靠,对不同产地牛肝菌鉴别效果最佳。

表3 SVM对不同数据集的分类结果Table 3 Prediction accuracy of SVM based on different data matrixes
3 结 论
采用红外光谱法和紫外光谱法采集不同产地绒柄牛肝菌的光谱信息,选择2D+Savitzky-Golay平滑(15 点平滑)和2D+SNV+MSC预处理方法分别对样品红外光谱和紫外光谱进行优化处理,减少噪音干扰。对优化后的光谱数据进行初级融合,并通过PLS-DA提取VIP大于1的波段进行中级数据融合。通过PLS-DA和SVM判别分析分别建立分类鉴别模型,并比较红外光谱、紫外光谱、低级融合和中级融合技术对不同产地绒柄牛肝菌的鉴别效果。结果显示:中级数据融合建立PLS-DA和SVM判别模型对未知样品产地的预测正确率分别为81.25%、96.88%,鉴别效果优于其他技术,表明中级融合对不同产地牛肝菌区分效果最佳,且SVM判别模型与PLS-DA模型相比更加稳定、可靠。中级融合技术结合SVM建立鉴别模型,能够准确、有效区分不同产地绒柄牛肝菌,为快速鉴别野生食用菌提供有效方法,对食品评价具有重要意义。
参考文献:
[1] 李涛, 王元忠, 李荣春. 绒柄牛肝菌子实体营养成分分析[J]. 食用菌学报, 2008, 15(4): 70-72. DOI:10.3969/j.issn.1005-9873.2008.04.013.
[2] CHEUNG P C K. The nutritional and health benefits of mushrooms[J].Nutrition Bulletin, 2010, 35(4): 292-299. DOI:10.1111/j.1467-3010.2010.01859.x.
[3] RADZKI W, SLAWINSKA A, JABLONSKA-RYS E, et al.Antioxidant capacity and polyphenolic content of dried wild edible mushrooms from Poland[J]. International Journal of Medicinal Mushrooms, 2014, 16(1): 65-75. DOI:10.1615/IntJMedMushr.v16.i1.60.
[4] 杨天伟, 张霁, 史云东, 等. 红外光谱结合多元统计分析快速鉴别不同种类牛肝菌[J]. 食品科学, 2015, 36(24): 116-121. DOI:10.7506/spkx1002-6630-201524020.
[5] WANG X M, ZHANG J, WU L H, et al. A mini-review of chemical composition and nutritional value of edible wild-grown mushroom from China[J]. Food Chemistry, 2014, 151: 279-285. DOI:10.1016/j.foodchem.2013.11.062.
[6] LI Y, ZHANG J, LI T, et al. Geographical traceability of wild Boletus edulis based on data fusion of FT-MIR and ICP-AES coupled with data mining methods (SVM)[J]. Spectrochimica Acta Part A: Molecular and Biomolecular Spectroscopy, 2017, 177: 20-27. DOI:10.1016/j.saa.2017.01.029.
[7] 杨天伟, 李涛, 张霁, 等. 不同产地, 种类牛肝菌的紫外光谱鉴别分析[J]. 食品工业, 2015, 36(9): 208-212.
[8] 杨天伟, 李涛, 张霁, 等. 不同产地绒柄牛肝菌紫外指纹图谱鉴别分析[J]. 食品工业科技, 2015, 36(9): 301-305. DOI:10.13386/j.issn1002-0306.2015.09.057.
[9] LI Y, ZHANG J, ZHAO Y L, et al. Characteristic fingerprint based on low polar constituents for discrimination of Wolfiporia extensa according to geographical origin using UV spectroscopy and chemometrics methods[J]. Journal of Analytical Methods in Chemistry,2014, 2014: 519424. DOI:10.1155/2014/519424.
[10] LIAN B, ZANG J P, HOU W G, et al. PCR-based sensitive detection of the edible fungus Boletus edulis from rDNA ITS sequences[J].Electronic Journal of Biotechnology, 2008, 11(3): 102-109.DOI:10.4067/S0717-34582008000300011.
[11] KHALEGHI B, KHAMIS A, KARRAY F O, et al. Multisensor data fusion: a review of the state-of-the-art[J]. Information Fusion, 2013,14(1): 28-44. DOI:10.1016/j.inffus.2011.08.001.
[12] VERA L, ACEÑA L, GUASCH J, et al. Discrimination and sensory description of beers through data fusion[J]. Talanta, 2011, 87: 136-142.DOI:10.1016/j.talanta.2011.09.052.
[13] WU Z, XU E, LONG J, et al. Comparison between ATR-IR, Raman,concatenated ATR-IR and Raman spectroscopy for the determination of total antioxidant capacity and total phenolic content of Chinese rice wine[J]. Food Chemistry, 2016, 194: 671-679. DOI:10.1016/j.foodchem.2015.08.071.
[14] PIZARRO C, RODRÍGUEZ-TECEDOR S, PÉREZ-DELNOTARIO N, et al. Classification of Spanish extra virgin olive oils by data fusion of visible spectroscopic fingerprints and chemical descriptors[J]. Food Chemistry, 2013, 138(2): 915-922. DOI:10.1016/j.foodchem.2012.11.087.
[15] DEARING T I, THOMPSON W J, RECHSTEINER C E, et al.Characterization of crude oil products using data fusion of process raman, infrared, and nuclear magnetic resonance (NMR) spectra[J].Applied Spectroscopy, 2011, 65(2): 181-186.
[16] BIANCOLILLO A, BUCCI R, MAGRÌ A L, et al. Data-fusion for multiplatform characterization of an Italian craft beer aimed at its authentication[J]. Analytica Chimica Acta, 2014, 820: 23-31.DOI:10.1016/j.aca.2014.02.024.
[17] DI ANIBAL C V, CALLAO M P, RUISÁNCHEZ I.1H NMR and UV-visible data fusion for determining Sudan dyes in culinary spices[J].Talanta, 2011, 84(3): 829-833. DOI:10.1016/j.talanta.2011.02.014.
[18] DOESWIJK T G, SMILDE A K, HAGEMAN J A, et al. On the increase of predictive performance with high-level data fusion[J].Analytica Chimica Acta, 2011, 705(1): 41-47. DOI:10.1016/j.aca.2011.03.025.
[19] BORRÀS E, FERRÉ J, BOQUÉ R, et al. Data fusion methodologies for food and beverage authentication and quality assessment: a review[J]. Analytica Chimica Acta, 2015, 891: 1-14. DOI:10.1016/j.aca.2015.04.042.
[20] MARTINS A R, TALHAVINI M, VIEIRA M L, et al. Discrimination of whisky brands and counterfeit identification by UV-Vis spectroscopy and multivariate data analysis[J]. Food Chemistry, 2017, 229: 142-151.DOI:10.1016/j.foodchem.2017.02.024.
[21] PAULI E D, BRUNS R E, SCARMINIO I S. UV-Vis spectral fingerprinting and chemometric method applied to the evaluation of Camellia sinensis leaves from different harvests[J]. Analytical Methods, 2016, 8(41): 7537-7544. DOI:10.1039/C6AY02462A.
[22] ZHAO Y L, ZHANG J, JIN H, et al. Discrimination of Gentiana rigescens from different origins by Fourier transform infrared spectroscopy combined with chemometric methods[J]. Journal of AOAC International, 2015, 98(1): 22-26. DOI:10.5740/jaoacint.13-395.
[23] QI L M, ZHANG J, ZUO Z T, et al. Determination of iridoids in Gentiana rigescens by infrared spectroscopy and multivariate analysis[J]. Analytical Letters, 2017, 50(2): 389-401. DOI:10.1080/00 032719.2016.1178757.
[24] 姚森, 张霁, 李杰庆, 等. 利用FTIR和化学计量学对牛肝菌亲缘关系的研究[J]. 河南农业科学, 2017, 46(1): 110-115. DOI:10.15933/j.cnki.1004-3268.2017.01.020.
[25] LI Y, ZHANG J, LI T, et al. Ultraviolet spectroscopy used to fingerprint five wild-grown edible mushrooms (Boletaceae) collected from Yunnan, China[J]. Journal of Spectroscopy, 2016, 2016: 7813405.DOI:10.1155/2016/7813405.
[26] CHONG I G, JUN C H. Performance of some variable selection methods when multicollinearity is present[J]. Chemometrics and Intelligent Laboratory Systems, 2005, 78(1): 103-112. DOI:10.1016/j.chemolab.2004.12.011.
[27] FAGHIHIAN H, SABOKROOH E. Immobilization of ligands on surfactant coated alumina for removal of cations[J]. Iranian Journal of Chemistry and Chemical Engineering, 2012, 31(4): 1-9.
[28] LAWAETZ A J, CHRISTENSEN S M U, CLAUSEN S K, et al. Fast,cross cultivar determination of total carotenoids in intact carrot tissue by Raman spectroscopy and partial least squares calibration[J]. Food Chemistry, 2016, 204: 7-13. DOI:10.1016/j.foodchem.2016.02.107.
[29] OUSSAMA A, ELABADI F, PLATIKANOV S, et al. Detection of olive oil adulteration using FT-IR spectroscopy and PLS with variable importance of projection (VIP) scores[J]. Journal of the American Oil Chemists’Society, 2012, 89(10): 1807-1812. DOI:10.1007/s11746-012-2091-1.
[30] MA H, WANG J, CHEN Y, et al. Rapid authentication of starch adulterations in ultrafine granular powder of Shanyao by near-infrared spectroscopy coupled with chemometric methods[J]. Food Chemistry,2017, 215: 108-115. DOI:10.1016/j.foodchem.2016.07.156.
[31] BONA E, MARQUETTI I, LINK J V, et al. Support vector machines in tandem with infrared spectroscopy for geographical classification of green arabica coffee[J]. LWT-Food Science and Technology, 2017,76: 330-336. DOI:10.1016/j.lwt.2016.04.048.
[32] LI X, XU K, ZHANG Y, et al. Optical determination of lead chrome green in green tea by Fourier transform infrared (FT-IR) transmission spectroscopy[J]. PLoS ONE, 2017, 12(1): e0169430. DOI:10.1371/journal.pone.0169430.
[33] BALABIN R M, SAFIEVA R Z, LOMAKINA E I. Gasoline classification using near infrared (NIR) spectroscopy data: comparison of multivariate techniques[J]. Analytica Chimica Acta, 2010, 671(1):27-35. DOI:10.1016/j.aca.2010.05.013.
[34] POULADZADEH P, SHIRMOHAMMADI S, BAKIROV A, et al.Cloud-based SVM for food categorization[J]. Multimedia Tools and Applications, 2015, 74(14): 5243-5260. DOI:10.1007/s11042-014-2116-x.
