Tol2转座子介导斑马鱼rps26基因附近增强子捕获及注解分析
2018-04-12桑亚通沈丹陈伟产舒恒顾浩高波宋成义
桑亚通,沈丹,陈伟,产舒恒,顾浩,高波,宋成义
扬州大学 动物科学与技术学院 农业与农产品安全国际合作联合实验室,江苏 扬州 225009
增强子是能强化转录起始的一段DNA序列,最早是由Benerji在SV40DNA中发现[1],随后在病毒及真核生物基因组中均发现了增强子元件的存在。已有研究表明增强子在基因表达调控中通过介导转录因子与靶基因的结合,参与基因的表达调控,发挥重要作用。与启动子不同的是,增强子对基因的调控不具有方向性,且不受两者距离的影响,无论位于基因的上游或下游,甚至基因的内含子中,均可调控靶基因的表达[2-3]。因此,增强子的获得对于研究基因表达调控模式具有重要意义,但传统方法进行增强子注解效率低下。研究证实增强子捕获技术是一种有效的增强子注释方法[4],最早在 1979年 Casadaban等以LacZ为目的基因,将一个启动子缺失的乳糖操纵子载体通过噬菌体感染随机整合到大肠杆菌基因组上,最终获得了一些特异性表达的基因[5]。受此研究启发,研究人员对启动子捕获载体进行改造,将一个报告基因和最小启动子融合取代缺失启动子的报告基因,该启动子不能单独驱动报告基因的表达,而当插入位点附近存在增强子元件时,则启动子被激活从而驱动报告基因的表达,这种方法即为增强子捕获。首个转座子介导的增强子捕获载体是基于P转座子设计的,以β-半乳糖苷酶基因为报告基因,成功应用于果蝇的增强子捕获研究[6]。之后,转座子介导增强子捕获技术广泛应用于各种动物的增强子研究。Grabher等[7]在青中成功应用了以转座子为介导的增强子捕获技术,Balciunas等[8]利用SB转座子,在斑马鱼中进行增强子捕获研究,获得了9种具有不同的组织或器官特异性GFP表达模式的斑马鱼品系。除此之外,研究人员对增强子捕获系统进行改进,利用酵母双元杂交系统 (Gal4-UAS) 进行增强子捕获。目前,二元的Gal4-UAS系统也已经成功应用于斑马鱼和果蝇的增强子捕获研究[9-11]。
斑马鱼作为研究脊椎动物的模型,具有养殖方便、繁殖周期短、产卵量大、胚胎体外受精、体外发育、胚体透明等优点,已成为生命科学研究的重要工具。近年来,Tol2转座子已成功应用于斑马鱼的增强子捕获研究[12]。本研究通过sp-PCR、原位杂交和比较基因组学等技术手段对本实验室已经建立的Tol2转座子介导的增强子捕获品系 (TK4) 进行鉴定,以期解析所捕获的增强子。
1 材料与方法
1.1 材料
Tuebingen斑马鱼购自国家斑马鱼资源中心;Tol2转座子介导的增强子捕获转基因斑马鱼 F1代TK4系为本实验室制备。
基因组提取试剂盒、纯化试剂盒、切胶回收试剂盒购自宝生物工程有限公司;限制性内切酶Sau3AⅠ、T4 DNA连接酶、退火缓冲液均购自NEB有限公司;无RNA酶DNaseⅠ、RNAsein、NaOH、链酶蛋白酶、多聚甲醛、PTU、HEPES、Tween20、tRNA、肝素钠、苯酚、氯仿、HCl、BCIP、NBT等购自Sigma公司;地高辛标记RNA混合液、蛋白酶 K、羊抗地高辛标记物抗体等购自Roche公司;转录缓冲液、T7 RNA聚合酶等购自Promega公司;去离子甲酰胺购自CarloErba;羊血清、无水甲醇、NaCl、KCl、MgCl2、无水乙醇等购自国药集团化学试剂有限公司。
1.2 TK4系斑马鱼的来源与荧光检测
TK4系由本实验室前期通过Tol2转座子介导制备的稳定增强子捕获品系F1代,该转基因增强子捕获载体中含一个 krt4 (keratin4) 迷你启动子和GFP表达盒 (图1A)。将TK4系与TU系野生型斑马鱼进行交配,获得F2代胚胎,在E3溶液中培养至特定的发育时期以备荧光检测。在体式荧光显微镜 (M165FC,Lecia,德国) 下观察不同时期的胚胎GFP的表达并做好记录。本实验中观察的胚胎GFP的表达时期分别是6 hpf、24 hpf、48 hpf、3 dpf、4 dpf、5 dpf。
1.3 转基因在基因组上插入位点鉴定
参照文献[13],通过sp-PCR进行转基因在基因组上插入位点鉴定。首先根据转座元件序列设计两轮PCR引物,接头序列参考文献[13],引物序列及接头序列如表1所示。然后进行sp-PCR,具体步骤如下:采用 TaKaRa的基因组提取试剂盒提取饲养 20 d的 F2代斑马鱼的基因组,用Sau3AⅠ对基因组进行酶切,以产生 GATC末端进行后续接头连接。50 µL反应体系为:基因组DNA 5 µL,10×NEB 缓冲液 5 µL,Sau3AⅠ3 µL,双蒸水补齐至50 µL。将反应体系置于37 ℃过夜处理,之后用DNA片段纯化试剂盒 (TaKaRa公司) 进行纯化并用45 µL双蒸水洗脱。接头连接50 µL 体系包括:基因组酶切 37 µL,10× T4 DNA连接酶缓冲液5 µL,接头6 µL,T4 DNA连接酶(400 U/µL) 2 µL;连接反应条件:16 ℃,16 h。其中,使用 SPLNK-BOT 2 µL,SPLNK-GATC-TOP 2 µL,退火缓冲液46 µL进行接头合成。反应程序为95 ℃、3 min;自然降至室温。之后进行两轮PCR,其中第一轮PCR体系 (50 µL) 为:连接基因组 DNA 10 µL,双蒸水 11 µL,2×Taqmix 25 µL,SPLINK 1 2 µL,Tol2/ SP1R 2 µL。PCR 扩增程序:94 ℃预变性 5 min;94 ℃ 30 s,60 ℃ 30 s,72 ℃2 min,35个循环;最后72 ℃延伸10 min。第二轮PCR体系 (50 µL) 为:第一轮 PCR 产物1 µL,双蒸水 20 µL,2×Taqmix 25 µL,SPLINK 2 2 µL,Tol2/SP2R 2 µL。PCR 扩增程序:94 ℃预变性5 min;94 ℃ 30 s,60 ℃ 30 s,72 ℃ 90 s,35 个循环;最后 72 ℃延伸 10 min。反应完成后,将PCR产物在 1.2%琼脂糖凝胶电泳中进行电泳检测并切胶回收,测序。
1.4 增强子注解
将测序获得的染色体侧翼 DNA序列通过BLASTN与 ENSEMBL的斑马鱼基因组数据库(GRCz10) 进行比对。为了检测整合位点附近的增强子元件,再从Ensembl浏览器上下载斑马鱼Danio rerio、青Oryzias latipes、东方红鳍Takifugu rubripes、秀美花Poecilia formosa、罗非鱼Oreochromis niloticus、矛尾鱼 Latimeria chalumnae、非洲爪蟾Xenopus tropicalis、鸡Gallus gallus、大鼠Rattus norvegicus、小鼠Mus musculus、人Homo sapiens的整合位点侧翼序列上下游各50 kb区域的基因组序列 (www.ensembl.org),并将所得到的序列进行比对分析 (http://genome.lbl.gov/vista/mvista/submit.shtml)。
1.5 原位杂交
根据插入位点处的内源基因rps26编码序列设计反义RNA原位杂交探针,探针rps26-001以及探针rps26-201引物序列 (表2)。采用地高辛和T7 RNA聚合酶标记合成反义RNA探针。参考文献[14]进行斑马鱼胚胎原位杂交,通过体式显微镜 (M165FC,Lecia,德国) 采集图片。
2 结果与分析
2.1 F2代胚胎不同发育时期GFP表达
将 TK4系F1与TU系斑马鱼进行杂交,收集胚胎于 E3培养液中培养至特定的发育时期进行观察并拍照记录。结果如图1所示,在胚胎发育至6 hpf未观察到GFP表达 (图 1B),在胚胎发育至24 hpf时观察到GFP表达,表达部位主要集中于头部和躯干 (图 1C)。随着发育时间的延长,组织器官逐渐分化,48 hpf时GFP表达部位主要集中于头部、眼睛、下颌、脊髓和肌肉等(图 1D)。从 48 hpf发育至 5 dpf,GFP表达部位基本稳定,主要集中于中脑、后脑、眼睛、下颌和脊髓,肌肉的GFP荧光表达强度逐渐减弱(图 1E–G)。

表1 sp-PCR引物序列与接头序列[13]Table 1 sp-PCR primers and linker sequences[13]

表2 探针rps26-001与rps26-201引物序列Table 2 Probe rps26-001and rps26-201 primer sequences
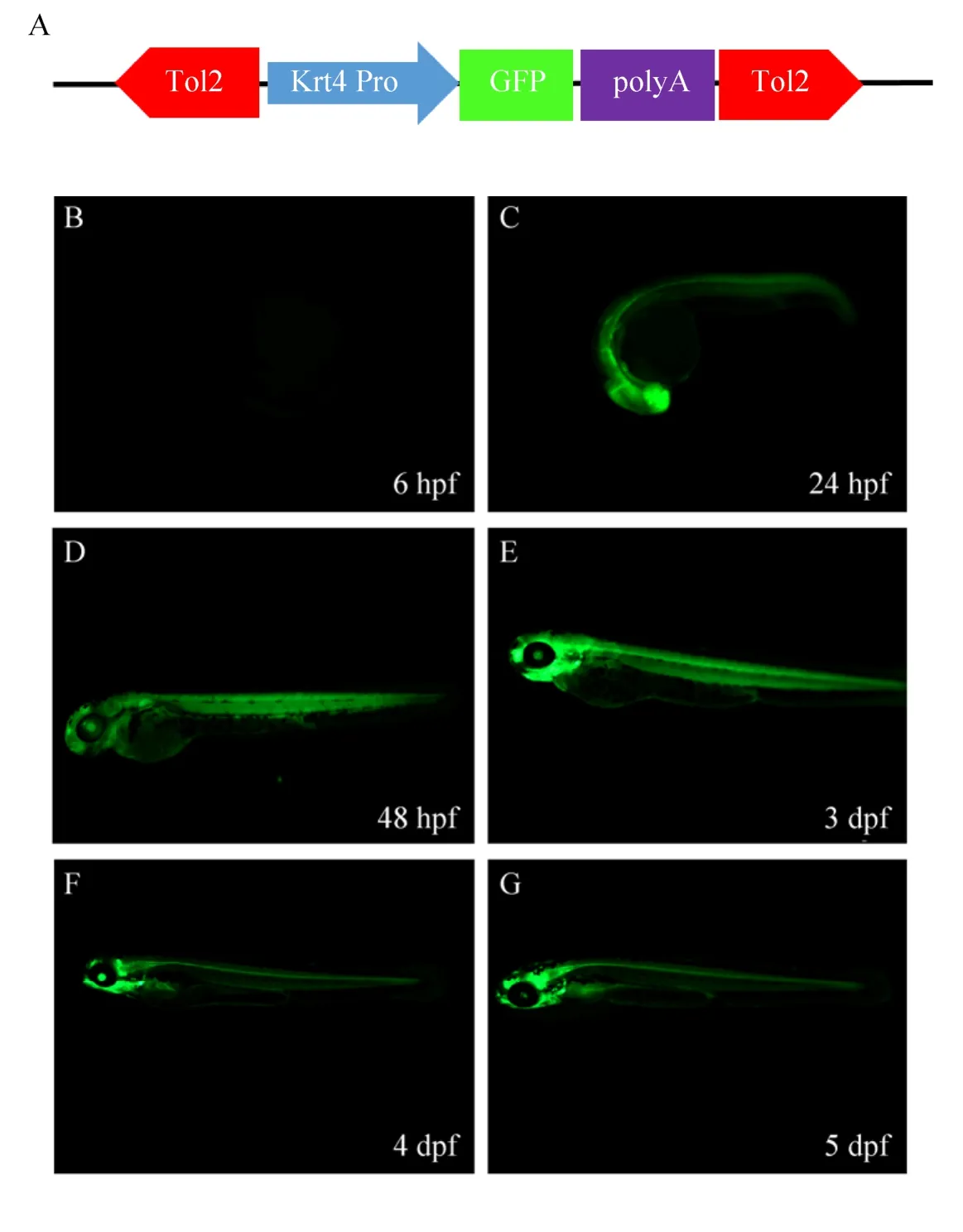
图1 TK4增强子捕获转基因斑马鱼不同发育时期的荧光表达情况 (A:TK4增强子捕获品系转基因构件示意图;B–G为不同发育阶段胚胎荧光检测)Fig. 1 GFP-expression of TK4 enhancer trapping transgenic zebrafish at different developmental stages. (A)Transgenic components of TK4 enhancer trapping line. (B–G) GFP-expression at different developmental stages.
2.2 增强子捕获转基因斑马鱼的增强子注解
将测序结果中得到的Tol2转座子插入位点的侧翼DNA序列在ENSEMBL的斑马鱼基因组数据库 (GRCz10) 中进行比对,结果显示捕获载体插入23号染色体rps26基因的intron1中,且报告基因的插入方向与rps26基因的方向相反。从Ensembl浏览器上下载斑马鱼、青、东方红鳍、秀美花、罗非鱼、矛尾鱼、非洲爪蟾、鸡、大鼠、小鼠、人的插入位点侧翼序列上下游各 50 kb区域的同源基因组序列以及注解文件,并将所得到的序列进行比对分析,结果如图 2所示,载体插入位置为黑色箭头所指。在100 kb的基因组范围内有 7个基因,分别为arf3a、wnt10b、wnt1、rps26、IKZF4、dnajc22和lmbr1l。在rps26基因的下游有2个CNS,其中CNS1较CNS2相似序列的保守度 (Identity)更高,相似序列的宽度 (Width) 更广,并且这两个元件在真骨鱼类 (青、东方红鳍、秀美花和罗非鱼) 中高度保守,但是在两栖类 (非洲爪蟾)、鸟类 (鸡) 和哺乳类 (小鼠、大鼠和人)中均不保守。结果提示这两个CNS可能为潜在的增强子。

图2 插入位点侧翼区比较基因组学分析Fig. 2 Comparative genomic analysis of the flank sequences of the insertion site. Dr: Danio rerio; Tr: Takifugu rubripes; Ol: Oryzias latipes; Pf: Poecilia formosa; On: Oreochromis niloticus; Lc: Latimeria chalumnae; Xt: Xenopus tropicalis; Gg: Gallus gallus; Rn: Rattus norvegicus; Mm: Mus musculus; Hs: Homo sapiens; UTR: untranslated region;CNS: conserved non-coding sequence.
2.3 原位杂交
由于载体的插入位点是在rps26基因的1号内含子中,为了验证TK4的GFP表达模式是否与rps26基因表达模式一致,进行了受精至3 dpf斑马鱼早期胚胎原位杂交实验。斑马鱼rps26基因存在两个可变剪切转录本,分别为rps26-001和rps26-201,根据这两个转录本分别设计原位杂交探针,检测rps26基因在斑马鱼早期胚胎中的表达特性。结果显示,两个转录本在单细胞受精卵中 (0.2 hpf) 已经启动表达,提示存在母源性表达;而rps26-001在0.75 hpf时表达有所减弱,至4 hpf时表达又开始增强,rps26-201在 0.75 hpf表达明显高于rps26-001,表明rps26-201的表达启动要早于rps26-001;在4 hpf至48 hpf的发育过程中两个转录本均有表达;在发育至24 hpf时,表达呈现全身性的特点,无明显组织和细胞特异性,而发育至48 hpf时,表达部位主要集中于头部及中脑,呈现明显的组织特异性;发育至3 dpf时,组织特异性表达更加明显,主要集中于中脑-后脑连接处、眼眶以及原肾管等部位表达,各阶段rps26-201的表达信号要高于rps26-001。rps26-001和rps26-201的表达在早期胚胎 (受精至24 hpf)中呈现无明显组织和细胞特异性,且启动较早,而GFP在6 hpf时尚无表达信号,至24 hpf则呈现一定的组织特异性,两者并不一致,但后期rps26-001和rps26-201的表达与GFP表达模式部分相似,包括主要集中在脑部,特别是中脑信号比较强。

图3 rps26-001和rps26-201转录本在不同发育阶段斑马鱼胚胎中原位杂交Fig. 3 In situ hybridization for rps26-001和rps26-201 transcripts at different stages of zebrafish embryos.
3 讨论
转座子是在基因组内可自主移动的一段DNA序列,最早是由 McClintock在研究玉米籽粒颜色遗传时发现的[15]。而Tol2转座子则是由Koga等在研究白化青鱼时发现[16],属于 hAT转座子超家族。目前,Tol2转座子因其可携带较长的插入片段等优势,已广泛应用于斑马鱼、爪蟾、鸡和小鼠等模式脊椎动物的研究中[12,17–19]。最近研究表明,Tol2转座子已成功应用于斑马鱼的功能基因[20]和增强子捕获[21-23]的研究中。传统的增强子捕获技术需要耗费大量的人力物力,为了分离到单性状的个体,需要制备大规模的G3代突变群体,除此之外,传统方法很难分离得到增强子。目前在斑马鱼上进行增强子捕获研究最有效的方法是利用转座子介导的增强子捕获技术,这一技术为研究相关基因的调控模式提供了很好的工具。近年来,利用转座子介导的增强子捕获技术获得插入突变体库进而对相关器官功能、疾病等的研究已取得较大进展。Parinov等[12]利用Tol2转座子介导斑马鱼增强子捕获技术,建立突变品系,研究斑马鱼发育的相关功能基因。Xue等[24]通过Tol2转座子介导的大规模增强子诱捕筛选到 26个血管特异表达绿色荧光蛋白(EGFP) 报告基因的转基因斑马鱼系,其中有一些品系在胚胎的某些特异血管结构中表达绿色荧光蛋白。最终获得EGFP报告基因受hhex或ets1a基因增强子控制的转基因斑马鱼品系,而hhex和ets1a基因对血管与血细胞前体的发育具有重要作用,因此,该品系为深入研究这两个基因在血管与血液发育中的作用机制提供了新的机遇。Huang等[25]利用Tol2转座子系统介导的增强子捕获技术构建得到了心肌特异性表达GFP的转基因斑马鱼,且在心房和房室管中观察到了特异性表达的GFP。
本课题组采用Tol2转座子介导的增强子捕获技术获得了大量斑马鱼插入突变体,通过报告基因GFP可确定捕获的增强子和内源基因时空表达特性,大大提高了筛选效率。我们已通过表型筛选建立了若干个突变品系,本研究所用的TK4系斑马鱼表型为头部和躯干具有较为明显的GFP表达,通过 sp-PCR的方法成功地克隆到了Tol2转座子插入位点的侧翼基因组序列,揭示了TK4系的插入位点是rps26基因的1号内含子,并且报告基因的插入方向与基因相反。同时结合比较基因组学手段,通过跟其他物种进行同源比对,发现在rps26基因下游有2个潜在的增强子信号(CNS1和CNS2),其中CNS1较为明显,在鱼类中保守性较强。在 CNS1的上下游有很多基因,包括arf3a、wnt10b、wnt1、rps26、IKZF4、dnajc22和lmbr1l。已有研究表明,Wnt1在小鼠的中脑和前后脑的形成中起重要作用,在斑马鱼中需要wnt1和wnt10b来维持中脑和后脑连接处的Pax2.1和Fgf8的阈值水平[26]。rps26基因编码的核糖体蛋白就是核糖体小亚基40S的组成部分,该蛋白属于rps26e核糖体蛋白家族[27]。IKZF4也称转录因子Eos,是锌指蛋白转录因子ikaros家族中的一员。作为负性调节因子可以参与许多基因的表达调控[28]。有研究表明,Eos位于细胞核中,在中枢神经和周围神经系统的发育过程中扮演重要角色[29]。Eos对淋巴系细胞发育和形成过程也有重要作用[30]。dnajc22作为果蝇wurst基因的同源物,目前研究较少,而果蝇wurst基因编码一种 J结构域跨膜蛋白,该蛋白是果蝇气管尺寸和气道清除的必要调节因子[31]。对于基因arf3a和lmbr1l的功能及其表达特性,目前尚未见报道。目前关于这些基因的表达调控机理也尚未见报道。而该品系的建立及注释对于进一步研究这些基因的功能特点及其表达调控机制提供了很好的模式动物。Gharbi等[32]所捕获的转基因斑马鱼品系中,通过原位杂交验证了捕获的内源性基因kctd15a与转基因斑马鱼荧光蛋白表达模式基本一致,而且在后期的发育过程中也有着相似的时空表达特征。在 Liu等[4]所捕获的转基因斑马鱼品系中,通过对EGFP和捕获的内源性基因设计反义 RNA探针来检测内源性基因的表达模式比较,发现EGFP的表达模式与所捕获到的内源性基因基本一致。在刘帅军等[33]所捕获的转基因斑马鱼品系中,通过原位杂交验证了捕获的内源性基因denraa与转基因斑马鱼GFP表达模式基本一致。而本研究中通过比较TK4的GFP表达模式与rps26基因原位杂交结果,发现两者表达模式存在相似之处,但也并不完全一致,提示两者可能既接受共同的增强子调控,但也存在不同增强子调控,所获得的 2个潜在的增强子 (CNS1和 CNS2) 可能对附近的基因 (包括rps26) 发挥差异的时空表达调控作用,但需进一步深入研究验证。
4 结论
本研究首次获得了rps26基因的2个潜在增强子调控元件,揭示了rps26基因表达特性及其与潜在增强子调控的可能联系。研究结果为深入理解和研究rps26基因调控机制提供重要参考;同时本研究所尝试的增强子研究综合技术手段(包括转座子介导的增强子捕获、sp-PCR、比较基因组学和原位杂交等) 也为后基因组学时代解析基因表达调控提供方法参考。
[1]Banerji J,Rusconi S,Schaffner W. Expression of a β-globin gene is enhanced by remote SV40 DNA sequences. Cell,1981,27(2): 299–308.
[2]Spilianakis CG,Lalioti MD,Town T,et al.Interchromosomal associations between alternatively expressed loci. Nature,2005,435(7042): 637–645.
[3]Lomvardas S,Barnea G,Pisapia DJ,et al.Interchromosomal interactions and olfactory receptor choice. Cell,2006,126(2): 403–413.
[4]Liu CY,Song GL,Mao L,et al. Generation of an enhancer-trapping vector for insertional mutagenesis in zebrafish. PLoS ONE,2015,10(10): e0139612.
[5]Casadaban MJ,Cohen SN. Lactose genes fused to exogenous promoters in one step using a Mu-lac bacteriophage:in vivoprobe for transcriptional control sequences. Proc Natl Acad Sci USA,1979,76(9):4530–4533.
[6]Bellen HJ,O’Kane CJ,Wilson C,et al. P-elementmediated enhancer detection: a versatile method to study development inDrosophila. Genes Dev,1989,3(9):1288–1300.
[7]Grabher C,Henrich T,Sasado T,et al. Transposonmediated enhancer trapping in medaka. Gene,2003,322: 57–66.
[8]Balciunas D,Davidson AE,Sivasubbu S,et al. Enhancer trapping in zebra fi sh using the Sleeping Beauty transposon. BMC Genomics,2004,5(1): 62.
[9]Bellen HJ. Ten years of enhancer detection: lessons from the fly. Plant Cell,1999,11(12): 2271–2281.
[10]Distel M,Wullimann MF,Köster RW. Optimized Gal4 genetics for permanent gene expression mapping in zebrafish. Proc Natl Acad Sci USA,2009,106(32):13365–13370.
[11]Asakawa K,Kawakami K. TheTol2-mediated Gal4-UAS method for gene and enhancer trapping in zebrafish. Methods,2009,49(3): 275–281.
[12]Parinov S,Kondrichin I,Korzh V,et al.Tol2transposon-mediated enhancer trap to identify developmentally regulated zebrafish genesin vivo. Dev Dyn,2004,231(2): 449–459.
[13]Potter CJ,Luo LQ. Splinkerette PCR for mapping transposable elements inDrosophila. PLoS ONE,2010,5(4): e10168.
[14]Thisse C,Thisse B. High-resolutionin situhybridization to whole-mount zebrafish embryos. Nat Protoc,2008,3(1): 59–69.
[15]McClintock B. Chromosome organization and genic expression. Cold Spring Harb Symp Quant Biol,1951,16: 13–17.
[16]Koga A,Suzuki M,Inagaki H,et al. Transposable element in fish. Nature,1996,383(6595): 30.
[17]Hamlet MRJ,Yergeau DA,Kuliyev E,et al.Tol2transposon-mediated transgenesis inXenopustropicalis.Genesis,2006,44(9): 438–445.
[18]Sato Y,Kasai T,Nakagawa S,et al. Stable integration and conditional expression of electroporated transgenes in chicken embryos. Dev Biol,2007,305(2): 616–624.
[19]Keng VW,Ryan BJ,Wangensteen KJ,et al. Efficient transposition ofTol2in the mouse germline. Genetics,2009,183(4): 1565–1573.
[20]Clark KJ,Urban MD,Skuster KJ,et al. Transgenic zebrafish using transposable elements. Methods Cell Biol,2011,104: 137–149.
[21]Asakawa K,Suster ML,Mizusawa K,et al. Genetic dissection of neural circuits byTol2transposonmediated Gal4 gene and enhancer trapping in zebrafish.Proc Natl Acad Sci USA,2008,105(4): 1255–1260.
[22]Fisher S,Grice EA,Vinton RM,et al. Evaluating the biological relevance of putative enhancers usingTol2transposon-mediated transgenesis in zebrafish. Nat Protoc,2006,1(3): 1297–1305.
[23]Kondrychyn I,Teh C,Garcia-Lecea M,et al. Zebrafish Enhancer TRAP transgenic line database ZETRAP 2.0.Zebrafish,2011,8(4): 181–182.
[24]Xue YL,Xiao A,Wen L,et al. Generation and characterization of blood vessel specific EGFP transgenic zebrafishvia Tol2Transposon mediated enhancer trap screen. Prog Biochem Biophys,2010,37(7): 720–727.
[25]Huang W,Deng Y,Dong W,et al. The effect of excess expression of GFP in a novel heart-specific green fluorescence zebrafish regulated bynppaenhancer at early embryonic development. Mol Biol Rep,2011,38(2): 793–799.
[26]Lekven AC,Buckles GR,Kostakis N,et al.Wnt1andwnt10bfunction redundantly at the zebrafish midbrain-hindbrain boundary. Dev Biol,2003,254(2):172–187.
[27]Hou YL,Sun B,Hou WR. cDNA cloning and sequence analysis of ribosomal protein S26 gene (rps26) from the giant panda. J Beijing Norm Univ: Nat Sci,2010,46(2):177–181 (in Chinese).侯怡铃,孙冰,侯万儒. 大熊猫核糖体蛋白 S26亚基基因 (rps26) 的cDNA克隆及序列分析. 北京师范大学学报: 自然科学版,2010,46(2): 177–181.
[28]Caballero R,Setien F,Lopez-Serra L,et al.Combinatorial effects of splice variants modulate function of Aiolos. J Cell Sci,2007,120(Pt 15):2619–2630.
[29]Bao JX,Lin HN,Ouyang YN,et al. Activity-dependent transcription regulation of PSD-95 by neuregulin-1 and Eos. Nat Neurosci,2004,7(11): 1250–1258.
[30]Pan F,Yu H,Dang EV,et al. Eos mediates Foxp3-dependent gene silencing in CD4+regulatory T cells. Science,2009,325(5944): 1142–1146.
[31]Behr M,Wingen C,Wolf C,et al. Wurst is essential for airway clearance and respiratory-tube size control. Nat Cell Biol,2007,9(7): 847–853.
[32]Gharbi N,Zhao XF,Ellingsen S,et al. Zebrafish enhancer trap line showing maternal and neural expression ofkctd15a. Dev Growth Differ,2012,54(2):241–252.
[33]Liu SJ,Shen D,Zhong JH,et al. The annotation of enhancer-trapping mediated by the SB transposon in zebrafish. Biotechnol Bull,2017,33(5): 153–158 (in Chinese).刘帅军,沈丹,钟继汉,等. SB转座子介导的斑马鱼增强子捕获注解分析. 生物技术通报,2017,33(5):153–158.
