玉米光合突变体hcf136 (high chlorophyll fluorescence 136)的转录组分析
2018-04-10吴庆飞丁泽红李平华杜柏娟
吴庆飞 秦 磊 董 雷 丁泽红 李平华 杜柏娟
1 山东农业大学农学院, 山东泰安271018; 2 河南农业大学农学院, 河南郑州450002; 3 中国热带农业科学院热带生物技术研究所, 海南海口571101
光合作用是绿色植物在可见光的照射下, 将CO2和水转化为有机物贮存能量, 并释放出O2的过程, 是植物碳同化和生物量积累的重要途径。根据CO2固定途径的不同, 高等植物可被分为 C3植物、C4植物和景天酸代谢植物(CAM), 其中 C4植物 (如玉米)的光合作用由叶肉细胞和维管束鞘细胞共同参与完成。C4植物通过特异性定位于叶肉细胞的磷酸烯醇式丙酮酸羧化酶(PEPC)完成CO2的原初固定,生成的四碳双羧酸被转移到维管束鞘细胞脱羧重新释放出CO2, 参与卡尔文循环, 形成碳水化合物[1]。与C3植物相比, C4植物的叶绿体结构和功能具有二型性[2]。玉米叶肉细胞叶绿体富含基粒, 具有高PSII活性, 有利于线性电子传递与NADPH的合成, 但缺少 Rubisco活性, 不能积累淀粉[3-4]; 而维管束鞘细胞叶绿体不形成基粒, 缺少PSII活性[5-6], 主要进行环式光合电子传递合成 ATP, 积累核酮糖-1,5-二磷酸, 包含大量的淀粉颗粒。正是由于这种叶绿体结构及光合酶的特异分化, C4植物具有CO2同化率高,水、氮利用率高, 光呼吸弱等特点, 特别是在高温、干旱等逆境条件下比C3植物小麦、水稻等具有较高的生物产量[7-8]。因此, 研究 C4光合调控机制, 将其转移到C3作物, 提高C3作物的光合效率和籽粒产量,一直是国内外光合研究领域的前沿问题。
筛选和挖掘调控叶肉细胞叶绿体与维管束鞘细胞叶绿体特异分化的关键基因, 对深入了解C4光合机理至关重要。玉米是典型的 C4植物, 是研究 C4光合的模式材料, 利用玉米光合突变体解析叶绿体发育机理对理解C4光合具有重要意义, 然而已鉴定的玉米叶肉细胞和维管束鞘细胞缺陷突变体很少[9-11]。Covshoff等[12]利用AC/DS转座子系统筛选鉴定了一个玉米光合突变体hcf136, 该突变体叶肉细胞叶绿体不能形成基粒且丧失了PSII活性, 而维管束鞘叶绿体细胞发育无较大变化。在hcf136突变体叶肉细胞中, 几乎检测不到 PSII反应中心的核心亚基,而 PSI的活性不受影响; 突变体幼苗的捕光复合体主要以单聚体的形式存在, 而野生型中则以典型的三聚体形式存在, 此外, 突变体中编码 PSII核心复合体核心亚基(如psbB、psbH、psbN、psbT)与cytb6复合体(petB、petD)的psbB-psbH-psbT-petB- petD多顺反子的形成受阻。表明突变体中HCF136功能缺失, 致使 PSII的组装过程与稳定状态受到破坏, 该功能与之前报道的拟南芥同源基因ATHCF136功能相一致[13]。此外, Covshoff等[12]利用微阵列(Microarray)技术对突变体和野生型叶肉细胞和维管束鞘细胞进行转录组分析, 发现PSII缺失对叶肉细胞及维管束鞘细胞转录组的变化是特异的, 只有一小部分的基因变化趋势是一致的, 表明转录调控在C4光合两种细胞生化过程中起着至关重要的作用。但由于玉米基因组测序在2008年尚未完成, 并非所有表达基因都含括在 Microarray芯片上, 并且由于Microarray本身的局限性, 基因家族成员间存在交叉杂交现象, 因而有必要利用新技术进一步全面分析hcf136的转录组变化情况。
高通量测序技术的发展为转录组研究提供了新的契机。新一代转录组测序技术(RNA-Seq)几乎能够检测到细胞中全部的转录本, 可以在全基因组水平上提供所有基因的表达信息, 进而通过有效的基因剔选, 使人们可以迅速获得大量在某一特定组织或细胞中优势表达的基因, 以及在某个生物学过程中显著富集的基因; 这对于研究特定组织和细胞的生物学功能具有重要的指导意义[14]。与微阵列技术相比, RNA-Seq具有动态范围更大、背景噪音更低、能检测和定量先前未知的转录本及亚型等三大优点,因而 RNA-Seq技术在玉米 C4光合研究中被广泛应用并取得了一定进展[15-17]。本研究利用RNA-Seq转录组测序技术, 分析不同光强(高光480 µmol m-2s-1和低光80 µmol m-2s-1)下生长的野生型和hcf136突变体不同叶片部位的基因表达情况, 探究HCF136对光合作用的影响, 以期增加对C4光合调控机理的认识, 并为进一步深入研究HCF136功能提供参考。
1 材料与方法
1.1 材料种植及取样
随机选取160粒B73遗传背景的玉米HCF136+/-自交的种子, 置于 7 cm×7 cm×8 cm (长×宽×高)红色育苗盆, 再将80个育苗盆分别置高光与低光培养箱中。高光光强为480 µmol m-2s-1, 低光光强为 80 µmol m-2s-1; 光照时间 8:00—20:00, 温度 30℃; 黑夜时间20:00至次日8:00, 温度21℃。
种植 6 d后, 分别提取高光和低光条件下种植幼苗的叶片基因组 DNA并进行 PCR鉴定, 以确定突变体、杂合体及野生型。PCR引物为Mutator特异性引物(Mu1: 5'-GCCTCCATTTCGTCGAATCCC-3'; Mu2: 5'-GCCTCTATTTCGTCGAATCCG-3') 及HCF136基因特异性引物(HCF136-F: 5'-ACCCCTC CCATGGTATGACG-3'; HCF136-R: 5'-TTGCACCG CTAATGCCACTT-3')。根据基因型(PCR 鉴定)的结果, 参照江芳等[18]的方法选择hcf136突变体和野生型的叶片进行RNA-Seq取样: 种植8 d后, 于上午10:00切取幼苗第2叶暴露于光下部分(+4 cm, 从叶尖向下量4 cm, 切取0.5 cm)及未见光叶片部分(-1 cm, 第1叶叶枕部位向下, 切取0.5 cm), 立即用液氮冻存, 待RNA提取建库。
1.2 光合参数及叶绿素含量的测定
高光条件下播种12 d后, 对突变体、杂合体及野生型的发育成熟的第3叶, 选用CIRAS-2便携式光合测定系统(PP Systerms, 美国)测定光合参数, 包括净光合速率(Pn)、气孔导度(Gs)、胞间CO2浓度(Ci)、蒸腾速率(E), 测定条件为光强 1600 µmol m-2s-1,CO2浓度为400 µmol mol-1。用80%丙酮4℃黑暗浸泡叶片72 h, 取提液按照Porra[19], 用UV-1601紫外分光光度计(Shimadzu, 日本)测定叶绿素含量。采用FMS-2型便携脉冲调制式荧光仪(Hansatech, 英国)测定叶绿素荧光。30 min暗适应后, 用极其弱的测量光测定叶片初始荧光Fo, 接着打开强的饱和脉冲光(8000 µmol m-2s-1), 作用0.7 s, 测定暗适应下的最大荧光Fm。
1.3 RNA的提取及RNA-Seq文库的构建
采用TRIzol (Invitrogen, USA)法提参照说明书取叶片总 RNA, 提取过程中所用器具和耗材都经过RNase free处理。为尽量消除个体间的差异, 以5株相同基因型植株混合取样, 共 3次生物学重复。以琼脂糖凝胶电泳检测总 RNA完整性, NaNo-Drop1000 (微量紫外分光光度计)检测 RNA 的纯度和浓度。参照Wang等[20]方法构建RNA-Seq的文库。由北京贝瑞和康生物科技有限公司提供转录组测序,测序平台为 Illumina HiSeq 2500。
1.4 RNA-Seq数据的整合与分析
测序后得到的原始 reads, 参照 Jiang等[18]的方法, 利用FASTX-toolkit去除含有接头、重复的和测序质量很低的 reads后, 得到 clean reads, 并用FastQC检测测序的质量。采用tophat软件(线程=6,碱基错配=2)将clean reads比对到Phytozome网站上B73基因组(AGPv3版本)。采用 cuffdiff软件(线程=8, 允许基因组校正‘-frag-bias-correct’)计算基因表达量, 用FPKM (Fragments per kb per million fragments)表示, 其中 FPKM>1的基因被认为是表达的基因。利用Edge R[21]软件根据野生型和突变体的比较(FDR<0.01)确定差异表达基因。利用 MapMan软件(Fisher exact test,P< 0.05)参照 Li等[22]进行功能富集分析。
1.5 荧光定量PCR
对每个样品的总 RNA利用 SuperRT cDNA Synthesis Kit (购于北京康为世纪生物科技有限公司)反转录试剂盒合成cDNA第1链。利用UltraSYBR Mixture (High ROX)试剂盒(购于北京康为世纪生物科技有限公司)在Bio-Rad公司荧光定量PCR仪上采用SYBR green I荧光染料法进行qRT-PCR。以玉米中的 18S rRNA基因作为内参基因[23], 并设计内参引物(Zm18S-F: 5'-CAATGGAGATGGCTCGACTT-3';Zm18S-R: 5'-GTTGCACTGCGAGCATACAT-3')采用相对定量 2-ΔΔCt法分析结果。共 3次生物学重复, 4次技术重复, 参照Li等[22]的qRT-PCR引物。
2 结果与分析
2.1 HCF136基因突变导致玉米幼苗黄化死亡
hcf136突变体最初由美国俄勒冈大学的 Alice Barkan教授实验室筛选所得, 该突变体是由Mutator转座子插入引起。在高光条件下, 种植8 d后, 可以发现突变体叶片明显呈现淡绿色表型(图1-A), 大约17 d左右突变体死亡, 而野生型生长正常(图1-B)。
为了进一步确定Mutator插入位点, 选用基因特异引物及Mutator特异引物对突变体进行PCR鉴定(图 1-C), PCR产物测序发现Mu转座子插入GRMZM2G102838第2外显子(在ATG下游409 bp处), 并造成9 bp插入位点的正向重复(图1-D)。此位点不同于Covshoff等[12]发现的Ac/Ds突变体(插入位点在GRMZM2G102838第6外显子)。此外PCR鉴定(图1-C)结果表明在高光下出苗的80棵幼苗中,21棵黄化苗全为突变体, 59棵绿苗中有35棵为杂合体, 24棵为野生型。卡方检验(21∶35∶24=1∶2∶1;χ2= 1.475 <χ20.05=5.99;P>0.95)表明, 该黄化表型确实为孟德尔遗传隐性单基因控制的性状。
2.2 HCF136功能缺失影响了玉米幼苗的光合作用
突变体叶绿素a、b的含量明显低于野生型(与其黄化表型相一致), 净光合速率为负值, 且胞间CO2浓度明显高于野生型(表1)。表明该突变体只能进行呼吸作用, 是一个典型的光合功能缺失突变体,这也解释了为何hcf136突变体幼苗致死。此外, 野生型中Fv/Fm为0.81左右, 而突变体的Fv/Fm几乎为0, 说明在突变体中PSII的活性丧失, 这与之前报道的玉米hcf136Ds插入突变体结果相一致, 表明hcf136作为一个辅助因子在PSII组装过程中扮演着重要角色[12]。
2.3 RNA-Seq及差异表达基因功能富集分析
共构建了24个RNA-Seq文库, 每个样本包含3个生物学重复。但低光野生型-1 cm和+4 cm各有一个库构建失败, 因而最终只有 22个库用于后继分析, 除低光野生型样品只有 2个生物学重复外,其余样品均3个生物学重复。各样本生物学重复之间的相关性较高, 相关系数为 0.968~0.996。测序后共得到315 M原始reads, 在移除含有接头、重复和低测序质量的 reads后共得到了 210 M干净的 reads,其中大约 91% (191 M)被比对到玉米 B73 (AGPv3)基因组外显子上, 表明测序质量良好, 数据可用于后续分析。

图1 hcf136突变体的表型及基因型鉴定Fig.1 Phenotype and gene identification in the hcf136 mutantA: 种植8 d后的玉米幼苗, WT代表野生型, HET代表杂合体, hcf136为突变体; B: 种植17 d后的幼苗; C: hcf136突变体的PCR鉴定,用HCF136基因特异引物(F, R)与Mutator特异引物(Mu1, Mu2)进行PCR, M为DL2000 marker; D: Mu转座子的插入位置。下画线部分为插入造成的9 bp正向重复序列。A: Eight_day seedlings; WT, HET, and hcf136 represent the wild type, heterozygous, and mutant, respectively; B: 17_day seedlings;C: hcf136 confirmed and sequenced by PCR with specific primers of HCF136 gene (F, R) and Mutator (Mu1, Mu2), M is DL2000 marker;D: Mutator transposon insertion site in HCF136, and the underlines shows 9 bp forward repeat sequence caused by Mu insertion.
共有24 874个基因在玉米叶片表达。对野生型和突变体在不同光照处理及不同发育阶段的叶片切段进行转录组差异比较分析发现, 共有8949个基因差异表达(图2), 其中+4 cm高光(5552) > -1 cm高光(5000) > +4 cm 低光(476) > -1 cm 低光(185)。可以明显地看出, 在高光条件下, 无论是+4 cm部位还是-1 cm部位差异基因的个数都显著多于在低光条件下的差异基因个数, 表明光照强度对HCF136的功能有着极显著的影响。而且, 在相同光照条件下, +4 cm的基因差异个数均多于-1 cm, 说明叶片不同的发育阶段对光照响应的敏感度不同。此外, 在 4种条件下表达量均发生显著差异变化的基因共有 30个(表 2), 约占差异基因总数的 0.3%。其中包括HCF136, 表明这些基因与HCF136关系较为紧密。
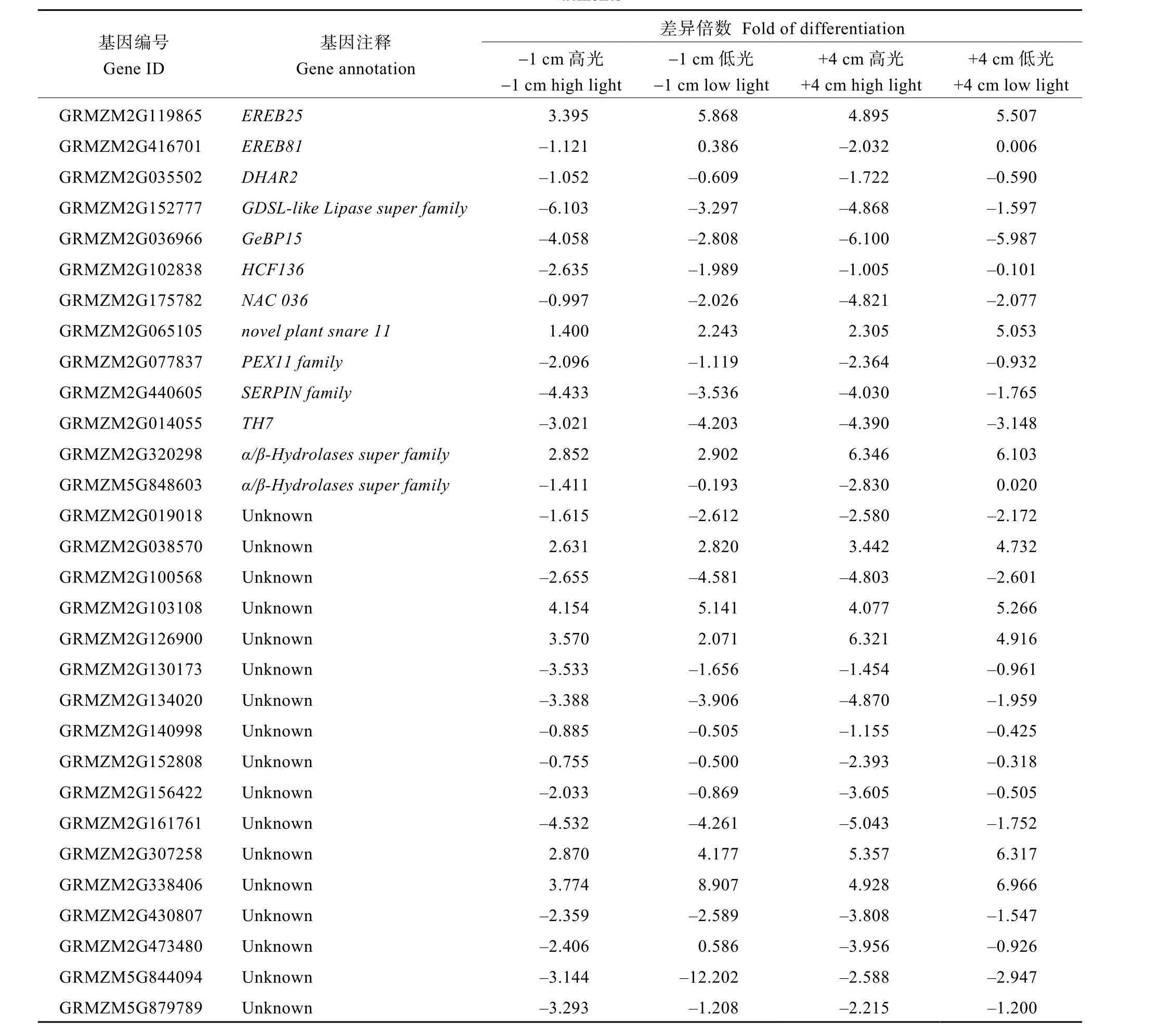
表2 野生型和突变体植株中显著差异表达的基因Table 2 Differentially expressed genes in wild type (WT) and mutant (hcf136) in both -1 cm and +4 cm under both high and low light treatments
如表3所示, 在突变体-1 cm部位, 高光条件下上调和下调的基因个数分别为2979个和2021个,低光条件下上调和下调的基因个数分别为 70个和115个, 2种光照条件下均上调和下调的基因个数分别为60个和41个; 而在突变体+4 cm部位, 高光条件下上调和下调的基因个数分别为 2838个和2714个, 低光条件下上调和下调的基因个数分别为347个和129个, 2种光照条件下均上调和下调的基因个数分别为 212个和 117个。就突变体转录组整体变化趋势而言, 除低光条件下-1 cm部位下调基因个数多于上调基因个数, 其他处理条件下的转录组变化均呈现上调基因个数多于下调基因个数。

表3 不同条件下差异表达基因的上调或下调数量Table 3 Number of up-regulated or down-regulated differentially expressed genes under different treatments
由图3可以明显地看出, 在高光条件下(无论+4 cm部位还是-1 cm部位)富集的代谢通路要显著多于低光条件下富集的代谢通路。且在高光条件下, +4 cm部位和-1 cm部位中, 非生物胁迫途径富集的最为显著, 其次是细胞壁代谢途径, 这可能是高光条件下突变体发生了光抑制引起的一些次级代谢反应。+4 cm部位中氨基酸代谢降解、主要碳水化合物代谢。降解、转运糖等途径在高光和低光上调的基因中显著富集; 而次要碳水化合物的代谢(棉籽糖家族、胼脂质家族)、线粒体电子传递等途径在下调的基因中显著富集; 且-1 cm部位中, 卡尔文循环及四吡咯化合物的合成等途径均在高光及低光中下调的基因中富集, 表明HCF136基因的缺失直接影响光合作用及碳水化合物的代谢过程。此外发现光呼吸途径只在-1 cm低光中上调的基因中富集, 说明这些基因的功能可能与发育部位及光照强度关系紧密。
2.4 光合作用通路的变化
HCF136作为PSII的组装因子, 其功能的缺失,使得突变体不能形成正常的 PSII, 无法进行正常的线性电子传递; 但 PSI的发育几乎不受影响, 且保持着正常的环式电子传递功能。因此,HCF136在光合作用中扮演着重要的角色。我们十分关注突变体中, 与光合作用相关基因的转录变化情况。由于+4 cm部位叶片发育成熟, 是光合作用的主要部位, 我们着重分析了该部位的转录变化情况。我们发现,无论在高光还是低光生长条件下,hcf136突变体+4 cm部位, 只有2个参与光合卡尔文循环的基因磷酸核酮糖激酶(PRK, GRMZM2G463280)与NADP-苹果酸脱氢酶(NADP-ME, GRMZM2G085019)表达显著上调, 而并未发现其他明显下调的基因, 尤其是我们特别关心的PSII相关基因转录水平未发生较大变化; 然而, 许多参与蛋白质降解的基因显著上调,这表明在突变体中, PSII复合体的缺失可能是蛋白翻译后的降解所致, 而非转录水平变化所引起。同时参与叶绿体合成的TOC159(GRMZM2G389118)基因的表达在突变体中明显上调, 有可能在缺失HCF136导致蛋白积累受阻情况下, 植物体通过提高TOC159的表达, 促进叶绿体蛋白的转运, 从而维持叶绿体合成。
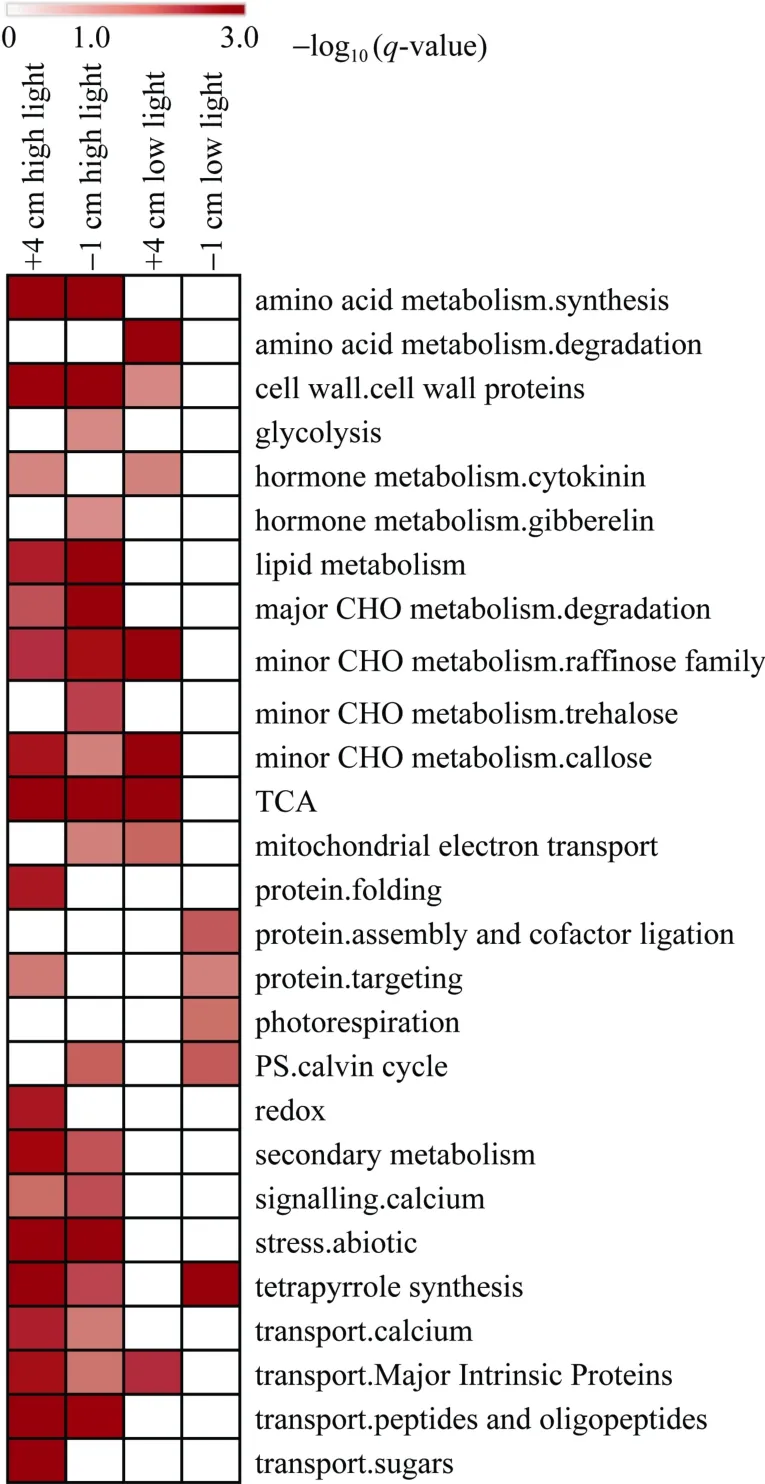
图3 差异表达基因功能富集热图Fig. 3 Functional enrichment heat map of differentially expressed genes红色表示被富集的功能途径, 红色越深代表被富集的越显著。Red color indicates the functional pathway being enriched.The deeper color, the more significant enrichment.
糖代谢通路与野生型相比发生了明显的变化(表4)。淀粉合成基因ADPGLC-PPase大亚基(APL2,GRMZM2G027955)显著下调。而参与淀粉降解的CINV2家族基因(GRMZM2G118737、GRMZM2 G170842)、BMY8(GRMZM2G035749)和BAM6(GRMZM5G803981)、糖苷水解酶基因RS6(GRMZM2G127147)等均显著上调。与此同时, 一些糖转运蛋白(如 GRMZM2G160460)的表达也显著上升。这表明, 在突变体中HCF136的缺失主要抑制了淀粉与糖的合成, 并加速其降解, 使植株不能得到供应其生长的足够养分。

表4 光合相关基因的表达变化Table 4 Expression changes of genes related to phtosynthesis
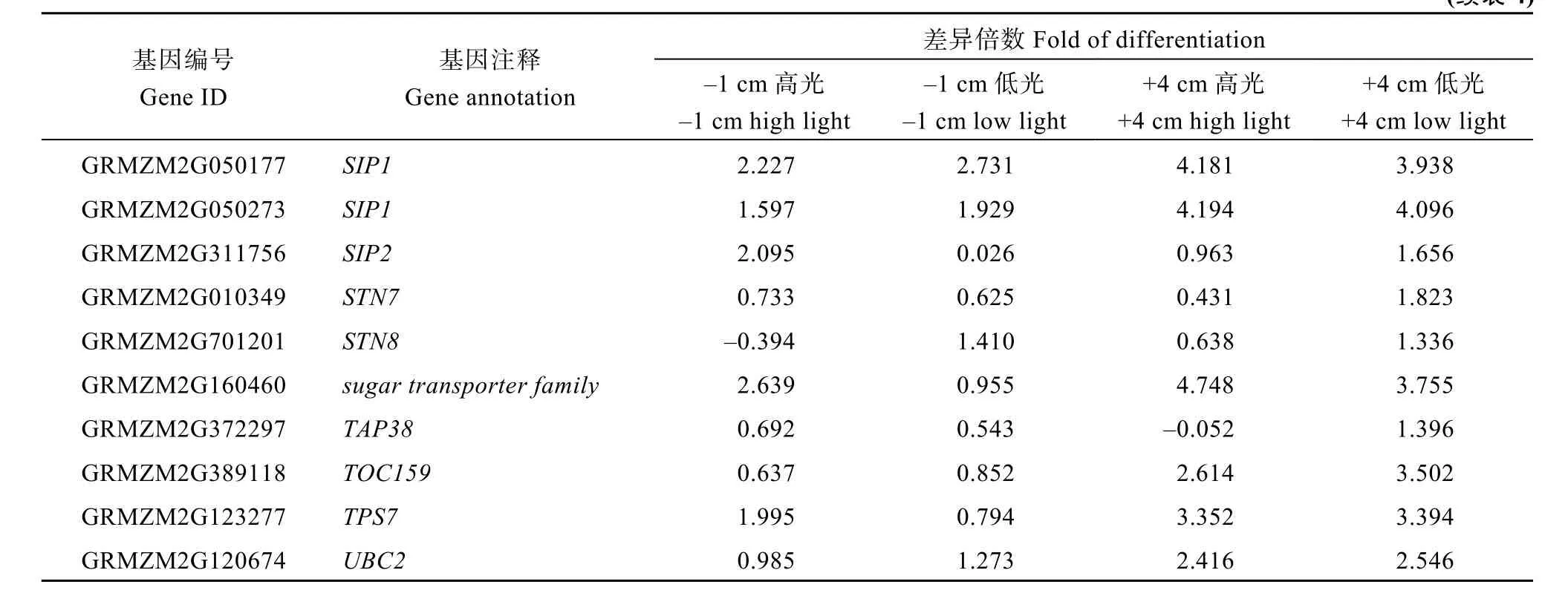
(续表 4)
Cu2+对叶绿体电子传递表现出两类抑制作用, 即依靠Fd的NADP光还原和PSII氧化侧的光还原[24-25]。有意思的是, 我们发现在突变体中, 许多铜离子转运蛋白尤其 GRMZM2G129399表达上调极为显著,在+4 cm部位, 高光和低光条件下分别上调128倍和50倍(表4)。表明在hcf136突变体中, 可能通过Cu2+的大量聚集破坏了PSII的电子传递, 继而加速其外排作用以期达到体内离子平衡作用。
2.5 HCF1 36功能缺失对转录因子的表达的影响
统计分析发现在+4 cm部位(不论高光和低光条件下), 共有31个转录因子的表达发生显著性差异变化(表5)。其中包含一些与光合相关显著下降的转录因子: 光敏色素相互作用因子PIL5(GRMZM2G 016756)与PIL6(GRMZM2G065374), 调控叶绿素合成的锌指结构转录因子GATA21(GRMZM2G 031983)、调控叶绿体特异分化的GLK1(GRMZM2G 026833)等。此外, 参与调控生长发育的NAC4(GRMZM5G898290)、调控内质网融合的RL1(GRMZM2G049378)、及参与水杨酸代谢的MYB59(GRMZM2G048136)等均呈现出显著下调。还发现一些显著上调的转录因子, 诸如调控光信号途径的RGA1(GRMZM2G023872)、参与光调控的bZIP转录因子HY5(GRMZM2G137046)、调控玉米根后期发育的转录因子NAC1(GRMZM2G114850)等。
在 4种处理条件下, 共有 4个转录因子的转录水平发生了显著变化: 包括参与乙烯信号通路的AP2家族的两个转录因子(GRMZM2G119865和GRMZM2G416701)调控生长发育的NAC036(GRMZM2G175782)及 1个参与细胞分裂素负调控的GeBP家族转录因子(GRMZM2G036966); 其中,相对于其他3个下调的转录因子, 只有1个AP2家族的转录因子(GRMZM2G103108)显著上调, 表明HCF136功能的缺失对其分别具有直接的正调控和负调控作用。值得一提的是, 在-1 cm (不论高光和低光条件), 发生差异变化的转录因子只有 5个, 除上述 4个转录因子外, 还有 1个参与幼胚发育的EMB2219(GRMZM2G426154)表达上调(表 5)。
2.6 荧光定量PCR验证RNA-Seq结果
为了进一步验证RNA-Seq结果的可靠性, 我们对15个不同的基因进行了qRT-PCR定量分析。对qRT-PCR与RNA-Seq结果进行相关性分析, 发现-1 cm部位的R2= 0.832, +4 cm部位的R2= 0.845, 说明RNA-Seq结果良好。
3 讨论
玉米是典型的C4植物, 其光合作用存在于两种形态和功能不同的光合细胞之中, 即维管束鞘细胞和叶肉细胞, 而C3植物的光合作用只存在于叶肉细胞之中。正是由于这两种光合细胞的特异分化, C4植物具有CO2同化率高, 水、氮利用率高, 光呼吸弱等特点, 特别是在高温、干旱等逆境条件下比C3植物小麦、水稻等具有较高的生物产量[7-8]。一直以来,解析C4植物2种光合细胞特异分化的调控机理都是光合研究的热点。而玉米HCF136基因的转录主要特异定位于叶肉细胞, 该突变体叶肉细胞叶绿体不能形成基粒且丧失了PSII活性, 而维管束鞘叶绿体细胞发育无较大变化。因此, 玉米hcf136突变体对了解两种光合细胞特异分化的过程至关重要[12]。

图4 RNA-Seq与qRT-PCR结果比较Fig. 4 Comparison of results between RNA-Seq and qRT-PCRFold of differentiation代表log2 (突变体表达量/野生型表达量)。Fold of differentiation is expressed as log2 (mutant expression level/WT expression level).

表5 野生型及突变体转录因子表达比较Table 5 Expression comparison of transcription factors between wild type and mutant (hcf136)
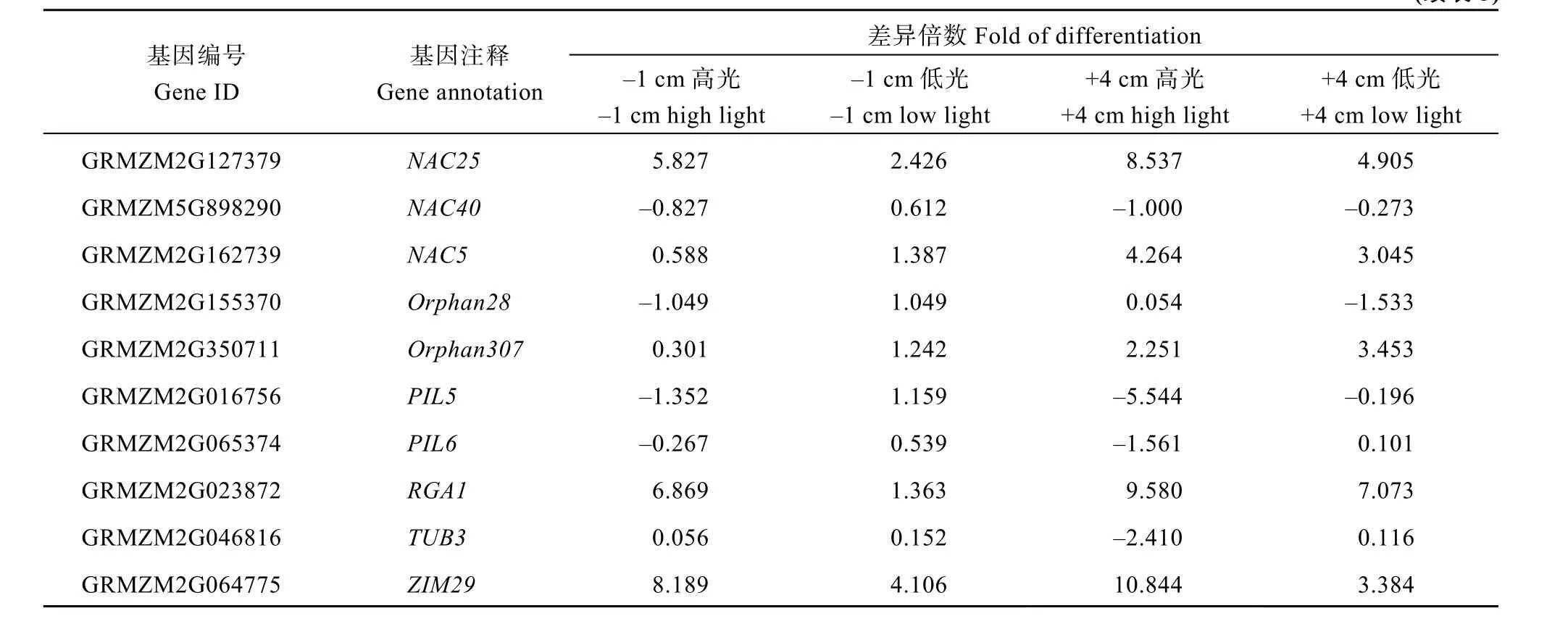
(续表 5)
美国俄勒冈大学的Alice Barkan教授向我们提供了一个hcf136突变体, 由Mu转座子插入引起, 为Covshoff等[12]发现的插入在HCF136基因第6外显子的Ac/Ds突变体的等位突变。该突变体幼苗叶片呈现淡绿色, 在2~3周间死亡。突变体的叶绿素a、b的含量明显低于野生型(与其黄化表型相一致), 净光合速率为负值, 且胞间 CO2浓度比野生型明显升高(表 1), 表明该突变体只能进行呼吸作用, 是一个典型的光合功能缺失突变体。此外, 野生型中Fv/Fm为0.81左右, 而突变体的Fv/Fm几乎为0, 说明在突变体中PSII的活性丧失, 这也与之前的报道相一致,表明HCF136作为一个辅助因子在PSII组装过程中扮演着重要角色[12]。
在hcf136突变体+4 cm部位(无论高光和低光条件), 只发现参与卡尔文循环的磷酸核酮糖激酶(PRK, GRMZM2G463280)与 NADP-苹果酸脱氢酶(GRMZM2G085019) 2个基因表达显著上调, 而并未发现其他明显下调的基因。尤其是我们特别关心的PSII相关基因转录水平未发生较大变化, 而参与蛋白质降解的许多基因显著上调, 表明在突变体中,PSII复合体的缺失不是转录水平变化所引起, 这也与之前的报道相一致[26]。而突变体中变化最明显的是参与淀粉降解的主要基因显著上调, 表明在突变体中HCF136的缺失主要抑制了淀粉与糖的合成,并加速其降解, 使得植株不能得到供应其生长足够的养分, 继而呈现死亡表型。
此外, 突变体中许多铜离子转运蛋白尤其GRMZM2G129399表达上调极为显著, 在+4 cm部位, 高光和低光条件下分别上调128倍和50倍。表明在hcf136突变体中, 可能通过Cu2+的大量聚集破坏了PSII的电子传递, 继而加速其外排作用以达到体内离子平衡。
在突变体中, 无论是+4 cm部位还是-1 cm部位差异基因的个数, 都显著多于在低光条件下的差异基因个数, 表明光照强度对HCF136的功能有着极为显著的影响。究其原因, 可能与HCF136自身的特性相关: 正常情况下, 光合作用过程中, 叶绿素荧光产量只占吸收光能的1%~2%, 而当涉及光合过程的关键基因产生突变时, 叶绿素吸收的光能无法有效用于光合作用, 导致荧光产量上升, 表现为高叶绿素荧光表型HCF (highchlorophyll fluorescence)[27-28]。造成高叶绿素荧光表型的突变, 主要由光合电子传递阻断引起[29]。而HCF136功能的缺失使PSII不能正常组装, 在高光胁迫下, PSII的损伤-修复循环不能正常进行。相比低光条件, 高光条件下突变体中+4 cm部位与-1 cm部位差异表达基因更多。
转录因子对玉米光合的调控起着重要作用。我们发现在+4 cm 部位(不论高光和低光条件下)一些转录因子发生显著变化, 如调控叶绿素合成的锌指结构转录因子GATA21、调控叶绿体特异分化的GLK1等都显著下调, 而调控光信号途径的RGA1、参与光调控的bZIP转录因子HY5、调控玉米根系后期发育的转录因子NAC1的表达也显著上调, 表明突变体中一系列生长发育过程受到影响。同时, 在4种处理条件下, 共有 4个转录因子的转录水平发生了显著变化: 包括参与乙烯信号通路的AP2家族的2个转录因子, 调控生长发育的NAC036、及1个参与细胞分裂素负调控的 GeBP家族转录因子, 表明这 4个转录因子不受光强的诱导, 且与HCF136功能紧密相关。这些差异表达转录因子的调控作用有待于进一步研究, 并将有助于了解玉米两种光合细胞特异分化的机理。
4 结论
hcf136突变体中PSII相关基因的转录未发生明显差异, 表明PSII复合体的缺失是非转录水平变化所引起。突变体中淀粉合成受阻, 糖降解、糖转运及 Cu2+转运等代谢过程加剧, 且一些转录因子表达发生显著变化。该结果在转录水平上揭示了HCF136基因的功能及其在玉米C4光合作用中的重要性, 为进一步揭示C4光合机理提供了理论依据。
致谢:感谢美国俄勒冈大学的 Alice Barkan教授给予的hcf136突变体及在实验中提供的帮助。
[1] Furbank R T, Hatch M D. Mechanism of C4photosynthesis: the size and composition of the inorganic carbon pool in bundle sheath cells.Plant Physiol, 1987, 85: 958-964
[2] Hatch M D. C4photosynthesis: a unique blend of modified biochemistry, anatomy and ultrastructure.Biochim Biophy Acta,1987, 895: 81-106
[3] Edwards G E, Franceschi V R, Ku M S, Voznesenskaya E V,Pyankov V I, Andreo C S. Compartmentation of photosynthesis in cells and tissues of C4plants.J Exp Bot, 2001, 52: 577-590
[4] Majeran W, Friso G, Ponnala L, Connolly B, Huang M, Reidel E.Structural and metabolic transitions of C4leaf development and differentiation defined by microscopy and quantitative proteomics in maize.Plant Cell, 2010, 22: 3509-3542
[5] Gregory R P, Droppa M, Horváth G, Evans E H. A comparison based on delayed light emission and fluorescence induction of intact chloroplasts isolated from mesophyll protoplasts and bundle-sheath cells of maize.Biochem J, 1979, 180: 253-256
[6] Takabayashi A, Kishine M, Asada K, Endo T, Sato F. Differential use of two cyclic electron flows around photo-system I for driving CO2-concentration mechanism in C4photosynthesis.ProcNatl Acad Sci USA, 2005, 102: 16898-16903
[7] Richard R A. Selectable traits to increase crop photosynthesis and yield of grain crops.J Exp Bot, 2000, 51: 447-458
[8] Zhu X G, Long S P, Ort D R. What is the maximum efficiency with which photosynthesis can convert solar energy into biomass.Curr Opin Biotechnol, 2008, 19: 153-159
[9] Stern D B, Hanson M R, Barkan A. Genetics and genomics of chloroplast biogenesis: maize as a model system.Trends Plant Sci, 2004, 9: 293-301
[10] 李保珠, 赵孝亮, 彭雷. 植物叶绿体发育及调控研究进展. 植物学报, 2014, 49: 337-345 Li B Z, Zhao X L, Peng L. Research advances in the development and regulation of plant chloroplasts.Chin Bull Bot, 2014, 49:337-345 (in Chinese with English abstract)
[11] Belcher S, Williams-Carrier R, Stiffler N, Barkan A. Large-scale genetic analysis of chloroplast biogenesis in maize.Biochim Biophys Acta, 2015, 1847: 1004-1016
[12] Covshoff S, Majeran W, Liu P, Kolkman J M, van Wijk K J,Brutnell T P. Deregulation ofmaize C4photosynthetic development in a mesophyll cell-defective mutant.Plant Physiol,2008, 146: 1469-1481
[13] Plucken H, Muller B, Grohmann D, Westhoff P, Eichacker L A.The HCF136 protein is essential for assembly of the photosystem II reaction center inArabidopsis thaliana.FEBS Lett, 2002, 532:85-90
[14] Schuster S C. Next-generation sequencing transforms today’s biology.Nat Methods, 2008, 5: 16-18
[15] Chang Y M, Liu W Y, Shi A C, Shen M N, Lu C H, Lu M Y.Characterizing regulatory and functional differentiation between maize mesophyll and bundle sheath cells by transcriptomic analysis.Plant Physiol, 2012, 160: 165-177
[16] John C R, Smith-Unna R D, Woodfield H, Covshoff S, Hibberd J M. Evolutionary convergence of cell-specific gene expression in in-dependent lineages of C4grasses.Plant Physiol, 2014, 165:62-75
[17] Tausta S L, Li P, Si Y T, Gandotra N, Liu P, Sun Q. Developmental dynamics of Kranz cell transcriptional specificity in maize leaf reveals early onset of C4-related processes.J Exp Bot, 2014, 65:3543-3555
[18] 江芳, 丁泽红, 董雷, 李平华. 玉米光合突变体bsd2(bundle sheath defective II)的转录组分析. 植物生理学报, 2016, 52:1214-1222 Jiang F, Ding Z H, Dong L, Li P H. Transcriptome analysis on the maize photosynthetic mutantbsd2 (bundle sheath defective II).Plant Physiol J, 2016, 52: 1214-1222 (in Chinese with English abstract)
[19] Porra R J. The chequered history of the development and use of simultaneous equations for the accurate determination of chlorophyllaandb.Photosynth Res, 2002, 73: 149-156
[20] Wang L, Si Y, Dedow L K, Shao Y, Liu P, Brutnell T P. A low cost library construction protocol and data analysis pipeline for Illumina-based strand-specific multiplex RNA-seq.PLoS One, 2011,6: e26426
[21] Robinson M D, McCarthy D J, Smyth G K. Edge R: a bioconductor package for differential expression analysis of digital gene expression data.Bioinformatics, 2010, 26: 139-140
[22] Li P, Ponnala L, Gandotra N, Wang L, Si Y, Tausta S L, Kebrom T H, Provart N, Patel R, Brutnell T P. The developmental dynamics of the maize leaf transcriptome.Nat Genet, 2010, 42:1060-1067
[23] Suresh V K, Tellabati M, Nelli R K, White G A, Perez B B,Sebastian S, Slomka M J, Brown I H, Stephen P D, Kin C C. 18S rRNA is a reliable normalisation gene for real time PCR based on influenza virus infected cells.Virl J, 2012, 9: 230
[24] Yruela I, Montoya G, Picorel R. The inhibitory mechanism of Cu(II) on the photosystem II electron transport from higher plants.Photosynth Res, 1992, 33: 227-233
[25] Ouzounidou G, Mousbakas M, Karataglis S. Responses of maize(Zea maysL.) plants to copper stress: IR growth, mineral content and ultrastructure of roots.Environ Exp Bot, 1995, 2: 167-176
[26] Meurer J, Plücken H, Kowallik K V, Westhoff P. A nuclear-encoded protein of prokaryotic origin is essential for the stability of photosystem II inArabidopsis thaliana.EMBO J,1998, 17: 5286-5297
[27] Maxwell K, Johnson G N. Chlorophyll fluorescence: a practical guide.J Exp Bot, 2000, 51: 659-668
[28] Meurer J, Meierhoff K, Westhoff P. Isolation of high-chlorophyllfluorescence mutants ofArabidopsis thalianaand their characterization by spectroscopy, immunoblotting and Northern hybridization.Planta, 1996, 198: 385-396
[29] Varotto C, Pesaresi P, Maiwald D. Identification of photosynthetic mutants ofArabidopsisby automatic screening for altered effective quantum yield of photosystem II.Photosynthetica, 2000,38: 497-504
