精子与肿瘤细胞融合模型的建立
2018-04-09靳广毅林桂淼刘好许改霞王晓梅
陈 强 靳广毅 林桂淼刘 好许改霞王晓梅*
(1. 深 圳大学医学院生理学教研室,广东 深圳 518060;2. 深圳大学光电子器件与系统教育部重点实验室,广东 深 圳 518060)
多年以来,人们普遍接受男性体细胞表现为XY基因型,而女性则表现为XX基因型。随着分子生物学和遗传学研究方法的进步,科学家在许多女性的不同组织和器官中均发现了含有Y染色体的细胞,这些细胞被称为微嵌合体(microchimerism)。
关于微嵌合体的形成机制目前学术界存在不同的观点。Nelson[1-2]认为微嵌合体可能来源于通过胎盘屏障而滞留于母体的男性胎儿细胞,或者来源于男性胎儿游离DNA与母体体细胞的融合。然而这种观点在随后的一些临床和实验研究中受到极大的挑战,因为许多研究发现微嵌合体不仅存在于孕育过男胎的母亲,也存在于未受孕过的女性[3-5]。
近年来,微嵌合体在许多女性多个组织和器官中的发现提示精子与女性体细胞发生融合的可能性,而一些女性肿瘤组织相对于周边正常组织有较高的嵌合细胞比例提示精子可能更容易与肿瘤细胞发生融合[6-7]。然而,目前尚未见精子与肿瘤细胞体外融合的实验报道。据此,我们以小鼠精子和肿瘤细胞系为研究对象,体外建立精子与肿瘤细胞融合模型,并对该模型的生物特征进行了初步研究。
1 材料与方法
1.1 主要试剂和仪器
本研究中使用的主要仪器和试剂包括:FV10i型共聚焦显微镜(Olympus),35 mm培养皿(Corning Costar),滤孔直径40 μm的过滤皿(Thermo Scientific),嵌套式的培养皿/板(Corning Costar),35 mm共聚焦皿(皿底玻璃片直径10 mm,MatTek),DAPI(Sigma)以及DiI羰花青染料(Beyotime)等。
1.2 肿瘤细胞培养
实验中所用细胞系(表1)均为深圳大学医学院纳米磁实验室保存,细胞维持及冻存、复苏均按照美国模式培养物集存库(American Type Culture Collection,ATCC) 推荐的方法进行。

表1 实验中涉及的细胞系及其培养条件
1.3 精子与肿瘤细胞的共培养基
以RPMI 1640和Ham’s F10的混合培养基为基础,我们合成了一种新型的供精子与体细胞或肿瘤细胞共培养的培养基,并命名为STCM(sperm-tumor co-culture medium)。在STCM中加入不同浓度的胎牛血清白蛋白(bovine serum albumin,BSA)分别配制成获能液(含3%BSA)和共培养液(含0.3% BSA)。我们对STCM和传统使用的RPMI 1640在支持肿瘤生长及支持精子与肿瘤细胞融合特性(融合率和融合时间)进行了比较实验。
1.4 小鼠精子的获取及获能处理
取8~10周龄雄性C57BL/6小鼠(购自广东省医学实验动物中心,实验动物检疫编号44007200016312),饲养于SPF级动物房,半昼半夜,自由取食、取水。经1周左右适应期后的动物用于精子获取。实验前一天准备获能液于35 mm培养皿中,并将培养皿置于37℃、CO2体积分数为5%的培养箱内平衡过夜。
常规切取小鼠附睾组织,并置于平衡过夜后的获能液中,在无菌条件下剪碎附睾组织后置于滤孔直径为40 μm的过滤皿中过滤,将过滤后的精子吸入1.5 mL EP管并置于培养箱内静置,使精子上游15 min,具体过程参照世界卫生组织(World Health Organization,WHO)推荐的步骤[8]进行。取出EP管,吸取上层运动活跃的精子用于共培养实验。
1.5 共培养前的准备
共培养实验前将待实验细胞接种于35 mm培养皿,细胞接种密度为30~40/mm2,待单个集落的细胞数量为4~8个时进行共培养实验。共培养实验前以Hanks液清洗接种细胞的培养皿3次,每次5 min,然后向培养皿内添加500 μ L共培养基,准备与精子共培养。
1.6 精子与肿瘤细胞的共培养及对照研究
取5 μL经上游处理后的精子于精子分析仪进行精子计数。另取适量精子(精子与肿瘤细胞的配比为40∶1~50∶1)置于接种肿瘤细胞的35 mm培养皿并补足液体总量至1.5 mL。然后迅速将培养皿置于37 ℃、CO2体积分数为5%的培养箱内进行共培养。实验设置0.5、1、1.5和2 h共4个观察点,于倒置相差显微镜下观察细胞融合情况并进行拍照。
本研究设立了空白对照和嵌套式培养两个对照研究。嵌套式的培养皿/板能够在屏蔽精子和肿瘤细胞直接接触的同时而使两种细胞处于同一培养体系中。
1.7 嵌合型肿瘤细胞的共聚焦显微镜成像
将肿瘤细胞接种于35 mm共聚焦皿。肿瘤细胞与精子的共培养实验按常规进行。实验共持续4 h,每隔30 min设立1个观察点。在每个观察点首先常规固定残存于培养皿底部的肿瘤细胞,然后以0.1 mg/mL DAPI和10 μmol/L DiI羰花青染料分别染色细胞核和细胞膜10 min。以FV10i型共聚焦显微镜进行成像。三维共聚焦成像时,首先进行分层扫描,然后以成像系统自带软件进行三维重建,进行单个三维共聚焦成像需进行30~40层的分层图像。
1.8 精子与肿瘤细胞融合率的估算方法
以MTT法测得的空白对照组D(490)估计参与共培养实验的肿瘤细胞总数;以共培养后残存的贴壁肿瘤细胞的D(490)估计非嵌合型肿瘤细胞的数量。这样,1-D(490)残存肿瘤细胞/ D(490)空白对照组即为精子与肿瘤细胞的融合率。MTT实验按常规方法进行。
1.9 统计学方法
精子与肿瘤细胞的融合率以均数±标准差的形式表示,以方差分析(ANOVA)的方法进行融合率的比较。所有数据的统计均以SPSS 16.0软件进行,以α=0.05为检验水准。
2 结果
2.1 精子与肿瘤细胞的融合过程
当获能精子加入共培养体系数分钟后,精子便可聚集于HeLa细胞(经多个肿瘤细胞系检测结果相似,在此仅以HeLa细胞为例进行说明)的侧向周围,并主动地贴附于HeLa细胞(图1A);当共培养时间达到1 h或以上时,部分精子能够穿越HeLa细胞的细胞膜而进入细胞内,精子的尾部尚残存于肿瘤细胞外(图1B);经过约1.5 h我们观察到呈球形改变的嵌合细胞(图1C);经过约2 h的共培养,镜下可以发现球形漂浮的肿瘤细胞,可见残存的精子尾于细胞外摆动,并推动细胞在培养液上层游动(图1D)。

图1 精子与肿瘤细胞融合过程
2.2 融合细胞的共聚焦显微镜成像
经过1.5~2 h的共培养后我们采用共聚焦显微镜成像的方法确认了精子与HeLa细胞的融合现象,如图2所示。我们从3个不同方位(矢状面、冠状面和侧面)进行了拍摄,均证实了精子已经进入HeLa细胞内部。同时我们发现个别肿瘤细胞能够与两个精子同时发生融合(图2A),但多数的肿瘤细胞通常只与1个精子融合(图2B)。此外我们发现对于共培养时间超过3 h 的残存贴壁细胞进行三维重建并不能找到融合细胞。
2.3 共培养实验及对照研究
鉴于先前实验观察到的融合细胞离壁漂浮生长的特性,我们对空白对照组、嵌套式培养组及直接共培养组实验后残存细胞总数进行了检测。结果发现(图3):空白对照组和嵌套式培养组残存细胞的D(490)显著高于直接共培养组(p<0.05),而前两者残存细胞的D(490)之间并无显著差异(P=0.71)。
2.4 精子与肿瘤细胞融合模型的优化
新合成的STCM与RPMI 1640培养基比较发现,应用两种培养基进行肿瘤细胞培养时可以得到类似的肿瘤细胞生长曲线(图4A),但分别应用两种培养基进行精子与肿瘤细胞(HeLa、MCF-7和H441细胞系)融合实验时,与RPMI 1640培养基相比,STCM能够显著提高精子与肿瘤细胞的融合率(p<0.05,图4B),如HeLa细胞在RPMI 1640培养基中与小鼠精子的融合率为13.2%±2.9%,而在STCM中的融合率升高到21.4%±4.1%,后者显著高于前者,应用MCF-7和H441细胞系可以得到相似的结论。而且我们发现STCM能够在一定程度上加速精子与肿瘤细胞(HeLa细胞)的融合(图4C)。
3 讨论
早在100多年前Austin[9]就发现精子存在于多种动物的子宫黏膜。随后的多项研究表明:含有Y染色体的微嵌合体可存在于女性的肝脏[10]、 甲状腺[3,7]、 脑[11], 输卵管上皮[12]、 外周血淋巴细胞[13-15]以及造血和免疫细胞[16]。
关于精子与体细胞融合的体外研究报道可以追溯到上世纪70年代。1971年,Sawicki和Koprowski[17]报道在仙台病毒的作用下,兔精子能够和多种体细胞相融合。1974年Bendich在《Science》杂志上发表文章[18]证实小鼠精子能够穿透中国仓鼠成纤维细胞并形成嵌合细胞,这种嵌合细胞携带精子的遗传信息并能够在体外传代培养。次年,Higgins等[19]用大鼠的精子获得了相似的实验结果并在体外连续培养嵌合细胞达50代以上。1999年Takao等[20]发现位于卵母细胞外的卵巢颗粒细胞能够被精子穿透而形成嵌合细胞。2009年发表于《Reproduction》杂志的一篇报道[21]发现不同种属来源的肾细胞也能够和精子发生融合。

图2 嵌合型肿瘤细胞的共聚焦显微镜成像

相对于体细胞,精子可能更容易与肿瘤细胞发生融合,因为一些临床研究发现:相对于周边的正常组织,女性肺腺癌[6]和 甲状腺癌[7]组织中存在更高比例的嵌合细胞。然而,迄今为止我们尚未看到精子与肿瘤细胞体外融合的实验研究。
我们的研究以小鼠精子和多个不同种属来源的肿瘤细胞系建立共培养体系,并对精子与肿瘤细胞的融合特征进行了初步研究。采用镜下连续观察的方法,我们发现精子与肿瘤细胞的融合多发生于共培养后的1.5~2 h内,而且我们发现与精子融合的肿瘤细胞贴附能力下降,漂浮于培养基中,并在精子尾部的摆动下产生了一定的运动能力(图1)。采用共聚焦显微镜三维重建的方式我们证实了精子与肿瘤细胞融合的可能性(图2)。同时,我们发现,精子与肿瘤细胞的融合多发生于共培养的1.5~2 h内,对于共培养3 h后残存的贴壁细胞进行共聚焦显微镜观察,并没有发现融合细胞。我们推测可能是共培养3 h后绝大多数的融合细胞均漂浮于培养基的上层,因而在贴壁细胞中难于发现。
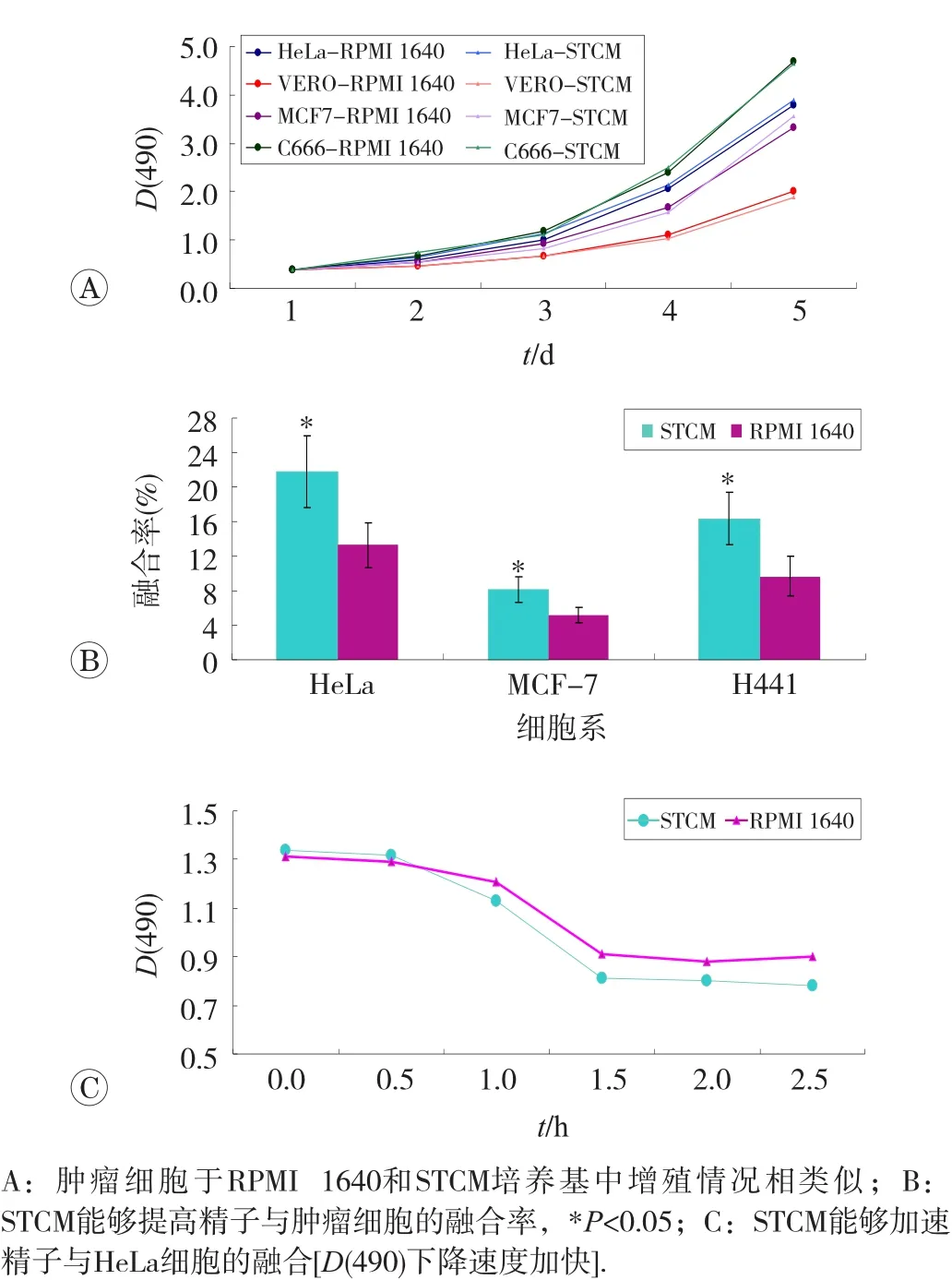
图4 STCM对精子与肿瘤细胞融合模型的优化
为排除与精子直接接触外的其他因素所导致的肿瘤细胞贴附能力的降低,我们设立了空白对照和嵌套式共培养对照研究。嵌套式共培养体系可以避免精子与肿瘤细胞的直接接触,而使两种细胞共存于同一培养体系中。结果发现,空白对照组和嵌套式培养组的残存细胞总数显著多于直接共培养组,而前两者的残存细胞总数之间并无显著差异(图3),说明非直接与精子接触不会导致肿瘤细胞贴附能力的降低,直接共培养组贴壁细胞数量的减少应为精子与肿瘤细胞融合形成的嵌合细胞的贴附能力降低而漂浮生长所致。
我们的实验结果显示:①精子与肿瘤细胞共培养2 h后光镜下可见悬浮于培养基中的嵌合细胞;②共聚焦三维成像实验提示共培养3 h后绝大多数的融合细胞已经悬浮于培养液中;③嵌套式共培养体系的对照研究显示精子与肿瘤细胞的直接接触、融合以及嵌合细胞贴附能力的降低是导致共培养后残存肿瘤细胞数量降低的直接原因,故此我们提出估算精子与肿瘤细胞融合率的方法,即以共培养3 h后残存肿瘤细胞的数量来衡量精子与肿瘤细胞的融合率。如果以MTT法估算精子与肿瘤细胞的融合率(R),那么,R=1-D(490)残存肿瘤细胞/ D(490)空白对照组×100%。
为了建立更为稳定和高效的精子与肿瘤细胞共培养体系,我们以新合成的STCM代替常规使用的RPMI 1640培养基。STCM培养基在参考常用于精子培养的HTF、TYH、BWW及BO培养基主要成分的基础上,以RPMI 1640和Ham’s F10的混合培养基为基础,添加精子培养的必需成分,使该培养基在满足细胞培养的前提下,最大化的提供精子培养所需营养成分,从而适合精子与体细胞或肿瘤细胞共培养。我们的实验结果显示STCM培养基在不影响肿瘤细胞生长和贴附的情况下(图4A),能够极大地提高精子与肿瘤细胞的融合率(图4B),并且能够在一定程度上加速精子与肿瘤细胞的融合(图4C)。这些结果提示STCM作为一种新合成的培养基更适合用于精子与肿瘤细胞融合实验研究。
综上所述,精子与体细胞或者肿瘤细胞融合是一种奇特的生物学现象。虽然我们很早就认识到了该种现象的存在,但对于它存在的价值和意义却知之甚少。在本研究中我们建立了一种稳定的精子与肿瘤细胞融合的模型,并且以多种方式证实了精子与肿瘤细胞体外融合的现象,提出了精子与肿瘤细胞融合率的估算方法,并采用新型的STCM培养基对融合模型进行了优化。虽然如此,仍然有许多的相关问题亟待解决,比如,哪些相关因素决定了精子与肿瘤细胞的融合率?精子与肿瘤细胞的融合机制与受精机制有哪些区别和联系?精子与肿瘤细胞融合后会导致肿瘤细胞生物学性状发生哪些改变?相信利用我们建立的稳定的精子与肿瘤细胞融合模型上述问题在不久的将来都会得到合理的解释与答案。
[1] NELSON J L. Microchimerism:expanding new horizon in human health or incidental remnant of pregnancy?[J]. Lancet,2001,358(9298):2011-2012.
[2] NELSON J L. Microchimerism in human health and disease[J].Autoimmunity,2003,36(1):5-9.
[3] SRIVATSA B, SRIVATSA S, JOHNSON K L, et al.Microchimerism of presumed fetal origin in thyroid specimens from women: a case-control study[J]. Lancet, 2001, 358(9298):2034-2038.
[4] YAN Z,LAMBERT N C,GUTHRIE K A,et al. Male microchimerism in women without sons:quantitative assessment and correlation with pregnancy history[J]. Am J Med,2005,118(8):899-906.
[5] BRODSKY S V,IVANOV I. Spermatozoa-somatic cell fusion--a mechanism for microchimerism formation[J]. J Theor Biol,2009,259(1):190-192.
[6] O'DONOGHUE K,SULTAN H A,AL-ALLAF F A,et al.Microchimeric fetal cells cluster at sites of tissue injury in lung decades after pregnancy[J]. Reprod Biomed Online,2008,16(3):382-390.
[7] CIRELLO V,RECALCATI M P,MUZZA M,et al. Fetal cell microchimerism in papillary thyroid cancer:a possible role in tumor damage and tissue repair[J]. Cancer Res, 2008,68(20):8482-8488.
[8] World Health Organization. WHO laboratory manual for the examination and processing of human semen[M]. 5th ed. WHO Press,2010.
[9] AUSTIN C R. Entry of spermatozoa into the fallopian-tube mucosa[J]. Nature,1959,183(4665):908-909.
[10] STEVENS A M,MCDONNELL W M,MULLARKEY M E,et al. Liver biopsies from human females contain male hepatocytes in the absence of transplantation[J]. Lab Invest, 2004,84(12):1603-1609.
[11] CHAN W F,NELSON J L. Microchimerism in the human brain:more questions than answers[J]. Chimerism,2013,4(1):32-33.
[12] PACEY A A,HILL C J,SCUDAMORE I W,et al. The interaction in vitro of human spermatozoa with epithelial cells from the human uterine (fallopian) tube[J]. Hum Reprod,1995,10(2):360-366.
[13] ASHIDA E R,SCOFIELD V L. Lymphocyte major histocompatibility complex-encoded class II structures may act as sperm receptors[J]. Proc Natl Acad Sci USA,1987,84(10):3395-3399.
[14] MIKHAIL M A, M'HAMDI H, WELSH J, et al. High frequency of fetal cells within a primitive stem cell population in maternal blood[J]. Hum Reprod,2008,23(4):928-933.
[15] GILMORE G L,HAQ B,SHADDUCK R K,et al. Fetalmaternal microchimerism in normal parous females and parous female cancer patients[J]. Exp Hematol,2008,36(9):1073-1077.
[16] KHOSROTEHRANI K,BIANCHI D W. Multi-lineage potential of fetal cells in maternal tissue:a legacy in reverse[J]. J Cell Sci,2005,118(Sup 8):1559-1563.
[17] SAWICKI W,KOPROWSKI H. Fusion of rabbit spermatozoa with somatic cells cultivated in vitro[J]. Exp Cell Res,1971,66(1):145-151.
[18] BENDICH A, BORENFREUND E, STERNBERG S S.Penetration of somatic mammalian cells by sperm[J]. Science,1974,183(4127):857-859.
[19] HIGGINS P J,BORENFREUND E,BENDICH A. Appearance of foetal antigens in somatic cells after interaction with heterologous sperm[J]. Nature,1975,257(5526):488-489.
[20] TAKAO Y,FUJIWARA H,YAMADA S,et al. CD9 is expressed on the cell surface of human granulosa cells and associated with integrin alpha6beta1[J]. Mol Hum Reprod,1999,5(4):303-310.
[21] MATTIOLI M,GLORIA A,MAURO A,et al. Fusion as the result of sperm-somatic cell interaction[J]. Reproduction,2009,138(4):679-687.
