甜菜碱改善水稻高GC含量DNA序列的PCR扩增
2018-04-08王同同张利平尹康权
王同同 张利平 尹康权
(1. 河北大学生命科学学院,保定 071002;2. 中国科学院微生物研究所植物基因组学国家重点实验室,北京 100101)
水稻是人类最主要的粮食作物之一。随着地球人口日益增长,人们对于粮食需求也日益增高,因此研究水稻增产、抗病、抗旱等各个领域均具有重要意义。自1988年以来,科学家发明了聚合酶链式反应(Polymerase chain reaction,PCR)技术[1],之后被广泛应用于分子生物学研究领域。对水稻DNA序列的PCR扩增是水稻各个研究领域的基础技术。然而相比拟南芥等双子叶模式植物,水稻基因组有更多的高GC含量DNA序列。高GC 含量DNA序列容易形成二级结构,使得DNA模板变性会很困难,同时退火时引物与模板结合受阻,不容易配对或容易形成错配从而导致扩增效率很低或者扩增失败[2],这使得许多的水稻DNA序列用常规的PCR方法难以扩增。已知使用Two-Step PCR[3-4]和Slow-Down PCR[5-6]可以促进高GC含量DNA序列的扩增,但是在我们以往实验中发现这两种方法对于许多水稻的高GC含量DNA序列扩增并无明显作用。已有文献报道甘油、二甲基亚砜、甲酰胺、核苷酸类似物7-deaza dGTP、dITP、牛血清白蛋白(BSA)、Triton X-100、乙二醇和1,2-丙二醇等添加剂可促进高GC含量DNA序列的PCR扩增[7-16]。这些添加剂不仅可以阻止模板和引物的二级结构的形成,还可以提高引物在温度低于其熔解温度的情况下与模板链的退火机会[17-19]。我们在研究中发现扩增高GC含量水稻DNA序列时在PCR反应体系中加入甜菜碱(Betaine)能够明显提高PCR产物扩增的特异性和产率。通过对3个高GC含量的水稻基因片段LOC_Os08g06050(GC 含量 75.4%)、LOC_Os01g01019(GC含量64.2%)和LOC_Os01g11080(GC含量73.9%)进行PCR扩增,在反应体系中加入不同浓度的甜菜碱,发现甜菜碱浓度在1-2 mol/L时能够明显改进PCR扩增效果。同时,用二甲基亚砜和甘油作为PCR添加剂同样地扩增这3个高GC含量水稻基因片段,发现二者虽然也可以表现出促进效果,但都不如甜菜碱明显。此外,还使用了不同的DNA聚合酶对LOC_Os01g01019基因片段进行PCR扩增,发现加入甜菜碱都能增强特异性,并且提高PCR产物的收率。
1 材料与方法
1.1 材料
实验中水稻品种为日本晴野生型水稻;Fastpfu DNA聚合酶购自北京全式金生物技术有限公司;Taq DNA聚合酶购自天根生化科技(北京)有限公司;Phusion超保真 DNA聚合酶和Q5 高保真DNA聚合酶购自New England Biolabs(NEB)公司;甜菜碱(Betaine)、二甲基亚砜(DMSO)和甘油(Glycerin)试剂均购自Sigma-Aldrich公司;快捷型植物基因组DNA提取系统试剂盒购自天根生化科技(北京)有限公司。
1.2 方法
1.2.1 水稻DNA提取 利用快捷型植物基因组DNA提取系统试剂盒来提取水稻叶片基因组DNA作为PCR模板。利用NanoDrop 2000分光光度计测定DNA浓度。
1.2.2 引物 本实验挑选了3个高GC含量的水稻基因片段:LOC_Os08g06050(部分序列,片段大小为399 bp,GC含量75.4%)、LOC_Os01g01019(部分序列,片段大小为1 016 bp,GC含量64.2%)和LOC_Os01g11080(部分序列,片段大小为1 034 bp,GC含量 73.9%),并设计引物由北京六合华大基因科技股份有限公司合成。
水稻基因片段LOC_Os08g06050引物序列为:
正向引物AGCGGAAACCTGGGCGAGTTGTC,反向引物CCGACGAGACGTCGTCGCGC;
水稻基因片段LOC_Os01g01019引物序列为:
正向引物AGGACGTTGACAGGTGCTGGAG,反向引物ACTGCTCAACCGCATGCCCATG;
水稻基因片段LOC_Os01g11080引物序列为:
正向引物ATGGGCCACGCATCCACCGACC,反向引物TCTCCGGCGAGCTCACTCACCAC。
1.2.3 PCR扩增体系及条件 Taq DNA聚合酶反应体系(50 μL)包括 5 μL 10×Taq 缓冲液(Mg2+free),4 μL 2.5 mmol/L dNTP Mixture,4 μL 50 mmol/L MgCl2,10 μmol/L 正向引物与反向引物各 1 μL,100 ng 水稻基因组DNA,2.5 U Taq DNA 聚合酶,反应体系中分别加入不同浓度的甜菜碱(反应终浓度为0-2.75 mol/L),二甲基亚砜(反应终浓度为0-20%)或者甘油(反应终浓度为0-20%)。3个基因的PCR反应条件均为:95℃预变性3 min;95℃变性30 s,65℃退火30 s,72℃延伸70 s,30个循环;72℃延伸5 min。
Phusion 超保真DNA聚合酶反应体系(50 μL)包括 10 μL 5×Phusion HF 缓冲液,4 μL 2.5 mmol/L dNTP Mixture,2 μL 50 mmol/L MgCl2,10 μmol/L正向引物与反向引物各1 μL,100 ng 水稻基因组DNA,2.5 U Phusion超保真DNA聚合酶,向反应体系中加入终浓度为1.5 mol/L的甜菜碱。PCR反应条件为:98℃预变性1 min;98℃变性20 s,65℃退火20 s,72℃延伸35 s,30个循环;72℃延伸5 min。
Q5高保真DNA聚合酶反应体系(50 μL)包括 10 μL 5×Q5 反应缓冲液,4 μL 2.5 mmol/L dNTP Mixture,2 μL 50 mmol/L MgCl2,10 μmol/L 正向引物与反向引物各1 μL,100 ng 水稻基因组DNA,2.5 U Q5高保真DNA聚合酶,向反应体系中加入终浓度为1.5 mol/L的甜菜碱。PCR反应条件为:98℃预变性1 min;98℃变性10 s,65℃退火30 s,72℃延伸35 s,30个循环;72℃延伸2 min。
Fastpfu DNA聚合酶反应体系(50 μL)包括10 μL 5×Fastpfu 缓 冲 液,4 μL 2.5 mmol/L dNTP Mixture,2 μL 50 mmol/L MgCl2,10 μmol/L 正向引物与反向引物各1 μL,100 ng 水稻基因组DNA,2.5 U Fastpfu DNA聚合酶,向反应体系中加入终浓度为1.5 mol/L的甜菜碱。反应条件为95℃预变性3 min;95℃变性30 s,65℃退火30 s,72℃延伸35 s,30个循环;72℃延伸5 min。
以上PCR反应均进行两次重复实验。
1.2.4 PCR产物条带分析 取PCR产物,用1%的琼脂糖凝胶进行电泳,电泳后用JY04S-3C型凝胶成像分析系统(北京君意东方电泳设备有限公司)进行扫描成像,并用ImageJ软件分析PCR产物条带灰度值,通过Excel软件统计汇总并做图表分析。
2 结果
2.1 不同浓度甜菜碱下水稻高GC含量DNA片段的PCR扩增
以水稻DNA为模板,用Taq DNA聚合酶对3个高GC含量的水稻基因片段LOC_Os08g06050(GC含 量 75.4%)、LOC_Os01g01019(GC含 量 64.2%)和LOC_Os01g11080(GC含量73.9%)进行PCR扩增,在反应体系中分别加入终浓度为0、0.25 mol/L、0.5 mol/L、0.75 mol/L、1.0 mol/L、1.25 mol/L、1.5 mol/L、1.75 mol/L、2.0 mol/L、2.25 mol/L、2.5 mol/L和2.75 mol/L的甜菜碱。在扩增LOC_Os01g11080基因片段时,甜菜碱浓度在1-2.25 mol/L时对于PCR扩增具有明显的增强效果;在扩增LOC_Os01g01019基因片段时,甜菜碱浓度在0.75-2 mol/L时对于PCR扩增具有明显的增强效果;在扩增LOC_Os08g06050基因片段时,甜菜碱浓度在0.75-2 mol/L时对于PCR扩增具有明显的增强效果(图1)。分析本实验中3个高GC含量的水稻基因片段的扩增情况,发现甜菜碱浓度在1-2 mol/L时能够明显提高扩增产量和产物特异性。
2.2 不同增强剂对于水稻高GC含量DNA片段PCR的影响
在利用Taq DNA聚合酶对3个高GC含量的水稻基因片段LOC_Os08g06050(GC含量75.4%)、LOC_Os01g01019(GC含 量64.2%) 和LOC_Os01g11080(GC含量73.9%)进行PCR扩增时,分别添加了浓度梯度为0%、2.5%、5%、7.5%、10%、12.5%、15%、17.5%及20%的甘油和浓度梯度为0%、2.5%、5%、7.5%、10%、12.5%、15%、17.5%及20%的二甲基亚砜。在扩增LOC_Os01g11080基因片段时,7.5%-15%的甘油以及7.5%-12.5%的二甲基亚砜表现出明显的增强效果;在扩增LOC_Os01g01019基因片段时,7.5%-10%的甘油以及5%-10%的二甲基亚砜表现出明显的增强效果;在扩增LOC_Os08g06050基因片段时,7.5%-10%的甘油以及5%-10%的二甲基亚砜表现出明显的增强效果(图2)。
综合分析图2中3个基因片段的扩增结果,发现二甲基亚砜浓度在7.5%-10%时,甘油浓度在7.5%-10%时表现出了最明显的增强效果。为了比较不同增强剂对PCR反应的增强效果,选择了10%的甘油和10%的二甲基亚砜与甜菜碱进行平行比较。在利用Taq DNA聚合酶对3个高GC含量的水稻基因片段LOC_Os08g06050、LOC_Os01g01019和LOC_Os01g11080进行PCR扩增时,分别添加了终浓度为10%二甲基亚砜、终浓度为10%的甘油以及终浓度为1.5 mol/L的甜菜碱。实验结果(图3)显示虽然二甲基亚砜和甘油能够促进水稻高GC含量片段的扩增,但是促进效果都没有甜菜碱明显。
2.3 甜菜碱对不同DNA聚合酶PCR的影响
为了验证甜菜碱是否对于不同的DNA聚合酶催化的水稻高GC含量DNA序列的PCR反应均有增强作用,分别用Taq DNA聚合酶、Phusion 超保真DNA聚合酶、Q5 高保真DNA聚合酶以及Fastpfu DNA聚合酶对LOC_Os01g01019基因片段进行PCR扩增,并向各个反应体系中加入终浓度均为1.5 mol/L的甜菜碱。结果(图4)表明,对于这几种DNA聚合酶,甜菜碱都起到了不同程度促进效果,不仅提高了PCR产量,而且增强了产物特异性。
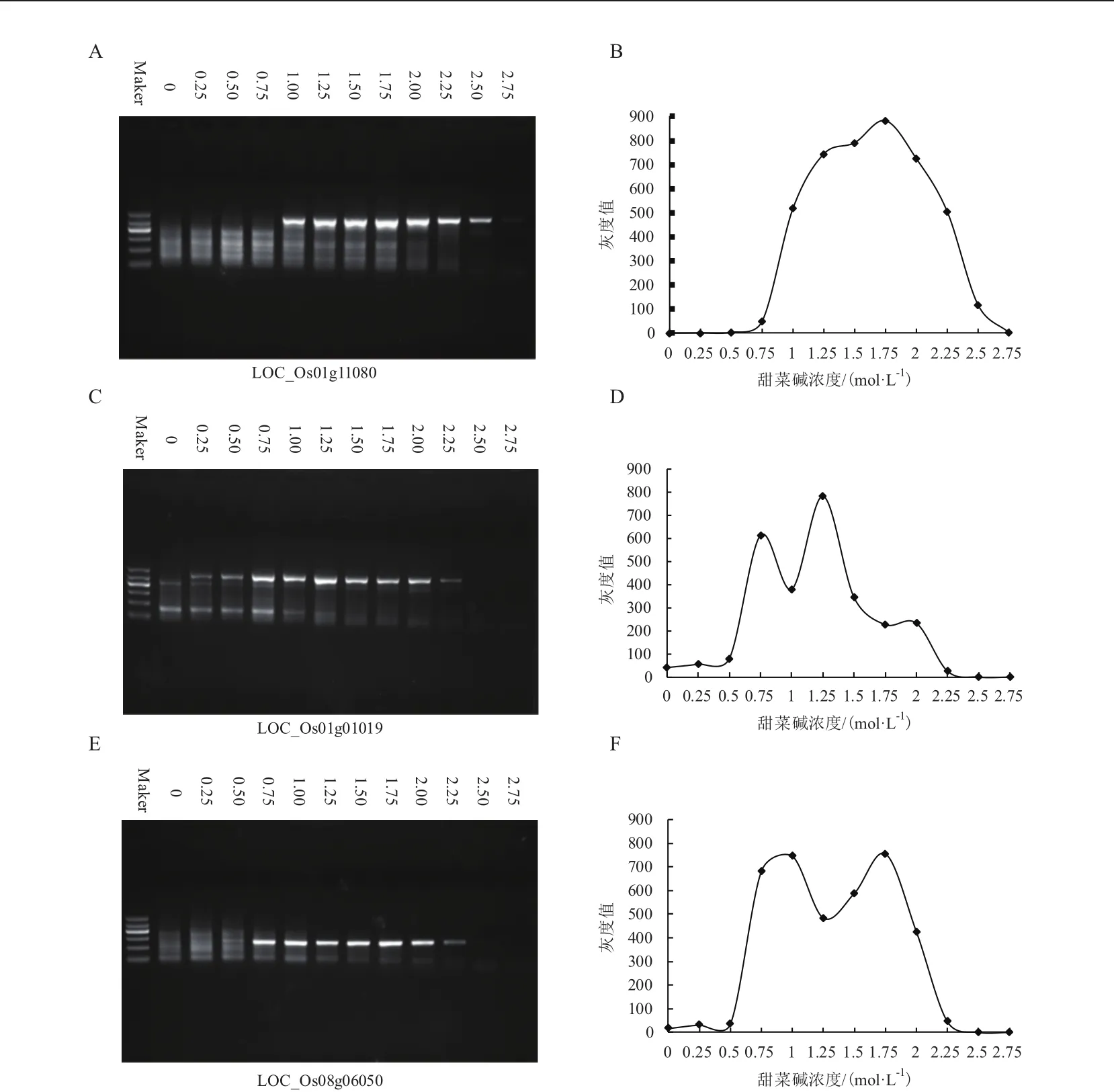
图1 不同甜菜碱浓度对水稻高GC含量DNA片段PCR的影响
3 讨论
水稻的基因组序列富含高GC含量的序列。高GC 含量DNA序列形成的二级结构比较稳定,使得DNA模板变性会很困难,同时在退火时引物与模板结合受阻,容易形成错配从而导致扩增效率很低或者扩增失败[2]。而且在PCR扩增的第一个循环时,高GC含量的模板DNA序列中的单股DNA容易形成分子内部的茎环结构,DNA聚合酶扩增时可能会跳过茎环结构,从而导致PCR产物缺少这段结构的序列[20],因此用传统的PCR方法来准确扩增水稻的高GC含量DNA序列非常困难。
在PCR反应体系中加入甜菜碱能够削弱二级结构的影响,促进PCR反应的进行[9-10,21]。但是其对PCR的作用又具有双刃剑的特点,如图1所示,加入甜菜碱的浓度过低时,不足以起到削弱二级结构的作用;而过高的甜菜碱浓度会抑制PCR反应,同样使得扩增失败。分析本实验中3个高GC含量的水稻基因片段的扩增情况,发现甜菜碱浓度在1-2 mol/L时能够明显提高扩增产量和产物特异性。但是对于不同的GC含量的DNA序列,在PCR扩增时所需的最适甜菜碱浓度也会有所差异,因此对于所有的高GC含量水稻基因序列的PCR扩增,本实验未能得出一个最适的甜菜碱浓度。

图2 不同浓度的甘油和二甲基亚砜对水稻高GC含量DNA片段PCR的影响

图3 不同增强剂对于水稻高GC含量DNA片段PCR的影响
此外,除了甜菜碱还有如二甲基亚砜、甘油等多种PCR增强剂都可以削弱高GC含量DNA序列二级结构的影响。通过比较10%二甲基亚砜、10%甘油和终浓度1.5 mol/L的甜菜碱发现对于水稻的高GC含量DNA序列的PCR扩增,甜菜碱的增强效果最为明显。有研究表明,将不同的PCR增强剂相互混合也可以有效地增强高GC含量基因序列的PCR扩增效果[22-24],但其对于水稻高GC含量DNA序列的PCR扩增是否具有增强作用还需要进一步深入研究。此外,甜菜碱对于长片段的PCR扩增也有增强作用,它能减少甚至消除长片段PCR扩增中的非特异扩增,对于这一点,二甲基亚砜和甘油等其他添加物则无明显作用[25-27]。因此,如果遇到长片段的水稻高GC含量序列,甜菜碱将表现出更为明显的增强优势。
目前,市面上的DNA聚合酶种类比较丰富,不同的DNA聚合酶对于水稻高GC含量DNA序列的扩增结果也不相同。在不加入甜菜碱时,Taq DNA聚合酶和Phusion超保真DNA聚合酶均对LOC_Os01g01019基因片段扩增失败,Q5高保真DNA聚合酶和Fastpfu DNA聚合酶虽然能在一定程度上扩增得到目的片段,可是扩增特异性明显降低。加入甜菜碱之后,对于4种DNA聚合酶,PCR扩增产量以及扩增特异性均明显增强。这表明甜菜碱对于多种DNA聚合酶作用下的水稻高GC含量序列的PCR扩增均有增强作用。

图4 甜菜碱对不同DNA聚合酶PCR的影响
4 结论
对于水稻高GC含量DNA序列的PCR扩增,加入终浓度为1-2 mol/L的甜菜碱能够起到显著的增强作用。这种增强作用明显高于二甲基亚砜和甘油,并且对于多种DNA聚合酶作用下的水稻高GC含量序列的PCR扩增均有增强作用。
[1]Saiki RK, Gelfand DH, Stoffel S, et al. Primer-directed enzymatic amplification of DNA with a thermostable DNA polymerase[J].Science, 1988, 239(4839):487-491.
[2]Mamedov TG, Pienaar E, Whitney SE, et al. A fundamental study of the PCR amplification of GC-rich DNA templates[J].Computational Biology and Chemistry, 2008, 32(6):452-457.
[3]Schuchard M, Sarkar G, Ruesink T, et al. Two-step“hot”PCR amplification of GC-rich avian c-myc sequences[J].Biotechniques, 1993, 14(3):390-394.
[4]Henry RJ, Oono K. Amplification of a GC-rich sequence from barley by a two-step polymerase chain reaction in glycerol[J]. Plant Molecular Biology Reporter, 1991, 9(2):139-144.
[5]Frey UH, Bachmann HS, Peters J, et al. PCR-amplification of GC-rich regions:‘slowdown PCR’[J]. Nature Protocols, 2008, 3(8):1312.
[6]Bachmann HS, Siffert W, Frey UH. Successful amplification of extremely GC-rich promoter regions using a novel ‘slowdown PCR’technique[J]. Pharmacogenetics and Genomics, 2003, 13(12):759-766.
[7]Chakrabarti R, Schutt CE. The enhancement of PCR amplification by low molecular-weight sulfones[J]. Gene, 2001, 274(1):293-298.
[8]Sun Y, Hegamyer G, Colburn NH. PCR-direct sequencing of a GC-rich region by inclusion of 10% DMSO:application to mouse c-jun[J]. Biotechniques, 1993, 15(3):372.
[9]Jensen MA, Fukushima M, Davis RW. DMSO and betaine greatly improve amplification of GC-rich constructs inde novosynthesis[J]. PLoS One, 2010, 5(6):e11024.
[10]Henke W, Herdel K, Jung K, et al. Betaine improves the PCR amplification of GC-rich DNA sequences[J]. Nucleic Acids Research, 1997, 25(19):3957-3958.
[11]Sarkar G, Kapelner S, Sommer SS. Formamide can dramatically improve the specificity of PCR[J]. Nucleic Acids Research,1990, 18(24):7465.
[12]Turner SL, Jenkins FJ. Use of deoxyinosine in PCR to improve amplification of GC-rich DNA[J]. Biotechniques, 1995, 19(1):48.
[13]Dierick H, Stul M, De Kelver W, et al. Incorporation of dITP or 7-deaza dGTP during PCR improves sequencing of the product[J]. Nucleic Acids Research, 1993, 21(18):4427.
[14]McConlogue L, Brow MAD, Innis MA. Structure-independent DNA amplification by PCR using 7-deaza-2’-deoxyguanosine[J].Nucleic Acids Research, 1988, 16(20):9869.
[15]Kreader CA. Relief of amplification inhibition in PCR with bovine serum albumin or T4 gene 32 protein[J]. Appl Environ Microbiol, 1996, 62(3):1102-1106.
[16]杨霞, 刘芳, 孟良玉, 等. 乙二醇和1, 2-丙二醇增强富含GC碱基人类基因组DNA模板的PCR扩增[J]. 中国生物工程杂志, 2009, 29(3):69-73.
[17]Weissensteiner T, Lanchbury JS. Strategy for controlling preferential amplification and avoiding false negatives in PCR typing[J]. Biotechniques, 1996, 21(6):1102-1109.
[18]Dutton CM, Christine P, Sommer SS. General method for amplifying regions of very high G+C content[J]. Nucleic Acids Research,1993, 21(12):2953-2954.
[19]Sahdev S, Saini S, Tiwari P, et al. Amplification of GC-rich genes by following a combination strategy of primer design, enhancers and modified PCR cycle conditions[J]. Molecular and Cellular Probes, 2007, 21(4):303-307.
[20]Viswanathan VK, Krcmarik K, Cianciotto NP. Template secondary structure promotes polymerase jumping during PCR amplification[J]. Biotechniques, 1999, 27(3):508-511.
[21]白雪源, 孟令英, 崔红, 等. 甜菜碱促进高 GC 含量 p16 基因的 PCR 扩增[J]. 中华医学遗传学杂志, 2000, 17(3):224-225.
[22] Baskaran N, Kandpal RP, Bhargava AK, et al. Uniform amplification of a mixture of deoxyribonucleic acids with varying GC content[J]. Genome Research, 1996, 6(7):633-638.
[23]Musso M, Bocciardi R, Parodi S, et al. Betaine, dimethyl sulfoxide,and 7-deaza-dGTP, a powerful mixture for amplification of GC-rich DNA sequences[J]. The Journal of Molecular Diagnostics, 2006,8(5):544-550.
[24]Ralser M, Querfurth R, Warnatz HJ, et al. An efficient and economic enhancer mix for PCR[J]. Biochemical and Biophysical Research Communications, 2006, 347(3):747-751.
[25]吴斌, 杨威, 李强, 等. 甜菜碱提高长距离PCR扩增效率的研究[J]. 实用医学杂志, 2008, 24(17):2924-2926.
[26]Hengen PN. Optimizing multiplex and LA-PCR with betaine[J].Trends in Biochemical Sciences, 1997, 22(6):225-226.
[27]Chen XQ, Zhang XD, Liang RQ, et al. Betaine improves LA-PCR amplification[J]. Chinese Journal of Biotechnology, 2004, 20(5):715-718.
