利用RNA-seq分析诱导性系统抗性(ISR)产生时匍匐翦股颖转录因子的表达变化
2018-03-27牛奎举马晖玲
史 毅,牛奎举,马晖玲
(甘肃农业大学 草业学院/草业生态系统教育部重点实验室/甘肃省草业工程实验室/中-美草地畜牧业可持续发展研究中心,甘肃 兰州 730070)
植物根际益生菌(Plant growth-promoting rhizobacteria,PGPR)是可寄生在植物根际并且具有促进植物生长能力的一类细菌的总称[1-2]。PGPR被发现有促生能力后,相关报道指出在植物根部施加根际益生菌可提高植物的抗病能力[3-6]。2003~2004年,Ryu等[7-8]首次报道PGPR的挥发性化合物可作为诱导剂,促进拟南芥的生长并且对抗病性有帮助,同时分析了PGPR挥发性化合物的成分,发现2,3-丁二醇是主要组成物,证明了人工合成2,3-丁二醇与PGPR挥发性化合物有同样的促生和抗病诱导效果[7-8]。从而开启了众多植物学家利用PGPR挥发性化合物诱导植物抗病性的研究[9]。
匍匐翦股颖褐斑病病原为立枯丝核菌(Rhizoctoniasolani),多发于夏季,该菌是一种土壤习居菌,为无性繁殖,传播迅速,冷暖季型禾本科草坪草上该病害均发生[10]。为了控制草坪病害,杀菌剂的使用越来越频繁,但大量使用杀菌剂不但成本高而且严重污染环境。所以,研究人员开始致力于发现安全的替代品来控制草坪病害。PGPR挥发性化合物是一种极有前景的植物真菌病害控制剂,然而其诱导植物产生抗病性的作用机理尚不清楚,对其作用机理的深入了解是将相关化合物应用于实际的前提。
植物的诱导抗性主要分两类,植物系统获得抗性(Systemic acquired resistance,SAR)和植物诱导系统抗性(Induced systemic resistance,ISR)。SAR通常报道为由水杨酸信号通路和NPR1(Nonexpresser of PR genes)基因的诱导产生,并且基于植物病程相关蛋白(Pathogenesis-Related genes,PR)的诱导而起作用[11]。而ISR通常与茉莉酸/乙烯(JA/ET)信号通路有关[12]。PGPR及其挥发性化合物诱导产生的植物抗病性大多报道为ISR类型,通过JA/ET通路传播信号,引起植物全面广谱的抵抗能力。然而利用转录组学分析单子叶植物被细菌挥发性化合物处理后引起植物抗病性机理的研究报道较少。
抗病诱导过程中,植物基因表达的变化,是诱导抗病性产生的根本原因,而基因表达的变化由调控转录因子(Transcript factors,TFs)与位于基因启动子前的顺式表达元件的结合而产生。TFs在植物生长发育,以及逆境胁迫发挥重要作用。
通过对匍匐翦股颖被诱抗剂2,3-丁二醇处理前后的转录组进行比较分析,发掘匍匐翦股颖ISR抗性相关转录因子,并研究其表达模式,将为诱抗剂应用于实际和匍匐翦股颖抗病基因资源的应用奠定基础。
1 材料和方法
1.1 植物材料制备及处理
匍匐翦股颖(Agrostisstolonifera)商用品种“PennA4”,由北京克劳沃公司提供。种子经浸泡消毒后自然风干,以灭菌的沙土混合物(1∶2)为基质,种于240 mL组培瓶中,每瓶均匀撒入0.25 g种子,封口膜遮盖,在甘肃农业大学草业学院恒温培养室中培养,温度为20℃昼/18℃夜,光周期为16 h昼/8 h夜。生长2周后,用注射器分别将10 mL浓度为150,250,350,450, 和550 μmol/L的2,3-丁二醇溶液施加在匍匐翦股颖植株贴近土壤部位,以蒸馏水为对照,每个处理4个重复。
1.2 病原菌接种及病情分析
采用Cortes-barco等[13]的接菌物制备方法,利用麦粒培养基制备立枯丝核菌接菌物,并将其打碎研磨成粉末。2,3-丁二醇处理后第7 d对植物材料进行接菌,接菌时,先从瓶口向内喷水雾,使植物叶片湿润,然后向叶片撒播接菌物粉末,每瓶0.1 g,使其均匀分布在瓶内植物的叶片上。封口膜遮盖培养于恒温培养室,培养条件同前,每天观察病情。以目测发病叶片的发病面积总和占所有叶片面积的百分比为病症面积百分比,以病原菌菌丝生长面积占所有叶片面积的百分比为菌丝所占面积,以此2个指标共同评价草坪草病情。
1.3 丁二醇对病原菌直接抑制作用测试
将生长在PDA培养基的立枯丝核菌切下5 mm菌块,置于添加0,50,500,1 000,5 000 μmol/L 2,3-丁二醇的PDA培养基上,每个浓度设4个重复,平板置于28℃培养9 d,每天观测真菌生长状况。
1.4 测序样品准备和RNA提取
植物材料使用2,3-丁二醇处理前(0 d),处理后1 d,处理后4 d,处理后7 d即接菌前,分别取样,各4个重复,每瓶内所有叶片为一个样,剪取叶片用锡箔纸包裹后迅速置于液氮中低温保存。所有样品用植物RNA提取试剂盒(OMEGA)提取RNA。经琼脂糖凝胶电泳鉴定,测量浓度,RIN(RNA integrity number)指数后选择完整性好,纯度浓度高,RIN>6.3的RNA样品用于建库测序。
1.5 建库和测序
每个时间点的RNA样品,分别由4个重复样品混合而成,用于构建cDNA库。测序使用Illumina HiSeq平台,由诺禾致源公司(中国,北京)完成。
1.6 转录组组装
测序得到的原始数据(raw reads)使用FASTQC确定待分析数据(clean reads)质量,Trinity用于转录本的从头组装(De novo assembly),然后使用CD-HIT(http:∥weizhongli-lab.org/cd-hit/)选取相似类中最长的作为唯一基因,得到所有基因序列集合(Unigene)作为分析基因表达量的参考序列[14](reference sequences)。
1.7 差异表达基因(Differential expression genes,DEGs)数字分析及荧光定量PCR分析
用测序得到的各库clean reads映射(map)于组装得到的参考转录组,然后使用DEGseq对各Unigene在各库的reads数量(均一化后)进行分析,以|log2(foldchange)|>1,qvalue<0.005为阈值选取差异表达基因。使用iTAK[15](http:∥bioinfo.bti.cornell.edu /tool/itak)软件查找所有差异表达基因中的转录因子。选取抗病相关转录因子基因20个,引物设计见表1,使用SYBR Green染料进行荧光定量PCR,利用ΔΔCT法得到各基因在各植物样本中的表达量。20 mL反应体系,SYBR 10 m,上下游引物各0.8 mL,模板5 mL,去离子水3.4 mL。反应条件95℃ 10 min;40 cycles为95℃ 15 s,57℃ 1 min。
2 结果与分析
2.1 立枯丝核菌侵染后匍匐翦股颖病情分析
2,3-丁二醇处理后的植株与对照出现褐斑病害的症状以及时间并无区别,直到接菌后的第4 d,不同浓度丁二醇处理植株以及对照之间病情仍无显著区别,接菌后第7及第10 d,丁二醇处理过的植株的疾病症状显著少于对照(图1)。尤其250 μmol/L 2,3-丁二醇比其他浓度表现出对病症更大的抑制作用。丁二醇对病原菌直接抑制作用测试显示添加在PDA平板中的2,3-丁二醇,无论浓度高低,对立枯丝核菌生长无影响(图2,3)。
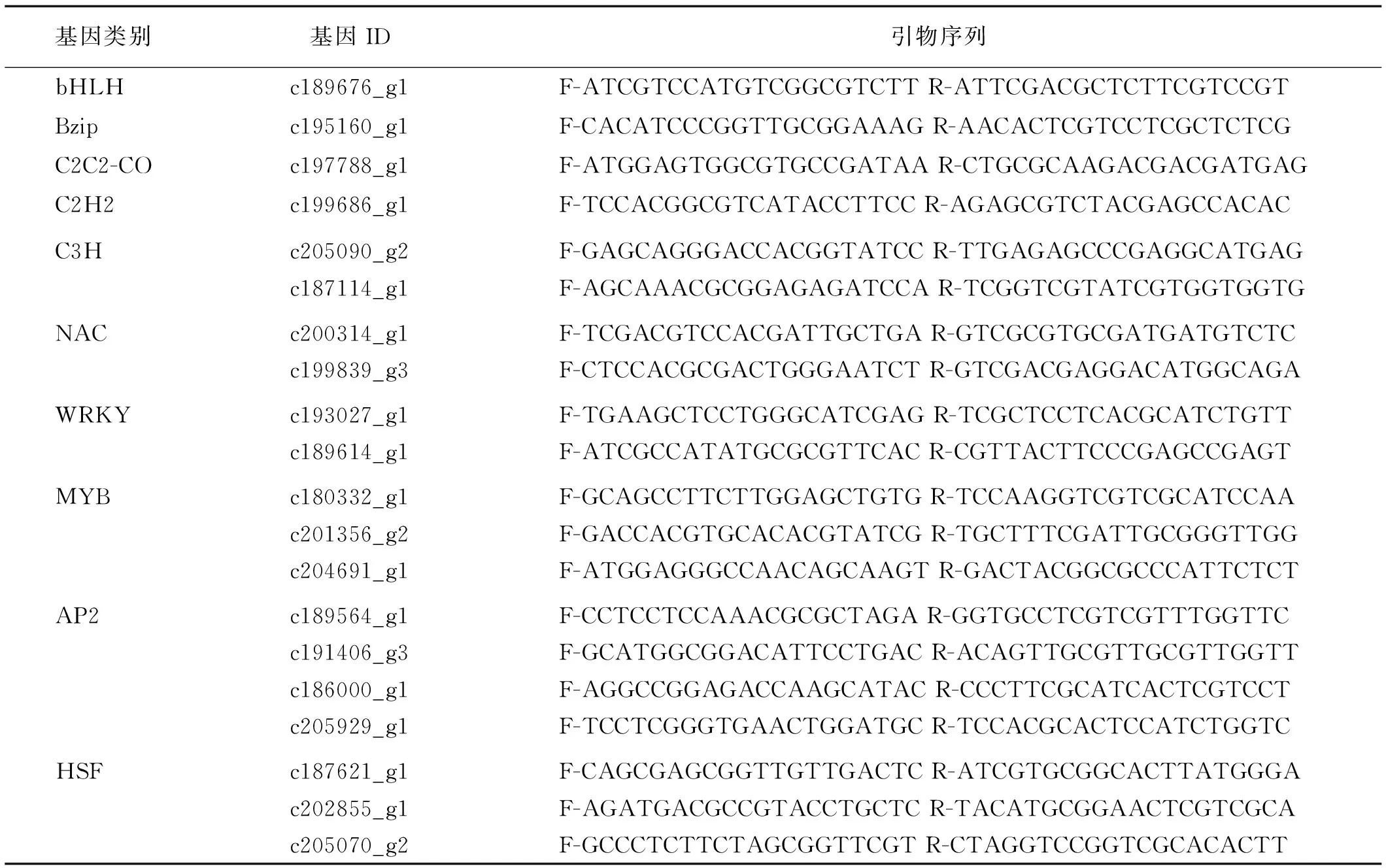
表1 qRT-PCR验证基因选择及引物设计

图1 接菌10 d后的匍匐翦股颖发病状况Fig.1 The disease occurrence of creeping bentgrass after inoculation for 10 days注:图中左上,250 μmol/L 2,3-丁二醇处理未接菌植株;左下,水处理未接菌植株;右上,250 μmol/L 2,3-丁二醇处理接菌植株;右下,水处理接菌植株

图2 高浓度丁二醇在PDA培养基上处理下立枯丝核菌的生长Fig.2 Effect of 2,3-butanediol in high concentration on R.solanidevelopment on PDA medium
基于以上病情分析及丁二醇直接作用于病原菌的

图3 不同浓度2,3-丁二醇处理下叶片的显症面积和菌丝覆盖面积Fig.3 Effect of 2,3-butanediol on plant leaf percent area of lesions and mycelium注:图中左为各浓度2,3-丁二醇对叶片显症面积的不同影响;右为各浓度丁二醇对立枯丝核菌
结果,丁二醇对病原菌无直接影响,而是可诱导植物自身产生抗病性,且250 μmol/L 2,3-丁二醇诱导作用最明显,所以,植物转录组测序及分析采用250 μmol/L 2,3-丁二醇作为处理剂。
2.2 转录组组装及差异基因分析
利用Illumina 2×150 bp 双端测序共测得258,606和402条reads,产生25Gb数据,去除接头和低质量序列后,得到252,117和292条高质量待分析reads。经Trinity从头组装后,得到466,761条转录本。过半数的转录本长度为700 bp,组装结果显示N50=1 100。选择334,212条转录本(所有转录本的71.60%)作为Unigene,平均长度573 bp,N50=791 bp。比较得到各库相较于0DPT,有一定的上调表达和下调表达基因(图4)。可以看出,经2,3-丁二醇处理后随着时间变化,差异表达基因逐渐增多,且下调表达基因明显多于上调基因。
2.3 差异基因的转录因子分类
利用iTAK软件从所有差异表达基因中挑选得到32个表达量发生变化的转录因子。其中下调表达转录因子明显多于上调表达转录因子。上调表达基因中的转录因子主要归类为bHLH,MYB,NAC和C2C2-CO-like等转录因子家族,还有3个基因可被定义为Orphans转录因子,即功能不明确的转录因子。下调表达的转录因子可归类至13个转录因子家族,其中最多的是HSF家族,其次为AP2-EREBP家族(表2)。

图4 经2,3-丁二醇处理1,4和7 d后匍匐翦股颖转录组差异表达基因Fig.4 The number of DEGs of creeping bentgrass transcriptome caused by 2,3-butanediol on 1 d,4 d and 7 d after treatment
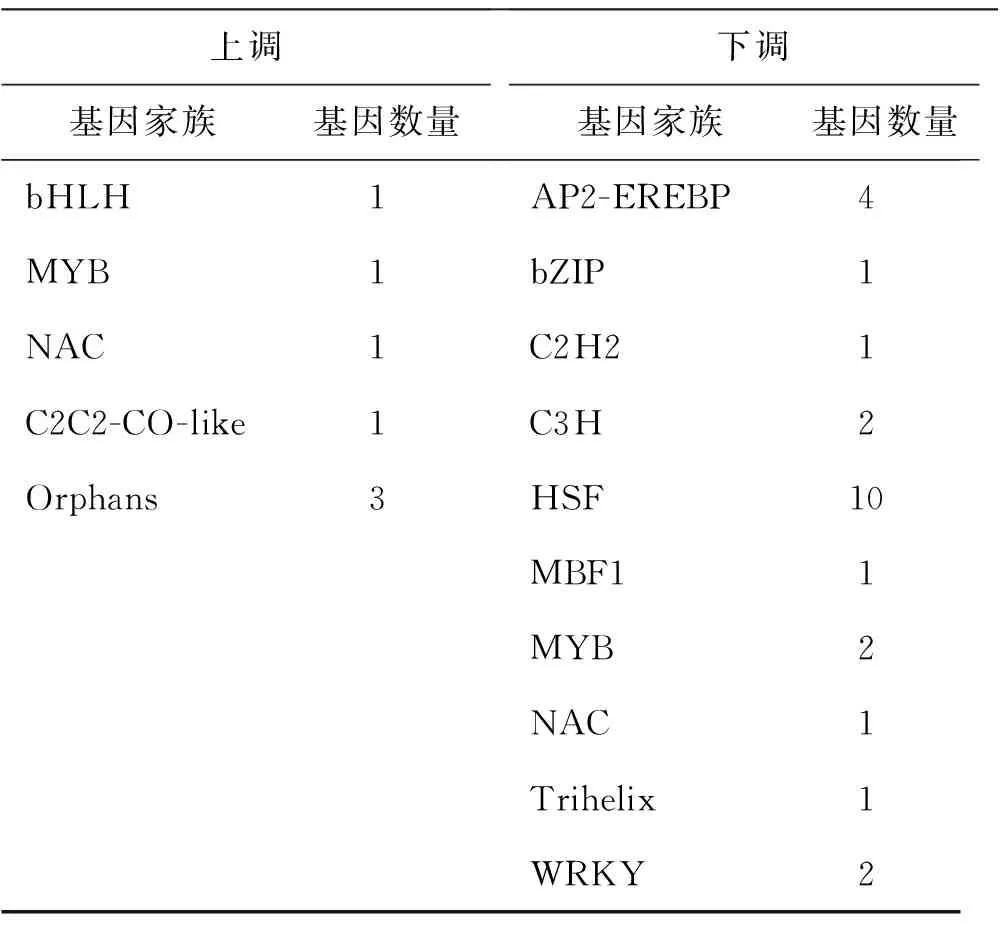
表2 匍匐翦股颖差异基因中转录因子家族信息
2.4 抗病相关差异转录因子表达变化
差异表达基因可以注释到的转录因子中,大多为与植物抗逆和抗病相关的转录因子,但仍有一些未见与逆境相关的报道,试验中挑出与植物抗病相关的转录因子,并对其在各时间点的表达量进行了分析。结果表明,在4个时间点呈持续上调的转录因子有bHLH,C2C2-CO-like,1个NAC和1个MYB转录因子。在4个时间点呈现上调再下调表达的有WRKY和3个AP2-EREBP家族转录因子。呈显著下调的是Bzip,C2H2,C3H,1个NAC,HSF的10个转录因子(图5)。

图5 匍匐翦股颖差异表达转录因子中抗病相关转录因子表达模式Fig.5 The expression pattern of defense related TFs in the DEGs of creeping bentgrass transcriptome
2.5 qRT-PCR 验证
为了验证RNA-seq的准确性,采用实时荧光定量PCR对20个转录因子相关基因在各时间点的相对表达量进行了分析(图6)。选取的20个基因中包括与抗病相关转录因子的所有家族,其中10个HSF转录因子表达趋势一致,所以只选取3个做验证。MYB转录因子的c201356_g2转录本的实际表达量未像RNA-seq分析结果表现出的先下调再恢复的趋势,但整体呈下调后平稳的趋势,可以认为两种结果相似。AP2-EREBP转录因子的c191406_g3和c186000_g1转录本虽不像RNA-seq结果在第4 d呈明显上调,而是在第1 d就出现显著上调,但整体趋势仍是先上调后下调,与RNA-seq一致。HSF转录因子的c202855_g1转录本实际表达量表现出先下调后恢复,与RNA-seq结果稍显不一致,但最终表达量为小幅度下调。所以经20个基因的实时荧光定量验证,实际表达量与RNA-seq分析一致,说明RNA-seq分析结果有较高的可信度。
3 讨论
诱抗剂诱导植物产生对病原体的广谱抗性主要是由于其对植物基因表达产生的影响,转录因子是植物细胞内基因表达调控的关键因子,也称反式作用因子,是指能够与基因启动子区域中顺式作用原件发生特异性相互作用的DNA结合蛋白,通过他们之间以及与其他相关蛋白之间的相互作用激活或抑制某些基因的转录[16]。试验发现2,3-丁二醇处理后,匍匐翦股颖对草坪草褐斑病病原R.Solani产生了诱导性抗性,为了对其诱导机理有一定了解,利用RNA-seq方法对匍匐翦股颖叶片在丁二醇处理前后的不同时间点样品的转录组进行测序分析,找到差异表达基因,并利用生物信息学软件找到差异基因中的转录因子,进而分析差异表达的转录因子如何调控匍匐翦股颖诱导抗性的产生。

图6 部分匍匐翦股颖抗病相关差异表达转录因子qRT-PCR验证表达量Fig.6 TheqRT-PCR of part of the defense related TFs of creeping bentgrass
差异基因数量分析发现,随着丁二醇处理时间的延长,差异基因数量逐渐增多,由处理1 d的上调表达49个基因、下调表达141个基因增长至处理7 d后的上调表达149个、下调表达272个基因。且不管处理时间为几天,下调表达基因始终多于上调表达基因。这与Pan等[17]的研究结果一致,他用外源细胞分裂素处理麻风树2,4和22 h后对其进行转录组测序,与未处理植株相比其表达下调基因明显多于表达上调基因。2,3-丁二醇虽与细胞分裂素不是同一种物质,但其同样有促进植物生长的作用,外源化合物处理植物出现下调基因明显多于上调基因并不少见,在多种植物的外源处理诱导研究中均有发现。下调基因可能引起某些物质表达量的减少,但不代表调控能力的下降,下调的调控物质有可能减少对某些基因的抑制作用,反而起到上调的作用。
转录因子根据DNA结合结构域的差异,可以分成不同的基因家族,如C2H2锌指蛋白,bZIP,MYB,WRKY,NAC和HSF。研究中发现的差异表达转录因子,并且有过与植物抗病相关报道的包括以下家族:bHLH,Bzip,C2C2-CO-like,C2H2,C3H,NAC,WRKY,MYB,AP2-EREBP和HSF。
上调表达的转录因子有bHLH,C2C2-CO-like,1个NAC,和1个MYB。其中bHLH转录因子全称为Basic helix-loop-helix碱性-螺旋-环-螺旋转录因子家族。这一类转录因子在植物中扮演重要的角色,它们常常比较保守,有研究表明bHLH转录因子参与拟南芥等模式植物的所有与茉莉酸信号调节途径相关的生物进程中[18]。该家族包含一个MYC2转录因子,它参与主要的茉莉酸相关调节,并且是ISR诱导途径中的主要的转录调控因子[19]。研究中bHLH转录因子的上调表达,可估计与植物ISR诱导呈正相关。C2C2-CO-like,C2H2和C3H转录因子是主要的锌指蛋白转录因子家族。锌指蛋白转录因子被报道与植物逆境响应的调节相关[20]。其中C2H2被报道可调控PGPR诱导植物过程中的基因转录[21]。研究中只有C2C2-CO-like转录因子上调表达,而C2H2和C3H转录因子均为下调表达。这有可能是由于植物体尚未遇到病原菌侵染,所以相关转录因子只有一部分上调表达。NAC蛋白是一种植物特有的转录因子家族,被证明有多重生物学功能[22]。有报道证明拟南芥NAC蛋白ATAF2可抑制病程相关基因的表达[23]。然而另有研究指出植物过表达两个NAC蛋白可调节茉莉酸诱导的抗性相关基因的表达[24]。研究中,两个NAC转录因子在2,3-丁二醇处理后,一个呈上调表达而另一个下调表达,此结果正好与PGPR诱导植物产生ISR的信号途径有关,上调表达的NAC转录因子可能抑制了病程相关蛋白的表达,并提高了其他茉莉酸相关抗性基因的表达,从而使植物产生ISR。MYB转录因子最早发现于欧芹的抗病相关基因苯丙氨酸解氨酶(PAL)和4-香豆酸辅酶A连接酶(4CL)的调节过程中[25],证明有一类MYB转录因子与植物抗病是正调节关系。研究中2个上调表达的MYB转录因子可能属于这一类。
试验中呈先上调再下调表达的有WRKY和3个AP2-EREBP家族转录因子。WRKY转录因子家族被证明是拟南芥抗病转录调节过程中所必须的,然而除了一些WRKY转录因子是基础抗性和植物R-基因调节所必须的,还有一部分WRKY与植物抗性呈负调节关系[26]。研究中2个WRKY转录因子呈先上调后下调表达,可能是因为这2个WRKY转录因子为短期响应因子,而在其情况下呈现负调节作用。
呈显著下调的是Bzip,C2H2,C3H,1个NAC和10个HSF转录因子,其中Bzip转录因子包含一个基本的保守结构域和一个亮氨酸发卡结构 (leucine zipper),该家族分为几个大类,有大量的证据表明,该转录因子家族参与调控生物及非生物胁迫相关代谢途径,其中D类Bzip转录因子被报道参与植物病程相关蛋白表达的诱导过程[27],即植物SAR的产生。然而丁二醇诱导的植物ISR与病程相关蛋白并无关系,所以研究中一个Bzip基因的下调表达也正好可以证明这一点。HSF和AP2-EREBP转录因子均为植物逆境情况下诱导表达的常见转录因子[28-29],然而在研究中10个HSF转录因子和4个AP2-EREBP,除了3个AP2基因早期呈现出先上调后下调的趋势,其余均呈显著下调表达,可能是没有病原菌的介入,2,3-丁二醇直接施入尚不足以使植物产生应激反应。这也是本研究的新的发现,尚未有研究报道过PGPR或其挥发性化合物的施加会引起AP2-EREBP和HSF转录因子的大幅度下调表达。
4 结论
经过2,3-丁二醇的喷施诱导,匍匐翦股颖植株可产生对草坪草褐斑病病原立枯丝核菌的抗性,继而通过对匍匐翦股颖被诱抗剂2,3-丁二醇处理前后的转录组进行比较分析,发掘匍匐翦股颖ISR抗性相关转录因子,并研究它们的表达模式。发现诱导过程中,随着诱导时间递增,差异表达基因增多,其中32个转录因子相关基因可归类为13个转录因子家族。抗病相关的转录因子27个可归类为bHLH,Bzip,C2C2-CO-like,C2H2,C3H,NAC,WRKY,MYB,AP2-EREBP和HSF共10个基因家族。这10个家族的转录因子表现为不同的表达模式,共同作用于匍匐翦股颖的ISR诱导。同时实时荧光定量PCR验证了转录组测序的准确性。结论为植物诱抗剂的实际应用和匍匐翦股颖抗病基因资源的应用奠定了基础。
[1] Kloepper J W,Schroth M N,Miller T D.Effects of rhizosphere colonization by plant growth-promoting rhizobacteria on potato plant development and yield [J].Phytopathology,1980,70(11):1078-1082.
[2] Glick B R.The enhancement of plant growth by free-living bacteria [J].Can J Microbiol,1995,41:109-117.
[3] Kloepper J W,Metting F B J.Plant growth-promoting rhizobacteria as biological control agents[J].Brenners Encyclopedia of Genetics,1992:353-355.
[4] Loon L C V,Bakker P A H M,Pierterse C M J.Systemic resistance induced by rhizosphere bacteria [J].Annual Review of Phytopathology,1998,36(1):453-483.
[5] Kloepper J W,Rodriguez-Kabana R,Zehnder G W,etal.Plant root-bacterial interactions in biological control of soilborne diseases and potential extension to systemic and foliar diseases [J].Australasian Journal of Plant Pathology,1999,28:27-33.
[6] Cleyet-Marcel J C,Larcher M,Bertrand H,etal.Plant growth enhancement by rhizobacteria[M]∥J-F Morot-Gaudry.Nitrogen Assimilation by Plants,Physiological,Biochemical and Molecular Aspects.Enfeld,N H:Science Publishers,2001:185-197.
[7] Ryu C M,Farag M A,Hu C H,etal.Bacterial volatiles promote growth in Arabidopsis[J].Proceedings of the National Academy of Sciences,2003,100(8):4927-4932.
[8] Ryu C M,Farag M A,Hu C H,etal.Bacterial volatiles induce systemic resistance in Arabidopsis[J].Plant physiology,2004,134(3):1017-1026.
[9] 马源,马晖玲,刘兴菊.2R,3R-丁二醇和2,3-丁二醇诱导匍匐翦股颖抗病性的比较[J].草原与草坪,2017,37(1):79-83.
[10] Smiley R W,Dernoeden P H,Clarke B B.Compendium of turfgrass diseases[M].Minnesota:American Phytopathological Society,2005.
[11] Kachroo A,Venugopal S C,Lapchyk L,etal.Oleic acid levels regulated by glycerolipid metabolism modulate defense gene expression in Arabidopsis[J].Proceedings of the National Academy of Sciences of the United States of America,2004,101(14):152-157.
[12] Ton J,Pieterse C M J,Loon L C V.The Relationship Between Basal and Induced Resistance in Arabidopsis[M].Springer US:Multigenic and Induced Systemic Resistance in Plants,2006:197-224.
[13] Cortes-barco A M,Hsiang T,Goodwin P H.Induced systemic resistance against three foliar diseases of Agrostis stolonifera by (2R,3R)-butanediol or an isoparaffin mixture[J].Annals of Applied Biology,2010,157(2):179-189.
[14] Grabherr M G,Haas B J,Yassour M,etal.Full-length transcriptomeassembly from RNA-seq data without a reference genome [J].Nature Biotechnology,2011,29(7):644-652.
[15] Pérez-Rodríguez P,Riano-Pachon D M,Corrêa L G G,etal.PlnTFDB:updated content and new features of the plant transcription factor database [J].Nucleic acids research,2010,38(suppl_1):822-827.
[16] 张椿雨,龙艳,冯吉,等.植物基因在转录水平上的调控及其生物学意义[J].遗传,2007,29(7):793-799.
[17] Pan B Z,Chen M S,Ni J,etal.Transcriptome of the inflorescence meristems of the biofuel plant Jatropha curcas treated with cytokinin [J].BMC genomics,2014,15(1):974.
[18] Goossens J,Mertens J,Goossens A.Role and functioning of bHLH transcription factors in jasmonatesignalling[J].Journal of Experimental Botany,2016:440.
[19] Pieterse C M,Zamioudis C,Berendsen R L,etal.Induced systemic resistance by beneficial microbes[J].Annual Review of Phytopathology,2014,52(52):347-375.
[20] Chen W,Provart N J,Glazebrook J,etal.Expression profile matrix of Arabidopsis transcription factor genes suggests their putative functions in response to environmental stresses[J].Plant Cell,2002,14(3):559-574.
[21] Cartieaux F,Contesto C,Gallou A,etal.Simultaneous interaction of Arabidopsis thaliana with Bradyrhizobium Sp.strain ORS278 and Pseudomonas syringaepv.tomato DC3000 leads to complex transcriptome changes[J].Molecular plant-microbe interactions,2008,21(2):244-259.
[22] Olsen A N,Ernst H A,Leggio L L,etal.NAC transcription factors:structurally distinct,functionally diverse[J].Trends in plant science,2005,10(2):79-87.
[23] Delessert C,Kazan K,Wilson I W,etal.The transcription factor ATAF2 represses the expression of pathogenesis-related genes in Arabidopsis[J].Plant Journal for Cell & Molecular Biology,2005,43(5):745-757.
[24] Bu Q,Jiang H,Li C B,etal.Role of the Arabidopsis thaliana NAC transcription factors ANAC019 and ANAC055 in regulating jasmonic acid-signaled defense responses[J].Cell Research,2008,18(7):756-767.
[25] Logemann E,Parniske M,Hahlbrock K.Modes of Expression and Common Structural Features of the Complete Phenylalanine Ammonia-Lyase Gene Family in Parsley[J].Proceedings of the National Academy of Sciences of the United States of America,1995,92(13):5905-5909.
[26] Eulgem T,Somssich I E.Networks of WRKY transcription factors in defense signaling[J].Current Opinion in Plant Biology,2007,10(4):366-371.
[27] Jakoby M,Weisshaar B,Dröge-Laser W,etal.bZIP transcription factors in Arabidopsis[J].Trends in Plant Science,2002,7(3):106-111.
[28] Hao H T,Zhao X,Shang Q H,etal.Comparative Digital Gene Expression Analysis of the Arabidopsis Response to Volatiles Emitted by Bacillus amyloliquefaciens[J].Plos One,2016,11(8):158-162.
[29] Wu C.Heat shock transcription factors:structure and regulation[J].Annual Review of Cell & Developmental Biology,2003,11(11):441-469.
