CRISPR/cas系统及其在家禽上的应用研究进展
2018-03-20黄玲玲黄小红卢立志
黄玲玲,黄小红,陈 黎,卢立志*
(1.福建农林大学动物科学学院,福建福州 350002;2.浙江省农业科学院畜牧兽医研究所,浙江杭州 310023)
基因编辑技术是生物科学研究的一种非常重要的工具,基因编辑技术的发现开启了生物基因人工修饰的大门。锌指样核酸酶(Zinc-Finger Nucleases,ZFN)和转录激活因子效应物核酸酶(Transcription Activator-Like Effectors Nucleases,TALEN)技术分别作为第1代和第2代核酸内切酶,已被广泛地用于基因组定点修饰功能。CRISPR/cas(Clustered Regularly Interspaced Short Palindromic Repeats and CRISPR-Associated Proteins)是继二者之后出现的一种新的基因组靶向修饰技术,CRISPRs 最先引起科学界的注意是其存在于许多细菌和古细菌间隔排列的串联重复序列(CRISPRs),其转录的产物crRNA(CRISPR-derived RNA)与CRISPR相关蛋白特异性抑制外来质粒或噬菌体侵入,具有精确抵御外源入侵的作用,后来被证明是一种获得性免疫系统[1]。作为最新发展起来的基因编辑技术,CRISPR/cas因其简单、方便、高效的特点正被广泛运用于基因治疗、基因功能研究和建立与人类疾病相关的动物模型等方面的研究。近年来随着CRISPR系统技术不断被深入开发,一些新效应蛋白被发现,本文就CRISPR/cas系统分类、原理及新效应蛋白发现和cas9(S. Pyogenes)多种变构体技术开发研究进展及在家禽上的应用进行综述。
1 CRISPR 系统分类及作用机制
1.1 CRISPR系统分类 根据cas基因的组成和效应蛋白的种类,CRISPR系统被分为了2 类 6型,超过19个亚型[2-4](图1)。Class 1为用多个效应蛋白复合物干扰靶基因的CRISPR/cas 系统,包括Ⅰ、Ⅲ和Ⅳ型;Class 2为用单一效应蛋白干扰靶基因的CRISPR/cas系统,包括Ⅱ、Ⅴ和VI型。
Ⅰ型系统包括6 种不同亚型,系统特征性蛋白是cas3 蛋白,它包含1个 HD水解酶结构域和1个类DExH 解旋酶结构域,所有亚型均编码crRNA引导复合物 Cascade(CRISPR-Associated Complex for Antiviral Defe nse),包括cas1、cas2、cas3等多个蛋白的复合物,这个复合物具有RNA核酸内切酶活性,参与precrRNA转录过程;Type III系统特异性蛋白cas10,其具有RNA内切酶蛋白活性,与TypeⅠCascade功能相似,参与crRNA的剪切和成熟,包括 Type III-A csm和 Type III-B cmr 2种类型,既可以靶标DNA,又可以靶标RNA;Ⅳ型缺少cas1、cas2蛋白,替代的多效应蛋白复合物有cas5、cas7、csf1蛋白,csf1是该系统的特征性蛋白,根据是否有解旋酶DinG可以分为2种亚型[5-7]。
Class2 类较Class 1类系统结构更简单、清楚。CRISPR/cas9为 Class2 Ⅱ 型 系 统,cas9 包 含 RuvC和 HNH(His-Asn-His domain)2个 结 合 域, 切 割位点邻近crRNA互补序列下游的原间隔区相邻基序(Protospacer Adjacent Motif,PAM),其中HNH切割与 crRNA 互补的DNA链,而RuvC切割非互补链,产生平末端双链切口(Double Stand Break,DSB)。Ⅴ型包括RuvC以及核酸内切酶结合域(Nuc)构成的效应蛋白cas12a、cas12b、cas12c,产生粘性末端双链切口,该型在pre-crRNA加工过程对tracrRNA需求不同[8-9]。VI包 括 2个 HEPN(Higher Eukaryote and Prokaryote Nucleotide-Binding)结合域,具有RNA核酸内切酶活力,介导靶向RNA编辑系统,由crRNA互补配对的ssRNA活化HEPN结合域,引导非序列特异性剪切[10]。
1.2 CRISPR作用机制 无论哪种类型的CRISPR系统,其抵御外源遗传物质的防御过程原理都大致相同,以目前研究最为清楚的 CRISPR/cas9 系统为例(图2):在整合阶段,CRISPR 系统会捕捉噬菌体或外源的DNA,以cas9识别PAM(NGG)序列作为挑选间隔序列的条件,由cas1、cas2及Ⅱ型其他因子介导将间隔序列整合到CRISPR中的重复序列之间;在组装阶段,间隔序列转录形成pre-CrRNA在cas9与RNaseⅢ及其他因子作用下,与反向激活crRNA(Trans-Activating crRNA,tracrRNA)互补配对组装形成crRNA-tracRNA-Cas9复合物;在干扰阶段,识别后准确地切除或降解靶定的序列,这个过程由RNA-蛋白酶复合物执行,crRNA识别与其碱基互补配对序列,cas9将其进行切割,造成双链断裂,激活细胞的非同源末端连接(Non-Homologous End Joining,NHEJ)或同源重组(Homologous Recombination,HR)2种修复机制,从而实现基因的插入或缺失[11]。
2 CRISPR新型效应蛋白
2.1 RNase活性的Lshc2c2、Fncas9 随着对细菌免疫系统基础机制研究的深入,Type II (Fncas9)和Type VI(c2c2)被发现具有切割RNA链活性的功能,拓宽了CRISPR系统靶向范围。c2c2在细菌Leptotrichia shahii被发现,识别位点是3´端原间隔序列单核苷酸(PFS)A、U、C,它在ssRNA结合位点外的不同距离上剪切ssRNA。至今,c2c2还未应用于真核生物细胞中[10]。另一个具有相似RNA序列识别功能的核酸内切酶Fncas9(Francisella novicida)于2013年被发现 ,2015年被运用于抑制人肝癌细胞中丙型肝炎病毒HCV(Hepatitis C Virus)的增殖,Price等[13]设计了rg RNA(RNA-Targeting Guide RNA )靶定HCV正负义链 5´、3´UTR区,抑制RNA病毒基因复制和蛋白翻译来抑制病毒在细胞上的增殖,令人惊奇的是点突变在Fncas9 的Ruv C 和HNH 功能域激活位点(D11A 和H969A),并没有降低抑制HCV病毒效率,而点突变在富含精氨酸序列ARM(Arginine-Rich Motif)R59A位点(cas9与crRNA相互结合作用),显著降低了抑制HCV病毒效率,表明Fncas9系统介导的RNA引导RNA序列剪切作用的机理值得更深入的研究。

图1 CRISPR/cas 系统分类[3,11]
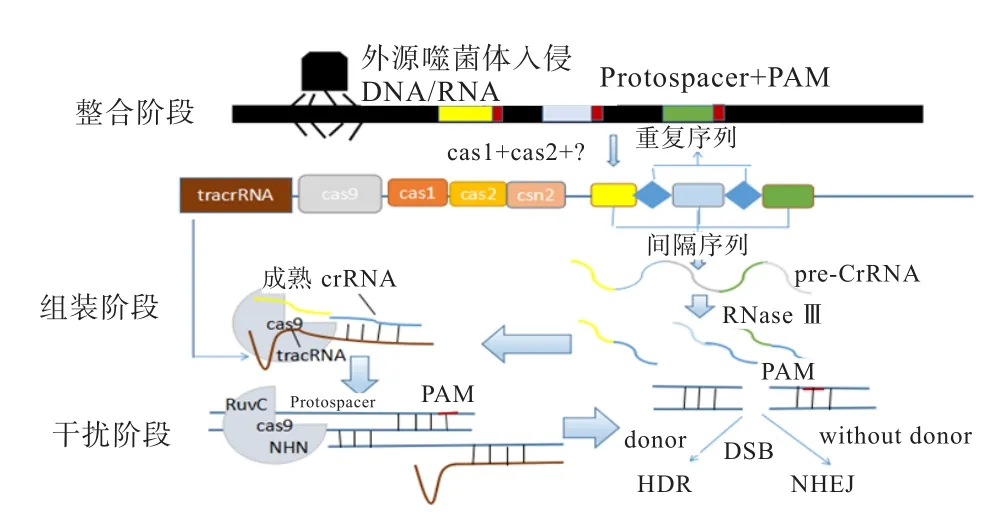
图2 自然环境下抵御外源遗传物质的防御原理(CRISPR/cas9)[12]
2.2 小分子效应蛋白 近年发现,与cas9同源形式的Staphylococcus aureus(Sacas9)、Streptococcus thermophilus(St1cas9)和Neisseria meningitidis(Nmcas9),分子量分别为3.2、3.4、3.2 kb,比Spcas9(4.2 kb)较小,同属于Type II免疫系统。由于Spcas9蛋白较大,运转到真核生物上有时候需要包装成病毒,而小分子效应蛋白更容易被转运到生物体内,这些cas9同源效应蛋白较Spcas9需要更长的PAM (间隔序列临近基序),缩小靶点范围,但也减少了脱靶位点数量[14-16]。
2015年张锋小组发现的 CRISPR/ cpf1 属于 2 类Ⅴ型系统,包括Francisella novicida(Fncpf1)、Acidaminococcus(Ascpf1)、Lachnospiraceae bacterium(Lbcpf1),而cpf1 效应蛋白同时具DNA和RNA内切酶活性,因此初级转录产物pre-crRNA由cpf1自身加工,不需要tracrRNA的参与,cpf1剪切DNA所需的crRNA长度仅为42~44 nt,只需替换CRISPR 中的间隔序列就可以靶定不同基因,cpf1的发现扩大了CRISPR系统基因组编辑的靶点范围,尤其是富含AT的基因组[17-18]。
3 Spcas9各种变构体
3.1 提高特异性变构体 Spcas9-nickase单切口酶是一种Spcas9变体,通过突变其中1个催化亚基HNH或RuvC形成的效应物,而dSpcas9是Spcas9 2个结构域RuvC的D10A位点和HNH的H840A 位点同时突变形成的新变体,有RNA介导DNA结合能力而无内切酶活性,用荧光或其他蛋白标签标记,可用于活体DNA成像,该变体与FokI核酸酶融合,形成FokI-dcas9,降低脱靶概率,提高准确性[19]。Spcas9-CD是dSpcas9酶N端与胞苷脱氨酶融合形成新变体,这个变体直接介导C转变U,在DNA复制期间,A结合U,T结合A,最后导致 C-T(或G-A)转变,是一个碱基编辑工具。Komor等[20]在体内外验证了BE3(APOBEC-XTEN-dcas9)的碱基特异性编辑效率比Spcas9通过HDR介导编辑效率高,并靶定载脂蛋白E(APOE4 )在112和158位氨基酸碱基 C-T转变,为治愈晚发性阿尔茨海默病提供可能,为未来基因治愈建立方法。Kleinstiverd等[21]开发了Spcas9-HF1,破坏Spcas9:sgRNA复合物与DNA结合位点(N497,R661,Q695,Q926),降低对非靶位点DNA剪切能力,提高Spcas9效应蛋白在基因组水平上靶向特异性。
3.2 转录水平调控变构体 dSpcas9可以融合适当效应域,调控基因转录激活或抑制(CRISPRa,CRISPRi)。Larson等[22]在大肠杆菌和人细胞上单独利用dSpcas9,设计sgRNA靶定蛋白编码区序列或UTR区,使dcas9-sgRNA复合物结合到DNA链上,阻止了RNA聚合酶(RNAP)靠近,阻止转录延伸,另设计sgRNA靶定在启动子RNAP结合位点(-35和-10)或转录因子结合位点(TFBS),使dcas9-sgRNA复合物结合到起始位点,阻止RNAP或转录因子与DNA链结合,阻止转录的起始[29-31]。
Piatek等[23]将dSpcas9的C端与激活效应域EDLL和TAL融合,靶定目标基因启动子序列Bs3:uidA(即Bs3启动子启动uida报告基因)和PDS基因启动子,提高了uida/PDS在植物细胞表达水平,并融合dcas9与SRDX抑制效应域得到dcas9:SRDX,靶定PDS基因启动子,均能有效地抑制PDS基因表达水平。Gilbert等[24]融合转录抑制域KRAB domain of Kox1得到dCas9-KRAB,并转染至人HEK293细胞,抑制转录水平上的表达,同时结合转录激活域VP64和p65均不同程度激活了报告GFP基因在人HEK293细胞转录水平的表达。
3.3 表观调控变构体 dSpcas9也可通过靶定基因序列甲基化来调控基因表达。Liu等[25]利用dspcas9 蛋白分别与Tet1及Dnmt3a融合建立了有效介导基因组去甲基化(dcas9-Tet1)和甲基化(dcas9-Dnmt3a)的方法,利用dcas9-Tet1介导神经细胞BDNF基因启动子 IV和MyoD基因增强子去甲基化,提高了BDNF/MyoD基因表达水平;利用dcas9-Dnmt3介导miR-290的CTCF锚定靶点甲基化,阻止招募CTCF因子结合成环,从而暴露了增强子,提高了临近环Nlrp12基因表达水平。Vojta等[26]利用dcas9-Dnmt3a系统靶定BACH2和IL6ST基因启动子CpG 岛使其甲基化,从而下调了这2个基因的表达水平,为研究这2个基因表达与炎症性肠道疾病免疫球蛋白N端糖基化之间可能存在的联系奠定基础。Choudhury等[27]利用dcas9-Tet1系统介导BRCA1基因启动子去甲基化,上调BRCA1基因表达水平。有研究报道,沉默BRCA1基因会提高得十几种类型癌症的概率[28-30],为未来治疗癌症提供可能。
4 CRISPR/cas在家禽上的应用
4.1 发育模型建立 胚胎发育是一个极其复杂的生物过程,往往需要利用一些实验模型系统进行研究胚胎发育机制,与其他动物模型相比,鸡胚由于易于手术操作,具有来源方便、经济实用且可同时进行大样本实验观察等优点,一直是发育生物学研究的经典实验模型,具有重要的实验价值[31]。Véron等[32]利用CRISPR技术靶定转录因子PAX7(主要表达在早期胚胎神经管背部细胞),利用Tet-on系统和Tol2转座系统,将表达cas9蛋白和2条靶向PAX7的sgRNA通过转座酶整合到鸡胚基因组中,加入多西环素TRE promoter启动cas9蛋白表达,得到大片段稳定敲除PAX7基因的鸡胚,为后续研究PAX7基因在胚胎发育过程中功能研究奠定基础。
随着对胚胎干细胞(ESC)分化机制的研究,人们发现更多调控ESC向原生殖细胞(PGC)分化的关键基因。Zuo等[33]通过CRISPR/cas9技术敲除C1EIS(Chromosome1 Expression in SSC)基因发现,视黄酸(Retinoic Acid,RA)诱导ESC向SSC分化过程中敲除组ESC表面标记基因nanog和sox2表达量显著高于对照组和过表达组,相反精原干细胞SSC表面标记基因integrina6和stm8表达量显著低于对照组和过表达组,验证了C1EIS基因在雄性生殖细胞分化过程中起重要作用。
4.2 生产性状改良 由于禽类多为卵生动物,受精卵紧密结合于表面蛋黄,而且未受精的卵子也很难取出,因此禽类不可能像哺乳动物那样通过核移植获得基因编辑个体,因此转基因禽类的发展比较缓慢。Bai等[34]利用自己构建的stCRISRP/cas9系统和SSA-RPG 双报告载体系统,在DF-1细胞系上也成功地敲除了过氧化物酶体增生物激活受体(PPAR),线粒体ATP酶合成体ε亚基(ATP5E)和OVA基因,为后续应用CRISPR改良性状建立方法。Oishi等[35]利用CRISPR/cas9系统,借助嘌呤霉素及博来霉素富集高效敲除了卵清蛋白(OVA)和卵类黏蛋白(OVM)的鸡PGC细胞,同时将敲除鸡OVM基因的PGC细胞注射入鸡胚的血管中,得到了精子是嵌合体的转基因公鸡。将此公鸡的后代通过杂交获得OVM基因敲除的纯合体转基因鸡,这是首次利用CRISPR/cas9系统制备的转基因鸡。Caitlin等[36]利用CRISPR/cas9系统分别设计2条sgRNA靶定GFP(通过Tol2转座系统整合外源GFP)和DMRT1基因(DMRT1定位在Z染色体上,之前研究报道表明与性别发育有关[37]),通过精子携带基因转移SMGT(Sperm Mediated Gene Transfer)得到后代基因敲除鸡,为生产改良性状家禽提供思路。胡曼等[38]利用CRISPR/cas9技术敲除肌肉生成抑制素(Myostatin,MSTN)为未来改良畜禽瘦肉率、改善肉质性状奠定基础。Kim等[39]利用CRISPR/cas9技术在鹌鹑成肌细胞上对MyoD基因进行敲除,降低了MyoD基因的表达水平,研究MyoD在肌肉分化过程中的调控作用,深入了解肌肉增殖分化机制,为改善肉质性状作铺垫。
5 结 语
目前,CRISPR/cas 系统相关的基因组编辑技术已被应用于多个研究领域,包括基因功能研究、基因治疗、疾病模式动物的构建及畜禽遗传改良方面的运用等,但同时也存在许多技术局限性,如细胞毒性和脱靶效应等。为降低脱靶效应应慎重靶点筛选,适当缩小sgRNA长度,设计成对sgRNA结合单切口酶或dcas9-fokI融合酶等。
对于临床应用的安全性,科学家们也迫切地在大自然中寻找可以控制CRISPR/cas系统的开关蛋白,继Bondy-Denomy等[40]、Pawluk等[41]在铜绿假单胞菌分别发现了关闭type I-F 和 I-E系统“anti-CRISPR”小分子蛋白(50~150个氨基酸),这些阻滞蛋白分子量小,可以随意通过核孔进出作用于效应蛋白。Rauch等[42]发现了关闭TII-A(目前应用最广效应蛋白Spcas9)的“anti-CRISPR”蛋白AcrIIA4,并在大肠杆菌和人细胞上成功抑制了Spcas9/dSpcas9靶向编辑效率。此外,Shin等[43]在体内外验证了 AcrIIA4蛋白抑制Spcas9编辑效率,并利用AcrIIA4成功降低了HBB、VEGF基因的脱靶效率。随着CRISPR系统的深入研究和开发,期待未来CRISPR系统会得到更广阔的发展和应用。
[1] Jinek M, Chylinski K, Fonfara I,et al. A programmable dual-RNA -guideDNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337(6096): 816-821.
[2] Makarova Y I, Wolf O S, Alkhnbashi F,et al. An updated evolutionary classification of CRISPR–Cas systems[J]. Nat Rev Microbiol, 2015, 13(11): 722-736.
[3] Shmakov O O, Abudayyeh K S, Makarova Y I,et al. Discovery and functional characterization of diverse Class 2 CRISPR–Cas systems[J]. Mol Cell, 2015, 60(3): 385-397.
[4] D Burstein L B, Harrington S C, Strutt A J,et al. New CRISPR–Cas systems from uncultivated microbes[J]. Nature, 2017,542(7640): 237-241.
[5] Makarova K S, Haft D H, Barrangou R,et al. Evolution and classification of the CRISPR-Cas systems[J]. Nat Rev Microbiol, 2011, 9: 467-477.
[6] Marraffini L A, Sontheimer E J. CRISPR interference: RNA-directed adaptive immunity in bacteria and archaea[J]. Nat Rev Gene, 2010, 11(3):181-190.
[7] Rath D, Amlinger L, Rath A,et al. The CRISPR-Cas immune system: biologymechanisms and applications[J]. Biochimie,2015, 117: 119-128.
[8] Deltcheva E, Chylinski K, Sharma C M,et al. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III[J]. Nature, 2011, 471: 602-607.
[9] Yamano T, Nishimasu H, Zetsche B,et al. Crystal structure of Cpf1 in complex With guide RNA and target DNA[J]. Cell,2016, 165(4): 949-962.
[10] Abudayyeh O O, Gootenberg J S, Konermann S,et al. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector[J]. Science, 2016, 353: 5573.
[11] East-Seletsky A O, Connell M R, Knight S C,et al. Diversity,classification and evolution of CRISPR-Cas systems[J].Microbiology, 2017, 37: 67-78.
[12] Cong L. CRISPR:Groundbreaking technology for RNA-guided genome engineering[J]. Analytical Biochem, 2017, 532: 87-89.
[13] Price A A, Sampson T R, Ratner H K,et al. Cas9-mediated targeting of viral RNA in eukaryotic cells[J]. Proc Natl Aca Sci USA, 2015, 112(19): 6164-6169.
[14] Ran F A, Cong L, Yan W X,et al. In vivo genome editing using Staphylococcus aureus Cas9[J].Nature, 2015, 520(7546): 186-191.
[15] Kleinstiver B P, Prew M S, Tsai S Q,et al. Engineered CRISPRCas9 nucleases with altered PAM specificities[J]. Nature, 2015,523(7561): 481-485.
[16] Hou Z,Zhang Y, Propson N E,et al. Efficient genome engineering in human pluripotent stem cells using Cas9 from Neisseria meningitidis[J]. Proc Natl Acad Sci, 110(39): 15644-15649.
[17] Kim H K, Song M, Lee J,et al. In vivo high-throughput profiling of CRISPR-Cpf1 activity[J]. Nat Methods, 2017,14(2): 153-159.
[18] Kleinstiver B P, Tsai S Q, Prew M S,et al. Genome-wide specificities of CRISPR-Cas Cpf1 nucleases in human cells[J].Nat Biotechnol, 2016, 34(8): 869-874.
[19] Ran F A, Hsu P D, Lin C Y,et al. Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity[J]. Cell, 2013, 154(6): 1380-1389.
[20] Komor A C, Kim Y B, Packer M S,et al. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage[J]. Nature, 2016, 533(7603): 420-424.
[21] Kleinstiver B P, Pattanayak V, Prew M S ,et al. High-fidelity CRISPR-Cas9 nucleases with no detectable genome-wide offtarget effects[J]. Nature, 2016, 529(7587): 490–495.
[22] Larson M H, Gilbert L A, Wang X,et al. CRISPR interference(CRISPRi) for sequence-specific control of gene expression[J].Nat Protoc, 2013, 8(11): 2180-2196.
[23] Piatek A, li Z, Baazim H,et al. RNA-guided transcriptional regulation in planta via synthetic dCas9-based transcription factors[J]. Plant Biotechnol, 2015, 13(4): 578-589.
[24] Gilbert L A, Larson M H, Morsut L,et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes[J]. Cell, 2013, 154(2): 442-451.
[25] Liu X S, Wu H, Ji X,et al. Editing DNA methylation in the mammalian genome[J]. Cell, 2016, 167(1): 233-247.
[26] Vojta A, Dobrinic P, Tadic V,et al. Repurposing the CRISPRCas9 system for targeted DNA methylation[J]. Nucleic Acids Res, 2016, 44(12): 5615-5628.
[27] Choudhury S R, Cui Y, Lubecka K,et al. CRISPR-dCas9 mediated TET1 targeting for selective DNA demethylation at BRCA1 promoter[J]. Oncotarget, 2016, 7(29): 46545-46556.
[28] Marot D, Opolon P, Brailly-Tabard S. The tumor suppressor activity induced by adenovirus-mediated BRCA1 overexpression is not restricted to breast cancers[J].Gene Ther.2006, 13(3): 235–244.
[29] Fan S, Wang J A, Yuan R Q. BRCA1 as a potential human prostate tumor suppressor: modulation of proliferation, damage responses and expression of cell regulatory proteins[J].Oncogene. 1998, 16(23): 3069–3082.
[30] Thompson D, Easton D F, Consortium BCL. Cancer Incidence in BRCA1 mutation carriers[J]. J Natl Cancer Inst. 2002,94(18): 1358–1365.
[31] 王恒, 殷慧群, 章孝荣, 等. 鸡胚模型在生物研究中的应用进展[J]. 生命科学, 2009, 21(2): 312-315.
[32] Véron N, Qu Z, Kipen P A,et al. CRISPR mediated somatic cell genome engineering in the chicken[J]. Dev Biol, 2015,407(1): 68-74.
[33] Zuo Q, Jin K, Wang Y,et al. CRISPR/Cas9-mediated deletion of c1eis inhibits chicken embryonic stem cell differentiation into male germ cells[J]. J Cell Biochem, 2017, 118(8): 2380-2386.
[34] Bai Y, He L, Li P,et al. Efficient genome editing in chicken DF-1 cells using the CRISPR/Cas9 system[J]. Gene, 2016, 6(4):917-923.
[35] Oishi I, Yoshii K, Miyahara D,et al. Targeted mutagenesis in chicken using CRISPR/Cas9system[J]. Sci Rep, 2016, 6:23980.
[36] Cooper C A, Challagulla A, Jenkins K A. Generation of gene edited birds in one generation using sperm transfection assisted gene editing (STAGE)[J]. Transgenic Res, 2017, 26(3): 331-347.
[37] Smith C A, Roeszler K N, Ohnesorg T, The avian Z-linked gene DMRT1 is required for male sex determination in the chicken[J]. Nature, 2009, 461(7261): 267-271.
[38] 胡曼, 康倩倩, 胡晓湘, 等. CRISPR/Cas9系统在鸡MSTN上的效率验证[J]. 中国家禽, 2016, 38(7): 5-9.
[39] Kim S W, Lee J H, Park B C,et al. Myotube differentiation in clustered regularly interspaced shortpalindromic repeat/Cas9-mediated MyoD knockout quail myoblast cells[J]. Asian-Australas J Anim Sci, 2017, 30(7): 1029-1036.
[40] Bondy-Denomy J, Pawluk A, Maxwell,et al. Bacteriophage genes that inactivate the CRISPR/Cas bacterial immune system[J]. Nature, 2013, 493(7432): 429-432.
[41] Pawluk A, Bondy-Denomy J, Cheungn V H,et al. A new group of phage anti-CRISPR genes inhibits the typeI-E CRISPRCas system of Pseudomonas aeruginosa[J]. Mbio, 2014, 5(2):e00896.
[42] Rauch B J, Silvis M R, Hultquist J F,et al. Inhibition of CRISPR-Cas9 with bacteriophage proteins[J]. Cell, 2017,168(1-2): 150-158.
[43] Shin J, Jiang F, Liu J J,et al. Disabling Cas9 by an anti-CRISPR DNA mimic[J]. Sci Adv, 2017, 3(7): e1701620.
