基于26S rRNA D1/D2区序列对不同时期大曲中酵母菌的分离与鉴定
2018-03-13窦晓杨建刚曹新志马莹莹郭家秀张琦苏畅
窦晓,杨建刚,曹新志,马莹莹,郭家秀,张琦,苏畅
(四川理工学院生物工程学院,四川自贡 643000)
大曲作为中国白酒发酵过程中的发酵剂,可以给发酵过程提供微生物、前体风味物质以及各种酶类[1~3]。酵母菌作为大曲微生物中的主要组成部分之一,对大曲的功能的形成有着不可或缺的作用。传统的酵母菌株鉴定方法建立在菌株的形态、生理特性和生物化学特性质差异之上。然而这些特征会受培养条件的影响,在某种程度上具有不确定性,为确保鉴定结果的可靠性,鉴定一株菌经常需要完成 50~100项实验[4]。费时费力且重复性不高。
核糖体RNA基因(rRNA)存在于所有细胞生物中并具有相同的起源和功能,因而可以反映所有物种间具有可比性的进化史。而且rDNA中有些片段具有高度的序列同源性或保守性,从而可为不同物种间的系统学比较提供研究参考点。
Kurtzman & Robnett[5,6]和 Fell等[7]测定了所有已知酵母种类的26S rRNA D1 D2区序列,发现在这一600 bp左右的片段中,种内序列变异小于等于1%,而种间序列差异大于 1%。目前这一标准已被国际酵母分类学界普遍接受,从而也使酵母菌的菌种鉴定变得相对容易。近年来,以核酸为研究对象的分子生物学技术以其简便、准确等优点逐渐应用到酵母菌的分类鉴定中。
单链构象多态性(Single Strand Conformation Polymorphism,SSCP)技术是基因突变分析中最常用的方法之一。其理论基础是单链DNA具有序列特异性的二级或三级构象。一个或多个碱基的差异能影响其构象,在非变性聚丙烯酰胺凝胶电泳中,构象的改变表现为单链DNA迁移率的改变,根据各样品的单链迁移率,就可以把突变体和非突变体区分开。该方法最初应用于人类基因点突变的检测中[8,9],由于其在检测碱基差异方面的高分辨力,而广泛应用于人类和植物病原真菌种的鉴定和菌株分析中[10,11]。
本研究通过对不同时期大曲中分离的酵母菌的26S rRNA基因D1/D2区序列进行分析,在GenBank数据库中进行同源序列搜索(BLAST),比较供试菌株与已知酵母菌相应序列的同源性。依据分离鉴定的结果对大曲生产过程中的酵母菌的演替规律进行了初步探究。
1 材料与方法
1.1 材料
1.1.1 样品
大曲取自泸州老窖制曲生态园,对生产过程中0、3、5、7、10、25、90 d的大曲进行采集。在形态学观察的基础上,挑取了260株酵母进行26S rRNA基因D1/D2区的序列测序与比对。
1.1.2 仪器试剂
基因扩增仪(GT9612)购自杭州柏恒科技有限公司;离心机(CF15R)购自日立集团;电泳仪(JK600C)购自北京君意东方电泳设备有限公司;凝胶成像系统Alpha Innotech购自美国Alpha公司;Taq PCR Master Mix,引物NL1,NL4均购自北京博迈德生物技术有限公司。
1.2 方法
1.2.1 酵母菌分离
在泸州老窖制曲生态园中采取第0、3、5、7、10、25、90 d的大曲,每个时期的大曲杂碎混匀后取50 g加入到450 mL无菌水里面充分混匀后,吸取1 mL混合液到装有9 mL无菌水的试管中,依次做成10-2,10-3,10-4,10-5,10-6五个梯度,然后再用YPD培养基(酵母粉10 g/L,葡萄糖20 g/L,蛋白胨20 g/L,琼脂20 g/L,自然pH)和PDA培养基(马铃薯200 g/L,葡萄糖20 g/L,琼脂20 g/L,自然pH)进行平板分离。
1.2.2 总DNA的提取
在涂布的平板上挑取单菌落再划线纯化,分离纯的酵母菌株。用Makimura等[12]方法提取菌体总DNA。在斜面上挑取少量酵母菌体(一环),置于一已灭菌的1.5 mL的离心管内。加入100 μL裂解液(100 mM Tris,30 mM EDTA,0.5% SDS,pH 8.0 15P灭菌30 min备用),100 ℃水浴15 min。加入100 μL 2.5M的醋酸钾后(室温冷却后),置于冰上30 min至60 min。在4 ℃下13000 r/min离心5 min,上清液移至一新1.5 mL离心管内(200 μL枪)。加入等体积的氯仿异戊醇(24:1),剧烈震荡10 min(上下摇动,除去蛋白),13000 r/min,离心15 min,吸取100 μL上清液至另一只灭菌的1.5 mL离心管中。重复上一步,直到两相之间没有沉淀,将上清液移至另一只灭菌的 1.5 mL离心管中(可省略)。加入等体积(100 μL)的冷的异丙醇,放置-20 ℃静置15 min(沉淀DNA)。13000 r/min离心15 min。用100 μL 70%乙醇洗涤沉淀。重复上一步骤。将沉淀置于真空干燥器中抽干乙醇(65 ℃烘干 30 min或65 ℃水浴)。加入 50 μL已灭菌的 MILLI-Q 水(ddH2O),在4 ℃下溶解2 h,-20 ℃保存备用。
1.2.3 PCR扩增和测序
PCR扩增26S rRNA D1 D2区,PCR扩增反应所用引物参照 Kurtzman等[5],正向引物 NL1:5′-GCATATCGGTAAGCGGAGGAAAAG-3′,反向引物 NL4:5′-GGTCCGTGTTTCAAGACGG-3′。PCR 采用25 μL体系,扩增反应条件:95 ℃ 5 min;94 ℃ 45 s,55 ℃ 45 s,72 ℃ 45 s,循环 35 次;72 ℃ 8 min。PCR反应产物的琼脂糖电泳检测:取2 μL PCR扩增原液点样于 1%的琼脂糖凝胶,电泳,溴化已锭(EB)染色后在紫外灯照射下确定是否扩出所要片段。26S D1 D2区扩增出单一明亮条带,片段大小约为500~600 bp。
1.2.4 DNA序列分析方法
DNA序列分析:用DNA Star软件并结合DNA正反向序列图谱对DNA序列进行拼接和校正。将校正后的序列在国际核酸数据库(http:www.ncbi.nlm.nih.gov.blast)中进行同源序列搜索,初步确定受试菌株的分类地位,一般在D1 D2区可以按如下经验值判断:(1)与最近缘种的模式菌株相似率为 100%,可确定为同一种;(2)与最近缘种的模式菌株的相似率<98%,可初步确定为新种;(3)与最近缘种的模式菌株的相似率在99%~100%之间,它们的关系要视不同的情况而定,除考虑序列差异外,还应考虑生理生化性状差异。
SSCP分析:用 DCodeTM 突变检测电泳仪进行SSCP检测。参照使用说明配置丙稀酰胺,甲叉双丙稀酰胺(37.5:1)的8%凝胶。把PCR产物稀释2~5倍,取5 μL(大约150~300 ng)与等量的上样缓冲液(0.05%溴酚兰,0.05%二甲苯青,95%甲酰胺,4% 0.5 mol EDTA,pH 8.0)混匀,在95 ℃下变性10 min,迅速放在冰上,放置15 min。把变性后的样品依次加入到点样孔中,200 V电压10 ℃电泳16 h。电泳结束后将凝胶按照 Wallace[13]的方法进行硝酸银染色,用相机拍照保存。
2 结果与讨论
2.1 26S rRNA D1/D2区序列的扩增
在对分离的260株菌的26S rRNA D1/D2区序列进行了PCR扩增,下图为PCR扩增产物电泳图,显示产物的长度都在500~750 bp,这与酵母菌的D1/D2区域序列长度为500~600 bp左右相符合,说明扩增正常。电泳效果图见图1。

图1 26S rRNA D1/D2区序列PCR扩增产物电泳图Fig.1 PCR amplification product electrophoresis of 26S rRNA D1/D2 region sequence
2.2 26S rRNA D1/D2区序列的比对
对分离的260株菌的26SrRNA基因D1/D2区序列在 GenBank数据库中进行同源序列搜索(BLAST)后,将同源性大于 99%的菌株归为同一个种发现有101株菌与异常威克汉姆酵母(Wickerhamomyces anomalus)的同源性高于99%,故将这101株酵母菌鉴定为异常威克汉逊酵母(Wickerhamomyces anomalus);其中 52株菌与扣囊覆膜酵母(Saccharomycopsis fibuligera)的同源性大于99%,故鉴定为扣囊覆膜酵母(Saccharomycopsis fibuligera);11株菌与酿酒酵母(Saccharomyces cerevisiae)的相似度为 100%;Clavispora lusitaniae 18株;库德毕赤酵母(Pichia kudriavzevii)15株;假丝酵母(Candida glabrata)16株;Kluyveromyces marxianus 13株;Torulaspora delbrueckii 9株;生丝毕赤酵母(Hyphopichia burtonii)7株;Meyerozyma guilliermondii 3株;Pichia caribbica 2株;其余均为一株。表1为部分菌株在Genbank数据库中进行同源序列搜索后结果。

表1 部分菌株26S rRNA基因D1/D2区与模式菌株比对结果Table 1 Comparison of 26S rRNA gene D1 / D2 region of part of the strains with model strains
2.3 对不同种菌株的 26S rRNA基因D1/D2-SSCP图谱比较
为了进一步确定它们之间的种间差异,对部分不同菌株进行26S rRNA D1/D2-SSCP图谱分析比较,结果如图2所示。所比较株菌D1/D2-SSCP图谱都包括两条泳动慢的单链和一条泳动快的双链。双链位置的不同反映菌株间26S rRNA基因D1/D2区片段长度的差异,碱基序列的差异通过单链位置及两单链位置关系的差异反映在SSCP图谱上。因此,由图2可以看出不同类型株菌的核型差异很明显。

图2 部分菌株间的26S rRNA基因D1/D2-SSCP图谱比较Fig.2 Comparison of 26S rRNA D1 / D2-SSCP patterns among some strains
2.4 基于26S rRNA D1/D2区序列构建M-L系统树

图3 基于26S rRNA基因D1/D2区序列构建的M-L系统树Fig.3 M-L system tree based on 26S rRNA D1 / D2 regionsequence
用软件MEGA6.06对所有序列进行比对后,删除两端未对齐的碱基生成进化树,并进行1000次的Bootstraps检验。采用“Maximum Likelihood Tree”(M-L)方法显示进化树,结果见图。构建了22种不同菌株与其相似度最接近的模式菌株的M-L系统树如(图3),通过系统发育树对大曲中分离出的不同类型的酵母的种间亲缘关系进行了直观界定。
2.5 不同时期大曲酵母菌的分布规律
根据对260株酵母菌的26S rRNA D1/D2区序列比对鉴定的结果以及前期平板计数的结果对不同时期大曲中的酵母菌进行统计分析。如图4所示,在大曲生产初期酵母种群数量较多,在顶温区(7 d~10 d)种群减少,酵母数量最多,但种群开始减少,主要以异常威克汉姆酵母(Wickerhamomyces anomalus)和扣囊复膜酵母(Saccharomycopsis fibuligera)为主,成品大曲(90 d以后)酵母主要以扣囊复膜酵母(Saccharomycopsis fibuligera)为主。这可能是随着大曲培菌过程中温度的变化使酵母群落发生了相应的演替。
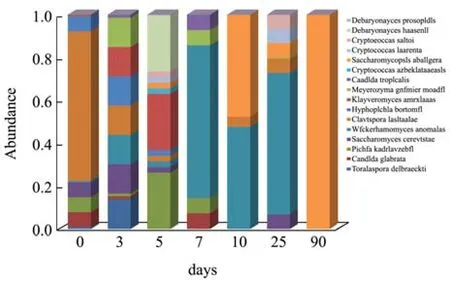
图4 不同时期大曲中酵母的分布Fig.4 Yeastdistributions in Daqu in different periods
3 讨论
3.1 本文通过传统的平板计数的方法对大曲培菌过程中的酵母菌的种群变化进行了初步的统计分析,发现顶温区(7~10 d)酵母数量最多,主要以异常威克汉姆酵母(Wickerhamomyces anomalus)和扣囊复膜酵母(Saccharomycopsis fibuLigera)为主,成品大曲(90 d以后)酵母主要以扣囊复膜酵母(Saccharomycopsis fibuligera)为主。成品大曲中的酵母菌种类相对单一,而它又能直接参与中国白酒的发酵过程,因此对成品大曲中含量最高的扣囊复膜酵母进行更深一步的的研究很有必要。但传统分离由于自身的局限性对各时期的优势菌株的体现并没有现代分子生物学那样系统直观,例如DGGE和高通量等方法。Zhang Liqiang等运用PCR-DGGE技术解析了不同类型大曲中微生物的组成差异[14];Zheng等运用高通量测序技术对30年窖龄和300年窖龄的泸州老窖窖泥微生物的多样性进行了系统分析[15];这些技术与传统的分离鉴定方法存在着很大的优势,可以简单直观地反映出某一时期某种物质中的可培养的以及不可培养的微生物的组成,但传统分离也存在着自身独有的优势,相比较现代分子生物学技术它可以得到活的菌株,为微生物的进一步研究提供基础。因此运用现代分子生物学技术手段来指导传统分离将是以后的发展方向。
3.2 随着DNA序列分析技术的日趋成熟和简易化,序列分析方法已经被广泛应用于酵母菌的分类鉴定以及系统学研究。而 26S rRNA基因D1/D2区序列在GenBank/EMBL/DDBJ等国际核酸序列数据库中的公布为酵母菌的分类鉴定带来了极大的方便[16]。
3.3 26S rRNA的Dl/D2区域位于大亚基的5′端,序列长度在600 bp左右,该段区域具有较高的变异率,可以用于亲缘关系较近的菌株之间的分类研究。此区域序列已建成了完备的分析数据库,用其进行酵母菌种的鉴定更为方便准确,其应用也最为广泛[17,18]。本实验也是依于此对大曲生产过程中不同时期的样品中分离出的酵母菌进行了26S rRNA基因D1 D2区的序列同源性分析,共分析鉴定了260株酵母菌。从而确定了大曲生产过程中不同时期的酵母菌的组成结构,初步掌握了大曲中酵母菌的演替规律,对研究传统中国白酒的功能微生物奠定了一定的基础。
[1]Xiao-Wei Zheng, Minoo Rezaei Tabrizi, M J Robert Nout, et al. Daqu-A traditional Chinese liquor fermentation starter [J].Journal of the Institute of Brewing, 2011, 177: 82-90
[2]Xiao-Wei Zheng, Zheng Yan, Bei-Zhong Han, et al. Complex microbiota of a Chinese “Fen” liquor fermentation starter(Fen-Daqu), revealed by cμLture-dependent and cμLtureindependent methods [J]. Food Microbiology, 2012, 31(2):293-300
[3]Chang-lu Wang, Dong-jian Shi, Guo-li Gong.Microorganisms in Daqu: a starter cμLture of Chinese [J].World J Microbiol. Biotechnol., 2008, 24(10): 2183-2190
[4]Lin C CS, Fung D Y. Conventional and rapid methods for yeast identification [J]. Crit. Rew. Microbiolo., 1987, 14(4):273-289
[5]Kurtzman C P, Robnett C J. Identification and phylogeny of ascomycetous yeasts from analysis of nuclear large subunit(26S) ribosomal DNA partial sequences [J]. Antonie van Leeuwenhoek, 1998, 73(4): 331-371
[6]Kurtzman C P, Robnett C J. Identification of clinically important ascomycetous yeasts based on nucleotide divergence in the 5' end of the large-subunit (26S) ribosomal DNA gene [J]. Journal of Clinical Microbiology, 1997, 35(5):1216-1223
[7]Fell J W, Boekhout T, Fonseca A, et al. Biodiversity and systematics of basidiomycetous yeasts as determined by larger subunit rDNA D1 D2 domain sequence analysis [J].International Journal of Bacteriology, 2000, 50(3): 1351-1371
[8]Orita M, Iwahana H, Kanazawa H, et al. Detection of polymorphisms of human DNA by gel electrophoresis as single strand conformation polymorphisms [J]. Proceedings of the National Academy of Sciences , 1989, 86(8):2766-2770
[9]Orita M, Susuki Y, Sekiya T, et al. Rapid and sensitive detection of point mutations and DNA polymorphisms using the polymerase chain reaction [J]. Genomics, 1989, 5(4): 874-879
[10]Kong P, Hong C, Richardson P A, et al. Single-strand conformation polymorphism of ribosomal DNA for rapid species differentiation in genus Phytophthora [J]. Fungal Genetics and Biology, 2003, 39(3): 238-249
[11]Kumar M, Shukla P K. Use of PCR targeting of internal transcribed spacer regions and single-stranded conformation polymorphism analysis of sequence variation in different regions of rRNA genes in fungi for rapid diagnosis of mycotic keratitis [J]. Journal of Clinical Microbiology, 2005,43(2): 662-668
[12]Makimura K, Murayama Y S, Yamaguchi H. Detection of a wide range of medically important fungal species by polymerase chain reaction (PCR) [J]. Journal of Medical Microbiology, 1994, 40(5): 358-364
[13]Wallace A J. SSCP heteroduplex analysis. In PCR mutationdetection protocols. Ed. By Theophilus BDM,Rapley R. 2000, 151-163
[14]Liqiang Zhang, Chongde Wu, Xiaofei Ding, et al.Characterization of microbial communities in Chinese liquor fermentation starters Daqu using nested PCR-DGGE [J].World Journal of Microbiology & Biotechnology, 2014,30(12): 3055-3063
[15]Qi Zheng, Bairong Lin, Yibin Wang, et al. Proteomic and high-throughput analysis of protein expression and microbial diversity of microbes from 30- and 300-year pit muds of Chinese Luzhou-flavor liquor [J]. Food Research International, 2015, 75: 305-314
[16]王泽举,王汝瑱,孙悦,等.几株酵母菌的分子系统学鉴定[J].食品科学,2012,15:195-200 WANG Ze-ju, WANG Ru-zhen, SUN Yue, et al. Molecular phylogenic identification of wine-related yeasts [J]. Food Science, 2012, 15: 195-200
[17]Fell J W, Boekhout T, Fonseca A, et al. Biodiversity and systematics of basidiomycetous yeasts as determined by large-subunit rDNA D1/D2 domain sequence analysis [J]. Int.J Syst. Evol. Microbiol., 2000, 50(3): 1351-1371
[18]Scorzetti G, Fell J W, Fonseca A, et al. Systematics of basidiomycetous yeasts: a comparison of large subunitD1/D2 and internal transcribed spacer rDNA regions [J]. FEMS Yeast Res, 2002, 2(4): 495-517
