马立克病病毒miR-M4-5p宿主靶基因cDNA文库的构建及鉴定
2018-03-13薛正飞李会珍马圣明宋利娜张改平
薛正飞,滕 蔓,李会珍,马圣明,3,宋利娜,张 雅,罗 俊,3,张改平*
(1. 河南农业大学,生命科学学院,郑州 450002; 2. 河南省农业科学院动物免疫学重点实验室,农业部动物免疫学重点实验室,河南省动物免疫学重点实验室,郑州 450002;3. 河南科技大学,动物科技学院,动物疫病与公共安全重点实验室,洛阳 471003)
马立克病(Marek’s disease,MD)是由马立克病病毒(Marek’s disease virus,MDV)感染鸡引起的一种肿瘤性免疫抑制性传染病[1]。MDV属于疱疹病毒科,α-疱疹病毒亚科,马立克病毒属。依据生物学特性的不同,MDV可分为血清1型(MDV-1)、血清2型(MDV-2)和血清3型(HVT)三种血清型[2],而在最新的病毒分类中又被重新命名为禽疱疹病毒2型(GaHV2)、禽疱疹病毒3型(GaHV3)和火鸡疱疹病毒Ⅰ型(MeHV1)[3]。其中,仅MDV-1具有致病性和致瘤性。目前,虽然可以用疫苗较好地预防MD肿瘤发生,但由于疫苗免疫压力及病毒自身进化等原因,MDV流行毒株的毒力正不断增强[4]。MDV感染鸡群后不仅导致宿主免疫抑制,而且可快速诱发肿瘤及大批鸡死亡,因此MDV感染被认为是研究疱疹病毒致病和致肿瘤的理想模型[5]。
microRNA(miRNA)是一类长度为21~25 nt的非编码小分子RNA,它们在细胞的发育与分化、细胞凋亡、肿瘤发生等许多生物学过程中调控蛋白编码基因的转录后表达水平[6-7]。 MDV-1共编码26个成熟体miRNA,其中miR-M4-5p与宿主miR-155具有相同的种子序列,是miR-155的同源物[8]。由于miR-155参与调控许多肿瘤的形成过程,被认为是致癌基因[9-13],miR-M4-5p可能也发挥着类似的重要作用。最近的研究表明miR-M4-5p在MDV致瘤性方面直接发挥着重要作用。敲除miR-M4-5p的MDV致病和致瘤能力显著减弱[14],但具体的作用机制仍然不是十分清楚。此前通过生物信息学软件预测和试验验证,我们已证实LTBP1是miR-M4-5p一个重要的宿主靶基因[15]。由于生物信息学方法预测miRNA的靶基因存在候选分子众多、筛选及鉴定工作量大等缺点,本研究采用hybrid-PCR的策略构建miR-M4-5p的宿主候选靶基因cDNA文库[16],通过双荧光素酶报告试验(dual fluorescence reporter assays, DLRA)分析miR-M4-5p与候选靶基因的相互作用,为进一步揭示miR-M4-5p的分子调控机制奠定基础。
1 材料与方法
1.1 细胞
HEK 293T细胞由河南省农业科学院动物免疫学重点实验室保存;鸡胚成纤维细胞(chick embryo fibroblast,CEF)分离自7~8日龄SPF鸡胚。
1.2 主要试剂、载体
TRIzol Reagent,购自Invitrogen公司;3′-Full RACE Core Set with PrimeScript RTase、PremixTaqTM、DNA限制性内切酶XhoⅠ、NotⅠ、PrimeScriptTMRT reagent kit with gDNA Eraser、DNA Ligation kit和pMD19-T(simple)vector,均购自宝生物工程(大连)有限公司;DLRA系统,购自Promega公司;Annealing Buffer for Oligos(5×),购自碧云天生物科技公司;psiCHECK-2和pcDNA6.2-miR-neg载体,由中山大学徐辉博士馈赠;pcDNA6.2-miR-M4-5p、pcDNA6.2-mut-miR-M4-5p质粒和菌种[15],由河南省农业科学院动物免疫学重点实验室构建并保存。
1.3 引物合成
本研究使用的DNA引物均使用Premier Primer 5.0软件设计,由生工生物工程(上海)股份有限公司合成。引物名称、序列等相关背景信息见表1。
表1 用于扩增或合成候选靶基因3′-UTR的引物
Table 1 Primers used for amplifying the wild type or mutated 3′-UTRs of putative mRNA targets
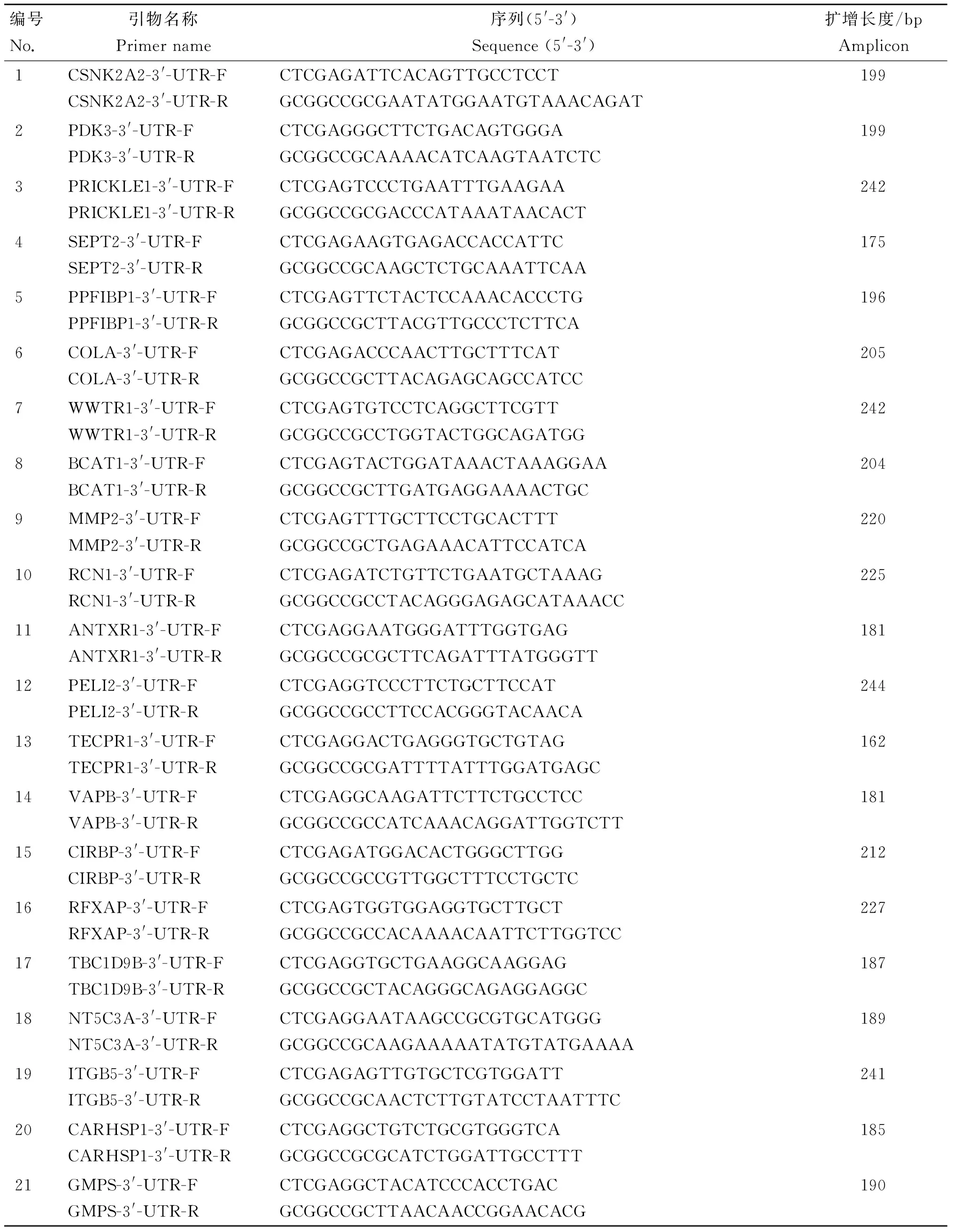
编号No.引物名称Primername序列(5'-3')Sequence(5'-3')扩增长度/bpAmplicon1CSNK2A2-3'-UTR-FCSNK2A2-3'-UTR-RCTCGAGATTCACAGTTGCCTCCTGCGGCCGCGAATATGGAATGTAAACAGAT1992PDK3-3'-UTR-FPDK3-3'-UTR-RCTCGAGGGCTTCTGACAGTGGGAGCGGCCGCAAAACATCAAGTAATCTC1993PRICKLE1-3'-UTR-FPRICKLE1-3'-UTR-RCTCGAGTCCCTGAATTTGAAGAAGCGGCCGCGACCCATAAATAACACT2424SEPT2-3'-UTR-FSEPT2-3'-UTR-RCTCGAGAAGTGAGACCACCATTCGCGGCCGCAAGCTCTGCAAATTCAA1755PPFIBP1-3'-UTR-FPPFIBP1-3'-UTR-RCTCGAGTTCTACTCCAAACACCCTGGCGGCCGCTTACGTTGCCCTCTTCA1966COLA-3'-UTR-FCOLA-3'-UTR-RCTCGAGACCCAACTTGCTTTCATGCGGCCGCTTACAGAGCAGCCATCC2057WWTR1-3'-UTR-FWWTR1-3'-UTR-RCTCGAGTGTCCTCAGGCTTCGTTGCGGCCGCCTGGTACTGGCAGATGG2428BCAT1-3'-UTR-FBCAT1-3'-UTR-RCTCGAGTACTGGATAAACTAAAGGAAGCGGCCGCTTGATGAGGAAAACTGC2049MMP2-3'-UTR-FMMP2-3'-UTR-RCTCGAGTTTGCTTCCTGCACTTTGCGGCCGCTGAGAAACATTCCATCA22010RCN1-3'-UTR-FRCN1-3'-UTR-RCTCGAGATCTGTTCTGAATGCTAAAGGCGGCCGCCTACAGGGAGAGCATAAACC22511ANTXR1-3'-UTR-FANTXR1-3'-UTR-RCTCGAGGAATGGGATTTGGTGAGGCGGCCGCGCTTCAGATTTATGGGTT18112PELI2-3'-UTR-FPELI2-3'-UTR-RCTCGAGGTCCCTTCTGCTTCCATGCGGCCGCCTTCCACGGGTACAACA24413TECPR1-3'-UTR-FTECPR1-3'-UTR-RCTCGAGGACTGAGGGTGCTGTAGGCGGCCGCGATTTTATTTGGATGAGC16214VAPB-3'-UTR-FVAPB-3'-UTR-RCTCGAGGCAAGATTCTTCTGCCTCCGCGGCCGCCATCAAACAGGATTGGTCTT18115CIRBP-3'-UTR-FCIRBP-3'-UTR-RCTCGAGATGGACACTGGGCTTGGGCGGCCGCCGTTGGCTTTCCTGCTC21216RFXAP-3'-UTR-FRFXAP-3'-UTR-RCTCGAGTGGTGGAGGTGCTTGCTGCGGCCGCCACAAAACAATTCTTGGTCC22717TBC1D9B-3'-UTR-FTBC1D9B-3'-UTR-RCTCGAGGTGCTGAAGGCAAGGAGGCGGCCGCTACAGGGCAGAGGAGGC18718NT5C3A-3'-UTR-FNT5C3A-3'-UTR-RCTCGAGGAATAAGCCGCGTGCATGGGGCGGCCGCAAGAAAAATATGTATGAAAA18919ITGB5-3'-UTR-FITGB5-3'-UTR-RCTCGAGAGTTGTGCTCGTGGATTGCGGCCGCAACTCTTGTATCCTAATTTC24120CARHSP1-3'-UTR-FCARHSP1-3'-UTR-RCTCGAGGCTGTCTGCGTGGGTCAGCGGCCGCGCATCTGGATTGCCTTT18521GMPS-3'-UTR-FGMPS-3'-UTR-RCTCGAGGCTACATCCCACCTGACGCGGCCGCTTAACAACCGGAACACG190

(转下页 Carried forward)
1.4 候选靶基因文库的构建
按照TRIzol RNA提取试剂盒说明书,提取CEF细胞的总RNA。用NanoDrop-2000全波长紫外分光光度计(Invitrogen公司)测定RNA浓度和纯度。按照3′-Full RACE Core Set with PrimeScriptTMRTase试剂盒说明书,将提取的RNA样品进行反转录合成cDNA,然后进行套式PCR反应。PCR反应中miR-M4-5p的种子序列特异性上游锚定引物序列为5′-GRRGGGTTCCGRTRCRGCRTTR-3′(由于miRNA在结合靶基因时,允许G:U配对,所以在设计合成特异性锚定引物时,将与U对应的碱基用R代替,表示A或G碱基)。取全部PCR反应液进行琼脂糖凝胶电泳分析3′ RACE PCR产物,用胶回收试剂盒回收介于150~1 000 bp的全部PCR产物,连接到pMD19-T载体并转化JM109感受态细胞铺板培养,挑选单克隆菌落扩大培养,用miR-M4-5p种子序列特异性锚定引物和inner引物(TaKaRa)进行菌液PCR鉴定,将阳性克隆菌的菌液送生工生物工程(上海)股份有限公司进行测序。分析测序结果,通过BLAST进行序列相似性比对分析,确定候选靶基因的名称和基因全序列。
1.5 双荧光素酶报告载体的构建
以CEF基因组cDNA为模板,优先扩增miRNA结合靶点在3′-UTR区域内的29个候选靶基因片段。PCR反应体系包括:ExTaq10 μL,候选靶基因3′-UTR上下游引物(表1,#1~29)各1 μL (10 μmol·L-1),CEF cDNA 1 μL(100 ng·μL-1)和ddH2O 7 μL。PCR程序:95 ℃ 5 min;98 ℃ 10 s,55 ℃ 30 s,72 ℃ 1 min,30个循环;72 ℃ 10 min。PCR产物用1.5%琼脂糖凝胶电泳分析。PCR产物经胶回收后,连接pMD19-T载体,转化JM109感受态细胞挑单菌落PCR鉴定阳性后送测序,将目的片段从测序正确的T载体上切割下来并回收,与同样酶切处理的psiCHECK-2回收载体产物4 ℃过夜连接,然后转化JM109感受态细胞,涂氨苄抗性平板培养过夜,挑单菌落送生工生物工程(上海)股份有限公司测序。
将合成的3′-UTR突变寡核苷酸(表1,#30~36)分别用ddH2O溶解至终浓度为50 μmol·μL-1,进行退火反应。反应体系包括: 无核酸酶水40 μL,寡核苷酸退火缓冲液(5×) 20 μL,正、反寡核苷酸链各20 μL (50 μmol·L-1)。反应程序: 95 ℃变性2 min,然后每90 s下降1 ℃直至25 ℃。同上所述,构建双荧光报告载体。
1.6 双荧光素酶报告试验
psiCHECK2-3′-UTR和pcDNA6.2-miR-M4-5p质粒各取300 ng,均匀混于12.5 μL无血清培养基中,按照48孔细胞板每孔1 μg Lipofectamine 2000和12.5 μL质粒的比例均匀混合,按Lipofectamine 2000说明书,将孵育后的Lipofectamine 2000和混合质粒(25 μL·孔-1)共转染汇合度为80%~90%的HEK 293T细胞。以pcDNA6.2-miR-neg为阴性对照,每组均设3个重复,在37 ℃、5% CO2培养箱中静置培养。
转染36 h后,将HEK 293T细胞培养基弃去,用PBS洗2次,然后用DLRA系统进行报告基因检测。首先用20 μL ddH2O稀释细胞裂解液裂解细胞,室温孵育15 min。然后将细胞裂解液转移至96孔白底透明酶标板中,每孔加入100 μL LAR II,轻柔混合均匀后用Ω酶标仪(OMG Labtech)测定萤火虫荧光素酶数值。最后向每孔加入100 μL Stop and Glo Reagent,轻柔混合均匀后,同上测定海肾荧光素酶数值。最后统计分析海肾荧光素酶/萤火虫荧光素酶活性比值。
2 结 果
2.1 miR-M4-5p宿主候选靶基因cDNA文库的构建
将hybrid-PCR产物电泳分析后割胶回收(图1),连接pMD19-T载体,转化E.coliJM109后铺板培养,挑取600个单菌落接菌活化后进行菌液PCR鉴定,共有580个菌落可以扩增出目的条带(图2),将这580个样品送样测序,共获得403个有效的基因序列。BLAST分析获得88个单一基因序列(表2)。其中,结合位点在3′-UTR的基因有63个,结合位点在CDS的基因有20个,结合位点在5′-UTR的基因有5个。结合位点在3′-UTR的63个基因中有29个基因的结合位点与miR-M4-5p的种子区完全互补(包括G:U)。我们优先选择这29个基因进行下一步的DLRA试验分析。
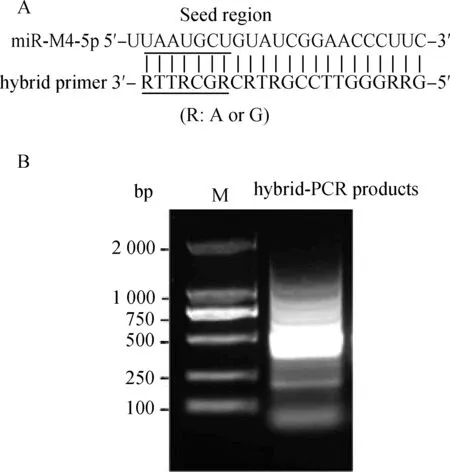
M. DNA marker图1 Hybrid-PCR特异性引物示意图(A)和hybrid-PCR产物电泳分析(B)Fig.1 Schematic of miR-M4-5p hybrid primer (A) and agarose gel electrophoresis of PCR products (B)
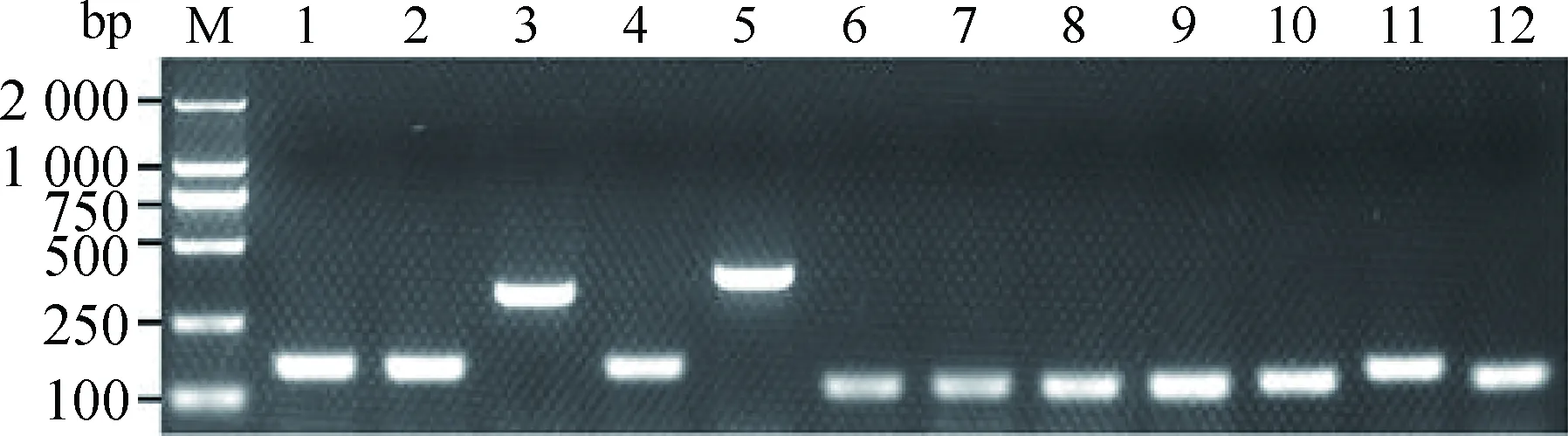
M. DNA相对分子质量标准; 1~12. 菌液克隆号M. DNA marker; 1-12. Clone number of bacterial fluid图2 Hybrid-PCR产物的部分菌液PCR产物电泳分析Fig.2 Identification of part hybrid-PCR products by agarose gel electrophoresis
表2 miR-M4-5p靶标文库候选基因列表
Table 2 Putative target mRNAs of miR-M4-5p
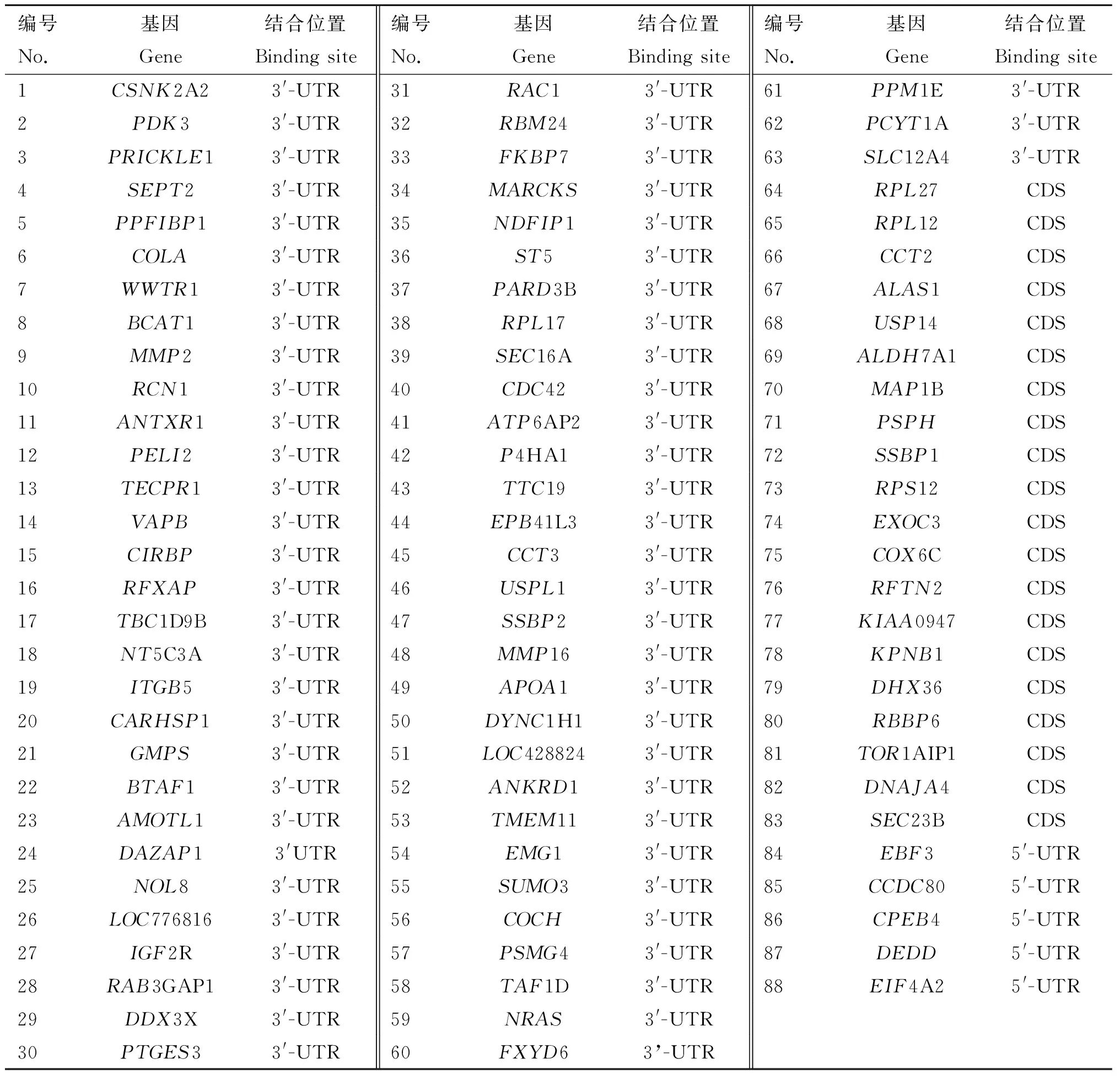
编号No.基因Gene结合位置Bindingsite编号No.基因Gene结合位置Bindingsite编号No.基因Gene结合位置Bindingsite1CSNK2A23'-UTR31RAC13'-UTR61PPM1E3'-UTR2PDK33'-UTR32RBM243'-UTR62PCYT1A3'-UTR3PRICKLE13'-UTR33FKBP73'-UTR63SLC12A43'-UTR4SEPT23'-UTR34MARCKS3'-UTR64RPL27CDS5PPFIBP13'-UTR35NDFIP13'-UTR65RPL12CDS6COLA3'-UTR36ST53'-UTR66CCT2CDS7WWTR13'-UTR37PARD3B3'-UTR67ALAS1CDS8BCAT13'-UTR38RPL173'-UTR68USP14CDS9MMP23'-UTR39SEC16A3'-UTR69ALDH7A1CDS10RCN13'-UTR40CDC423'-UTR70MAP1BCDS11ANTXR13'-UTR41ATP6AP23'-UTR71PSPHCDS12PELI23'-UTR42P4HA13'-UTR72SSBP1CDS13TECPR13'-UTR43TTC193'-UTR73RPS12CDS14VAPB3'-UTR44EPB41L33'-UTR74EXOC3CDS15CIRBP3'-UTR45CCT33'-UTR75COX6CCDS16RFXAP3'-UTR46USPL13'-UTR76RFTN2CDS17TBC1D9B3'-UTR47SSBP23'-UTR77KIAA0947CDS18NT5C3A3'-UTR48MMP163'-UTR78KPNB1CDS19ITGB53'-UTR49APOA13'-UTR79DHX36CDS20CARHSP13'-UTR50DYNC1H13'-UTR80RBBP6CDS21GMPS3'-UTR51LOC4288243'-UTR81TOR1AIP1CDS22BTAF13'-UTR52ANKRD13'-UTR82DNAJA4CDS23AMOTL13'-UTR53TMEM113'-UTR83SEC23BCDS24DAZAP13'UTR54EMG13'-UTR84EBF35'-UTR25NOL83'-UTR55SUMO33'-UTR85CCDC805'-UTR26LOC7768163'-UTR56COCH3'-UTR86CPEB45'-UTR27IGF2R3'-UTR57PSMG43'-UTR87DEDD5'-UTR28RAB3GAP13'-UTR58TAF1D3'-UTR88EIF4A25'-UTR29DDX3X3'-UTR59NRAS3'-UTR30PTGES33'-UTR60FXYD63’-UTR
UTR.非编码区;CDS.编码区
UTR. Untranslated regions; CDS. Coding sequence
2.2 双荧光素酶报告载体的构建及鉴定
利用表1所示的引物对#1-29,通过PCR从CEF基因组cDNA中扩增29个候选靶基因的部分3′-UTR片段(图3A),经测序分析确定目的序列与预测完全相符,分别提质粒进行双酶切后电泳割胶回收上述PCR产物,将纯化的3′-UTR PCR产物分别连接到psiCHECK2载体上,通过双酶切验证和测序鉴定,成功构建了29个psiCHECK2-3′-UTR报告载体(图3B)。
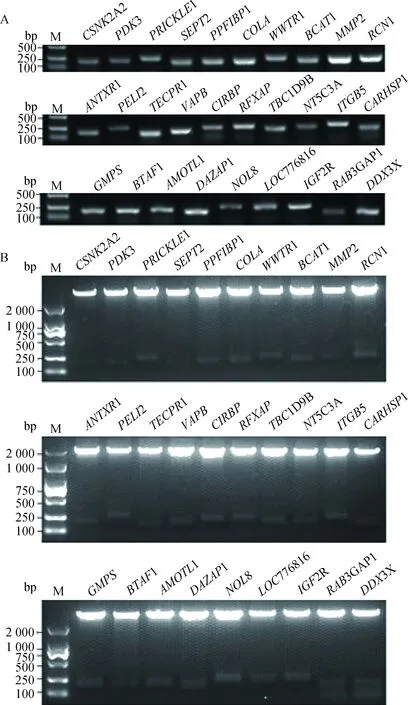
M. DNA marker 图3 候选靶基因3′-UTR的PCR扩增(A)和psiCHECK2-3′-UTR重组质粒酶切鉴定(B)Fig.3 The PCR amplification of 3′-UTRs of target genes (A) and identification of the recombinant psiCHECK2-3′-UTR plasmid by double enzyme digestion (B)
2.3 双荧光素酶报告试验
第一轮DLRA筛选,将psiCHECK2-3′-UTR质粒分别和pcDNA6.2-miR-M4-5p质粒或阴性对照质粒pcDNA6.2-miR-neg共转染HEK 293T细胞,36 h后用DLRA系统检测荧光值并计算相对荧光素酶活性(即海肾荧光素酶活性值/萤火虫荧光素酶活性值)。结果显示,在29个候选靶基因中,有19个基因的psiCHECK2-3′-UTR质粒与pcDNA6.2-miR-M4-5p质粒共转染HEK 293T细胞的海肾荧光素酶活性值/萤火虫荧光素酶活性值显著低于阴性对照组的比值(图4)。
第二轮DLRA筛选,将第一轮筛选到的19个候选基因再次重复试验,但同时加上psiCHECK2-3′-UTR质粒和pcDNA6.2-mut-miR-M4-5p质粒共转染组,DLRA结果显示有7个候选基因的psiCHECK2-3′-UTR质粒和pcDNA6.2-miR-M4-5p质粒共转染组的荧光比值与pcDNA6.2-miR-neg质粒或pcDNA6.2-mut-miR-M4-5p质粒对照组差异均显著(图5)。
第三轮DLRA筛选,首先构建第二轮中筛选获得的7个候选基因的3′-UTR突变体psiCKECK2重组质粒,然后同时用这些psiCHECK2-3′-UTR突变体质粒代替psiCHECK2-3′-UTR质粒进行DLRA试验。结果发现PRICKLE1、COLA、BCAT1、ANTXR1和TECPR1的3′-UTR突变后失去抑制效果,而TBC1D9B的3′-UTR突变后仍有抑制效果,NT5C3A的psiCHECK2-3′-UTR质粒和pcDNA6.2-miR-M4-5p质粒共转染组的荧光比值与阴性对照组差异不显著(图6)。三轮DLRA结果表明,PRICKLE1、COLA、BCAT1、ANTXR1和TECPR1可以确定为miR-M4-5p的宿主靶基因。
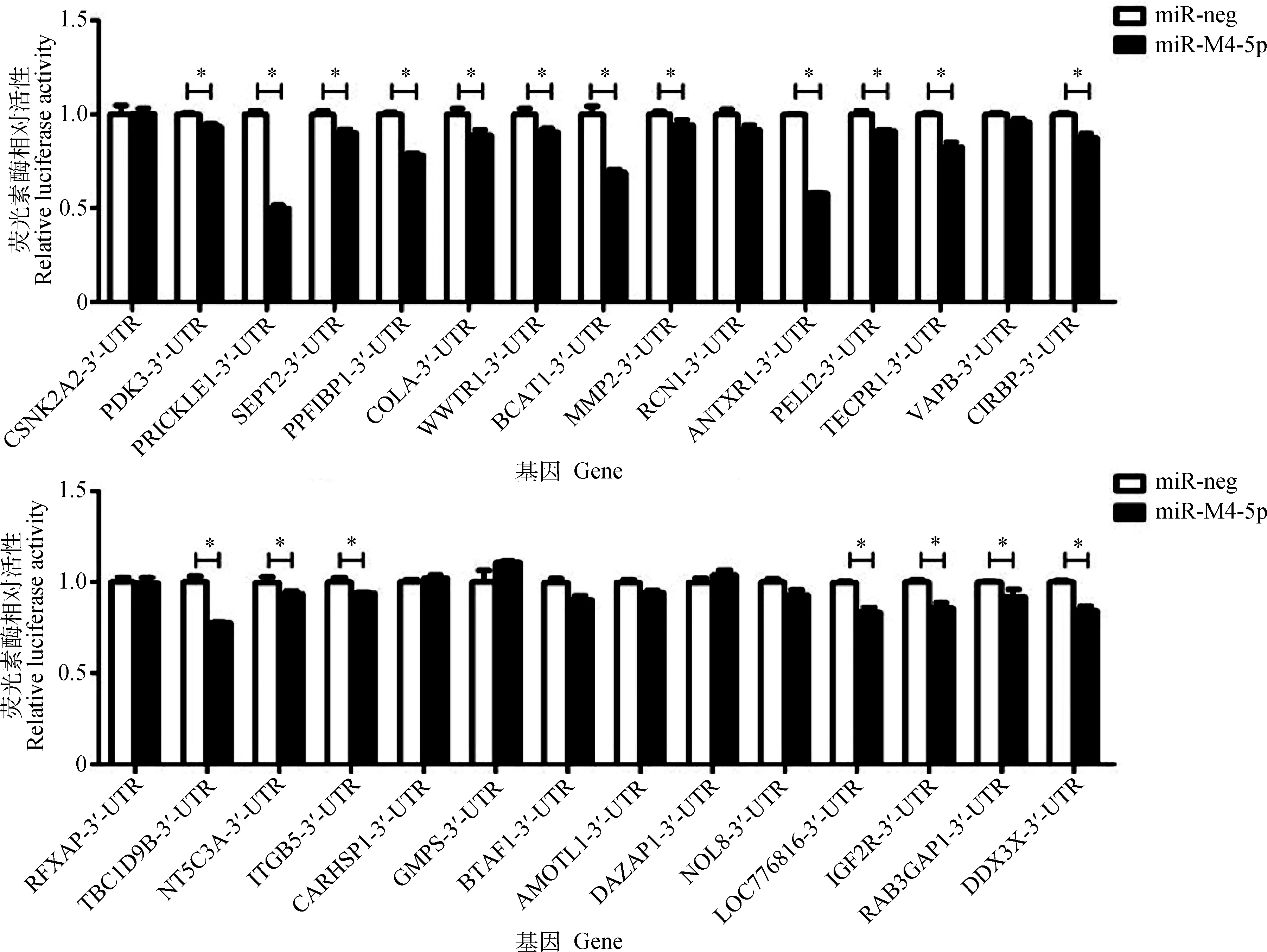
误差线表示三次独立重复试验的标准差。*.差异显著(P<0.05)Error bars indicate the standard deviation (SD) for triplicate experiments. *. Significant difference (P<0.05)图4 DLRA分析miR-M4-5p与候选靶基因3′-UTR的体外相互作用Fig.4 Analysis of the in vitro interactions between miR-M4-5p and the 3′-UTR of putative target genes utilizing dual luciferase reporter assay (DLRA)

误差线表示三次独立重复试验的标准差。*.差异显著(P<0.05)Error bars indicate the standard deviation (SD) for triplicate experiments. *. Significant difference (P<0.05)图5 DLRA分析miR-M4-5p与候选靶基因3′-UTR突变体的体外相互作用Fig.5 Analysis of the in vitro interactions between miR-M4-5p and the mut-3′-UTR of putative target genes utilizing dual luciferase reporter assay (DLRA)
3 讨 论
miRNA通过与不同靶基因相互作用,可以调控多种生物学过程。近年来,发现MDV编码的病毒miRNA在MD肿瘤的发生发展过程中可能发挥重要作用。之前的动物攻毒试验数据也显示,缺失miR-M4前体基因的MDV突变毒株与其亲本毒株相比致瘤率显著下降[14]。通过生物信息学软件预测和试验验证,已经证实了miR-M4-5p可在感染宿主体内靶向调控宿主基因LTBP1的表达,影响TGF-β的成熟及向胞外分泌的过程,进而抑制TGF-β信号通路来激活原癌蛋白c-MYC的过表达,最终可能促进诱导MD肿瘤的发生[15]。目前有多种软件可用于miRNA的靶基因预测与分析,但在具体分析时主要遵循以下几个原则,如miRNA与靶基因的互补性、靶基因在不同物种间的保守性、miRNA-mRNA之间形成双链的稳定性等。除了生物信息学方法之外,也开发出了很多其他有效的研究策略和方法[17]。用hybrid-PCR通过锚定引物寻找特定miRNA的候选靶基因的方法最早是在2011年提出来的[16],通过模拟miRNA在体内与mRNA的靶向结合调控过程,利用PCR扩增出特定的基因片段,结合生物信息学分析初步鉴定这些锚定扩增出来的候选基因,从而构建一个相对软件预测来说更为可信的候选靶标文库,大大降低了后期试验验证的工作量。如果再综合生物学软件预测结果可以更好地优化miRNA靶基因筛选与鉴定的实验流程,极大地提高研究效率。
针对MDV-1编码的miR-M4-5p,我们之前通过软件预测并参考相关文献已经找到了一个重要的靶基因LTBP1。本研究中我们又采用hybrid-PCR构建候选靶标cDNA文库,初步筛选鉴定的克隆中有63个候选靶基因的预测结合位点位于3′-UTR,其中只有25个候选靶基因的预测结合位点在CDS或5′-UTR。在这些候选靶基因中,尚未发现此前已报道鉴定的miR-M4-5p宿主靶基因,这可能与目前筛选鉴定的克隆数量有关,也可能与候选靶基因的表达丰度有关,因为hybrid-PCR过程可能会导致高丰度表达的基因掩盖低丰度基因。多数情况下,miRNA结合到靶基因mRNA的3′-UTR区域以发挥调控作用,因此我们首先关注靶标文库中结合位点在3′-UTR的63个候选靶基因。进一步分
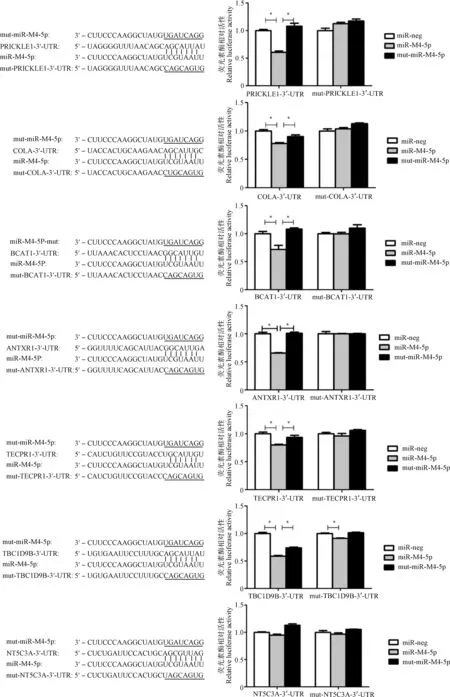
下划线序列为突变的种子序列或靶点序列;误差线表示三次独立重复试验的标准差。*.差异显著(P<0.05)The underlined nucleotide sequences indicate the mutated seed sequences or binding sites; Error bars indicate the standard deviation (SD) for triplicate experiments. *. Significant difference (P<0.05)图6 DLRA分析miR-M4-5p、mut-miR-M4-5p与候选靶基因3′-UTR及mut-3′-UTR的体外相互作用Fig.6 Analysis of the in vitro interactions between miR-M4-5p or mut-miR-M4-5p and the 3′-UTR or mut-3′-UTR of putative target genes utilizing dual luciferase reporter assay (DLRA)
析这63个候选靶基因的结合位点,发现有29个候选靶基因的预测靶点序列与miR-M4-5p种子区完全互补匹配,其他则不完全匹配。miRNA的种子区对其调节功能影响很大,即使一对碱基不互补都可能会显著影响其调节功能,所以我们优先针对与miR-M4-5p种子区完全互补的29候选靶基因进行下一步的DLRA鉴定工作。经过3轮筛选,逐步缩小筛选范围。第一轮筛选初步验证了19个候选靶基因可以与miR-M4-5p相互作用;第二轮筛选进一步验证了7个候选靶基因与miR-M4-5p的相互作用具有种子序列依赖性;而第三轮筛选通过种子序列及靶点双突变验证分析,最终将PRICKLE1、COLA、BCAT1、ANTXR1和TECPR1这5个基因鉴定为miR-M4-5p的宿主靶基因。
在这5种初步鉴定的宿主靶基因中,COLA编码的蛋白质目前尚无相关功能报道。ANTXR1是跨膜蛋白,其生理状态下的功能尚不十分清楚,可能参与细胞外基质稳态的维持[18-19]。PRICKLE1编码一种核受体,位于细胞膜表面,其对Wnt/β-catenin信号通路存在负调节作用[20]。miR-M4-5p对PRICKLE1的调控,可能进一步影响Wnt/β-catenin,从而改变多种基因表达,影响细胞的增殖和死亡。BCAT1编码支链氨基酸转移酶1,为支链氨基酸分解代谢过程中的关键酶,参与支链氨基酸的代谢过程,为组织细胞的生长代谢提供能量[21],miR-M4-5p对BCAT1的负向调控可能削弱宿主的体质和免疫能力,从而有利于MDV对宿主的感染。TECPR1编码的蛋白是一种有关选择性自噬的膜融合相关蛋白,在自噬小体的成熟及促进自噬小体与溶酶体融合方面起着重要的作用[22]。其可能参与MDV感染中对宿主的免疫抑制作用,从而有助于MDV在宿主体内的感染和增殖,逃避宿主的免疫保护。DLRA试验只是在体外验证了miR-M4-5p与宿主候选靶基因的相互作用,接下来仍需要利用实时荧光定量PCR、蛋白质印记等实验方法检测miR-M4-5p在体内是否对上述候选靶基因具有靶向调节作用,为进一步深入研究miR-M4-5p介导MDV感染及致瘤的分子机制奠定重要的基础。
4 结 论
通过hybrid-PCR建库及序列相似性比对分析,构建了miR-M4-5p的宿主候选靶基因cDNA文库;成功构建了29个候选靶基因的3′-UTR荧光素酶验证载体和突变载体;经过3轮DLRA分析表明PRICKLE1、COLA、BCAT1、ANTXR1和TECPR1可与miR-M4-5p在体外相互作用,初步鉴定为miR-M4-5p的宿主靶基因。
[1] SAIF Y M. 禽病学[M]. 苏敬良, 高 福, 索 勋, 译. 12版. 北京: 中国农业出版社, 2012: 129-151. SAIF Y M. Disease of poultry[M]. SU J L, GAO F, SUO X, translated. 12th ed. Beijing: China Agriculture Press, 2012: 129-151. (in Chinese)
[2] CALNEK B W. Pathogenesis of Marek’s disease virus infection[J].CurrTopMicrobiol, 2001, 255: 25-55.
[3] DAVISON A J, EBERLE R, EHLERS B, et al. The order herpesvirales[J].ArchVirol, 2009, 154(1): 171-177.
[4] WITTER R L. Increased virulence of Marek′s disease virus field isolates[J].AvianDis, 1997, 41(1): 149-163.
[5] OSTERRIEDER N, KAMIL J P, SCHUMACHER D, et al. Marek′s disease virus: from miasma to model[J].NatRevMicrobiol, 2006, 4(4): 283-294.
[6] BARTEL D P. MicroRNAs: genomics, biogenesis, mechanism, and function[J].Cell, 2004, 166(2): 281-297.
[7] FILIPOWICZ W, BHATTACHARYYA S N, SONENBERG N. Mechanisms of post-transcriptional regulation by microRNAs: are the answers in sight?[J].NatRevGenet, 2008, 9(2): 102-114.
[8] ZHAO Y G, YAO Y X, XU H T, et al. A functional MicroRNA-155 ortholog encoded by the oncogenic Marek′s disease virus[J].JVirol, 2009, 83(1): 489-492.
[9] FARAONI I, ANTONETTI F R, CARDONE J, et al.miR-155 gene: a typical multifunctional microRNA[J].BiochimBiophysActa, 2009, 1792(6): 497-505.
[10] HIGGS G, SLACK F. The multiple roles of microRNA-155 in oncogenesis[J].JClinBioinforma, 2013, 3: 17.
[11] KONG W, HE L, RICHARDS E J, et al. Upregulation of miRNA-155 promotes tumour angiogenesis by targeting VHL and is associated with poor prognosis and triple-negative breast cancer[J].Oncogene, 2014, 33(6): 679-689.
[12] TAM W, DAHLBERG J E. miR-155/BICas an oncogenic microRNA[J].GenesChromosomesCancer, 2006, 45(2): 211-212.
[13] TILI E, CROCE C M, MICHAILLE J J.miR-155: on the crosstalk between inflammation and cancer[J].IntRevImmunol, 2009, 28(5): 264-284.
[14] YU Z H, TENG M, SUN A J, et al. Virus-encoded miR-155 ortholog is an important potential regulator but not essential for the development of lymphomas induced by very virulent Marek’s disease virus[J].Virology, 2014, 448: 55-64.
[15] CHI J Q, TENG M, YU Z H, et al. Marek’s disease virus-encoded analog of microRNA-155 activates the oncogene c-Myc by targeting LTBP1 and suppressing the TGF-β signaling pathway[J].Virology, 2015, 476: 72-84.
[16] HUANG Y J, QI Y, RUAN Q, et al. A rapid method to screen putative mRNA targets of any known microRNA[J].VirolJ, 2011, 8: 8.
[17] DANGWAL S, BANG C, THUM T. Novel techniques and targets in cardiovascular microRNA research[J].CardiovascRes, 2012, 93(4): 545-554.
[18] YOUNG J A T, COLLIER R J. Anthrax toxin: receptor binding, internalization, pore formation, and translocation[J].AnnuRevBiochem, 2007, 76: 243-265.
[19] CULLEN M, SEAMAN S, CHAUDHARY A, et al. Host-derived tumor endothelial marker 8 promotes the growth of melanoma[J].CancerRes, 2009, 69(15): 6021-6026.
[20] CHAN D W, CHAN C Y, YAM J W P, et al. Prickle-1 negatively regulates Wnt/β-catenin pathway by promoting dishevelled ubiquitination/degradation in liver cancer[J].Gastroenterology, 2006, 131(4): 1218-1227.
[21] PEREBOOM T C, BONDT A, PALLAKI P, et al. Translation of branched-chain aminotransferase-1 transcripts is impaired in cells haploinsufficient for ribosomal protein genes[J].ExpHematol, 2014, 42(5): 394-403.e4.
[22] OGAWA M, YOSHIKAWA Y, KOBAYASHI T, et al. A Tecpr1-dependent selective autophagy pathway targets bacterial pathogens[J].CellHostMicrobe, 2011, 9(5): 376-389.
