CRISPR-Cas系统及其在结核分枝杆菌中的研究进展
2018-03-08翟小倩
翟小倩, 鲍 朗
(四川大学 华西医学中心感染免疫研究室, 成都 610041)
结核病是一种严重危害人类健康的慢性传染病,结核分枝杆菌(mycobacteriumtuberculosis)是其主要的致病源。 2016年WHO全球结核报告指出:2015年,世界范围内估计有1040万新发病例,新发病例中有120万(11%)HIV患者。有140万人死于结核病,其中40万是结核/HIV双重感染人群[1]。随着结核-HIV病毒共同感染病人数量增加以及结核多重耐药菌株流行,结核病的防治面临严峻挑战。因此,掌握结核病的发病机制是防治结核病的关键。
20世纪80年代,研究者在细菌基因组中首次发现了CRISPR-Cas获得性免疫系统。该系统在细菌抵御外源DNA入侵从而产生自我保护机制中发挥重要作用。迄今为止,CRISPR-Cas系统按作用机制中CAS蛋白的差异性分为I型、II型、III型三大类以及11种亚型[2]。结核分枝杆菌的CRISPR-Cas系统属于III-A型[3]。相关文献表明,结核III-A型CRISPR-Cas系统不仅是一种获得性免疫系统,还在结核毒力基因表达调控方面以及结核基因编辑方面均表现出重要作用,其功能呈现出多样性与复杂性。
1 结核III-A型CRISPR-Cas系统的简介以及作用机制
结核III-A型CRISPR-Cas系统由两大部分组成:CRISPR基因位点和CAS蛋白。CRISPR基因位点由前导序列、同向重复序列、间隔序列3部分组成。CAS蛋白由一系列保守的cas基因所编码,是整个系统的催化核心[2]。CRISPR-Cas系统通过降解入侵的外源DNA来抵御其对宿主细胞的干扰,同时对其产生特异性免疫记忆,预防同类外源DNA的再次入侵[4]。不同类型CRISPR-Cas系统的具体作用机制不同, 本文主要介绍结核III-A型CRISPR-Cas系统的具体作用机制,包括3大步骤:适应、表达、干扰[5](如图1)。
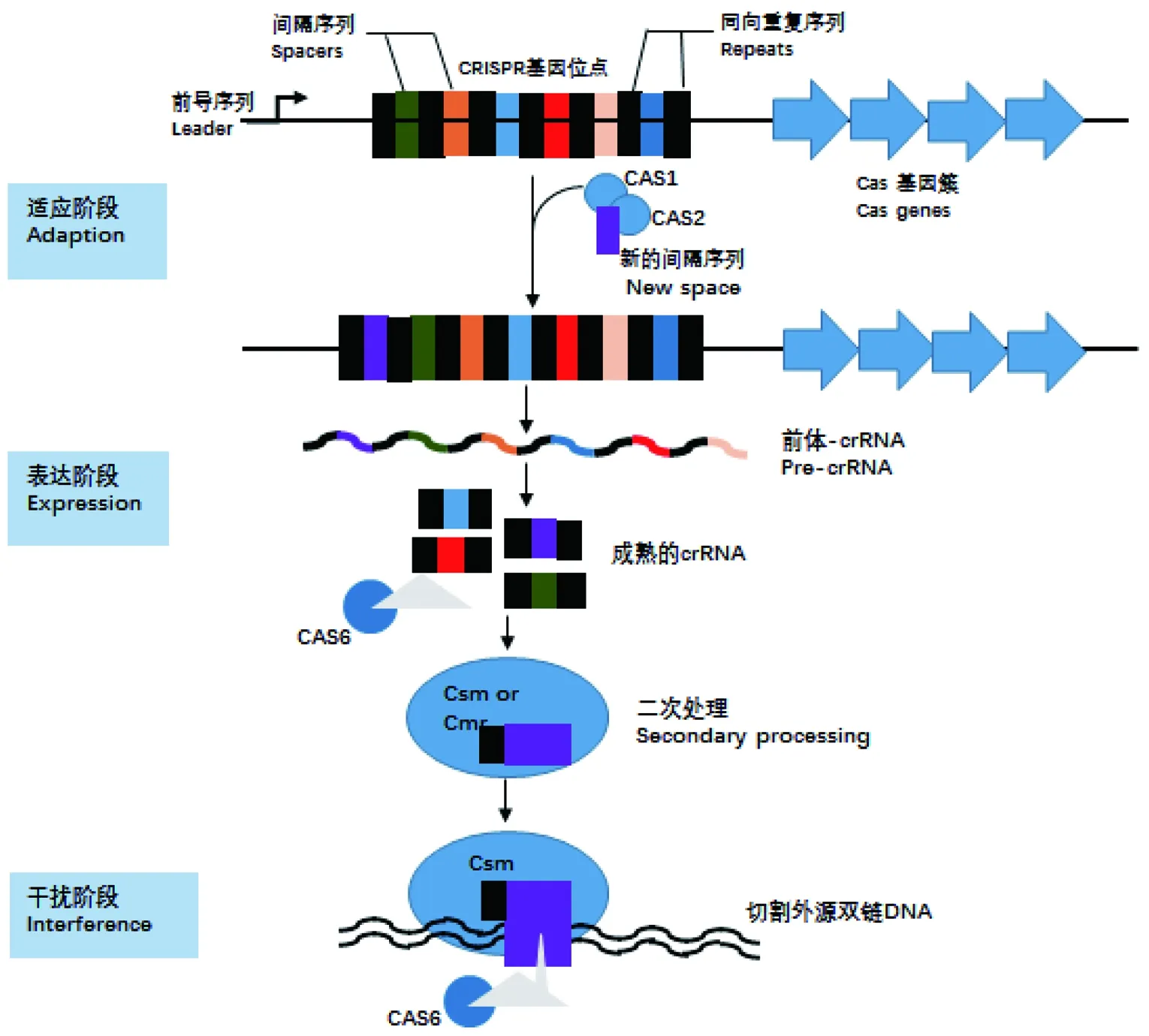
图1 结核Ⅲ-A型CRISPR-Cas系统的作用机制[5]Fig 1 The mechanism of Ⅲ-A type CRISPR-Cas system in mycobacterium tuberculosis[5]
第一步,适应阶段。由CAS1和CAS2组成的蛋白复合物裂解外源入侵的噬菌体和质粒中的原型间隔序列(proto-spacer),裂解后产生的新的间隔序列(new spacer)被整合到宿主基因组CRISPR位点的前导序列及第一个重复序列的近端。
第二步,表达阶段。新的间隔序列被整合到CRISPR位点后,从前导序列开始转录产生前体crRNA (Pre-crRNA),进一步通过CAS内核糖核苷酸酶(CAS6为核心)剪切重复序列,最终形成由单个间隔序列和切割剩余的部分重复序列组成的成熟crRNA8[7]。同时,产生的新的间隔序列,如果能够与CRISPR位点序列内部的短重复序列部分互补,将被整合到细菌基因组的CRISPR序列中,产生对外源DNA入侵的特异性免疫记忆。
第三步,干扰阶段。III-A型CRISPR-Cas系统的主要作用单元是CSM6蛋白。成熟的crRNA与CAS蛋白结合形成的复合体(Csm复合体),在成熟的crRNA的引导下,识别并结合在外源入侵的双链DNA中紧随原型间隔序列后的原型间隔序列毗邻基序(proto-spacers adjacent motif,PAM),进而CSM6(Rv2816编码)通过切割外源DNA,达到抵御外源不利因素入侵的效果[8]。总之,III-A型CRISPR-Cas系统的存在可以增强结核分枝杆菌的生存能力。
2 改造的 CRISPR-Cas9系统介导的基因编辑技术在结核分枝杆菌中的应用
CRISPR-Cas9系统是目前研究热门的新一代基因组编辑技术,属于CRISPR-Cas系统的第II型。CRISPR-Cas9系统只需要一种CAS9蛋白和两种RNA,即可实现对双链DNA的精确切割。其作用机制简单,容易进行体外设计和改造。即使宿主细胞内无该系统,也可利用改造的CRISPR-Cas9系统实现基因组编辑[9]。经过改造的CRISPR-Cas9系统又称为CRISPR干扰系统 (CRISPR interference),该系统应用了无核酸酶活性的CAS9蛋白(“dead” CAS9,dCAS9),dCAS9虽无核酸酶活性但可在RNA的指导下与特定靶向 DNA 序列结合[10]。将CRISPR干扰系统导入分枝杆菌后,dCAS9可以在分枝杆菌中稳定存在2周且不产生毒性,最终实现基因编辑[11]。研究表明,在结核分枝杆菌中应用CRISPR干扰系统能够有效抑制多种功能基因的表达,能够对多个基因进行同时敲除,也能够快速检测出必需基因[11]。该系统也可以沉默操纵子,结核groEL1位于groES下游, CRISPR干扰系统可直接通过沉默groESL1操纵子使groEL1的表达水平随groES表达水平下降而下降[12]。因此,CRISPR-Cas9技术的应用对推动结核分枝杆菌的研究有里程碑式的意义。
3 结核III-A型CRISPR-Cas系统在基因表达调控研究中的进展
结核III-A型CRISPR-Cas系统可以调控毒力基因的表达。Yosef等[5]认为CRISPR位点的转录和CAS蛋白都可以通过独立的基因调节控制细菌的毒力。Hatoum-Aslan和Maniv[13]已经证明CRISPR位点能够调节基因的表达。Sampson等[14]的研究发现弗朗西斯氏菌属的CAS9可以调节FTN_1103编码的脂蛋白的表达。这些研究使得人们开始关注CRISPR-Cas系统在结核分枝杆菌中调节基因表达的作用。最近有研究表明,结核中的非编码RNA可以影响结核基因调控网络。前文提到的crRNA就属于非编码RNA,而CRISPR基因位点是产生crRNA的主要来源,说明III-A型CRISPR-Cas系统在结核致病机制以及适应性机制中有重要作用[15]。更加深入研究CRISPR-Cas系统在基因表达中的调控作用,有助于探明结核分枝杆菌的作用机制从而实现结核病的防治。
4 结核III-A型CRSPR-Cas系统中cas基因的研究进展
全基因组测序发现,结核分枝杆菌有4 411 529个碱基,大约4000个基因[15]。深入研究单个基因以及蛋白的生物学功能或者基因之间的相互作用机制成为探明结核发病机制的热点。CAS蛋白家族作为CRSPR-Cas系统的催化核心,了解其各个基因及蛋白研究进展有助于更加全面地把握整个系统的作用,为进一步探明结核作用机制提供基础。CAS蛋白家族由9种cas基因组成:cas2、cas1、csm6、csm5、csm4、csm3、csm2、cas10和cas6,具体如图2[16]。这些CAS蛋白携带多种功能域,比如,核糖核酸酶、解螺旋酶、多聚酶等功能域以发挥作用[17]。

图2 分枝杆菌CRISPR-Cas系统的组成[16]Fig 2 The composition of CRISPR-Cas system of Mycobacterium[16]
4.1 Rv2816c-cas2
cas2对应结核基因组中的Rv2816c。CAS2蛋白是一种内核糖核苷酸酶,其二级结构以及潜在的残基对其自身的催化活性影响极大,为CRISPR-Cas系统功能正常发挥提供保障[18-19]。有研究者把cas2重组入耻垢分枝杆菌后,耻垢分枝杆菌的菌落表型以及生物膜形成发生明显改变。因而推测cas2的功能涉及调节细胞壁合成的相关基因表达,造成耻垢分枝杆菌细胞壁组成结构的改变,进而提高分枝杆菌自身毒力[20]。
4.2 Rv2817c-cas1
在CRISPR-Cas系统中,cas1是一种必需基因,也是序列最保守的cas基因,被认为是CRISPR-Cas系统的标志。cas1对应结核基因组中的Rv2817c,其表达的CAS1蛋白是一种金属依赖的核酸酶。CAS1蛋白通过和CAS2蛋白形成异聚体复合物从而获取外源基因的原型间隔序列,实现抵御外源DNA入侵的作用[21]。也有研究报道CAS1蛋白的表达和活性极有可能被程序性细胞死亡和休眠机制所密切调控和关联,具体机制不清楚[22-24]。
4.3 Rv2819c-Rv2820c
目前,Rv2819c和Rv2820c两种基因被认为与结核分枝杆菌的毒力与基因组进化有关。在III-A型CRISPR-Cas系统中的Rv2819c(csm5)和Rv2820c(csm4),其表达的两种CAS蛋白属于同一类蛋白家族,在处理前体-crRNA的转录过程,承担序列和结构特异的RNA酶的功能[25]。Rindi等[26]通过比较基因组学研究发现,csm5在结核减毒株 H37Ra中的表达情况较在结核标准毒力株H37Rv中明显下调,提示csm5可能与结核分枝杆菌的毒力有关。但分子流行病学研究发现,多重耐药流行的临床菌株——结核北京家族菌株RD207区缺失了7个cas,其中就包括csm5这个基因,其可能与结核家族种属中的基因组进化有关[27-29]。发生这种缺失现象的另一个基因Rv2820c在结核标准毒力株H37Rv中基因片段是完整的,但在结核北京家族株中其同源的Rv2820c却因为移码突变,导致氨基酸的编码改变产生基因截短,其长度只有原来的40%。Lam等[30]对截短的Rv2820c进行研究,发现无论是在体外还是体内实验中该基因均可以促进分枝杆菌的毒力。这种同源基因表现出不同表型差异的现象以及发生毒力改变是未来结核的研究热点。
5 总结与展望
综上所述,III-A型CRISPR-Cas系统作为获得性免疫系统,通过抵御外援干扰入侵使得结核分枝杆菌的生存能力增强。同时,其组成成分CRISPR基因位点以及cas基因在一定程度上有助于结核分枝杆菌的基因网络调控以及调节菌体毒力。该系统作用的复杂性与多样性使其研究更具意义。虽然结核北京家族株中存在不完整的CRISPR-Cas系统,使人推测CRISPR-Cas防御系统会随着进化而消亡[31],但是就目前的研究结果来看,这种基因组的进化是增强了CRISPR-Cas系统的作用,该系统在结核分枝杆菌中的作用是不容忽视的。未来,对CRISPR-Cas系统的某种基因的功能或者基因之间相互作用进行深入研究,可能会寻找到治疗靶点或者寻找到免疫原性极高的诊断指标。加之,改良的II型CRISPR-Cas9系统导入结核分枝杆菌中可以实现高效的基因编辑(敲除或沉默基因),利用该技术深入研究结核III-A型CRISPR-Cas系统将为解决结核病带来的困扰提供更多理论支持。
[1]WHO. Global tuberculosis report 2016 [EB/OL] . http://www. who. int/tb/ publications/global_report/en/.2016.
[2]MAKAROVA K S,HAFT D H,BARRANGOU R,et al. Evolution and classification of the CRISPR-Cas systems [J].Nat Rev Microbiol, 2011, 9(6):467-477.
[3]MAKAROVA K S, ARAVIND L, WOLF Y I, et al.Unification of Cas protein families and a simple scenario for the origin and evolution of CRISPR-Cas systems[J]. Biol Direct, 2011, 6:38.
[4]郑晓梅,张晓立,于建东, 等.CRISPR-Cas9介导的基因组编辑技术的研究进展 [J].生物技术进展,2015,5(1):1-9.
[5]YOSEF I, GOREN M G, QIMRON U. Proteins and DNA elements essential for the CRISPR adaptation process inEscherichiacoli[J]. Nucleic Acids Res, 2012, 40(12):5569-5576.
[6]CARTE J,WANG R,LI H,et al.Cas6 is an endoribonuclease that generates guide RNAs for invader defense in prokaryotes [J].Genes Dev, 2008, 22(24): 3489-3496.
[7]HATOUM-ASLAN A,MANIV I,SAMAI P,et al.Genetic characterization of antiplasmid immunity through a type III-A CRISPR-Cas system [J].J Bacteriol, 2014, 196(2): 310-317.
[8]GASIUNAS G,BARRANGOU R,HORVATH P,et al.Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria [J].Proc Natl Acad Sci USA, 2012, 109(39):E2579-E2586.
[9]QI L S, LARSON M H, GILBERT L A, et al.Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression [J].Cell, 2013, 152(5):1173-1183.
[10]CHOUDHARY E, THAKUR P, PAREEK M,et al. Gene silencing by CRISPR interference in mycobacteria[J]. Nat Commun, 2015, 6:6267.
[11]WESTRA E R, BUCKLING A, FINERAN P C. CRISPR-Cas systems:beyond adaptive immunity[J]. Nat Rev Microbiol, 2014, 12(5):317-326.
[12]ARAVINDHAN V, CHRISTY A J, ROY S, et al.Mycobacterium tuberculosis groE promoter controls the expression of the bicistronic groESL1 operon and shows differential regulation under stress conditions [J] . FEMS Microbiol Lett, 2009, 292(1):42-49.
[13]HATOUM-ASLAN A, MANIV I. Impact of CRISPR immunity on the emergence and virulence of bacterial pathogens[J]. Curr Opin Microbiol, 2013, 17:82-90.
[14]SAMPSON T R, SARO S D, LLEWELLYN A C, et al. A CRISPR/Cas system mediates bacterial innate immune evasion and virulence[J]. Nature, 2013, 497(7448): 254-257.
[15]ARNVIG K,YOUNG D.Non-coding RNA and its potential role in Mycobacterium tuberculosis pathogenesis [J] . RNA Biol, 2012, 9(4):427-436.
[16]COLE S T, BROSCH R, PARKHILL J, et al. Deciphering the biology ofMycobacteriumtuberculosisfrom the complete genome sequence[J]. Nature, 1998, 393(6685):537-544.
[17] HE L, FAN X, XIE J. Comparative genomic structures ofMycobacteriumCRISPR-Cas[J]. J Cell Biochem, 2012, 113(7):2464-2473.
[18]BELOGLAZOVA N, BROWN G, ZIMMERMAN M D, et al.A novel family of sequence-specific endoribonucleases associated with the clustered regularly interspaced short palindromic repeats[J]. J Biol Chem, 2008, 283(29):20361-20371.
[19]HAN D, KRAUSS G. Characterization of the endonuclease SSO2001 fromSulfolobussolfataricusP2[J]. FEBS Lett, 2009, 583(4):771-776.
[20]HUANG Q, LUO H, LIU M, et al. The effect ofMycobacteriumtuberculosisCRISPR-associated Cas2 (Rv2816c) on stress response genes expression,morphology and macrophage survival ofMycobacteriumsmegmatis[J]. Infect Genet Evol, 2016, 40:295-301.
[21]NUNEZ J K, KRANZUSCH P J, NOESKE J, et al. Cas1-Cas2 complex formation mediates spacer acquisition during CRISPR-Cas adaptive immunity [J]. Nat Struct Mol Biol, 2014, 21(6): 528-534.
[22]MAKAROVA K S, WOLF Y I, KOONIN E V. Comparative genomics of defense systems in archaea and bacteria [J]. Nucleic Acids Res, 2013, 41(8): 4360-4377.
[23]KOONIN E V, MAKAROVA K S. CRISPR-Cas: evolution of an RNA-based adaptive immunity system in prokaryotes [J]. RNA Biol, 2013, 10(5): 679-686.
[24]MAKAROVA K S ,ANANTHARAMAN V,ARAVIND L, et al. Live virus-free or die: coupling of antivirus immunity and programmed suicide or dormancy in prokaryotes [J]. Biol Direct, 2012, 7:40.
[25]HAURWITZ R E, JINEK M, WIEDENHEFT B, et al. Sequence and structure-specific RNA processing by a CRISPR endonuclease [J]. Science, 2010, 329(5997): 1355-1358.
[26]RINDI L , LARI N,GARZELLI C. Genes ofMycobacteriumtuberculosisH37Rv downregulated in the attenuated strain H37Ra are restricted toM.tuberculosiscomplex species[J]. New Microbiol, 2001, 24(3): 289-294.
[27]LI Q, WHALEN C C, ALBERT J M, et al. Differences in rate and variability of intracellular growth of a panel ofMycobacteriumtuberculosisclinical isolates within a human monocyte model[J]. Infect Immun, 2002, 70(11): 6489-6493.
[29]MANCA C,TSENOVA L, BERGTOLD A, et al. Virulence of aMycobacteriumtuberculosisclinical isolate in mice is determined by failure to induce Th1 type immunity and is associated with induction of IFN-alpha /beta [J]. Proc Natl Acad Sci U S A, 2001, 98(10): 5752-5757.
[30]LAM J T, YUEN K Y, HO P L, et al. Truncated Rv2820c enhances mycobacterial virulence ex vivo and in vivo [J]. Microb Pathog, 2011, 50(6): 331-335.
[31]HANEKOM M, GEY VAN PITTIUS N, MCEVOY C, et al.MycobacteriumtuberculosisBeijing genotype: a template for success [J]. Tuberculosis, 2011, 91(6): 510-523.
