大白菜抗根肿病的抗源筛选和分子标记鉴定
2018-03-08朱明钊张淑江章时蕃王晓武李国亮魏云晓岳丽昕孙日飞
朱明钊 张淑江 张 慧 章时蕃 李 菲 王晓武 武 剑 李国亮魏云晓 岳丽昕 孙日飞
(中国农业科学院蔬菜花卉研究所,北京 100081)
大白菜根肿病是由芸薹属根肿菌(Plasmodiophora brassicaeWoron.)侵染引起的专性寄生的世界性土传病害,主要危害十字花科作物,最早发现于地中海西岸和欧洲南部,现在许多国家都有发生(杨永林,1990)。根肿菌的休眠孢子可以在土壤中存活7 a以上(Karling,1968),因此,田间一旦受到污染,将长期不再适合十字花科作物的种植。通过田间管理和生物化学防治的方法不能从根本上防治根肿病,选育抗根肿病的大白菜新品种是最经济有效的方法(Kuginuki et al.,1999)。
迄今为止,在大白菜中至少有12个抗根肿病基因被定位出来,其中Crr2位于A01连锁群(Suwabe et al.,2003),CRc位 于 A02连 锁 群(Sakamoto et al.,2008),CRa、CRb、CRbkato、CRk、Crr3、PbBa3.1、PbBa3.3和Rcr1都位于A03连锁群(Hirai et al.,2004;Piao et al.,2004;Sakamoto et al.,2008;Matsumoto et al.,2012;Chen et al.,2013;Kato et al.,2013;Chu et al.,2014),Crr4位于 A06连锁群(Suwabe et al.,2006),Crr1位于A08连锁群(Suwabe et al.,2003);另外,Gao等(2014)将1个抗病基因定位到了与CRa相同的位置(Ueno et al.,2012),该基因可能与CRa是同一基因;王彤彤等(2012)将1个抗病基因定位到了A08连锁群并证明了该基因与Crr1不在同一区域,是一个新的抗病基因。
以往的基因定位工作只能定位1个或2个抗病基因,而对根肿菌分化的众多的生理小种,单个基因往往不起作用,因此,将多个抗病基因聚合到同一份材料当中是提高材料抗病的广泛性和耐久性的有效方法(Matsumoto et al.,2012;Hatakeyama et al.,2017)。本试验以根肿菌M01、M02和M03为病原菌菌源,对24份大白菜材料进行接种鉴定和分子标记鉴定,旨在筛选出抗多个根肿菌的抗源,并明确与之相关的抗病基因,以期为今后抗根肿病聚合育种工作奠定基础。
1 材料与方法
1.1 试验材料
供试24份大白菜材料(表1)保存于中国农业科学院蔬菜花卉研究所白菜课题组,以感病大白菜作为对照。供试根肿菌M01、M02和M03分别采自辽宁沈阳、云南昆明和河南郑州,根肿病菌根置于-20 ℃冰箱中保存备用。

表1 材料名称及来源
1.2 接种方法
1.2.1 菌液的制备 2017年3月8日在中国农业科学院蔬菜花卉研究所实验楼406进行,制备方法在Kawamura等(2017)的基础上进行了改进,取20 g保存于-20 ℃冰箱的根肿病菌根,室温下解冻后加入200 mL蒸馏水于搅拌器中搅碎,8层纱布过滤,滤液于4℃、4 000 r·min-1离心10 min,弃上清液,用无菌水悬浮沉淀,用血球计数板将浓度调至2×107个·mL-1,4 ℃冰箱保存备用。
1.2.2 接种 2017年3月在本所南圃场春化室进行,采用浸根法(柴阿丽 等,2015)接种。3月2日在铺有单层滤纸的培养皿中对大白菜种子进行催芽,6 d后将根系浸入配制好的根肿菌悬浮液中,30 min后将幼苗栽入提前装好基质并灌透底水的50孔穴盘中,每穴1株,每处理6穴,3次重复。42 d后进行病情调查。
1.3 病情调查
病 情 分 级 标 准 参 考 Kuginuki等(1999)、Suwabe等(2006)、Kato等(2013)的方法:0级,根部无肿大现象;1级,侧根根部有少量小肿瘤;2级,侧根根部有较大肿瘤或主根上有小肿瘤;3级,主根和侧根都出现严重的肿大(图1)。
病情指数计算及抗性评价标准参考Zhang等(2015)的方法:抗病(R),病情指数≤26;感病(S),病情指数>26。
病情指数=Σ(各病级株数×相应级数)/(调查总株数×3)×100

图1 根肿病病情分级标准
1.4 数据分析
采用SPSS软件对调查结果进行单因素方差分析:① 针对每1份材料对3种根肿菌的抗性表现进行方差分析;② 分别计算出24份材料对3种根肿菌病情指数的平均值,然后进行方差分析。
1.5 分子鉴定
1.5.1 DNA的提取 采集24份大白菜材料的幼嫩叶片,采用改良CTAB(十六烷基三甲基溴化铵)法(Murray & Thompson,1980)提取单株DNA,用Nanodrop 2000分光光度计测定DNA浓度。
1.5.2 分子标记检测 对24份大白菜材料进行分子标记鉴定,共用到22个标记,涉及10个基因。其中,标记HC352b-SCAR、SC2930-T-FW/-RV、SC2930-Q-FW/-RV、CRaim-T与 基 因CRa连 锁(Hayashida et al.,2008;Matsumoto et al.,2012;Ueno et al.,2012),标记 TCR05、TCR09、TCR79、TCR108、K-3与 基 因CRb连 锁(Piao et al.,2004;Zhang et al.,2014;Chen et al.,2016), 标记KBrH129J18R与基因CRbkato连锁(Kato et al.,2013), 标 记 B50-C9-FW/-RV、B50-6R-FW/-RV与 基 因CRc连 锁(Matsumoto et al.,2012), 标记 HC688-4-FW/-6-RV、HC688-4-FW/-7-RV与基 因CRk连 锁(Matsumoto et al.,2012), 标 记BRMS-088和BRMS-096分别与基因Crr1和Crr2连锁(Suwabe et al.,2003),标记OPC11-2S与基因Crr3连锁(Saito et al.,2006),标记M8、M9、M10与基因CR-A3连锁(Gao et al.,2014),标记BrID11683、BrID90269与1个尚未定位的基因连锁(王彤彤 等,2012)。所有引物均由生工生物工程(上海)股份有限公司合成。PCR扩增体系为10 μL:PCR mix 5 μL,1.0 μmol·L-1上下游引物各0.2 μL,50 ng·L-1模板 DNA 2 μL,ddH2O 补齐至10 μL。PCR扩增片段长度≥600 bp的标记利用1.5%的琼脂糖凝胶电泳分离,PCR扩增片段长度<600 bp的标记利用8%的聚丙烯酰胺凝胶电泳分离。
2 结果与分析
2.1 接种鉴定
从图2可以看出,感病对照CK对3种根肿菌都表现感病,说明本试验采用的接种方法准确可靠。24份大白菜材料中,有10份抗病材料,14份感病材料。其中,材料CR004、CR007、CR013、CR015和CR016对3种根肿菌都表现抗病,材料CR012、CR014和CR018对根肿菌M01和M02表现抗病,材料CR021只对根肿菌M01表现抗病,材料CR023只对根肿菌M02表现抗病。3个根肿病小种对24份大白菜材料平均病情指数的单因素方差分析结果表明:根肿菌M01和M02在24份材料中的致病力差异不显著,根肿菌M03在24份材料中的致病力与M01和M02差异显著,从材料CR014、CR017和CR018的表现也可以看出,根肿菌M01和M02之间的致病力差异不显著,而根肿菌M03的致病力与M01、M02差异显著。

图2 不同大白菜材料对3种根肿菌的抗性鉴定结果
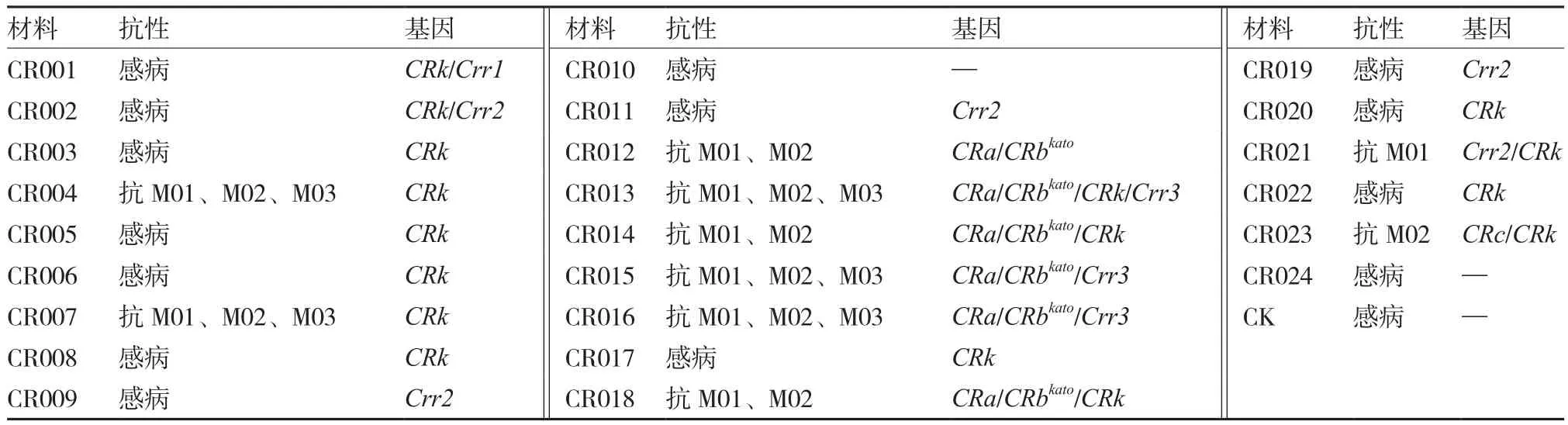
表2 不同大白菜材料的根肿病抗性及对应的抗性基因
2.2 分子标记鉴定
鉴定结果表明,本试验用到的22个分子标记中只有10个具有多态性,分别是SC2930-T-FW/-RV、SC2930-Q-FW/-RV、KBrH129J18R、B50-C9-FW/-RV、B50-6R-FW/-RV、HC688-4-FW/-6-RV、HC688-4-FW/-7-RV、BRMS-088、BRMS-096和OPC11-2S,共涉及7个基因,分别是CRa、CRbkato、CRc、CRk、Crr1、Crr2和Crr3。
由表2可以看出,感病材料中有的不含抗病基因,有的含有抗病基因CRk、Crr1或Crr2。材料CR012、CR014和CR018对根肿菌M01和M02表现出抗性,其中CR012含有基因抗病基因CRa和CRbkato,CR014和CR018含有抗病基因CRa、CRbkato和CRk; 材 料 CR004、CR007、CR013、CR015和CR016对根肿菌M01、M02和M03表现出抗性,其中CR004和CR007只含有抗病基因CRk,CR013含有抗病基因CRa、CRbkato、CRk和Crr3,CR015和CR016含有抗病基因CRa、CRbkato和Crr3;材料CR021只抗根肿菌M01且含有抗病基因Crr2和CRk,材料CR023只抗根肿菌M02且含有抗病基因CRc和CRk。
3 结论与讨论
明确根肿菌各生理小种在我国不同地区的分布,对提高抗病品种选育的效率具有非常重要的意义。根肿菌存在生理小种的分化,目前已经鉴定出的生理小种超过24个(孙保亚 等,2005)。我国主要的生理小种有2号、4号、7号、10号和11号,其中以4号生理小种为主(沈向群 等,2009;丁云花 等,2013)。彭沙莎等(2013)鉴定出湖南大白菜根肿菌生理小种有1号、4号、9号和13号;刘峰等(2013)鉴定出云南和西藏大白菜根肿菌生理小种有1号、2号、4号、6号、7号、10号、11号和12号;李宁等(2015)鉴定出陕西太白县根肿菌生理小种为7号。因此,明确抗源对不同生理小种或不同地区生理小种的抗性对育种工作十分重要。
分子标记在辅助选择育种、遗传多样分析和基因定位及克隆等方面的应用具有十分明显的优越性(McCouch et al.,1997;Liu et al.,2013)。孙保亚等(2005)从国外引进的大白菜品种中分离出48个抗根肿病大白菜自交系;朴钟云等(2010)通过分子标记辅助选择的方法加速回交选育,培育出大白菜优良自交系BJN3的9份抗根肿病近等基因系。Matsumoto等(2012)利用分子标记辅助选择和传统育种相结合的方法实现了3个抗根肿病基因CRa、CRc和CRk的聚合,并证明了聚合之后作物的抗病能力有所加强。本试验中,抗病基因CRk、Crr1和Crr2也存在于感病材料中,笔者推测这3个基因对材料的抗病性没有贡献。材料CR012、CR014和CR018对根肿菌M01和M02表现出抗性,其中CR012含有抗病基因CRa和CRbkato,CR014和CR018含有抗病基因CRa、CRbkato和CRk,由此推测材料对M01和M02的抗性可能与CRa或CRbkato有关,也可能与两者都有关。材料CR013、CR015和CR016对根肿菌M01、M02和M03都表现出抗性,其中CR013含有抗病基因CRa、CRbkato、CRk和Crr3,CR015和CR016含有抗病基因CRa、CRbkato和Crr3,推测基因Crr3对M03具有特异抗性。材料CR023只抗根肿菌M02且含有抗病基因CRc和CRk,因此认为基因CRc可能对M02具有特异抗性。CR004、CR007和CR021是3份比较特殊的材料,其中CR004和CR007对根肿菌M01、M02和M03都表现出抗性但只含有抗病基因CRk,CR021只抗根肿菌M01且含有抗病基因Crr2和CRk;由于本试验感病材料中也含有基因Crr2和CRk,因此推测这3份材料中可能还含有其他抗病基因,有待进一步研究。
本试验对24份大白菜材料进行接种鉴定,首次明确了它们对3个不同地区根肿菌的抗性,并筛选出10份抗病材料。同时,利用已经开发出的与抗病基因连锁的分子标记对24份材料进行了分子鉴定,明确了与材料抗病性相关的基因,对今后抗病材料的筛选和抗病基因的分子聚合育种具有较高的参考价值。
柴阿丽,朱发娣,王惟萍,石延霞,谢学文,李宝聚.2015.十字花科根肿病接种技术及发病条件研究.华北农学报,(S1):266-271.
丁云花,简元才,余阳俊,汪维红,耿丽华,康俊根.2013.我国8省市十字花科蔬菜根肿菌生理小种的鉴定.中国蔬菜,(16):85-88.
李宁,赵利民,鱼昭君,陈红红,惠麦侠.2015.大白菜品种对陕西省太白县根肿病的抗性鉴定.中国农学通报,31(34):173-176.
刘峰,张丽辉,姬广海.2013 .云南和西藏十字花科蔬菜根肿菌生理小种鉴定.中国蔬菜,(20):77-78.
彭沙莎,任佐华,黄小莉,刘敏捷,孙良菲,刘二明.2013.湖南十字花科作物根肿菌生理小种鉴定.长江蔬菜,(6):46-49.
朴钟云,吴迪,王淼,张腾.2010.大白菜抗根肿病近等基因系的分子标记辅助选育.园艺学报,37(8):1264-1272.
沈向群,聂凯,吴琼,张玉光,孟星河.2009.大白菜根肿病主要生理小种种群分化鉴定初报.中国蔬菜,(8):59-62.
孙保亚,沈向群,郭海峰,周永红.2005.十字花科植物根肿病及抗病育种研究进展.中国蔬菜,(4):34-37.
王彤彤,张淑江,章时蕃,李菲,张慧,孙日飞.2012.大白菜根肿病抗性基因的标记和定位.中国蔬菜,(14):31-35.
杨永林.1990.十字花科蔬菜根肿病抑菌型土壤初探.植物保护学报,17(2):127-131.
Chen J,Jing J,Zhan Z.2013.Identification of novel QTLs for isolatespecific partial resistance toPlasmodiophora brassicainBrassica rapa.PLoS One,8(12):e85307.
Chen L,Zhang X,Xu H.2016.Introgression of clubroot resistance into an elite pak choi inbred line through marker-assisted introgression breeding.Plant Breeding,135(4):471-475.
Chu M,Song T,falk K,Zhang X,Liu X,Chang A.2014.Fine mapping ofRcr1and analyses of its effect on transcriptome patterns during infection byPlasmodiophora brassicae.BMC Genomics,10.1186/1471-2164-15-1166.
Gao F,Hirani A H,Liu J,Liu Z,Fu G,Wu C,Peter B E,McVetty P B E,Li G.2014.Fine mapping a clubroot resistance locus in Chinese cabbage.J Amer Soc Hort Sci,139(3):247-252.
Hatakeyama K,Niwa T,Kato T,Ohara T,Kakizaki T,Matsumoto S.2017.The tandem repeated organization of NB-LRR genes in the clubroot-resistantCRblocus inBrassica rapaL.Molecular Genetics and Genomics.292(2):397-405.
Hayashida N,Takabatake Y,Nakazawa N.2008.Construction of a practical SCAR marker linked to clubroot resistance in Chinese cabbage,with intensive analysis ofHC352bgenes.Journal of the Japanese Society for Horticultural Science,77(2):150-154.
Hirai M,Harada T,Kubo N,sukada M,Suwabe K,Matsumoto S.2004.A novel locus for clubroot resistance inBrassica rapaand its linkage markers.Theoretical and Applied Genetics,108(4):639-643.
Karling J S.1968.The plasmodiophorales:including a complete host index,bibliography,and a description of diseases caused by species of this order.Molecular Biology & Evolution,22(3):582-588.
Kato T,Hatakeyama K,Fukino N,Matsumoto S.2013.Fine mapping of the clubroot resistance geneCRband development of a useful selectable maker inBrassica rapa.Breeding Sci,6(3):116-124.
Kawamura K,Shimizu M,Kawanabe T.2017.Assessment of DNA markers for seed contamination testing and selection of disease resistance in cabbage.Euphytica,213(1):28.
Kuginuki Y,Yoshikawa H,Hirai M.1999.Variation in virulence ofPlasmodiophora brassicaein Japan tested with clubrootresistant cultivars of Chinese cabbage(Brassica rapaL.ssp. pekinensis).European Journal of Plant Pathology,105(4):327-332.
Liu B,Wang Y,Zhai W,Deng J,Wang H,Cui Y,Cheng F,Wang X W,Wu J.2013.Development of InDel markers forBrassica rapabased on whole-genome re-sequencing.Theoretical and Applied Genetics,126(1):231-239.
Matsumoto E S,Ueno H S,Aruga D.2012.Accumulation of three clubroot resistance genes through marker-assisted selection in Chinese cabbage(Brassica rapassp. pekinensis).Journal of the Japanese Society for Horticultural Sciences,81(2):184-190.
McCouch S R,Chen X,Panaud O.1997.Microsatellite marker development,mapping and applications in rice genetics and breeding.Plant Molecular Biology,35:89-99.
Murray M G,Thompson W F.1980.Rapid isolation of high-molecular weight plant DNA.Nucleic Acids Research,8:4321-4325.
Piao Z Y,Deng Y Q,Choi S R.2004.SCAR and CAPS mapping ofCRb,a gene conferring resistance to Plasmodiophora brassicae in Chinese cabbage(Brassica rapassp. pekinensis).Theoretical and Applied Genetics,108(8):1458-1465.
Saito M,Kubo N,Matsumoto S,Suwabe K,Tsukada M,Hirai M.2006.Fine mapping of the clubroot resistance gene,crr3,inBrassica rapa.Theor Appl Genet,114:81-91.
Sakamoto K,Saito A,Hayashida N,Taguchi G,Matsumoto E.2008.Mapping of isolate-specific QTLs for clubroot resistance in Chinese cabbage(Brassica rapaL. ssp. pekinensis).Theoretical and Applied Genetics,117(5):759-767.
Suwabe K,Tsukazaki H,Lketani H.2003.Identification of two loci for resistance to clubroot(Plasmodiophoru brassicaeWorenin)inBrassica rapaL.Theoretical and Applied Genetics,107(6):997-1002.
Suwabe K,Tsukazaki H,Iketani H,Hatakeyama K,Kondo M,Fujimura M,Nunome T,Fukuoka H,Hirai M,Matsumoto S.2006.Simple sequence repeat-based comparative genomics betweenBrassica rapaandArabidopsis thaliana:the genetic origin of clubroot resistance.Genetics,173(1):309-319.
Ueno H,Matsumoto E,Aruga D,Kitagawa S,Matsumura H,Hayashida N.2012.Molecular characterization of theCRagene conferring clubroot resistance inBrassica rapa.Plant Mol Biol,80:621-629.
Zhang T,Zhao Z,Zhang C.2014.Fine genetic and physical mapping of theCRbgene conferring resistance to clubroot disease inBrassica rapa.Molecular Breeding,34(3):1173-1183.
Zhang H,Feng J,Zhang S.2015.Resistance toPlasmodiophora brassicaeinBrassica rapaandBrassica junceagenotypes from China.Plant Disease,99(6):776-779.
