SRAP技术分析海南降香檀遗传多样性
2018-03-05孟慧陈波杨云
孟慧 陈波 杨云


摘要 为探讨海南降香檀遗传多样性,以采自海南省东、西、南、北、中五个区域23个点185份降香檀单株材料为材料,采用SRAP分子标记对其遗传多样性进行分析。结果表明:利用筛选出的25对SRAP引物组合,扩增共检测到174个位点,多态性位点92个,多态率为52.8%。UPGMA聚类分析结果表明,185份海南降香檀种质材料与对照多裂黄檀遗传相似系数为0.56;在相似系数约为0.73处,可将收集到降香檀种质划分为4个类群。SRAP标记可较好地反映海南降香檀种质间的遗传多样性,为合理保护和利用海南降香檀种质资源提供科学依据与借鉴.并为海南降香檀分类系统和进化关系提供分子系统学依据。
关键词 SRAP;降香檀;遗传多样性
降香檀(Dalbergia odorifera T.Chen)为豆科黄檀属植物,是海南特有濒危树种,被列为国家二级重点保护野生植物[1-2]。其根、茎部干燥心材即为中药“降香”,具有行气活血、止痛止血的功效[3],是珍稀名贵南药。由于国产降香檀心材具有耐腐蚀、纹理细密,且散发芳香,经久不退等特点,是国家标准5属8类34种红木之一[4]。早在17世纪,海南省野生降香檀资源已被掠夺式砍伐[5]。现仅存的资源也被原产地附近居民采挖栽种于自家院落.导致原始森林中已很难找到成龄降香檀。由于野生资源的匮乏,心材的高附加值,导致海南、广东、广西、云南、福建、四川等地大量开展人工栽培降香黄檀林[6-12]。降香檀因地理分布的特点及特有的生长环境对其心材生长发育影响十分明显,分析降香檀遗传多样性对于降香檀种质资源的收集、品种选育、合理保护和利用具有重要的意义。
SRAP(序列相关扩增多态性)是一种新型的基于PCR的分子标记技术,该技术是通过独特的双引物设计对基因的ORFs (Open reading frames开放阅读框)的特定区域进行扩增,因不同个体以及物种的内含子、启动子与间隔区长度不同而产生多态性[13]。SRAP技术具有操作简便、重复性好、多态性高,共显性高、引物具有通用性、易于测序等优点,在动物、植物、微生物等学科中都广泛使用。其中,该技术在植物种质资源鉴定评价、遗传图谱构建、遗传多样性研究、基因定位、重要性状标记以及比较基因组学等方面广泛应用[14]。目前,有文献报道通过RAPD技术分析降香黄檀遗传多样性[15],而有关降香檀的SRAP分析尚未见报道。前人研究表明,SRAP技术在生态型进化史上比AFLP更具有一致性。在育种目标性状的评价方面明显优于RAPD标记,有较高的多态性标记比率,是评价遗传多样性、品种鉴定和系统发生的有效工具[16]。SRAP标记能够较好的开展遗传多样性分析,通过直接分析遗传物质的多态性作为不同居群的划分依据更为可行和客观,可以避免由于不同发育阶段和季节性生理生化差异而影响指标的准确性[17]。本研究利用筛选出的25对SRAP引物组合[18]对采自海南省东、西、南、北、中5个区域23个点185份降香檀单株材料进行遗传多样性分析,为合理保护和利用海南降香檀种质资源提供科学依据与借鉴,并为海南降香檀分类系统和进化关系提供分子系统学依据。
1 材料和方法
1.1 材料
1.1.1 植物材料
以采自海南省东、西、南、北、中五个区域23个点共185份降香檀(Dalbergia odorifera T.Chen)和1份外类群物种多裂黄檀(Dalbergia rimosaRoxb)的嫩叶进行遗传多样性分析,植物经中国医学科学院药用植物研究所海南分所资源室李榕涛老师鉴定无误,具体见表1。
1.1.2 引物材料
25对引物参考杨云等[18]对降香檀SRAP引物的筛选。
1.2 方法
1.2.1 DNA的提取方法与检测
降香檀基因组DNA提取参考杨云等[19]的方法。
1.2.2 PCR扩增与检测
PCR扩增反应体系的总体积为20μL,其中10×Taq Buffer 2μL、25 mmol/L MgCl2 1.6μL、4×dNTP Mixture 0.5μL、5 U/μL Tap DNA聚合酶0.2μL、左右引物浓度各2μL,约20 ng/μL基因组DNA 2μL。扩增程序为:94℃预变性3 min;94℃变性30 s,35℃退火45 s,72延伸45 s,5个循环;94℃变性30 s,50℃退火45 s,72延伸45 s,35个循环;72℃延伸10 min;4℃保存。PCR扩增产物采用8%非变性聚丙烯酰胺凝胶电泳检测PCR扩增产物,电泳结束后取下凝胶进行银染,染色程序参考张军等[20]的方法。
1.3 数据统计
以电泳照片记录谱带,按照相同迁移位上有多态性扩增带记为“1”,无带为“0”的方法,用Glyko Banscan 4.5软件辅助记录电泳谱带,多态性和重复性好的扩增带才被记录。聚类分析应用NTSYSpc 2.1数据分析软件进行,UPGMA方法聚类。
2 结果与分析
2.1 SRAP扩增结果
利用杨云等[16]筛选出的25对SRAP引物组合对185份种质材料进行PCR扩增,均获得带型丰富且清晰可辨的电泳图谱,共检测到174个位点,多态性位点92个,多态率为52. 8%,反映了海南降香檀种质具有较高的基因多样性水平。部分引物的电泳结果见图~2。
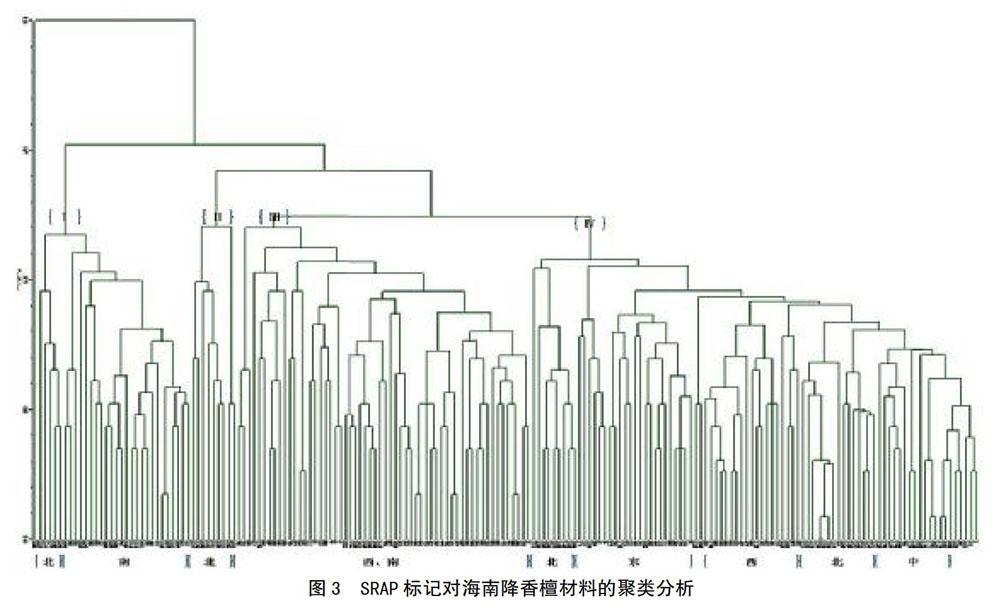
2.2 遺传多样性和聚类分析
根据SRAP标记对185份海南降香檀种质材料的遗传相似系数进行UPGMA聚类分析,得到聚类树状图(图3)。由图3可知,185份海南降香檀种质材料与对照多裂黄檀(编号186号)遗传距离较远,遗传相似系数为0.56。结果与余敏[21]提出的降香檀与多裂黄檀在黄檀属中有着较近的亲缘关系,但是二者又不属于同一植物类群的观点相同。5个行政区域23个自然居群的185份降香檀样品,总多态位点百分率是52.8%,说明降香檀种内遗传背景具有较丰富的多样性;在相似系数约为0.73处可将供试的185份海南降香檀种质材料划分为4个类群(即Ⅰ、Ⅱ、Ⅲ、Ⅳ),这与降香檀自然分布情况一致。
第Ⅰ类群主要为海南南部地区和北部地区的部分种质,这个类群又可划分为2个亚类群,南、北部地区的种质分别在2个不同亚类群中。
第Ⅱ类群除了南部地区的1份种质外(编号112号),其它8份全部为北部地区种质。
第Ⅲ类群主要包括了海南南部地區和西部地区的部分种质。
第Ⅳ类群包括了东、西、北和中部地区的部分种质,中部地区的大部分种质均聚在这个类群中,并且与其它地区的种质遗传距离较远。如果将这个类群再细划分,各地区的种质又分别在不同的亚类群中。
3 讨论
分子标记技术应用于降香檀遗传多样性检测方面的报道不多。杨新全等[22]利用RAPD分子标记技术对降香黄檀6个居群77个样品的遗传多样性进行研究,发现其多态位性百分率为54.55%。訾慧等[23]利用RAPD技术鉴别中药降香及其易混淆品山油柑,品种间出现了明显的多态性差异,每种药材均得到4~7条带,易混淆品山油柑与正品降香的带型不同,有明显的差异。本研究对185份海南降香檀种质材料进行了遗传多样性分析,结果表明,25对SRAP引物组合对185份海南降香檀种质材料进行PCR扩增,均获得带型丰富且清晰可辨的电泳图谱,多态率为52.8%,说明SRAP标记技术在海南降香檀种质的遗传多样性研究中有较高的检出率。
海南各地降香檀虽然被聚类为4大类群,但主要以北部地区和南部及西南部地区为主,这与海南降香檀野生分布集中在北部石山地区和南部及西南部地区相吻合,其余各地区种质基本来自于这两个大地区。
降香檀种质遗传多样性较为丰富,但来源主要为北部、南部及西南部的自然分布区。第Ⅲ类群中包含了南部和西部2个区域,这与降香檀历史记载的天然分布一致,分布在西南部和南部区域的种质资源来源基本相同;第IV类群中包含了北、东、西、中4个区域,唯独没有南部区域的种质资源,说明东、西、中区域的种源大多来自北部区域。北部区域由于海口的政治经济文化中心地位以及便利的交通,更利于降香檀种苗的运输和传播。其中,西部区域出现了南部和北部种质混杂的现象,主要原因是西部样品来源于儋州、白沙、昌江、东方等地,其中儋州等位于西北部,其种质主要来源于北部区域,而另一部分西部区域采样点位于西南部,和南部区域交叉种质交流频繁,所以导致西部区域种质出现南北混杂的现象。
本研究利用SRAP标记技术解析了海南地区降香檀种质遗传多样性,为我国降香檀种质资源调查、种质鉴别提供理论依据。但由于研究区域、采样数量的局限性,只能局部的反映降香檀种质的遗传多样性特点。因此,要全面系统地对降香檀种质遗传多样性研究还需要更大的采样量并结合多种研究方法,后续工作也正在开展。
参考文献
[1]中华人民共和国药典委员会,中国药典(一部)[S].北京:中国医药科技出版社.2015.
[2]韦直,陈德昭,陈邦余,等,中国植物志(第40卷)[M].北京:科学出版社,1994.
[3]陈焕镛,海南植物志[M].北京:科学出版社,1965.
[4]傅立国.中国植物红皮书——稀有濒危植物(第一册)[M].北京:科学出版社,1992.
[5]林励,丘金裕,蔡岳文,等,广东珍贵南药树种生产现状、问题及对策研究[J].中国现代中药,2013,15(2):127-130.
[6]罗文扬,罗萍,武丽琼,等.降香檀及其可持续发展对策探讨[J].热带农业科学,2009,29 (1):44.
[7]连辉明,张谦,殷祚云,等,广东降香黄檀人工林生长及寒害调查分析[J].中南林业科技大学学报.2014, 34(10):26-31
[8]郭文福,贾宏炎,降香黄檀在广西南亚热带地区的引种[J].福建林业科技,2006,33 (4):152.
[9]王立新,许丽萍,李倩,等,降香黄檀在普洱市引种试验初报,林业调查规划[J].2016,41(5):81-85.
[10]姚庆端,林清锦,郑志雷.福建省降香黄檀引种适宜区研究[J].福建林业科技,2012,39 (2):89-84.
[11]鲁洋,魏鹏,贾晨,等.降香黄檀在宜宾地区引种栽培试验初步评价[J].南方农业,2017,11(8):121-123.
[12]孟慧,谢彩香,杨云,等,降香黄檀产地适宜性分析[J].时珍国医国药,2010,21 (9):2 304-2306.
[13] Li G,Quiros C F.Sequence-related amplifiedpolymorphism (SRAP), a new marker system based ona simple PCR reaction: its application to mappingand gene tagging in Brassica [J]. Theor ApplGenet, 2001, 103: 455-461.
[14]刘中华,林志坚,李华伟.等,基于SRAP标记的甘薯遗传多样性与起源演化分析[J].植物遗传资源学报,2018,19 (3):468-477.
[15]杨新全,冯锦东,杨成民,等,濒危药用植物降香黄檀DNA提取及RAPD反应体系优化[J].热带林业,2007, 35 (1):47-49.
[16] Ferriol M, Pico B, Cordava P F.Molecular diversity of a germplasm collection of sqash (Cacurbitamoschata) determined by SRAP and AFLP makers [J].Crop Sci, 2004, 44: 653-664.
[17]钟军,王坤,仇萍,等.SRAP分子标记技术分析鱼腥草居群的遗传多样性[J].植物生理学通讯,2010, 46(3):210-216.
[18]杨云,孟慧,吴翼,等,降香黄檀SRAP分子标记的引物筛选[J].江西农业学报,2011,29(3):29-31.
[19]杨云,孟慧,魏建和,等,降香檀基因组DNA的提取方法研究[J].生物技术通讯,2009,20(3):383-385.
[20]张军,武耀廷,郭旺珍,等.棉花微卫星标记的PAGE/银染快速检测[J].棉花学报,2000,12(5):267-269.
[21]余敏.降香檀与多裂黄檀木材DNA提取及其rDNA-ITS序列分析[D].合肥:安徽农业大学,2012.
[22]杨新全,冯锦东,魏建和,等.我国特有濒危药用植物降香黄檀遗传多样性研究[J].世界科学技术-中医药现代化,2007, 9(2):73-76.
[23]訾慧,曹爱民,孟宪生,等.RAPD技术对重要降香的鉴别研究[J].辽宁中医药杂志,2009,36(1):103-105.
