基于SSR标记的我国咖啡种质资源遗传多样性分析及指纹图谱构建
2018-03-05王晓阳黄丽芳闫林周华陈金焕孙燕董云萍
王晓阳 黄丽芳 闫林 周华 陈金焕 孙燕 董云萍

摘要 为分析我国咖啡种质遗传背景,提高咖啡资源保护与利用效率,利用31个SSR分子标记对我国主要的92份咖啡种质进行遗传多样性检测。结果表明.92份咖啡种质遗传相似系数变幅为0.666~0. 997,平均为0. 85,其中小粒种咖啡种内平均遗传相似系数为0.923。在遗传多样性分析基础上,利用5个SSR标记构建了25个中粒种咖啡品系的DNA指纹图谱。研究表明,我国咖啡种质资源遗传基础非常狭窄,利用现有咖啡资源开展品种选育潜力有限,应加强对野生特异种质的引种与发掘利用。
关键词 咖啡;SSR;遗传多样性;指纹图谱
咖啡与可可、茶同列世界三大饮料作物,在世界热带农业经济、国际贸易及人们生活中占有重要地位。我国咖啡种植主要分布在云南、海南和四川等省,全国种植面积约3 000万hm2,总产量约12万t,是我国主要热带经济作物。
咖啡(Coffea Linn.)原产非洲埃塞俄比亚及刚果河流域,为茜草科咖啡属植物,该属包含大约100个种[1],其中,具有商业栽培价值的主要为小粒种(C.arabica L.)和中粒种(C. canephora Pierre ex A.Froelzner),其余种类主要作为种质资源和育种材料保存。我国咖啡资源主要来自于国外引种。目前,国内收集保存咖啡种质约700份,保存机构主要为云南省德宏热带农业科学研究所、中国热带农业科学院香料饮料研究所、云南省农业科学院热带亚热带经济作物研究所等科研單位(以下分别简称德宏所、香饮所、热经所),另有少量由咖啡企业自行引进保存,相当一部分种质存在重复保存。
我国咖啡种质资源引种渠道多元,保存较为分散,种质保存机构间相互引种且独立命名,难免存在种质重复引进、品种同种异名、异名同种,以及种质间亲缘关系不清等问题,给咖啡育种亲本选择及优良品种推广应用带来不便。对我国现有咖啡资源开展遗传多样性分析并进行指纹图谱构建,对于我国咖啡核心种质库构建、资源高效利用及品种推广具有重要意义。
国内相关学者利用形态学标记开展了咖啡种质资源遗传多样性研究[2],这种方法虽然简便、经济,但形态学标记数量有限,且易受环境条件的影响.黄丽芳[3]、闫林[4]等利用RAPD、ISSR等分子标记进行了咖啡种质资源遗传多样性分析及指纹图谱构建。RAPD、ISSR标记为随机性显性标记,结果可重复性有待提高。SSR分子标记为特异性共显性标记,稳定性好、结果可靠,非常适合遗传多样性分析及指纹图谱构建,现已广泛应用于水稻[5]、甘薯[6]、土豆[7]、大豆[8]、玉米[9]等许多作物的遗传多样性分析及指纹图谱构建。本研究利用SSR分子标记对我国主要咖啡种质资源进行遗传多样性分析,并对我国现有中粒种咖啡品种进行了DNA指纹谱图构建,为生产上品种真实性和纯度鉴定、优良品种推广应用提供技术支持,也为我国咖啡遗传改良、育种亲本选配、种质保护、核心种质库构建等提供科学依据。
1 材料与方法
1.1 材料
供试材料取自国内三大咖啡种质保存单位:德宏所、香饮所及热经所,共计92份,其中,小粒种59份、中粒种25份、大粒种8份(表1)。样本包括我国咖啡主栽品种、国外引进品种、自主选育品种及其它种质,基本涵盖了我国现有咖啡主要种质。采集咖啡种质叶片样本,利用硅胶干燥剂干燥保存。
1.2 方法
1.2.1 DNA提取
利用植物DNA提取试剂盒(品牌:TIAhGEN,产品型号:DP320)进行咖啡叶片样本DNA提取。
1.2.2 SSR标记检测
SSR引物来自相关文献发表的咖啡特异性SSR引物。PCR总反应体系为20μL,组分包括:2 ng/μL基因组DNA, 1.5 mmol/L MgCl2,0.2mmol dNTP,0.2μmol/L引物,0.03 U/μL Taq酶,1×PCR缓冲液。SSR-PCR扩增反应程序采用Touch-down PCR:94℃4min, 15个Touch-down循环(940C 15 s,600C 15 s,72℃ 30s,每次循环的退火温度降0.7℃),15个普通循环(94℃ 15s,49.5℃ 15 s,72℃ 30 s),最后于72℃延伸20 min。扩增产物于6%聚丙烯酰胺变性凝胶电泳分离检测,银染法显带分析。
1.2.3 数据统计及分析
统计SSR扩增条带,在相同位点上,有带记为“1”,无带记为“0”,利用Excel软件建立数据矩阵。利用NTSYS-pc2.10y分析软件通过加权平均法(UPMGA)计算遗传相似系数并进行聚类分析。筛选条带清晰、多态性强的SSR引物用于指纹图谱构建。对SSR标记扩增条带进行图形化处理;同一位点上有带用黑色方格表示.无带用白色方格表示,据此构建咖啡品系DNA指纹图谱。
2 结果与分析
2.1 多态性SSR引物筛选
通过相关文献检索.共获取410对咖啡特异性SSR引物[10-16]。利用小粒种(CIFC7963 (F6)、TYPICA)、中粒种(越南-1、泰国4-2)、中小粒杂交种(ARABUSTA)及大粒种(南顶)共6份种质对获得的410对SSR引物进行多态性筛选,共筛选出多态性SSR引物152对,从中继续筛选出条带清晰的多态性SSR引物31对(表2),用于后续遗传多样性分析及指纹图谱构建。
2.2 遗传多样性分析
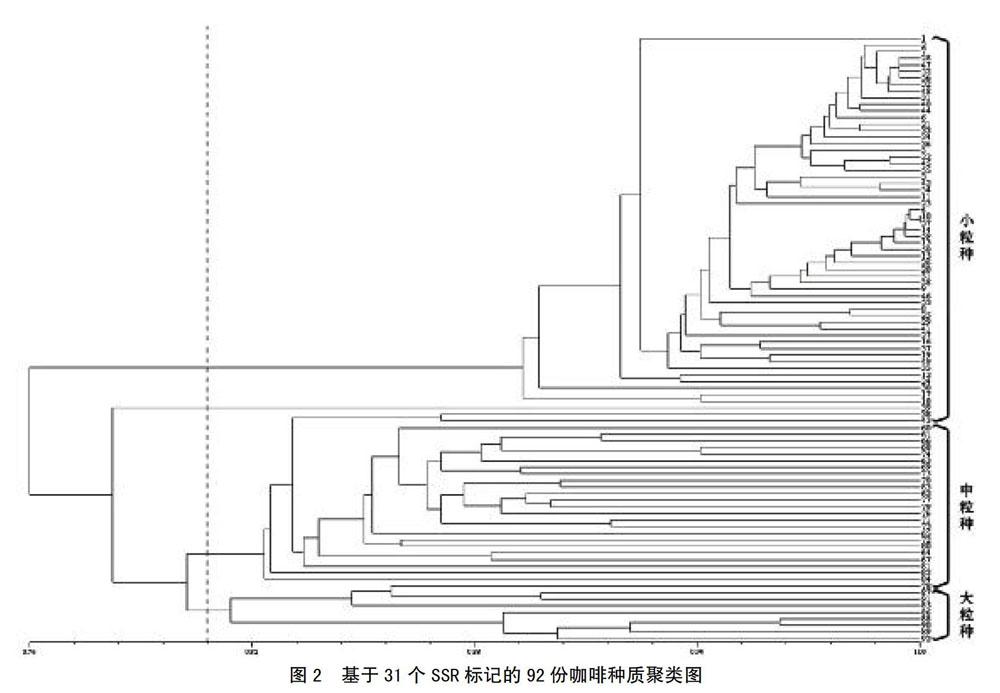
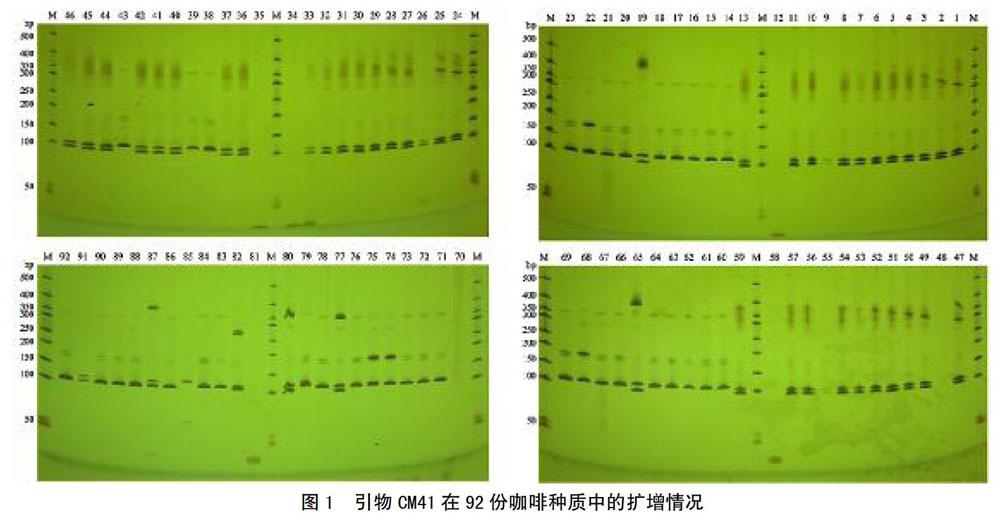
利用筛选出的31对SSR引物对92份咖啡种质进行了扩增检测(部分引物检测结果见图1)。通过遗传相似性分析,结果表明.92份咖啡种质遗传相似系数变幅为0. 666~0. 997 [‘CIFC7963 (F6)与‘德热199-1相似系数为1,二者可能存在同种异名情况],平均遗传相似系数为0. 85,大粒种、中粒种种内平均遗传相似系数分别为0. 849、0.85,小粒种种内平均遗传相似系数高达0. 923,表明我国小粒种咖啡种质资源遗传基础非常狭窄。进一步分析小粒种、中粒种、大粒种咖啡种间遗传相似系数,结果表明,种间平均遗传相似系数为0.764,在遗传相似系数分析基础上进行聚类分析,结果表明,在相似系数约0.8处可以大致将小粒种、中粒种、大粒种3个种分开(图2)。
2.3 指纹图谱构建
从31对SSR引物中筛选扩增条带清晰、稳定且多态性好的引物,对25个中粒种咖啡品系进行指纹图谱构建。结果表明,利用5对SSR引物(CMA41、CMA159、CM5、368、471,引物序列见表2)即可将25个中粒种咖啡品系完全区分开。5对SSR引物在25个中粒种品系中共扩增出20条清晰谱带,每对引物平均4条,CMA41引物扩增情况见图1。通过对SSR扩增条带进行图形化处理,获得了25个中粒种咖啡品系的SSR标记指纹图谱(图3)。
3 讨论
3.1 我国咖啡遗传多样性
本研究在国内首次利用SSR分子标记分析了我国咖啡种质资源的遗传多样性。92份咖啡种质平均遗传相似系数为0.85,其中小粒种种内平均相似系数高达0.923,表明我国咖啡种质资源尤其是小粒种种质资源遗传基础十分狭窄。咖啡在我国为外来引进作物,在国内无野生自然分布。我国现有咖啡种质很大一部分是为满足生产实际需求而引进的育成品种。目前世界现有咖啡育成品种主要来自于Typica、Bourbon、Timor hybrid及其变种等少数亲本材料,育成品种间遗传多样性十分狭窄,利用现有咖啡品种资源开展优良品种选育潜力十分有限。现有咖啡育成品种的遗传基础仅占世界所有咖啡种质资源遗传基础的很小一部分,加强对野生咖啡种质资源优异性状的发掘与利用对于现有咖啡品種遗传改良具有重要意义[17]。利用现有育成品种与野生特异性种质杂交,进而培育优良杂交品种已逐渐成为咖啡品种选育的趋势,目前,已培育出一系列具有高产、抗病、优质等优良性状的咖啡杂交新品种[18]。
3.2 咖啡分子标记指纹图谱构建
中粒种和小粒种作为世界两大咖啡商业栽培种,对其品种与种质资源开展指纹图谱构建具有重要意义。由于同属但不同种,二者形态学差异较大,成年植株一般肉眼即可对所属物种进行辨认区分.种子或幼苗也可利用种间特异性SSR标记进行快速鉴别[20],因此,只需分别对二者进行指纹图谱构建即可。
中粒种咖啡为异花授粉植物,相较自花授粉的小粒种咖啡,品种间遗传距离相对较大,更易于构建指纹图谱。本研究利用5对SSR引物构建了国内25个主要中粒种咖啡品系的DNA指纹图谱。5对SSR引物共扩增出20条带,条带数量较少,便于品种鉴定比对、可操作性强,且所有扩增条带清晰、稳定、可重复性好,对实际生产中优良品种鉴定与推广应用具有实际生产意义。利用12对SSR引物可将59份小粒种种质中的大部分区分开.但仍有相当一部分未能区分,这可能与种质间亲缘关系过近,甚至种质重复引进、同种异名有关,如其中的CIFC7963(F6)、P1、P2、P3、P4、P5、PT、T5715、T8667同为Catimor系列品种,亲缘关系十分接近,引种过程中也容易出现混淆。通过遗传多样性对现有小粒种咖啡种质进行亲缘关系分析,进而构建核心种质资源库,在此基础上进行小粒种核心种质指纹图谱构建更具可操作性,也更具实际应用意义。
参考文献
[1] Davis A P, Govaerts R, Bridson D M, et al. Anannotated taxonomic conspectus of the genus Coffea (Rubiaceae) [J]. Botanical Journal of theLinnean Society, 2010, 152(4): 465-512.
[2]周华,张洪波,夏红云,等.咖啡种质资源遗传多样性研究[J].热带农业,2015 (5):23-27.
[3]黄丽芳,董云萍,王晓阳,等.利用RAPD标记分析咖啡种质资源的遗传多样性[J].作物学报.2014, 35(12):2 313-2 319.
[4]闫林,黄丽芳,王晓阳,等.一种利用ISSR指纹图谱鉴定咖啡种质资源的方法[Z].中国.201510556607.2. 2018.
[5] Hoang Thi Hue, La Tuan Nghia, Hoang Tuyet Minh,et al. Evaluation of genetic diversity of local-colored rice landraces using SSR markers[J].International Letters of Natural Sciences, 2018,67: 24-34.
[6] Meng Yusha, Liu Qingchang. SSR fingerprinting of203 sweetpotato [Ipomoea batatas (L.) Lam.] varieties [J]. Journal of Integrative Agriculture,2018, 17 (1):86-93.
[7] Masahiro Kishine, Katsuji Tsutsumi, Kazumi Kitta.A set of tetra-nucleotide core motif SSR markersfor efficient identification of potato (Solanumtuberasum) cultivars [J]. Breeding Science, 2017,67 (5):544-547.
[8] Anchal Bisen, Dhirendra Khare, Priya Nair, etal. SSR analysis of 38 genotypes of soybean[Glycine Max (L.) Merr.] genetic diversity inIndia [J]. Physiol Mol Biol Plants, 2015, 21 (1) :109-115.
[9] Jhansi G, Surender M, Aparnna, et al. DNA fingerprinting of maize hybrids and development ofmolecular ID's using SSR markers their parentallines using simple sequence repeat markers [J].Research Journal of Agricultural Science, 2016, 6(1) : 43-47.
[10] Elisa S N, Edila V de R Von Pinho, Maria G G, etal. Development of microsatellite markers foridentifying Brazilian Coffea arabica varieties [J].Genetics and Molecular Biology, 2010, 33 (3):507-514.
[11] Poncet V, Dufour M, Hamon P, et al. Development ofgenomic microsatellite markers in coffeacanephora and their transferability to othercoffee species[J]. Genome, 2007, 50(12): 1156-1161.
[12] Hendre P S, Phanindranath R, Annapurna V, et al.Development of new genomic microsatellitemarkers from robusta coffee (Coffea cunephoraPieere ex A Froehner) showing broad cross-speciestransferability and utility in genetic studies[J]. BMC Plant Biology, 2008, 8(1) : 51.
[13] Cubry P, Musoli P, Legnate H, et al. Diversityin coffee assessed with SSR markers : structureof the genus Coffea and perspectives for breed-ing[J]. Genome, 2008, 51(1): 50-63.
[14] Musoli P, Cubry P, Aluka P, et al. Genetic differ-entiation of wild and cultivated populations:diversity of Coffea canephora Pierre in Uganda.[J]. Genome, 2011, 52(7): 634.
[15] Poncet V, Hamon P, Minier J, et al. SSR cross-am-plification and variation within coffe trees(Coffea spp.)[J].Genome, 2004, 47(6) :1071-1081.
[16] Baruah A, Naik V, Hendre P S, et al. Isolation andcharacterization of nine microsatellite markersfrom Coffea ababica L., showing wide cross-speciesamplifications [J]. Molecular Ecology Notes,2003, 3(4) : 647-650.
[17] Silvestrini M , Junqueira M G , Andrea C. Favarin,et al. Genetic diversity and structure ofEthiopian, Yemen and Brazilian Coffea arabicaL. ac-ces sions using microsatellites markers [J]. GeneticResources and Crop Evolution, 2007, 54 (6) : 1 367-1379.
[18] Bertrand B, Alpizar E, Lara L, et al. Performanceof coffea Arabica Fl hybrids in agroforestry andfull-sun cropping systems in comparison withAmerican pure line cultivars [J]. Euphytica,2011, 181(2) : 147-158.
[19] Habtamu Gebreselassie, Gizachew Atinafu, MeseretDegefa, et al. Arabica coffee (Coffea Arabica L.)hybrid genotypes evaluation for growthcharacteristics and yield performance undersouthern Ethiopian growing condition[J]. Acad.Res.J.Agri. Sci, 2018, 6(2): 89-96.
[20]王曉阳,董云萍,黄丽芳,等.利用SSR分子标记快速鉴别中粒种与小粒种咖啡[J].热带作物学报,2014, 35(4):622-627.
