应用DGGE技术研究玉米储藏过程中的霉菌群落时空分布特征
2018-02-09岳晓禹陈威风
岳晓禹,张 华,陈威风,邹 建,李 欣,杨 娜
(河南牧业经济学院食品工程学院,河南 郑州 450046)
玉米是我国农业生产的主要粮食作物和储粮品种之一。玉米在储藏过程中容易发生霉菌污染,造成严重的经济损失。霉菌污染不仅会导致储粮的颜色、味道、营养成分发生改变,食用价值降低,更会产生黄曲霉毒素、赭曲霉毒素、伏马毒素等多种真菌毒素,对食用者造成急性或者慢性的毒性危害,导致肝炎、肝癌等严重危害人体健康的疾病[1-2]。研究储粮中霉菌污染的发生规律,从而减少储粮霉菌污染,是提高我国粮食安全和食品安全的重要研究领域。
由于真菌毒素危害的严重性,多数研究者将关注点放在真菌毒素上,如对粮食中真菌毒素的污染现状及分布规律进行调查分析[3]、真菌毒素检测方法的建立[4-5]、真菌毒素脱毒方法的开发[6-7]等,而忽视了对霉菌群落本身的研究。霉菌与真菌毒素并非一一对应,一种霉菌可以产生多种毒素,而一种毒素也可能由多种霉菌产生,因此,并不能从储粮中毒素的分布来推测储粮中霉菌的种类。真菌毒素是霉菌的次级代谢产物,霉菌是真菌毒素的源头,研究粮食储存条件与霉菌群落之间的关系,根据实测数据建立储粮霉菌污染预测模型[8],从而采取相应措施减少霉菌污染,可以从根源上降低粮食中真菌毒素的含量。
目前我国关于粮食储存条件与霉菌群落组成之间的研究多数采用培养法进行,如钟雪美等[9]利用平板培养法研究了不同储粮方式的霉菌动态变化和空间格局变化;李听听等[10]通过培养分离鉴定法研究了玉米初始水分及相对湿度对储粮中霉菌群落的影响;吴红萍等[11]采用稀释涂布平板法和Biolog鉴定系统结合分析了海南省各市县储粮谷物霉菌菌相分布。在研究霉菌群落特征时,虽然培养法有利于霉菌菌种的鉴定,但也存在灵敏度低、误差大等缺点[12]。
在微生物群落研究中,分子生物学技术如变性梯度凝胶电泳(denaturing gradient gel electrophoresis,DGGE)等具有的巨大优势。DGGE技术的原理为序列不同的DNA分子在变性剂梯度变化的凝胶中因解链程度不同而获得不同的电泳迁移速率,因此可以通过DGGE将长度相同但序列不同的DNA进行分离[13]。利用通用引物扩增微生物特定DNA片段,再通过DGGE技术将其分离,即可获得样品中微生物群落的指纹图谱。DGGE技术已广泛应用于食品储藏中细菌群落的变化,如鲫鱼储藏过程中[14]、真空包装冷却鹿肉储藏过程中[15]、及生鲜南美白对虾储藏过程中细菌群落的变化[16]等,但在粮食霉菌群落研究中,分子生物学技术应用较少,仅有李听听等[17]利用DGGE技术研究了水分和环境湿度条件与粮食霉菌群落的相关性。为了研究玉米储藏过程中霉菌群落的发生规律,建立储粮霉菌污染预测模型积累理论和数据,本研究采用DGGE技术,对玉米在粮库储藏过程中霉菌群落进行研究,探讨储粮霉菌群落的时间和空间变化规律。
1 材料与方法
1.1 材料与试剂
从河南的储藏粮库中采集储藏玉米,玉米品种主要为郑单958、浚单20、先玉335、伟科702。
为研究储粮中霉菌群落的时间规律,分别采集了储藏1a 2 个月、储藏2a 2 个月、储藏3a 2 个月的不同储藏年限的玉米。为研究储粮中霉菌群落在粮库中的空间规律,采集了粮仓内储藏玉米样品。同一个粮仓设中心、4 个角5 个点,每个点的样品包含玉米粮堆的上中下层玉米,上层在粮面的10~20 cm处,中层在粮堆中间,下层在距底部20 cm处,仟样是先上后下逐层仟样,各点仟样数量一致。把采集到的玉米样品标记清楚后,放置-80 ℃冰箱保存备用。
丙烯酰胺、甲叉双丙烯酰胺 美国Amresco公司。
1.2 仪器与设备
KQ-250B超声波振荡器 中国上海早盈公司;H1850R低温高速离心机 湘仪离心机仪器有限公司;2720聚合酶链式反应(polymerase chain reaction,PCR)扩增仪 美国Biometra公司;DCodeTMUniversal Mutation Detection System DGGE仪、Gel Doc XR+凝胶成像分析系统 美国Bio-Rad公司。
1.3 方法
1.3.1 玉米表面霉菌基因组DNA提取
采用改良的十六烷基三甲基溴化铵(cetyltrimethyl ammonium bromide,CTAB)法提取玉米表面真菌[18-20],称取25 g玉米置于高压灭菌过的锥形瓶中,加入40 mL灭菌磷酸缓冲盐溶液(phosphate buffer saline,PBS),然后将装有样品的锥形瓶放入超声波振荡器中振荡1 h,将液体取出。其中20 mL放入离心管,4 ℃、2 000 r/min离心5 min。取上清液于新的离心管中,4 ℃、10 000 r/min离心10 min以沉淀菌体。加入65 ℃预热30 min的2% CTAB溶液2 mL,充分混匀,置65 ℃水浴1 h,期间适当摇匀,期间每隔5~10 min混匀一次,取出后,放至室温。之后按照酚-氯仿-异戊醇方法操作进行[17]。
1.3.2 真菌18S rRNA部分片段的扩增
利用DNAman软件分析并选用Ns7F和Ns8R引物对扩增真菌18S rRNA部分序列,带有GC夹的Ns7F:CGCCCGCCGCGCCCCGCGCCCGGCCCGCCGCC CCCGCCCCATAACAGGTCTGTGATGC和Ns8R:GCAGGTTCACCTACGGA[21],PCR产物的大小为353 bp。50 μL反应体系为:2×Pfu Master Mix 25 μL;上游引物GCNs7F 1 μL;下游引物Ns8R 1 μL;模板DNA 1 μL;Mg2+0.2 μL;ddH2O 21.8 μL。真菌18S rRNA-PCR扩增程序为:94 ℃预变性5min; 94 ℃变性1 min,52 ℃退火45 s,72 ℃延伸1 min,35 个循环;72 ℃延伸10 min;4 ℃保存。
1.3.3 扩增产物的DGGE和测序
DGGE使用8%的聚丙烯酰胺凝胶,18S rRNA的变性剂质量分数适合的范围为30%~60% (100%的变性剂中每100 mL中含有42 g尿素)。电泳运行条件:1×TAE电泳缓冲液,85 V电压,60 ℃恒温、恒压电泳12 h。电泳结束后用溴化乙锭染色法对凝胶进行染色0.5 h。将染色后的凝胶用凝胶成像分析系统拍照,获得DGGE图谱。将DGGE图谱上的主要条带分别进行切胶回收,每份回收产物使用不带GC夹子的引物进行PCR扩增反应,扩增产物进行Sanger测序。将测序所得的序列,与GenBank数据库进行BLAST相似性比对,以相似度最高的比对结果作为该电泳条带的物种注释。
1.3.4 霉菌群落多样性分析及统计分析
为分析储藏玉米中霉菌群落的分布规律,本研究对不同年份的样品和粮库中不同空间分布的样品中霉菌群落的多样性进行比较分析。凝胶成像系统获得的DGGE图谱利用Quantity One v4.6.9软件进行图谱整体背景去除、泳道背景去除、条带亮度分析,计算每个样品中各个条带亮度的相对值。将该数据导入PAST软件(v3.15)[22],利用PAST软件的multivariate/non-metric-MDS对数据进行非度量多维尺度(nonmetric multidimensional scaling,NMDS)作图分析,距离参数选择Bray-Curtis dissimilarity;利用PAST软件的multivariate/one-way-ANOSIM对数据进行ANOSIM分析,计算各组之间霉菌群落整体差异的显著性,距离参数选择Bray-Curtis dissimilarity;利用PAST软件的multivariate/Clustering/Neighbourjoining对各样品的霉菌群落进行聚类分析;将每个条带的物种注释和亮度相对值数据导入LEfSe在线分析工具(http://huttenhower.sph.harvard.edu/galaxy)[23],分析不同类型样品中具有显著差异的霉菌类群。
2 结果与分析
2.1 DGGE图谱分析
为了研究储藏玉米中霉菌群落的多样性及时空分布规律,本研究用GC-Ns7F和Ns8R对18S rRNA的部分序列进行PCR扩增,并将扩增产物进行DGGE分析,结果如图1所示。DGGE图谱中条带的亮度强弱表明相应霉菌物种丰度的高低。从DGGE图谱整体上看,不同储藏年限的样品中霉菌群落DGGE条带图谱具有比较明显的差异,储藏1 a的样品(15A~15E)霉菌群落中高亮度条带较多。而储藏2 a的样品(14A~14E)和储藏3 a的样品(13A~13E)霉菌群落中高亮度条带逐渐较少。表明玉米储藏过程中,优势菌群的多样性逐渐减少,这可能是由于玉米储藏过程中营养成分的变化及霉菌间相互竞争所导致的。而与储藏年份相比,粮库中不同空间位置样品的条带图谱比较相似,这可能是由于取样粮库中不同空间的环境条件较为一致。
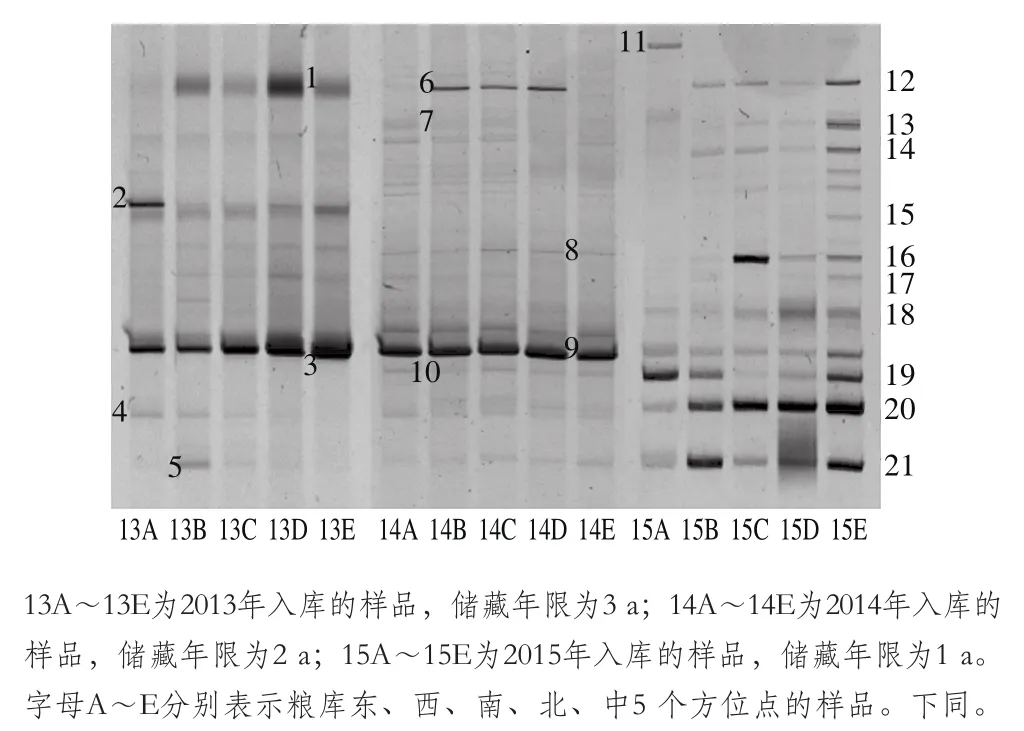
图1 不同年份及粮库不同空间位置的储藏玉米中霉菌群落的DGGE图谱Fig. 1 DGGE pattern of mold communities in stored corn for different years at different spatial locations in the depot
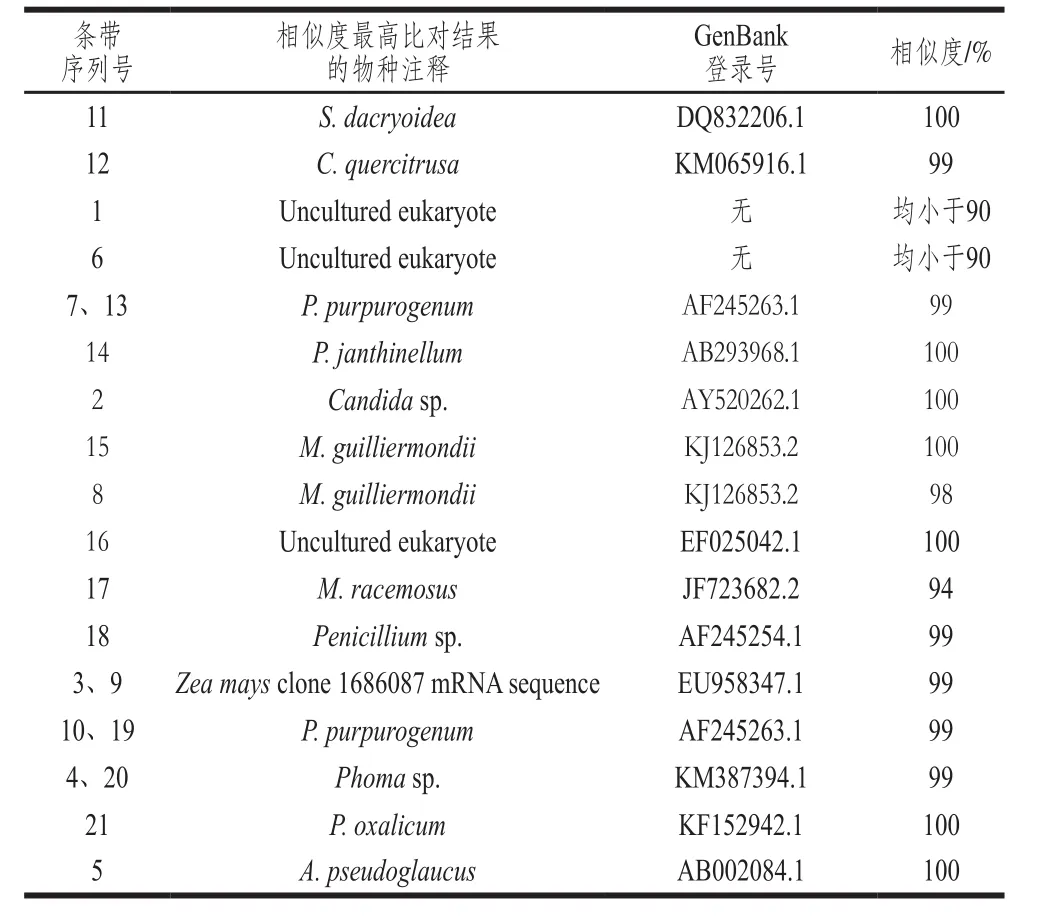
表1 DGGE图谱中的主要条带测序分析Table1 Sequencing of main DGGE bands
对DGGE图谱中的主要条带进行切胶回收、PCR扩增和Sanger测序,并通过BLAST与GenBank数据库比对,结果如表1所示。可以看出储藏3 a和储藏2 a的样品中,亮度最高的条带(DGGE图谱中标注为3和9)物种注释为Zea mays,即来源于玉米的的基因组的扩增产物。这可能是由于储藏年限长导致玉米细胞壁破损,致使霉菌基因组DNA提取结果中混有较多的玉米基因组DNA,由于不同DNA模板在PCR扩增中的竞争性,可能导致储藏2 a的样品和储藏3 a的样品的DGGE图谱中其他霉菌条带亮度较低。因此在后续多样性分析中,将所有样品注释为Z. mays的条带去除,重新计算其他条带的相对丰度。从霉菌种类上来看,此粮库中霉菌污染以青霉为主,如产紫青霉菌Penicillium purpurogenum、微紫青霉菌P. janthinellum、草酸青霉菌P. oxalicum及一株未鉴定到种的青霉物种Penicillium sp.,此外还发现了总状毛霉Mucor racemosus和假灰绿曲霉Aspergillus pseudoglaucus。此外还检测到部分非霉菌的真菌物种,如Sakaguchia dacryoidea、Candida quercitrusa、Meyerozyma guilliermondii。
2.2 储藏玉米中霉菌群落的时空分布规律分析
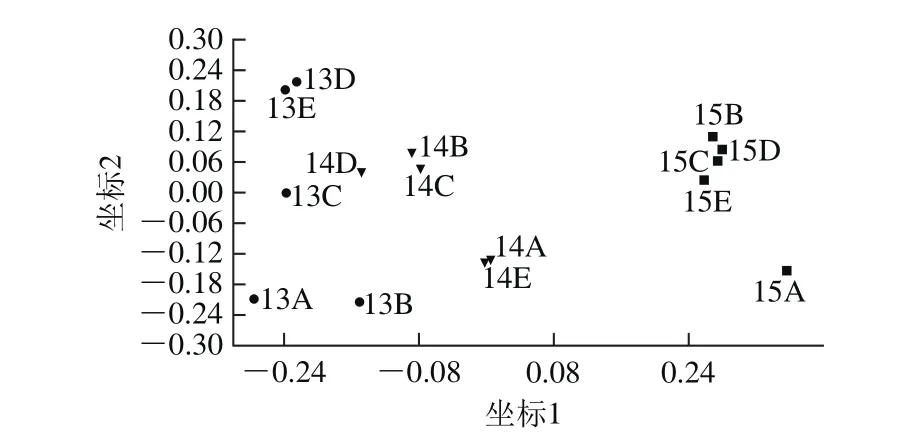
图2 不同储藏玉米样品中霉菌群落DGGE图谱的NMDS作图分析Fig. 2 NMDS plot for DGGE patterns of molds communities from different stored corn samples
为了进一步分析储藏玉米中霉菌群落的时间和空间分布规律,本研究将DGGE图谱中各真菌条带的相对丰度值输入PAST软件进行群落结构分析。如图2所示,NMDS可以在二维坐标中显示各个样品霉菌群落结构的相似性,图上两个样品点之间的距离越近,表明两个样品的霉菌群落结构越相似。从图2可以看出,储藏时间相同的样品中霉菌群落的点距离相对较近,出现聚类现象,而粮库中相同方位的样品的霉菌群落未出现聚类。该NMDS图谱的Stress值为0.137 2,表明该NMDS图谱具有较好的拟合度。利用one-way ANOSIM算法可以对上述聚类现象进行统计学分析以判定显著性,结果表明,当以储藏时间点为各样品分组依据时,储藏1 a的样品与储藏2 a的样品之间、储藏1 a的样品与储藏3 a的样品之间均具有显著性的分离聚类(P<0.05),而储藏2 a的样品与储藏3 a的样品之间的分离聚类不显著(P=0.35)。当以粮库空间位置为各样品的分类依据时,所有组之间分离聚类的结果均为不显著(P>0.05)。
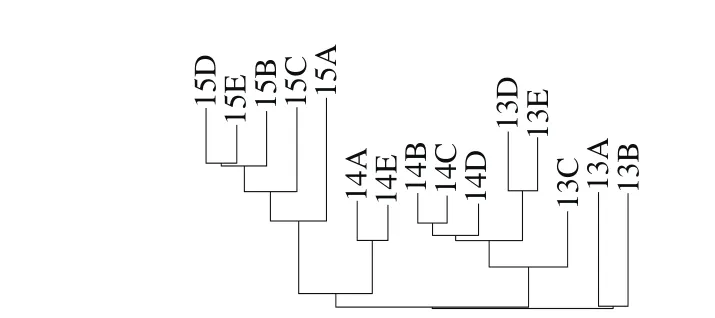
图3 不同储藏玉米样品中霉菌群落树状聚类图Fig. 3 Cluster dendrogram for mold communities from different stored corn samples
如图3所示,相同年份的样品在树状聚类图上的位置很近,表明他们的霉菌群落结构相似度较高。储藏2 a的样品中,有2 个样品在储藏1 a样品的分枝上,另外3 个样品在储藏3 a样品的分枝上,即储藏2 a的样品在树状聚类图上处在储藏1 a和储藏3 a的样品之间,呈现出过渡期的状态。由此可见,储藏玉米中霉菌群落的变化与其储藏时间具有较强的相关性,而与其在粮库中的空间位置相关性较小。
2.3 储藏玉米中霉菌群落与储藏时间的相关性特征
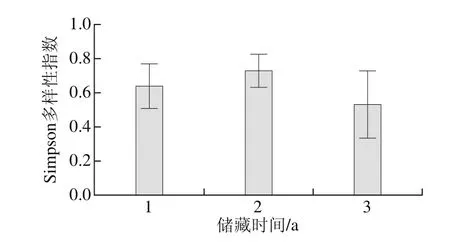
图4 不同储藏玉米样品中霉菌群落Alpha多样性Fig. 4 Alpha diversity of mold communities from different stored corn samples
采用微生物群落多样性研究中最为常用的Simpson指数作为样品内霉菌物种多样性(Alpha多样性)的评估指标,如图4所示。储藏2 a的样品中霉菌群落的物种多样性略高于其他两个储藏时间点的样品,但各组数据之间的差异均不显著。储藏2 a的样品多样性略高可能与其处于过渡期状态有关,过渡期状态的样品可能兼具储藏1 a和储藏3 a的霉菌群落的特征。
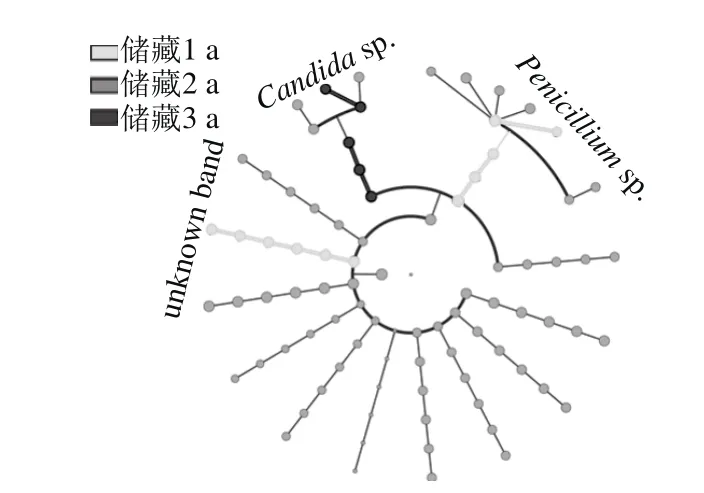
图5 不同储藏时间样品中霉菌群落的LEfSe分析Fig. 5 LEfSe analysis of mold communities from stored corn for different years
利用LEfSe在线分析工具可以分析各储藏时间点的样品的霉菌群落中的特征霉菌。如图5所示,Penicillium sp.及一个未测序的条带所代表的霉菌为储藏1 a样品的特征霉菌菌种,Candida sp.为储藏3 a样品的特征霉菌菌种,而储藏2 a的样品中没有发现特征霉菌菌种,这可能是由于储藏2 a的样品霉菌群落特征处于储藏1 a和储藏3 a样品霉菌群落特征的过渡期,因此该时间点的样品没有独有的霉菌种类。
3 讨 论
本研究团队在之前的报道中曾提出,依据预测微生物学,构建储粮中微生物生长预测模型,可以快速对储粮中微生物的生长情况进行判断,对储粮中病原微生物和腐败微生物的控制有重要作用[8]。霉菌是对储粮危害最为严重的微生物,对人类健康具有多方面的影响。为了建立储粮中霉菌群落生长预测模型,一方面要选择适当模型算法对霉菌群落生长进行模拟,另一方面需要实测的数据,对储粮霉菌群落的规律进行研究,并对各生长预测模型进行评估和优化。从本研究的结果可以看出,在生态环境条件均匀的同一粮仓中,不同空间位置的样品霉菌群落相似度较高,而不同储藏时间的样品霉菌群落具有较大的差异。因此在后续建立霉菌生长预测模型时,应将储藏时间设为主要因素之一,而将在粮库中的空间位置定为次要因素。
在传统的DGGE数据分析中,主要采取树状图聚类分析研究泳道带型的相似性[24-26]。本研究参照了16S rRNA扩增子测序中的数据分析方法[27-30],将NMDS、ANOSIM、Simpson多样性指数、LEfSe等分析引入了DGGE结果分析中,对DGGE数据进行了更加深入全面的研究。这些方法揭示了储藏1 a和储藏3 a的储藏玉米样品中霉菌群落具有较大的差异,而储藏2 a样品的霉菌群落处于过渡期状态,这表明粮库中玉米储藏的第2年可能是霉菌群落控制的关键时期。本研究发现储藏玉米中主要的污染霉菌为青霉,此外还包括毛霉和曲霉,这与孙武长等[3]报道的储粮中危害最普遍的霉菌为曲霉和青霉相一致。
为了建立准确可靠霉菌群落模型,还需对储粮霉菌群落进行更多的基础理论研究和数据收集。例如,不同粮食种类的霉菌群落可能不同,储粮表层和储粮内部的霉菌群落可能不同,不同温度、湿度的粮库中霉菌群落类型可能不同。应用本实验所采用的分子生物学方法,对上述问题进行深入研究,可以得到对储粮中霉菌群落特征的全面认识。
[1] EATON D L, JOHN D. The toxicology of aflatoxins: human health,veterinary, and agricultural significance[J]. Animal Feed Science &Technology, 1997, 68(3/4): 368-369.
[2] SORRENTI V, DI GIACOMO C, ACQUAVIVA R, et al. Toxicity of ochratoxin A and its modulation by antioxidants: a review[J]. Toxins,2013, 5(10): 1742-1766. DOI:10.3390/toxins5101742.
[3] 孙武长, 刘桂华, 杨红, 等. 粮食中真菌及真菌毒素污染调查[J]. 中国公共卫生, 2005, 21(12): 1532.
[4] 周玉庭, 任佳丽, 张紫莺. 粮食中霉菌污染检测方法现状及发展趋势[J]. 食品安全质量检测学报, 2016(1): 244-250.
[5] 沈飞, 吴启芳, 刘兵, 等. 粮食真菌毒素污染的无损检测方法研究进展[J]. 食品安全质量检测学报, 2014(8): 2372-2377.
[6] 成博伦, 赵玉濛, 胡锦涛, 等. 几种常见真菌毒素脱毒方法的研究进展[J]. 基因组学与应用生物学, 2015, 34(11): 2338-2344.DOI:10.13417/j.gab.034.002338.
[7] 王长才, 葛万军, 冼子君, 等. 真菌毒素生物脱毒机理的研究进展[J].饲料与畜牧: 新饲料, 2011(8): 41-43.
[8] 岳晓禹, 张恒业, 辛婷, 等. 储粮预测微生物模型的研究进展[J]. 中国粮油学报, 2012, 27(5): 118-123.
[9] 钟雪美, 王建有. 不同储粮方式的霉菌动态变化及空间格局研究[J].陕西农业科学, 1994(4): 7-9.
[10] 李听听, 陈伟, 李广富, 等. 不同储藏条件下玉米中霉菌对黄曲霉毒素B1的影响[J]. 食品与发酵工业, 2014, 40(6): 211-215. DOI:10.13995/j.cnki.11-1802/ts.2014.06.035.
[11] 吴红萍, 翟世博, 杜晨辉, 等. 海南省储粮稻谷的霉菌多样性分析[J].贵州农业科学, 2015, 43(4): 138-141.
[12] XU W. Functional nucleic acids detection in food safety[M]. Singapore:Springer Nature, 2016: 443-467. DOI:10.1007/978-981-10-1618-9.
[13] MUYZER G, SMALLA K. Application of denaturing gradient gel electrophoresis (DGGE) and temperature gradient gel electrophoresis(TGGE) in microbial ecology[J]. Antonie van Leeuwenhoek, 1998, 73(1):127-141.
[14] 李秀秀, 曾维伟, 陆兆新, 等. 鲫鱼贮藏过程中微生物菌相PCRDGGE分析及其防腐保鲜[J]. 食品科学, 2017, 38(5): 274-280.DOI:10.7506/spkx1002-6630-201705045.
[15] 施荷, 胡铁军, 秦凤贤, 等. 真空包装冷却鹿肉贮藏过程中的菌相变化[J]. 肉类研究, 2015, 29(4): 15-19.
[16] 赵飞, 肖莉莉, 张昭寰, 等. 基于PMA-PCR-DGGE对检测生鲜南美白对虾在贮藏过程中微生物多样性方法的优化[C]. 大连: 中国食品科学技术学会第十二届年会暨第八届中美食品业高层论坛, 2015:251-252.
[17] 李听听, 陈伟, 李广富, 等. 玉米储藏霉菌类群及玉米赤霉烯酮含量的研究[J]. 中国粮油学报, 2015, 30(12): 92-97.
[18] MELO S C, PUNGARTNIK C, CASCARDO J C, et al. Rapid and efficient protocol for DNA extraction and molecular identification of the basidiomycete Crinipellis perniciosa[J]. Genetics and Molecular Research, 2006, 5(4): 851-855.
[19] TAPIA-TUSSELL R, LAPPE P, ULLOA M, et al. A rapid and simple method for DNA extraction from yeasts and fungi isolated from Agave fourcroydes[J]. Molecular Biotechnology, 2006, 33(1): 67-70.
[20] 吴发红, 黄东益, 黄小龙, 等. 几种真菌DNA提取方法的比较[J]. 中国农学通报, 2009, 25(8): 62-64.
[21] 李听听. 玉米和小麦储藏中真菌多样性及真菌毒素的研究[D].泰安: 山东农业大学, 2015: 17-34.
[22] HAMMER Ø, HARPER D A T, RYAN P D. PAST: paleontological statistics software package for education and data analysis[J].Palaeontologia Electronica, 2001, 4(1): 1-9.
[23] SEGATA N, IZARD J, WALDRON L, et al. Metagenomic biomarker discovery and explanation[J]. Genome Biology, 2011, 12(6): R60.DOI:10.1186/gb-2011-12-6-r60.
[24] 王若宇, 杜丽平, 刘非, 等. PCR-DGGE分析西藏雪莲菌细菌多样性[J]. 现代食品科技, 2017, 33(2): 83-88. DOI:10.13982/j.mfst.1673-9078.2017.2.013.
[25] 邹欣涛, 万幸, 张韧, 等. 基于PCR-DGGE技术分析白木香结香前后内生真菌的变化[J]. 中药新药与临床药理, 2015(2): 221-225.DOI:10.19378/j.issn.1003-9783.2017.03.024.
[26] 柳尧波, 傅建功, 任素芳, 等. 应用PCR-DGGE分析发酵床养殖仔猪消化道菌群多样性及其演替[J]. 家畜生态学报, 2017, 38(3): 14-18.
[27] TIAN L F, CHEN S Y, LIU H Y, et al. In vivo effects of pichia pastorisexpressed antimicrobial peptide hepcidin on the community composition and metabolism gut microbiota of rats[J]. PLoS ONE, 2016, 11(10):e0164771. DOI:10.1371/journal.pone.0164771.
[28] GUO M Z, BAO Q, CHEN S Y, et al. Effects of neutrophils peptide-1 transgenic Chlorella ellipsoidea on the gut microbiota of male Sprague-Dawley rats, as revealed by high-throughput 16S rRNA sequencing[J].World Journal of Microbiology and Biotechnology, 2016, 32(3): 1-7.DOI:10.1007/s11274-015-1994-z.
[29] ZHANG X, ZHAO Y F, ZHANG M H, et al. Structural changes of gut microbiota during berberine-mediated prevention of obesity and insulin resistance in high-fat diet-fed rats[J]. PLoS ONE, 2012, 7(8): e42529.DOI:10.1371/journal.pone.0042529.
[30] LOOFT T, JOHNSON T A, ALLEN H K, et al. In-feed antibiotic effects on the swine intestinal microbiome[J]. Proceedings of the National Academy of Sciences, 2012, 109(5): 1691-1696. DOI: 10.1073/pnas.1120238109.
