一株尖孢镰刀菌细胞色素P450氧化酶基因的克隆及生物信息学分析
2018-02-05单丽红姜冬冬赵沙沙黄佳佳刘宏民
单丽红,姜冬冬,赵沙沙,黄佳佳,朱 丽,刘宏民
1)郑州大学药学院 郑州 450001 2)郑州大学新药研究开发中心 郑州 450001
屈螺酮及其类似物是一种高效、低毒、无不良反应的新一代甾体类避孕药,最早是由Schering公司于2000年开发的第4代口服避孕药[1],具有抗盐皮质激素和抗雄激素的作用,具有广阔的市场前景[2]。课题组筛选到一株对去氢表雄酮(dihydroepiandrosterone,DHEA)具有羟基化作用的霉菌,经分子生物学的方法鉴定为尖孢镰刀菌,暂编号为LZ22,目前国内外均未见其用于转化DHEA获得3β,7α,15α-三羟基雄甾-5-烯-17-酮的报道。在微生物作用下甾体发生的羟基化离不开细胞色素P450氧化酶(CYP)。CYP为一类亚铁血红素-硫醇盐蛋白超家族,因其还原态与一氧化碳结合后在450 nm处的特征吸收峰而得名[3]。CYP一直是医药学关注的重点,CYP在甾体类生物转化也成绩斐然[4]。对羟化酶重组菌的研究[5],一般集中在CYP基因的研究和改造。本课题组通过基因工程的手段,筛选到参与该催化过程的关键酶基因CYP6003A1。为了更好地阐述尖孢镰刀菌LZ22菌株7α,15α-羟化酶系统的分子催化机制,本研究通过数据库测序信息,采用PCR克隆了尖孢镰刀菌LZ22菌株的CYP基因,并利用生物信息学对其产物的结构特征进行了分析。
1 材料与方法
1.1菌株及试剂尖孢镰刀菌LZ22菌株(郑州大学药物研究院生物转化实验室自筛和保藏),Trizol试剂(北京博迈德生物公司),Taq DNA聚合酶(TaKaRa公司),PCR纯化试剂盒(康宁生命科学有限公司),cDNA合成试剂盒(TaKaRa公司)。培养基:葡萄糖3 kg/L,K2PO40.09 kg/L,蛋白胨1.2 kg/L,酵母膏0.1 kg/L。
1.2菌株培养30 ℃、190 r/min摇床培养菌株48 h后加入质量分数1% DHEA进行诱导,继续培养48 h。
1.3RNA提取、cDNA合成与PCR扩增Trizol法提取以DHEA为底物诱导48 h的尖孢镰刀菌的总RNA,反转录合成cDNA,取1 μL cDNA作为PCR扩增的模板。引物,上游5’-CGAAGAGAATGATT GAGAC-3’,下游5’-CGAAGGAAGAATGACAGAA-3’。PCR反应体系25 μL,包括上下游引物各1 μL,cDNA 1 μL,Taq DNA聚合酶12.5 μL ,水9.5 μL。反应条件:94 ℃预变性5 min;94 ℃ 30 s,60 ℃ 30 s,72 ℃ 30 s,35个循环。
1.4CYP6003A1基因的生物信息学分析通过软件拼接以及NCBI在线分析,得到CYP6003A1 cDNA全长以及对应的氨基酸序列。委托苏州金唯智公司测序,将CYP6003A1基因的氨基酸序列通过NCBI上的BLAST进行在线比对[6]。将序列数据以fasta格式形成一个文件后,用ClustalX2进行全序列自动比对。输出格式为Clustal格式(.aln);利用MEGA 6.0进行系统发育分析。采用MEGA 6.0的邻接法建树。利用ExPASy在线蛋白质分析软件ProtParam分析CYP6003A1基因编码的氨基酸;利用PROFsec网站预测CYP6003A1蛋白的二级结构;利用丹麦理工大学生物序列分析中心提供的蛋白质三级结构预测服务器CPHmodels运用神经网络进行同源建模预测蛋白质三级结构,并在Pymol中进行三维结构显示,创建高品质的蛋白质三维结构。
2 结果
2.1尖孢镰刀菌CYP基因的克隆从转录组数据库中发现一个编码CYP的基因,该基因的开放阅读框为2 073 bp,预测编码691个氨基酸的多肽。为克隆其完整的阅读框架,利用上述总RNA,通过反转录合成cDNA,并设计了一对特异性引物,扩增出大约3 500 bp的片段,电泳结果见图1,送苏州金唯智公司全长测序,结果表明基因序列大小为3 459 bp。
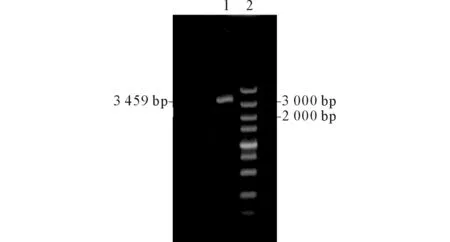
1:CYP6003A1全长基因片段;2:DNA Marker图1 尖孢镰刀菌CYP基因PCR产物凝胶电泳图
2.2尖孢镰刀菌CYP基因的序列分析将测序结果在NCBI数据库中进行BLAST搜索,发现尖孢镰刀菌CYP与下列真菌的CYP基因具有很高的同源性:尖孢镰刀菌EWZ38580.1、轮状镰刀霉菌EWG49946.1、燕麦镰刀菌KIL95759.1、梨孢镰刀菌OBS15561.1、禾生炭疽菌XP_008091343.1、弯颈孢霉KND86690.1、尖端赛多孢子菌XP_016646336.1、厚垣普奇尼亚菌OAQ60718.1、荚膜组织胞浆菌EEH05495.1;其中与尖孢镰刀菌氨基酸序列一致性最高,其高度保守区域见图2。BLAST比对结果显示,与CYP6003A1相似度前10个基因有3个是CYP基因,其余均是与CYP基因相关的基因(图3、4)。
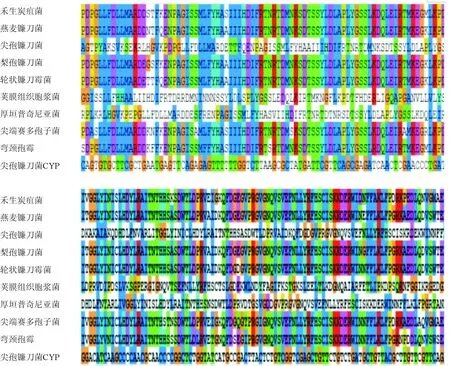
图2 尖孢镰刀菌CYP基因与其他真菌的CYP基因氨基酸序列的多重比对结果
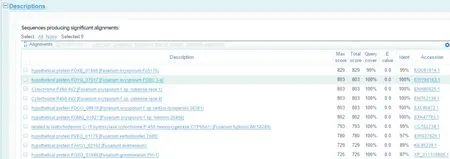
图3 CYP6003A1氨基酸序列BLAST比对结果
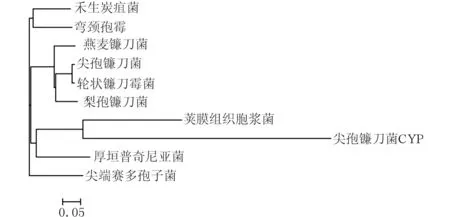
图4 尖孢镰刀菌CYP基因 与其他9个真菌基因之间的亲缘关系
2.3CYP6003A1氨基酸组成分析CYP6003A1蛋白氨基酸组成见表1。CYP6003A1等电点为6.78,蛋白质不稳定系数为34.47,属于稳定蛋白质。利用PROFsec网站预测的CYP6003A1蛋白的二级结构见图5。
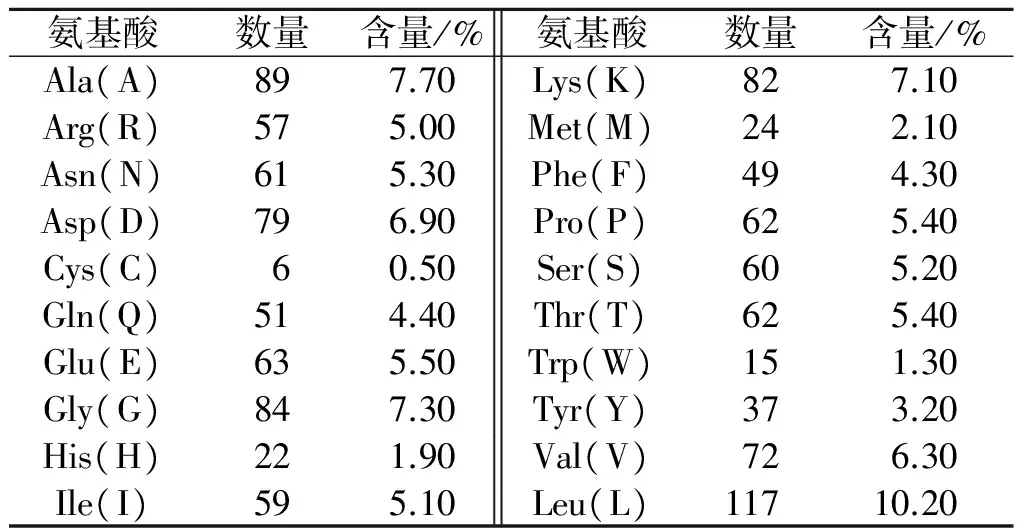
表1 CYP6003A1蛋白的氨基酸组成分析
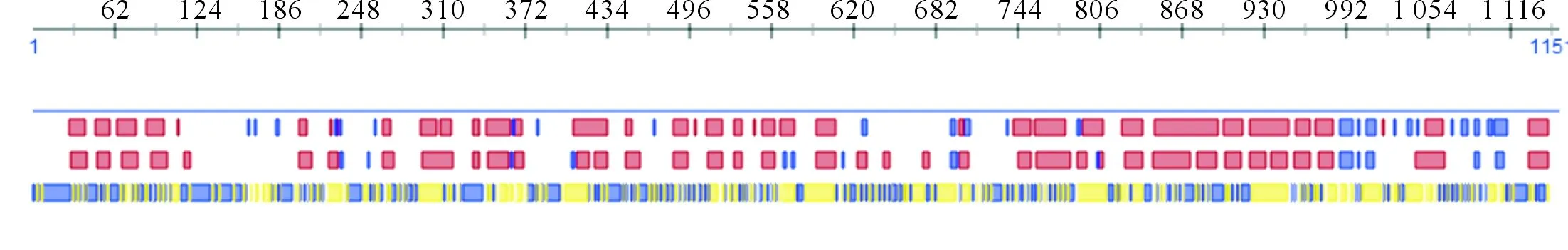
褐色:α-螺旋,38.31%;蓝色:β-折叠,3.91%;白色:β-转角,57.78%图5 CYP6003A1蛋白二级结构预测分析
2.4CYP6003A1蛋白结构模拟对CYP6003A1蛋白三级结构进行分析发现,该基因包含CYP典型功能区:离子对结合区和血红素结合位点,见图6。
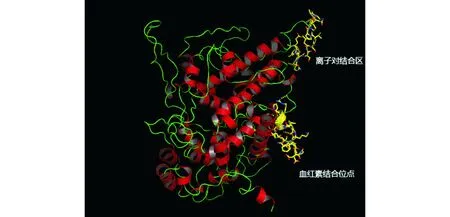
图6 CYP6003A1蛋白三级结构示意图
3 讨论
本研究用Trizol法提取以DHEA为底物诱导48 h的尖孢镰刀LZ22菌株的总RNA,通过PCR扩增克隆CYP基因并进行基因测序;运用生物信息学分析得到的cDNA序列[5],成功扩增出全长3 459 bp的cDNA片段,测序后将序列提交至NCBI进行BLAST,发现CYP6003A1相似度前10个基因有3个是CYP基因,其余均是与CYP基因相关的基因。生物信息学分析结果显示,该基因片段编码691个氨基酸的多肽,并对CYP6003A1蛋白结构进行Pymol分子模拟,将CYP6003A1与其他真菌该蛋白质进行了同源性及进化树分析。
屈螺酮是目前最好的第4代口服避孕药物,具有重要的商业价值[2]。课题组筛选到一株能高效转化DHEA 为7α,15α-二羟基DHEA的菌株,该产物可作为重要中间体合成屈螺酮,并且该路线简便易行,收率较高,有望进一步降低屈螺酮的制备成本。
CYP在生物转化合成屈螺酮关键中间体7α,15α-二羟基DHEA阶段发挥重要作用[7]。CYP是药物生物转化反应的主要代谢酶。越来越多的CYP被发现参与了多种化合物的生物转化[8],尤其是后修饰过程,使得CYP在微生物新药发掘领域脱颖而出[9]。
本研究通过克隆获得尖孢镰刀菌LZ22菌株CYP6003A1基因,并对其进行生物信息学分析,其结果一方面可为此类重组菌的构建奠定基础,另一方面也可进一步丰富甾体羟化酶的研究内容,同时也可为解决甾体药物羟基化反应收率低下的瓶颈问题提供新的思路和方法。
[1] 韩广甸,刘宏斌.新型女用口服避孕药屈螺酮的合成进展[J].中国药物化学杂志,2010,20(2):146
[2] 史建萍,孙亚楠,刘秀萍,等.屈螺酮炔雌醇预防育龄妇女宫腔镜下子宫内膜息肉切除术后复发的临床研究[J].解放军医药杂志,2015,27(11):106
[3] 任彭,刘兆平.细胞色素P450研究概况及其应用[J].食品与药品,2006,8(10):8
[4] SWIZDOR A,KOEK T,PANEK A,et al.Microbial Baeyer-Villiger oxidation of steroidal ketones using Beauveria bassiana:presence of an 11α-hydroxyl group essential to generation of D-homo lactones[J].Biochim Biophys Acta,2011,1811(4):253
[5] 赵位,王吴优,程建国,等.林麝肺炎克雷伯氏菌的分离鉴定及其kp05372基因的生物信息学分析[J].西北农林科技大学学报(自然科学版),2017,45(12):23
[6] 张霞,王配,霍金龙,等.版纳微型猪近交系BARD1基因克隆、mRNA表达及蛋白质功能生物信息学分析[J/OL].吉林农业大学学报.http://www.cnki.net/kcms/detail/22.1100.S.20170103.0924.001.html.DOI:10.13327/j.jjlau.2017.3291.
[7] SHAH SA,SULTAN S,HASSAN NB,et al.Biotransformation of 17α-ethynyl substituted steroidal drugs with microbial and plant cell cultures: a review[J].Steroids,2013,78(14):1312
[8] 年华,马明华,徐玲玲,等.细胞色素P450酶与药物代谢的研究进展与评价[J].中国医院用药评价与分析,2010,10(11):964
[9] 李众,张伟,李盛英.细胞色素P450酶与微生物药物创制[J].微生物学报,2016,56(3):496
