26个香菇菌株的遗传多样性分析
2017-12-26任海霞宫志远蒋志琴陆玉荣任鹏飞万鲁长
任海霞+宫志远+蒋志琴+陆玉荣+任鹏飞+万鲁长

摘要:香菇(Lentinus edodes)是世界上著名的食用菌之一,其菌株种类较多,遗传背景较为复杂。本研究利用ISSR-PCR技术,对26个香菇菌株进行DNA指纹分析,并使用NTSYS-PC软件对其遗传差异性进行聚类分析。结果表明,从106个随机引物中共筛选到9个引物,利用这9个引物对26个香菇菌株的DNA进行ISSR-PCR,检测到124个较为清晰的扩增位点,其扩增片段DNA分子量大小主要介于0.2~4.5 kb之间。通过聚类分析,发现相似水平在55%时出现种系分离;相似水平65%时,26个菌株聚为5个群组。本研究为山东省香菇遗传信息库的建立和遗传育种工作奠定了一定的基础。
关键词:香菇;ISSR-PCR;聚类分析;遗传多样性
中图分类号:S646.1+20.1文献标识号:A文章编号:1001-4942(2017)11-0020-05
Analysis of Genetic Diversity of 26 Lentinus edodes strains
Ren Haixia1, Gong Zhiyuan1, Jiang Zhiqin2, Lu Yurong2, Ren Pengfei1, Wan Luzhang1
(1.Institute of Agriculture Resources and Environment, Shandong Academy of Agricultural Sciences/
Key Laboratory of Wastes Matrix Utilization, Ministry of Agriculture, Jinan 250100,China;
2. Bazhou Science and Technology Development Exchange Center of Xinjiang, Bazhou 841000,China)
AbstractLentinus edodes is one of the most famous edible fungus species in the world. It has various strains and complex genetic background. In this study, we conducted DNA fingerprinting of 26 familiar Lentinus edodes strains by ISSR-PCR, and analyzed their genetic diversity by cluster analysis of NTSYS-PC software. The results showed that, 9 primers were selected from 106 random primers, which were used to conduct DNA ISSR-PCR in 26 Lentinus edodes strains, and 124 relatively clear sites were amplified with the DNA molecular weight ranging from 0.2 kb to 4.5 kb. Cluster analysis results showed that, species separation was appeared at the similarity level of 55%, and the 26 strains were mainly divided into five groups at the similarity level of 65%. This study laid certain foundations for establishing the genetic information library and genetic breeding of Lentinus edodes in Shandong Province.
KeywordsLentinus edodes;ISSR-PCR;Cluster analysis;Genetic diversity
香菇(Lentinus edodes)屬于真菌门,担子菌纲,伞蘑目,皮伞科,富含蛋白质、多种人体必需氨基酸、糖类、矿物质、维生素和脂类等营养物质,同时含有香菇多糖、香菇嘌呤、双链核糖核酸等免疫活性物质,具有极高的营养价值[1,2]。香菇是世界上第二大食用菌栽培菌种,其产量仅次于双孢菇。中国香菇栽培生产发展迅速,近年来在香菇的产销量上一直位居世界第一位[3]。随着产销量的增长,国内外对其栽培、育种、保鲜加工等方面高度重视,而香菇菌种是香菇生产中最重要的基础,因此,引进和选育优质、高产、抗逆性强的菌株有助于香菇产业的快速发展[4]。但是目前我国香菇引种比较混乱,菌株同名异物和同物异名现象频发,故对香菇菌株进行鉴定并确立其DNA指纹图谱将有助于从分子生物学角度准确识别菌种、了解其亲缘关系,从而有效利用不同香菇品种的优势进行育种,促进生产的发展[5]。
ISSR(inter-simple sequence repeat,简单序列重复区间扩增多态性)技术是在SSR和PCR基础上发展起来的一项技术,由Zietkiewicz等于1994年首次在植物研究中使用[6] ,由于操作简单、成本低、快速、灵敏、检测多态性能力强等特点而倍受青睐[7]。近几年该技术已逐步开始在食用菌种质资源研究、菌株鉴别和菌株遗传多样性分析等方面进行应用[8,9]。本研究采用ISSR-PCR方法,从106个随机引物中筛选出9个引物,对本实验室保存的26个香菇菌株进行遗传多样性分析,构建遗传相关聚类图,明确26个香菇菌株之间的亲缘关系,以便为香菇育种积累基础数据。endprint
1材料与方法
1.1供试菌株
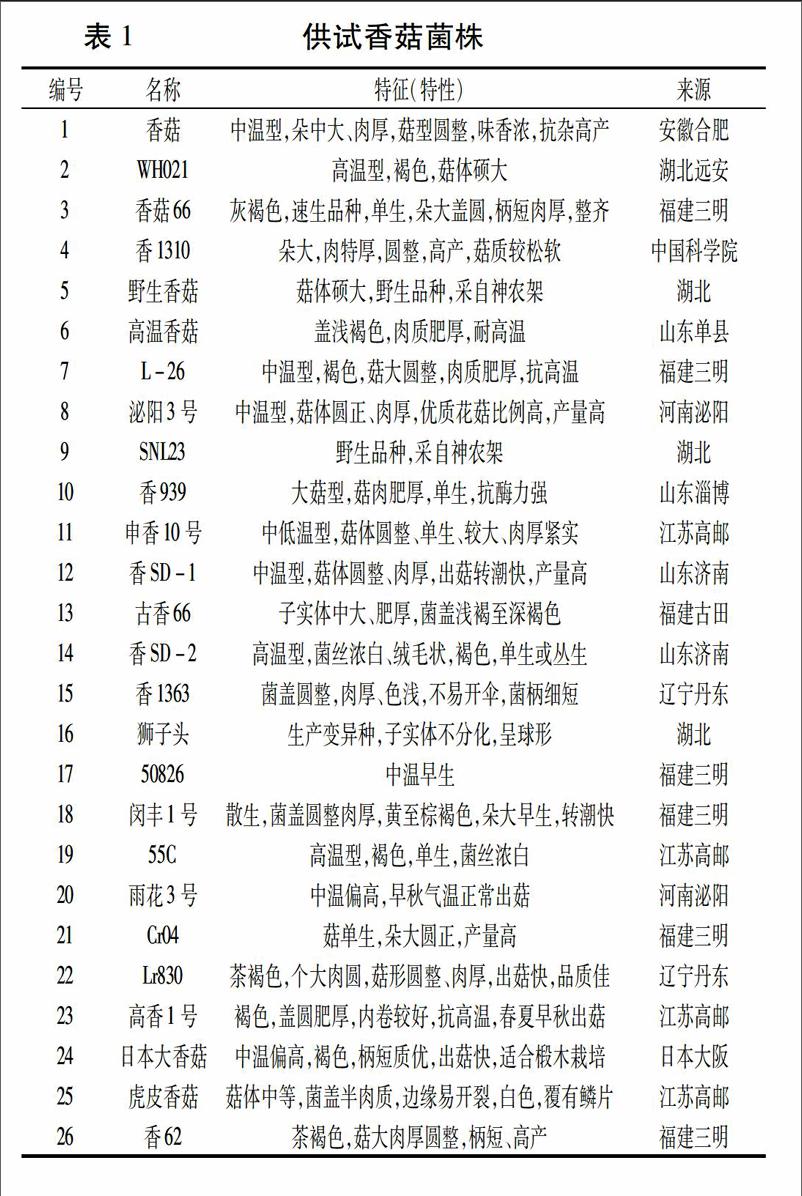
本试验所用的26个香菇菌株均由山东省农业科学院农业资源与环境研究所提供。各供试菌株的编号、名称、特征及来源见表1。
1.2培养基
PDA固体培养基:马铃薯200 g,葡萄糖20 g,KH2PO4 1 g,MgSO4·7H2O 1 g,蛋白胨3 g,酵母浸粉2 g,琼脂21 g,加水定容至1 000 mL,121℃灭菌30 min。
液体培养基:葡萄糖40 g,蛋白胨4 g,KH2PO4 3 g,MgSO4·7H2O 1.5 g,水1 000 mL,pH值7.0,121℃灭菌30 min。
1.3引物与试剂
试验所用引物序列来自UBC Primer Set #9(Microsatellite),均由上海生工生物工程技术服务有限公司合成。DNA Marker、dNTPs、Taq酶以及EB等其它试剂均为天根生化科技有限公司产品,皆为分析纯。
1.4香菇菌丝体培养及收集
挑取保藏香菇菌种接种于PDA斜面培养基上,25℃活化培养7~10天,再转接到液体培养基中,在25℃、150 r/min恒温摇床上培养12天。将培养液以3 000 r/min离心,弃上清液,用无菌蒸馏水洗涤菌丝3次,滤纸吸干残余水分,将所得菌丝体在-20℃保存备用。
1.5香菇菌丝DNA的提取
DNA提取采用改进的SDS法[10]。
1.6ISSR-PCR反应
反应体系为20 μL,其中含有1×PCR Buffer,1.2 mmol/L Mg2+,0.6 μmol/L引物,200 μmol/L dNTPs,模板DNA 50 ng,0.06 U/μL Taq 酶。
ISSR-PCR反应程序为94℃预变性3 min;94℃变性45 s,50℃退火50 s,72℃延伸2 min,35个循环;最后72℃延伸7 min,停止反应。4℃保存反应液备用。
1.7ISSR-PCR产物电泳检测
用1×TAE缓冲液配制浓度为1.2%的琼脂糖溶液,ISSR-PCR反应结束后,PCR管中各加4 μL溴酚蓝上样缓冲液,取13~15 μL至上样孔,以4~5 V/cm进行电泳。电泳结束后,用5 mg/mL EB溶液对琼脂糖凝胶染色20 min,再用去离子水脱色漂洗10 min,于凝胶紫外成像系统上观察、拍照[11]。
1.8ISSR-PCR产物电泳结果统计及处理分析
将所得的琼脂糖凝胶电泳图谱用Quantity One软件进行处理,对各泳道的DNA条带进行分子量标记并输出,以不同分子量DNA片段的条带有无作为多态性状,有记为1,无记为0,DNA凝胶电泳图谱即转换为[0,1]数字矩阵。然后利用NTSYS-PC软件进行聚类分析,构建26个香菇菌株的亲缘关系树状图。
2结果与分析
2.1ISSR-PCR的引物筛选
一般来说,ISSR-PCR扩增所用引物是随机且通用的,但是每个引物并非适合所有物种。由于引物是决定ISSR-PCR扩增成败及其特异性的关键因素,因此对于某具体物种来说,应尽可能地对多个引物进行筛选,以筛选出扩增多态性水平高并且重复性好的引物。本研究首先从所保存的菌株中随机选取香1310(菌株编号为4)和申香10号(菌株编号为11)2个菌株作为研究对象,以其DNA为模板,用随机选取的106个引物进行PCR扩增,筛选到9個多态性高、重复性好、带型清晰的引物,引物序列见表2。
2.226个香菇菌株ISSR-PCR扩增结果
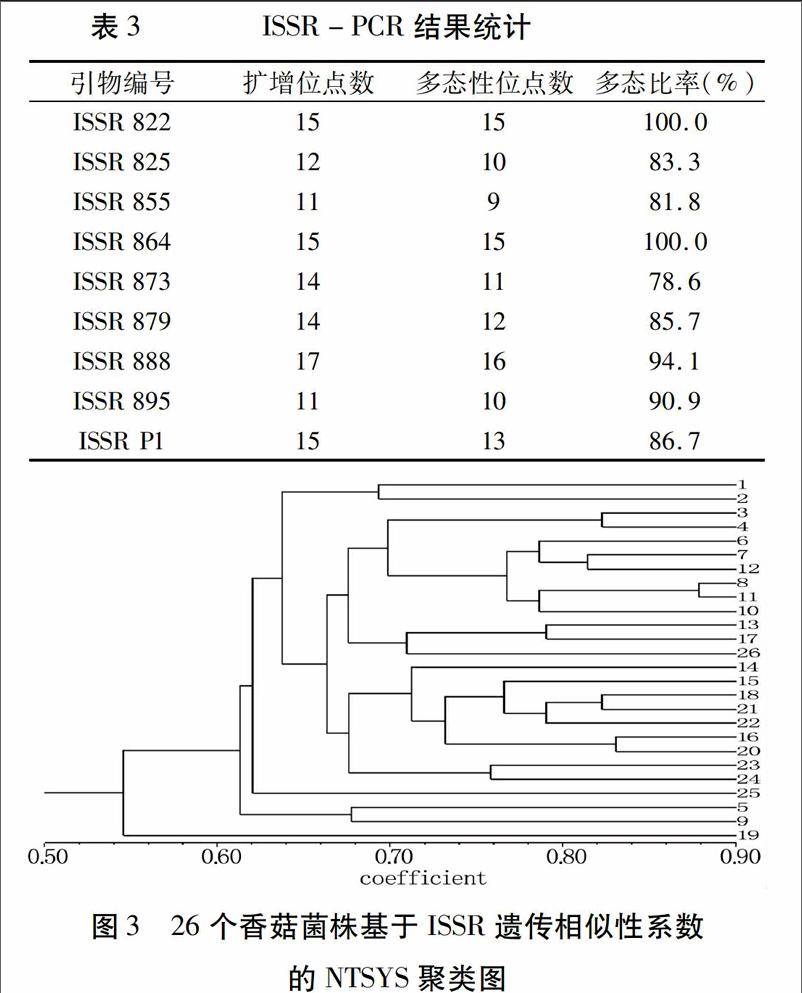
利用筛选到的9个引物对26个香菇菌株的DNA进行ISSR-PCR,共检测到124个扩增位点,其扩增片段DNA分子量大小主要介于0.2~4.5 kb之间。每个引物扩增的位点数介于11~17个,多态性比率介于78.6%~100.0%,9个引物扩增出的124个位点中,有111个为多态性,扩增位点平均多态性比率为89.5%(表3)。由此可见,本文所筛选的引物对香菇菌株的DNA扩增多态性丰富、稳定,可以用于香菇菌株间的亲缘关系分析,构建其亲缘关系聚类树状图。引物ISSR 873和ISSR 895对26个香菇菌株的ISSR-PCR扩增图谱见图1和图2。
M:Marker Ⅲ;B:空白;1~26:香菇菌株(见表1),下图同。
2.326个香菇菌株的聚类分析
利用NTSYS-PC软件对得到的26个香菇菌株的相似性[0,1]数字矩阵进行非加权配对算术平均法(UPMGA)聚类分析,得到这26个菌株的亲缘关系树状图,见图3。可以看出,供试的26个香菇菌株相似性在0.55~0.88之间。在0.65的相似水平上可以将26个香菇菌株分为五组,其中55C(菌株编号为19)、虎皮香菇(菌株编号为25)分别自成一组,野生香菇(菌株编号为5)和SNL23(菌株编号为9)聚成一组,香菇(菌株编号为1)和WH021(菌株编号为2)聚成一组,其余20个香菇菌株聚为一组。55C是一个野生种与生产种的杂交菌株,与其它香菇亲缘关系比较远,故自成一组;虎皮香菇与香菇同属香菇属,但是不同种,故也自成一组;野生香菇(菌株编号为5)和SNL23(菌株编号为9)都是采自神农架的野生香菇,故聚成一组;香菇(菌株编号为1)和WH021(菌株编号为2)都是中高温型、子实体较大的香菇,且与其它品种亲缘关系较远,故聚成一组;其余20个香菇菌株主要是常用的栽培菌株,故聚为一个大组。
3讨论与结论
分子生物学技术的发展为在分子水平上研究菌种的亲缘关系提供了技术保证。近年来,DNA技术已被广泛用于真菌的系统发育研究,对香菇品种进行菌株鉴定可有助于从分子生物学角度准确地识别菌种[12]。目前用于亲缘关系分析的DNA技术有很多,而ISSR技术具有操作简单、成本低、条件稳定、快速、灵敏、检测多态能力强、所需DNA模板量少等优点,目前已广泛应用于植物遗传多样性和品种鉴定的研究[13],在食用菌中的应用也日益广泛。本研究利用ISSR-PCR技术对26个香菇菌株进行亲缘关系聚类分析,结果显示,26个香菇菌株的多态性较为丰富,在65%的相似水平上,可以分为5大类:55C、虎皮香菇分别自成一类,野生香菇和SNL23同是神农架的野生采集品种,聚成一类,香菇和WH021聚成一类,其余20个香菇菌株聚为一类。说明野生香菇和栽培香菇具有较远的亲缘关系,各香菇菌株间的亲缘关系与其产地并无太大相关性。
本研究初步明确了本实验室保存的26个香菇菌株亲缘关系。但是由于香菇的栽培菌株特别多,栽培面积大,种植户、菌种厂相互引种的现象特别多,所以遗传背景比较复杂,仅采用一种分子生物学方法对菌株进行分析还远不能满足香菇遗传信息库和香菇遗传育种工作的技术需求,今后还需要采用一些必要的辅助方法如菌丝形态、子实体形态以及出菇条件等,对更多的菌株进行系统分析和研究[14]。
参考文献:
[1]江娟.八个香菇品种酯酶同工酶的研究[J].中国食用菌,2003,22(6):45-46.
[2]陈慧婵,裴斐,杨文建,等.金针菇、香菇和蛹虫草对小鼠体内抗氧化酶活性的影响[J].食品科学,2014,35(1):219-223.
[3]江洪涛,李伟芳.香菇出口生产技术综述[J].中国食用菌,2004,24(2):3-5,53.
[4]谢雪迎,任鹏飞,朱常香,等.24个香菇菌株的鉴定与分类研究[J].山东农业科学,2009(12):36-39.
[5]卓英,谭琦,陈明杰,等.香菇主要栽培菌株遗传多样性的AFLP分析[J].菌物学报, 2006, 25(2):203-210.(下转第44页)山 东 农 业 科 学2017,49(11):24~28Shandong Agricultural Sciences山 东 农 业 科 学第49卷第11期王宗文,等:不同棉花品种适宜机采性状比较研究DOI:10.14083/j.issn.1001-4942.2017.11.004
收稿日期:2017-06-18endprint
