柿属植物cpDNA分子标记开发及遗传变异研究
2017-12-19傅建敏索玉静张嘉嘉谭晓风
傅建敏 ,索玉静 ,张嘉嘉 ,谭晓风
(1.中南林业科技大学,湖南 长沙 410004;2. 中国林业科学研究院经济林研究开发中心,河南 郑州 450003;3. 国家林业局泡桐研究开发中心,河南 郑州 450003)
柿属植物cpDNA分子标记开发及遗传变异研究
傅建敏1,2,3,索玉静2,3,张嘉嘉2,3,谭晓风1
(1.中南林业科技大学,湖南 长沙 410004;2. 中国林业科学研究院经济林研究开发中心,河南 郑州 450003;3. 国家林业局泡桐研究开发中心,河南 郑州 450003)
基于柿属植物Diospyros spp.叶绿体全基因组测序结果,开发cpDNA分子标记,并利用16份柿属材料对其在种间及柿种内的可行性进行验证。结果表明: 4个cpDNA标记在16份柿属植物中分别检测到31、27、7、25个变异位点,简约信息位点分别为14、5、2、13个。分别对4个标记进行聚类分析表明: CP1标记可以将11份柿品种与5个柿的近缘种完全分离,且柿与油柿距离最近,表明该标记适用于柿属植物种间资源鉴定和遗传变异分析,但不适用于柿种内;CP2、CP3、CP4标记均不能将柿品种与柿近缘种完全区分,但在11份柿品种间均检测到了多个碱基突变、插入和缺失位点,且多数柿品种发生变异的位点不同,只有‘富有’和‘中柿一号’两个品种未检测到特异的变异位点,故可以考虑用于柿种内的遗传变异研究。
柿属植物;近缘种;cpDNA分子标记;遗传变异
柿属植物Diospyros spp.在长期栽培过程中分化出许多宝贵资源,急需开发出特异的分子标记对其种质资源及遗传多样性进行研究和评价。柿D.kaki Thunb.,作为柿属植物主要的果树栽培种,具有丰富的种质资源[1];但在长期的引种、杂交、嫁接等驯化过程中,很多品种出现了同物异名或同名异物等种质资源混杂不清的现象,且大多数品种间的亲缘关系也暂不清楚,为柿的育种工作带来极大的不便。近年来,运用EST-SSR[2]、SSR[3,4]、SRAP[5,6]、RAPD[7]、AFLP[8]等 分 子 标 记技术,极大地丰富了柿属植物种质资源的鉴定和评价工作;但就目前的情况,并未找到最合适的分子标记类型。此外,由于柿的起源进化情况暂不清楚,核基因组信息较少,且具有多套染色体组,而目前用于分析分子标记数据的常用数学模型一般只适用于二倍体,导致多倍体材料中许多遗传参数无法计算,所以基于核基因组的分子标记方法不能很好地运用于柿的遗传变异研究。而叶绿体基因组与核基因组相比,其分子量小、多拷贝、结构简单,有利于对其进行测序分析;并且,叶绿体基因组序列相对保守,突变类型主要表现为点突变、碱基插入或缺失;因此,探索开发有效的叶绿体基因组分子标记对柿属植物的种质鉴定、系统演化分析及遗传多样性分析等工作十分必要。
目前基于叶绿体DNA(cpDNA)的分子标记主要有PCR-RFLP标记[9]、cpSSR标记[10]和cpInDel标记[11]等。在叶绿体基因组变异的研究初期,PCR-RFLP标记被广泛应用于较高等级分类阶元的植物进化研究[12-14];cpSSR位点主要位于叶绿体基因组的非编码区[15-16],而叶绿体非编码区的DNA序列在群体间普遍存在变异,因此cpSSR标记被广泛应用于植物群体遗传变异的研究,目前多种重要经济植物相继开发出cpSSR标记[17-19]。InDel为含量最为丰富的多态性突变之一,兼具了各类遗传标记的优点。InDel标记的准确性高、变异稳定,可以避免由于特异性和复杂性而导致的后续分析模糊[20];InDel标记本质上属于长度多态性遗传标记,基于PCR扩增技术及电泳技术就可以对其进行分型。此外,InDel标记在种内和种间都有多态性,其通用性较强[21]。因此,InDel标记被广泛应用于物种进化关系研究、种质资源分析、基因定位及遗传图谱的构建等。
本研究基于5个柿属植物叶绿体全基因组测序的基础上[22],利用比对得到的InDel位点开发叶绿体基因组分子标记,并评价其用于柿属植物种间及种内遗传变异研究的可行性。
1 材料和方法
1.1 试验材料
本研究所用到的材料为柿的5个近缘种,及11个来自不同地区的柿品种(表1)。油柿采自陕西杨陵国家柿种质资源圃,其余4个种和所有柿品种采自中国林科院经济林研究开发中心柿种质资源圃。采取各样品的新鲜嫩叶,经液氮速冻后储存于-80℃冰箱备用。
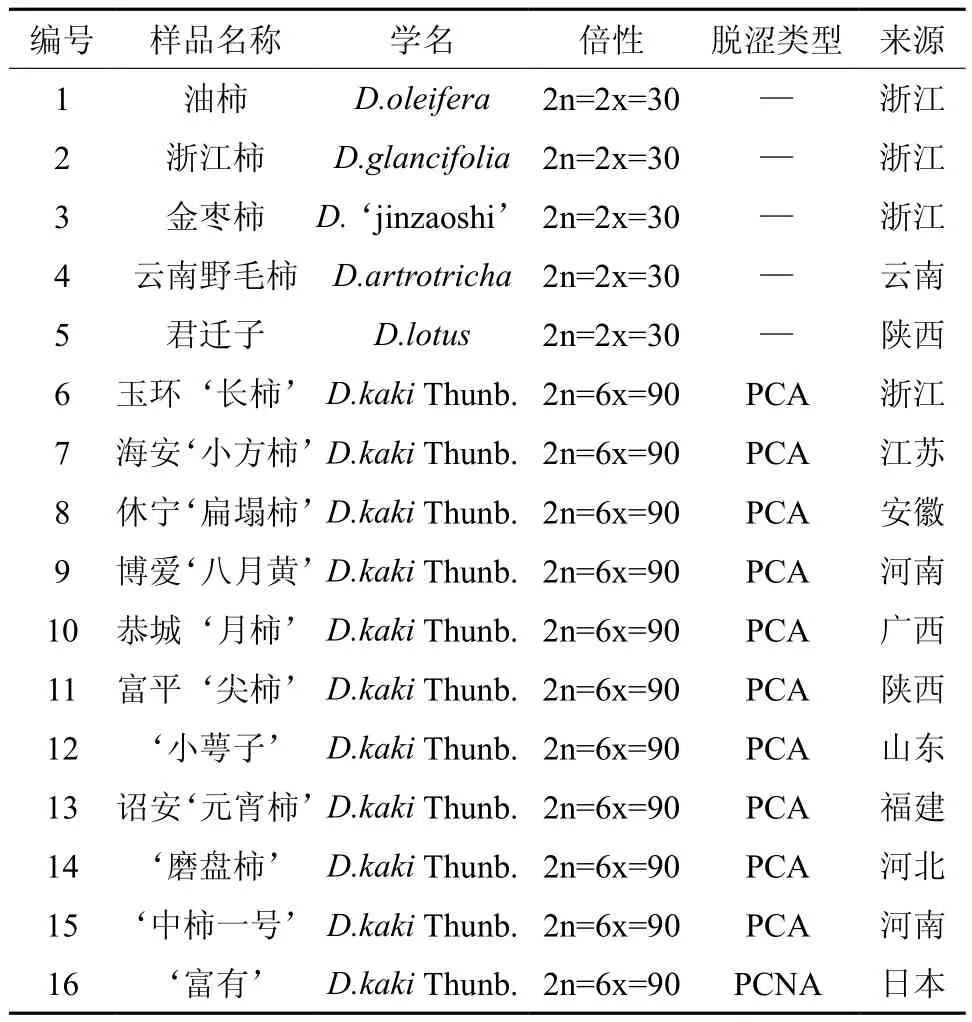
表1 试验材料†Table 1 The materials
1.2 引物设计
通过比对5个柿属植物叶绿体全基因组序列,选择差别在20 bp 以上、具有多态性的Indel位点,开发柿属叶绿体基因组分子标记。共筛选出3个符合要求的Indel位点,分别位于LSC区域的ycf4_cemA和trnT_trnL基因间隔区,以及位于SSC区的rps15基因内。其中,金枣柿、浙江柿和君迁子缺失位于ycf4_cemA区域的一段长为21 bp的序列,以及位于rps15区域的一段43 bp序列,而柿和油柿缺失位于trnT_trnL区域的一段28 bp的序列。此外,作为对照,选取柿属植物叶绿体基因组中的一段序列(位于LSC区域的trnH_psbA基因间隔区)共同开发叶绿体基因组分子标记。将以上3个Indel序列和1段叶绿体基因组序列连同侧翼序列提取出来,针对目的片段的上、下游,利用Primer 5.0软件设计特异引物,并用Oligo7.0软件对引物进行评价筛选,筛选出最优特异引物(表2),由生工生物工程(上海)有限公司合成。

表2 从柿属植物叶绿体基因组中开发的4个具有多态性的Indel标记Table 2 Details of 4 polymorphic Indel loci developed from Diospyros cpDNA
1.3 目的片段扩增与测序
采用CTAB法从16份被试材料中提取植物总DNA[23]。PCR扩增反应在bio-red公司PCR仪上进行。反应体系为25 μL,内含1×PCR缓冲液(含 1.5 mmol·L-1Mg2+),250 μmol·L-1dNTP,10 mmol·L-1Tris-HCl,50 mmol·L-1KCl,0.2 μmol·L-1primers,1.25 U Taq DNA聚合酶,40 ng DNA。反应程序为94℃预热3 min;94 ℃变性45 s,50.6~56.0 ℃退火1 min,72 ℃延伸75~150 s,共35个循环;最后72 ℃延伸5 min,4 ℃保温。PCR产物通过Gelred染色的2.0%琼脂糖凝胶电泳检测,Bio-red公司凝胶成像系统观察并记录谱带。利用UniQ柱型回收试剂盒(上海生工生物工程有限公司)回收纯化扩增产物,所得产物直接送上海生物工程有限公司,在3730型号自动测序仪上进行双向测序。
1.4 数据分析
应用BioEdit软件对所获得的4个区域进行序列拼接,并依据已测柿叶绿体基因组对所测区域进行手工校对。把校对后的序列导入到MEGA软件中,进行序列长度、G+C含量和变异位点分析。
2 结果与分析
用MEGA软件对16份柿属材料进行序列长度、G+C含量和变异位点分析,结果如表3所示:CP1标记扩增片段的长度为409 bp~427 bp,CP3标记的扩增片段最短,为234 bp~285 bp,CP4标记的扩增片段最长,为518 bp~571 bp。CP2、CP3、CP4标记是基于柿属植物叶绿体基因组中的Indel位点开发的,对扩增片段测序后比对发现,CP2标记扩增得到的片段中,金枣柿、浙江柿、君迁子、云南野毛柿都缺少一段21 bp的序列,CP3标记扩增得到的片段中,4个近缘种都缺少一段43 bp 的序列;而CP4标记扩增得到的片段中,柿与油柿均缺少一段28 bp的序列;将此3段缺失的序列分别与3个Indel序列进行比对,结果完全一致,即验证了此3个Indel位点存在的真实性。此外,基于叶绿体基因组trnH_psbA区开发的CP1分子标记,其扩增片段在柿与4个近缘种中无明显的长度差异。4个分子标记在11个柿品种中扩增片段测序序列长度也无明显差异。4个标记在16份柿属植物中扩增片段的平均G+C含量分别为35.4%、30.7%、32.2%和23.5%。CP1标记在16份柿属植物中检测到31个变异位点,其中简约信息位点为14个;CP2、CP3、CP4标记检测到的变异位点分别为27个、7个、25个,简约信息位点分别为5个、2个、13个。

表3 扩增片段序列信息Table 3 Sequence information of amplified fragments
利用MEGA软件对16份柿属植物的4个分子标记扩增序列进行遗传距离的估算和UPGMA聚类分析。结果如图1所示:基于CP1标记获得的聚类图(图1A)中,11份柿品种聚在一起,与5个柿的近缘种完全分离,且柿与油柿距离最近,其次是云南野毛柿,而浙江柿与君迁子聚为一支,金枣柿与柿距离最远,表明此标记可以较好的适用于柿属植物种间资源的鉴定。在基于CP2标记构建的聚类图(图1B)中,云南野毛柿和君迁子与部分柿品种聚为一类,而浙江柿和油柿与3个柿品种聚为一支,金枣柿单独为一支,表明此标记不能很好的区分柿及其近缘种。在基于CP3标记和CP4标记构建的聚类图(图1C和1D)中,云南野毛柿、浙江柿、君迁子、金枣柿等均能与柿品种区分开,而油柿与柿品种聚为一类,间接证明柿与油柿具有最近的亲缘关系。
此外,利用MEGA软件,针对11个柿品种的4个标记扩增片段的变异位点进行了分析。结果如表4所示:除了CP1标记在11个柿品种的扩增片段中未检测到变异位点外,另外3个标记都检测到了种内变异的位点。CP2标记共检测到6个变异位点,可以将11份柿品种分成两大类,即休宁扁塌柿、博爱八月黄、恭城月柿、海安小方柿、磨盘柿、小萼子、富有、中柿一号等8个品种为一类,在检测出的6个位点均无变异;而富平尖柿、玉环长柿、诏安元宵柿等3个品种为一类,富平尖柿和玉环长柿在489位点存在一个单核苷酸Indel(插入),富平尖柿在487位点和488位点发生G-C和A-G突变,而诏安元宵柿在487-489位点存在一个三核苷酸Indel(缺失)。CP3标记共检测到4个种内变异位点,其中恭城月柿在285-287位点发生三核苷酸Indel(插入),海安小方柿在275位点处发生了G-A突变,磨盘柿在285位点发生单核苷酸Indel(插入),富平尖柿在285和286位点发生二核苷酸Indel(插入)。CP4标记共检测到6个变异位点,休宁扁塌柿和博爱八月黄在578-580位点发生三核苷酸Indel(插入),且在580位点存在变异;恭城月柿在578和579位点发生二核苷酸Indel(插入);海安小方柿在567位点处发生A-T突变,在578位点发生单核苷酸Indel(插入);富平尖柿在578位点发生单核苷酸Indel(插入);磨盘柿、富有、中柿一号、玉环长柿、诏安元宵柿等在568位点发生T-A突变。综合3个标记,只有在富有和中柿一号两个品种内未检测到变异位点,其余品种均检测到发生变异的位点。
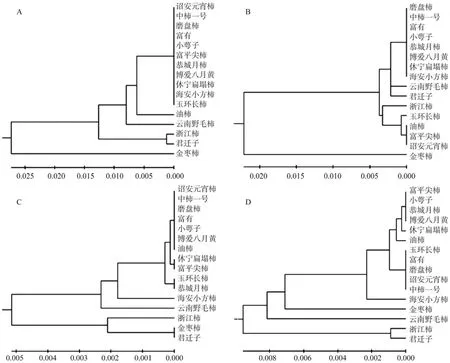
图1 基于4个叶绿体标记的16份柿属植物的聚类图Fig. 1 The dendrograms of clustering analysis of 16 Diospyros spp. based on 4 chloroplast markers
3 结论与讨论
目前应用于柿属植物遗传多样性研究的分子标记数量有限,尤其是适用于种内遗传变异研究的标记缺乏特异性,无法区分品种繁多的栽培柿。因此,开发更多有效的分子标记,对于研究柿的起源、多样性及演化具有重要意义。叶绿体基因组的结构和序列相对于核基因组而言比较保守,变异频率较低,常被用作植物种上的遗传多样性和系统进化研究[24,25]。此外,叶绿体基因组中某些进化速率较快的区域,也可用于探讨植物种间甚至种内不同基因型间的遗传关系[26,27]。胡德昌等[28]对9个柿品种cpDNA trnS-trnfM区域特异扩增并克隆测序,序列比对结果表明部分柿品种存在多个碱基突变,插入和缺失位点,相对于限制性内切酶酶切技术,cpDNA相应区域扩增片段测序得到的变异位点信息更为丰富有效。
本研究设计开发的4对叶绿体分子标记,在16份柿属植物中进行验证,发现用于序列比对的1786 bp 长度的测序序列中,总变异长度为90 bp,占被测序列总长的5.04%,表现出柿属植物种群内存在较高的遗传变异率。CP2、CP3、CP4标记是依据3个Indel位点开发的,其在柿与5个柿的近缘种中的扩增片段测序结果显示相应的Indel确实存在,即进一步验证了柿属植物叶绿体全基因组测序结果的准确性。此外,基于4个叶绿体基因组分子标记构建的聚类图提示:CP1分子标记在柿属植物种间的多态性良好,但在柿种内无多态性,故可以尝试用于柿属植物种间遗传多样性及起源等相关研究。CP2、CP3、CP4标记均无法将柿品种与柿的近缘种进行很好地区分,究其原因可能是由于用于开发标记的Indel位点在部分柿的近缘种间一致,导致开发的标记在种间的多态性较低,比如用于开发CP2标记的Indel是在柿和油柿中插入一段21 bp的序列,而在金枣柿、浙江柿、君迁子、云南野毛柿中都缺失该序列。但是,CP2、CP3、CP4标记在柿种内表现出一些位点变异,且大部分品种发生变异的位点不同,故可以考虑用于柿品种遗传多样性研究。
本研究开发出1对适用于柿属植物种类鉴定的叶绿体分子标记(CP1),及3对在柿品种间具有多态性位点的叶绿体分子标记(CP2、CP3、CP4),为柿属植物品种鉴定及遗传多样性分析提供了有力帮助。InDel作为新一代的遗传标记在群体遗传学、物种进化关系研究、基因定位、种子纯度等研究中独具优势,受到越来越多的关注。但是,InDel标记也存在一些不足,主要表现为其携带的遗传信息有限,要达到足够高的分辨效能,需要将更多的InDel位点进行联合分析。本研究开发出的CP2、CP3、CP4标记均在柿种内获得一些变异位点,说明其在柿种内品种间的遗传变异研究中具有较大的应用潜力,下一步的研究工作中,我们将继续开发更多的柿属叶绿体InDel分子标记,为柿种内的遗传多样性研究提供有效便捷的分子工具。
[1]孙 鹏, 李加茹, 段 伟,等. 河南省柿种质资源果实表型特征分析[J]. 经济林研究, 2015, 33(4):9-17.
[2]吴 硕, 傅建敏, 乌云塔娜, 等. 柿属EST-SSR引物设计及遗传多样性研究[J]. 中南林业科技大学学报,2012,32(3):152-157.
[3]Guo DL, Luo ZR. Microsatellite Isolation and Characterization in Japanese Persimmon (Diospyros kaki) [J]. Biochem Genet,2008, 46: 323-328.
[4]梁玉琴, 李芳东, 傅建敏, 等. 正交设计优化柿属植物SSRPCR反应体系[J]. 经济林研究, 2011, 29(4): 17-22.
[5]Guo DL, Luo ZR. Genetic relationships of some PCNA persimmons (Diospyros kaki Thunb.) from China and Japan revealed by SRAP analysis [J]. Genetic Resources and Crop Evolution, 2006, 53: 1597-1603.
[6]Jing ZB, Ruan XF, Wang R, et al. Genetic diversity and relationships between and within persimmon (Diospyros kaki)wild species and cultivated varieties by SRAP markers [J]. Plant Syst Evol Springer-Verlag Wien, 2013, 299(8):1485-1492.
[7]Mustafa A, Sezai E, Nalan Y, et al. The comparison of persimmon genotypes (Diospyros kaki Thunb.) by using RAPD and FAME data [J]. Roumanian Biotechnological Letters, 2008,13(4):3851-3858.
[8]Keizo Y, Chitose H, Akira K, et al. Relationship of European persimmon (Diospyros kaki Thunb.) cultivars to Asian cultivars characterized using AFLPs [J]. Genet Resour Crop Evol, 2008,55: 81-89.
[9]Besnard G, Berville A. On the choloroplast DNA variations in the olive (Olea europaea L.) complex: comparison of RFLP and PCR polymorphisms [J]. Theor Appl Genet, 2002, 104: 1157-1163.
[10]Powell W, Morgante M, Andre C. Hypervariable microsatellites provide a general source of polymorphic DNA markers for the chloroplast genome [J]. Curr Biol, 1995,5: 1023-1029.
[11]薛 宏, 易自力, 肖 亮, 等. 芒属植物叶绿体InDel标记的开发与应用[J]. 现代农业科技, 2015(7):153-155.
[12]Ito M, Yahara T, King RM, et al. Molecular phylogeny of Eupatorieae (Asteraceae) estimated from cpDNA RFLP and its implication for the polyploid origin hypothesis of the Tribe[J].Journal of Plant Research, 2000, 113(1):91-96.
[13]Magni CR, Ducousso A, Caron H, et al. Chloroplast DNA variation of Quercus rubra L. in North America and comparison with other Fagaceae[J]. Mol Ecol, 2005, 14(2): 513-24.
[14]Devos N, Raspé O, Oh SH, et al. The evolution of Dactylorhiza(Orchidaceae) allotetraploid complex: insights from nrDNA sequences and cpDNA PCR-RFLP data[J]. Molecular Phylogenetics & Evolution, 2006, 38(3):767-778.
[15]Jakobsson M, Sall T, Lind-Hallden C, et al. Evolution of chloroplast mononucleotide microsatellites in Arabidopsis thaliana [J]. Theoretical and Applied Genetics, 2007, 114:223-235.
[16]Provan J, Powell W, Hollingsworth PM. Chloroplast microsatellites: new tools for studies in plant ecology and evolution [J]. Trends in Ecology & Evolution, 2001, 16:142-147.
[17]Powell W, Morgante M, Mcdevitt r, et al. Polymorphic simple sequence repeat regions in chloroplast genomes:applications to the population genetics of pines[J]. Proceedings of the National Academy of Sciences, USA, 1995, 92:7759-7763.
[18]Cheng YJ, Vicente MCD, Meng HJ, et al. A set of primers for analyzing chloroplast DNA diversity in Citrus and related genera[J]. Tree Physiology, 2005, 25(6):661- 672.
[19]甄 贞, 曹庆芹, 杨 凯, 等. 栗属植物 cpSSR 标记技术的建立与体系优化研究[J]. 果树学报, 2007, 24(4): 557-560.
[20]杨 洁, 赫 佳, 王丹碧,等. InDel标记的研究和应用进展[J]. 生物多样性, 2016, 24(2):237-243.
[21]Väli U, Brandström M, Johansson M, et al. Insertiondeletion polymorphisms (Indels) as genetic markers in natural populations. BMC Genetics, 2008, 9: 715-720.
[22]Fu J, Liu H, Hu J, et al. Five Complete Chloroplast Genome Sequences from Diospyros: Genome Organization and Comparative Analysis[J]. Plos One, 2016, 11(7): 1-18.
[23]梁玉琴, 李芳东, 傅建敏, 等. 柿属植物基因组DNA提取方法比较[J].中南林业科技大学学报, 2012, 32(4): 170-173.
[24]Badenes ML, Parfitt DE. Phylogenetic relationships of the cultivated Prunus species from an analysis of chloroplast DNA variation[J]. Theor. Appl. Genet. 1995, 90: 1035-1041.
[25]Xu DH, Abe J, Kanazawa A, et al. Identification of sequence variations by PCR-RFLP and its application to the evaluation of cpDNA diversity in wild and cultivated soybeans [J]. Theor.Appl. Genet, 2001, 102: 683-688.
[26]King R, Ferris C. Choloplast DNA phylogeography of Alnus glutinosa (L.) Gaertn.[J]. Molecular Ecology, 1998, 7: 1151-1161.
[27]Wu W, Zheng YL, Chen L, et al. PCR-RFLP analysis of cpDNA and mtDNA in the genus Houttuynia in some areas of China[J].Hereditas, 2005, 142: 24-32.
[28]胡德昌, 张青林, 罗正荣. 部分柿品种叶绿体基因组 trnS-trnfM区域的序列变异[J].北京:中国科技论文在线,2007-02-05.
Exploitation of cpDNA molecular markers and genetic variation of Diospyros spp.
FU Jianmin1,2,3, SUO Yujing2,3, ZHANG Jiajia2,3, TAN Xiaofeng1
(1. Central South University of Forestry and Technology, Changsha 410004, Hunan, China; 2. Non-timber Forest Research and Development Center of CAF, Zhengzhou 450003, Henan, China; 3. China Paulownia Research Center, Zhengzhou 450003, Henan, China)
Based on the sequencing results, four cpDNA markers were developed, and 16 Diospyros plants were used to verify their feasibility. For the four cpDNA markers, 31、27、7、25 variable sites were detected,respectively;and the parsimony-informative sites were 14、5、2、13. Cluster analysis showed that CP1 marker could distinguish the five related species from 11 persimmon cultivars, indicating that CP1 was suitable for analyzing the genetic variations between different Diospyros species. While, CP2、CP3、CP4 failed to make a distinction between persimmon cultivars and their related species. Nonetheless, several single base mutations and Indels were found across the 11 persimmon cultivars, and most of the cultivars showed speci fi c variable sties, except for ‘Fuyuu’ and‘Zhongshi 1#’, this result suggested that CP2、CP3、CP4 markers could be used to analyze the genetic variations in persimmon cultivars.
Diospyros; related species; cpDNA markers; genetic variations
S727.3;S665.2
A
1673-923X(2017)06-0001-06
10.14067/j.cnki.1673-923x.2017.06.001
2016-11-27
国家“十二五”科技计划课题“涩柿种质资源收集评价与新品种选育”(2013BAD14B0502)
傅建敏,副研究员
谭晓风,教授,博导;E-mail:tanxiaofengcn@126.com
傅建敏,索玉静,张嘉嘉,等. 柿属植物cpDNA分子标记开发及遗传变异研究[J].中南林业科技大学学报,2017, 37(6):1-6.
[本文编校:吴 彬]
